干酪乳杆菌arat基因的敲除及其对苯乳酸合成代谢的影响
邓廷山, 宋丽莉, 孙 宇, 周茂超, 王 翠, 武国干*, 唐雪明*
(1.上海海洋大学 食品学院,上海 201306;2.上海市农业科学院 生物技术研究所,上海 201106;3.上海市农业科学院 上海市农业遗传育种重点实验室,上海 201106)
苯乳酸(phenyllactic acid,PLA)自2000年首次由Lavermicocca等[1]报道具有抑菌作用后,其所具有的优良抑菌性能,尤其是对致病菌与食物腐败菌的抑菌作用逐渐被发现和开发。PLA除了具有广谱抑菌特性,其安全性和稳定性也相较于目前广泛使用的化学防腐剂更加优良[2-3]。许多微生物可自身代谢产生PLA,而由于多数乳酸菌为GRAS微生物,以其为出发菌代谢产PLA在食品行业中无疑具有更好的发展前景[4]。天然的乳酸菌产PLA的量较低,通常不超过100 mg/L,这成为限制其广泛应用的关键因素[4]。国内外对于PLA代谢机制的研究较少,主要集中于高产菌株的筛选及培养基的优化[5-6],目前普遍认为PLA的代谢合成主要有两步:第一步通过芳香族氨基转移酶(Aromatic amino acid transferase,ArAT)的催化将苯丙氨酸(Phe)转化为苯丙酮酸(PPA), 第二步由PPA经过乳酸脱氢酶(Lactate dehydrogenase,LDH)的催化转化为PLA。该结论的得出来源于两步反应中的关键酶在体外进行的酶学研究,虽然ArAT与LDH在大肠埃希菌中进行的全细胞催化反应获得了极高的PLA的产量[7-8],但乳酸菌自身代谢产生PLA的量远不及此,其体内的代谢机制亟待进一步研究。CRISPR/Cas9基因编辑技术作为近年来的研究热点,在医学、检测、基因功能及代谢等多个领域得到了广泛的应用,在不断的研究和探索过程中逐渐开发和研究出多种Cas9蛋白的变体,以适用于不同类型的研究[9-10]。Song等[11]开发的质粒pLCNICK含有Cas9D10A,能够很好地运用于乳酸菌的基因编辑操作,并且该质粒具有较高的敲除效率,相对于传统同源重组的基因敲除方法具有快速高效的特点。本研究运用该基因编辑系统,对一株干酪乳杆菌的arat基因进行体内敲除,研究该基因的缺失对菌株产PLA及其代谢途径的影响,为乳酸菌的PLA合成代谢提供理论依据,为后续代谢途径调控的研究提供新的思路及方法。
1 材料与方法
1.1 材料
1.1.1 供试菌株及质粒 干酪乳杆菌(Lactobacilluscasei) 1.8727、大肠埃希菌DH10B菌株由上海市农业科学院生物技术研究所保藏,乳酸菌基因编辑质粒pLCNICK由上海理工大学惠赠。
1.1.2 培养基 ①MRS固体及液体培养基(OXOID)。②MMRS复苏培养基(mmol/L):MRS加终浓度蔗糖 500,MgCl220,CaCl22。③LB培养基(g/L):蛋白胨 10,酵母提取物 5, NaCl 10,固体培养基额外加入20 琼脂粉。必要时加入抗生素,其中MRS中红霉素终浓度为10 μg/mL,LB培养基中卡那霉素终浓度为50 μg/mL。
1.1.3 主要试剂与仪器设备ExTaq(TaKaRa),XbaⅠ、ApaⅠ快切酶(Thermo),色谱级苯乳酸、苯丙氨酸、甲醇(Aladdin),三氟乙酸(MACKLIN)。细菌基因组抽提试剂盒(天根生物科技有限公司),ClonExpress MultiS一步克隆试剂盒(南京诺唯赞生物科技有限公司),质粒小量抽提试剂盒(生工生物工程(上海)股份有限公司),引物合成及测序由生工生物工程(上海)股份有限公司完成。电转化仪(GenePulser Xcell,BIO-RAD);PCR仪(T100TM Thermal Cycler,BIO-RAD);凝胶成像系统(Gel DocTM EZ Imager,BIO-RAD);电泳仪(DYY-6C,北京市六一仪器厂);电热恒温培养箱(DHP-9272,太仓市华利达实验设备有限公司);恒温摇床(ZWY-240,上海智城分析仪器制造有限公司);离心机(MicroCL17,BIO-RAD);超净工作台(SW-CJ-IFD,苏州安泰空气技术有限公司);高效液相色谱仪(Agilent 1290 Infinity Ⅱ,美国安捷伦);色谱柱(XBridge C18,美国Waters);2 mm电转化杯(BIO-RAD)。
1.2 方法
1.2.1arat基因序列比对及引物设计 对NCBI数据库中3株已上传的干酪乳杆菌芳香族转氨酶基因arat进行比对分析,发现Lactobacilluscasei12A、Lactobacilluscasei52W、LactobacilluscaseiBL23三株菌的序列同源性为99%,以此为基础进行L.casei1.8727菌株arat基因的引物设计(表1)。

表1 引物列表
1.2.2arat-pLCNICK重组载体构建 提取L.casei1.8727基因组,并以该基因组为模板,使用3KF/3KR引物扩增包含arat基因及上下游各1 kb大小的目的片段,将该片段命名为3K片段。随后以该片段为模板,5′arat-FHa/3′arat-FHa;5′arat-RHa/3′arat-RHa引物分别扩增上游同源臂arat-FHa和下游同源臂arat-RHa。以pLCNICK为模板,以5′sgRNA/3′arat-sg引物扩增出靶向arat基因的sgRNA。以arat-RHa及sgRNA为模板,以AOlapF/2.1R为引物,利用Overlap PCR分别扩增出插入片段的下游部分,命名为RHa-sgRNA,纯化后再以其与arat-FHa进行一次Overlap PCR,以2.1F/2.1R为引物,最后扩增出插入片段,命名为arat-sg,该片段两段各含20 bp与pLCNINK质粒同源片段,用于一步克隆(图1A)。将pLCNICK转化DH10B感受态细胞,挑取阳性克隆接种至含卡那霉素的5 mL LB培养基过夜培养,按1%接种量接种至20 mL含卡那霉素的LB液体培养基培养12~16 h,抽提质粒后用XbaⅠ和ApaⅠ进行双酶切,胶回收载体片段。按照一步克隆试剂盒说明书配置反应体系,将arat-sg片段与线性化pLCNICK载体进行连接转化,涂布于含卡那霉素的固体LB培养基进行筛选,抽提质粒进行PCR验证及测序验证,构建得到的基因敲除质粒命名为arat-pLCNICK(图1B)。

图1 插入片段与基因编辑质粒的示意图 Fig.1 Schematic of insert fragment and gene editing plasmidA:插入片段arat-sg; B:arat-pLCNICK质粒图 A:Insert fragment arat-sg; B:plasmid map of arat-pLCNICK
1.2.3arat-pLCNICK电转化L.casei1.8727 制备L.casei1.8727感受态细胞[11],将5 μgarat-pLCNICK质粒与感受态细胞混合后转移到冰上预冷的2 mm电转化杯中,于冰上放置5 min。使用电转化仪,在2 000 V,400 Ω,25 μF条件下进行电击。立即加入1 mL MMRS,转入2 mL离心管中,于37 ℃静置2~3 h,然后涂布于红霉素抗性的MRS固体培养基,37 ℃培养2或3 d直至长出单菌落。
1.2.4L.casei1.8727Δarat敲除菌株的筛选鉴定 挑取单菌落接种于5 mL 含红霉素的MRS液体培养基,培养过夜,提取基因组,用3KF/3KR引物进行PCR验证,筛选出含有arat敲除菌的杂合菌,并在MRS固体培养基中连续划线传代两次,挑取二次划线的单菌落接种于5 mL MRS液体培养基中,再次提取基因组,用aratF/aratR及3KF/3KR两对引物进行PCR验证,并对3KF/3KR引物扩增的PCR片段测序,筛选出arat敲除菌株,命名为L.casei1.8727Δarat。
1.2.5 HPLC测定PLA与Phe产量 由于Phe是arat的直接底物,因此对PLA产物进行HPLC测定时,Phe的测定也至关重要,有研究表明Phe的添加可以增加PLA的产量[12-13]。发酵准备:制备12瓶20 mL MRS液体培养基,其中6瓶含终浓度2 g/L Phe。实验菌株:L.casei1.8727及L.casei1.8727Δarat。将提前活化好的两株菌种子液按1%(体积分数)接种量接种,两菌株均接种于MRS培养基与含终浓度2 g/L Phe的MRS培养基,每组三个平行重复,于37 ℃培养箱静置培养,每瓶发酵液分别于24、48、72 h取样1 mL,13 000 r/min离心10 min,0.22 μm滤膜过滤,上机测定。标准品制备:PLA与Phe标品分别于1 L容量瓶中配置成终浓度200、400 mg/L溶液。PLA稀释成5个梯度(mg/L):200、100、50、25、12.5;Phe稀释成5个梯度(mg/L):400、200、100、50、25。各标准品经0.22 μm滤膜过滤备用,混合标准品制备:将上述两种标准品按1∶1(体积比)混合。色谱条件:参考黄国昌等[14]的方法进行测定。色谱柱为XBridge C18,检测波长220 nm,进样量10 μL,流速1 mL/min,柱温30 ℃,流动相A液:0.05%三氟乙酸甲醇溶液,流动相B液:0.05%三氟乙酸水溶液,洗脱条件:0~20 min由A∶B从10%线性变化为100%,20~25 min保持100% A相,25~30 min A∶B从100%线性变化为10%。在进行各组发酵样品前,先对Phe与PLA的单个标准品进行上机测定,确定Phe与PLA的洗脱时间,随后将混合标准品与各组发酵液样品进行上机测定,同时将MRS培养基在相同色谱条件下测得的Phe与PLA含量作为空白对照。最后根据所得数据绘制标准曲线,并对各组发酵数据进行分析。
2 结果与分析
2.1 L. casei 1.8727 Δarat的鉴定
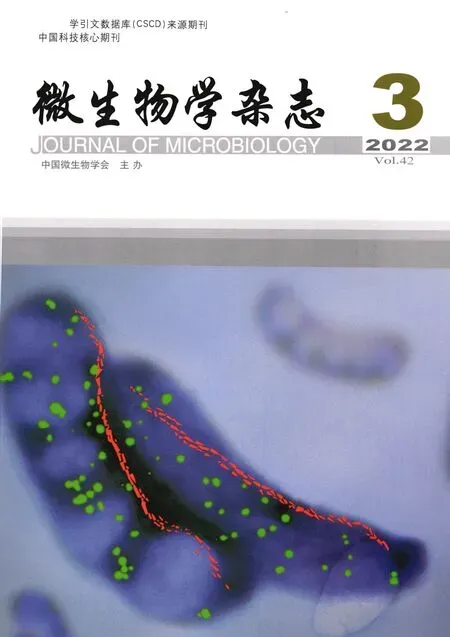
arat-pLCNICK质粒转化平板上挑取单菌落连续划线两次,再提取基因组后用3KF/3KR引物进行PCR鉴定,如图2A所示,敲除菌株扩增出2 kb大小的条带,与arat基因缺失后的大小相符。同时使用arat基因特异性引物对aratF/aratR进行PCR验证,如图2B所示,敲除菌株未扩增出目的条带。将图2A中敲除菌株的扩增片段割胶回收后进行测序验证,如图2C所示,测序所用引物为3KF/3KR,在片段两段进行单向测序,测序结果用DNAMAN软件与该3 kb片段进行比对,测序片段两端与原序列一致,显示arat基因已被敲除。
2.2 PLA与 Phe产量的HPLC测定
PLA在11~12 min之间出现洗脱峰且峰形较好(图3A),而PLA在7~8 min之间出峰(图3B),进行混合标样上机洗脱如图3C,洗脱峰分离效果及峰形较好,说明该洗脱方法对于同时分析PLA与Phe具有较好的效果。剩余混合标样按相同色谱条件进样分析,建立标准品回归曲线,如表2所示,表明回归曲线具有较高可信度与精确度。
各组发酵样品按相同色谱条件进样分析,所得结果如图4A、4B所示。MRS培养基作为空白对照经相同条件测定后发现其中含有约150 mg/L的Phe,而PLA的含量极低可以忽略,因此各组分Phe浓度数据减掉150 mg/L后进一步分析。图4A中在未添加Phe时,arat基因敲除后其PLA产量在各时期均有显著提高,而添加Phe后,PLA产量具有极显著提高,且在72 h时PLA产量提高了近66.7%。图4B中L.casei1.8727Δarat菌株的Phe产量相比于出发菌株具有显著性提高,且在72 h时其Phe产量提高近66.7%。数据表明L.casei1.8727菌株具有自身合成代谢Phe的能力,且Phe与PLA的产量随发酵时间的延长而逐渐积累,表明arat基因的敲除促进了PLA与Phe的合成。
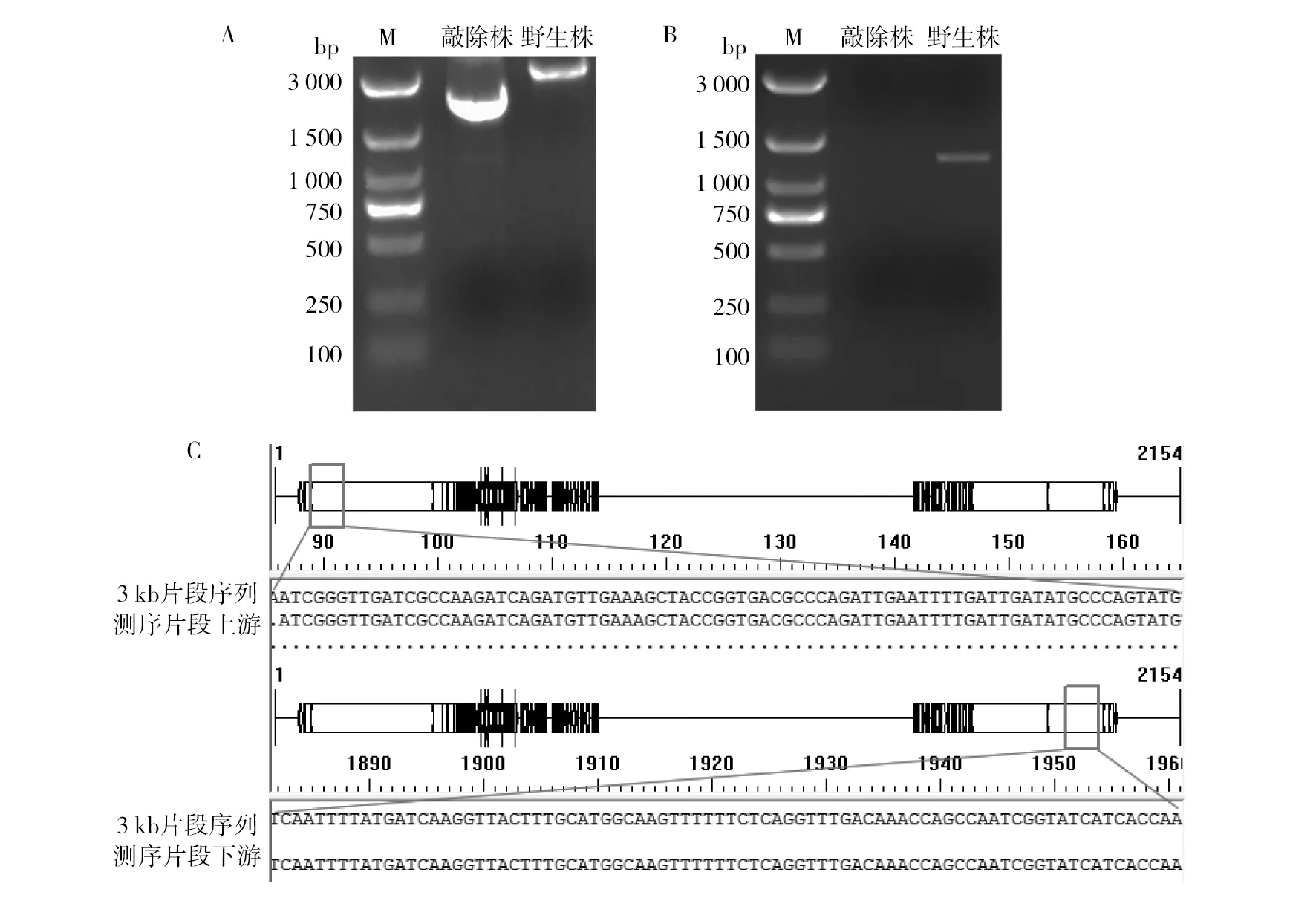
图2 arat基因敲除菌验证结果 Fig.2 Verification of arat gene knockoutA:同源臂引物验证; B:arat特异引物验证;C:测序验证A:Verification of primers for homologous arms; B:Verification of specific primers for arat; C:Sequencing validation

图3 单个标准品和混合标准品的色谱峰图 Fig.3 Chromatographic peak of single and mixed standardA:PLA的色谱峰图;B:Phe的色谱峰图;C:Phe与PLA的混合色谱峰图A:Chromatogram peaks of PLA; B:Chromatogram peaks of Phe; C:Chromatogram peaks of Phe and PLA

表2 Phe与PLA线性关系
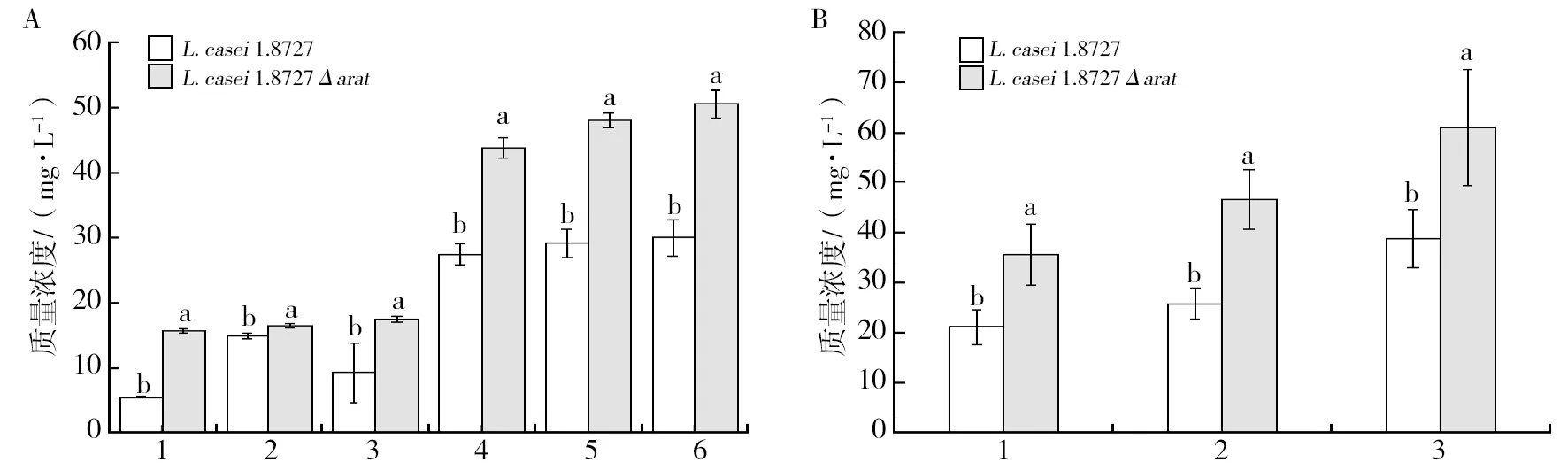
图4 各发酵样品Phe和PLA浓度数据分析柱状图Fig.4 The histogram of Phe and PLA concentration data analysis of each fermentation sampleA:PLA发酵数据柱状图, 1~3:两株菌分别在MRS培养基中发酵24、48、72 h; 4~6: 两株菌分别在含2 g/L苯丙氨酸的MRS培养基中发酵24、48、72 h。 B:Phe发酵数据柱状图, 1~3:两株菌分别在MRS培养基中发酵24、48、72 h。图A、B中a和b表示相同实验的两组数据有显著差异A:Histogram of PLA fermentation data, 1-3: The histograms of the two strains fermented in MRS medium for 24, 48 and 72 h; 4-6: The histograms of the two strains fermented in MRS medium containing 2 g/L Phe for 24, 48 and 72 h. B:Histogram of Phe fermentation data, 1-3: The histograms of the two strains fermented in MRS medium for 24, 48 and 72 h. In figure A and B,a and b indicate that there is a significant difference between two groups of data in the same experiment
3 讨 论
ArAT作为氨基转移酶中的一种,能够催化芳香族氨基酸与α-酮戊二酸之间的转氨反应,反应同时需要辅酶磷酸吡哆醛(pyridoxal-5-phosphate,PLP)的参与,该反应过程是可逆的[15]。ArAT是PLA代谢途径中的关键酶,因此研究ArAT对PLA合成代谢的影响具有重要意义。本研究通过实现对arat基因的敲除操作后发现arat缺失使PLA的产量提高了约66.7%,同时Phe的产量也比出发菌株提高约57.8%,该结果表明L.casei1.8727菌株能够自身代谢合成Phe。据相关研究可以推断Phe在ArAT的催化下生成苯丙酮酸PPA[16-17],在本研究中因为arat基因的缺失,可能导致Phe不能及时转化为PPA,从而导致Phe的积累。而PLA产量的提高,说明可能存在其他的氨基转移酶,或者PPA作为PLA的中间代谢底物时,其在细胞中的含量受其他路径的影响,使得在敲除arat基因后,并不能抑制PLA与Phe的合成代谢,而本研究中arat基因的缺失促进了PLA与Phe的合成,证明该反应步骤可能受到多个代谢途径的调控,在arat基因缺失后,很可能多个相关代谢路径的代偿最终使得PLA产量的提高。
有研究表明PLA的第一步转氨反应受多种因素的影响,如底物适用性,酶的活性,氨基受体α-酮戊二酸的可用性等[18-19]。通过酶学性质分析进一步确定该ArAT的底物适用性;不同种属的乳酸菌代谢合成PLA的能力具有很大差异[20-21],通过对多种不同种属来源的ArAT进行研究以判断酶活对转氨反应的影响;研究表明增加α-酮戊二酸浓度能显著提高PLA产量[21],α-酮戊二酸不仅作为该反应中的氨基受体,同时涉及多个生物反应过程,通过代谢工程调整其代谢流,使得α-酮戊二酸的体内含量提高,促进转氨反应的进行。
对于可能存在的其他合成PPA与Phe的代谢途径,在KEGG数据库中以干酪乳杆菌为种属来源的芳香族氨基酸代谢通路中发现酶E2.6.1.9参与Phe代谢,该酶名为组氨醇-磷酸氨基转移酶(Histidinol phosphate aminotranserase,HPA),该酶同样能以Phe为底物产PPA,在NCBI数据库中对多株干酪乳杆菌进行比对发现均存在HPA基因,该转氨酶很有可能共同参与干酪乳杆菌的PLA合成代谢。在近期的研究中,Sun等[22]通过基因组与转录组水平研究植物乳杆菌(Lactobacillusplantarum)LY-78合成PLA的代谢调控机制,并揭示了菌株中PLA生物合成的候选通路、操纵子和关键基因。据以上结论做出PLA可能的体内合成代谢途径,如图5所示。后续除了进一步研究ArAT的酶学性质之外,还需要研究组氨醇-磷酸氨基转移酶对PLA合成代谢的影响,同时结合代谢工程与转录组测序分析,进一步揭示PLA的合成代谢调控。总之,本研究发现对PLA合成代谢的研究具有重要意义,首次发现在干酪乳杆菌中arat基因的缺失能够促进PLA与Phe的合成代谢,同时本研究中所运用的基因编辑系统及方法能够很好地运用于干酪乳杆菌的基因敲除操作,为后续PLA代谢合成机制的进一步研究提供参考。
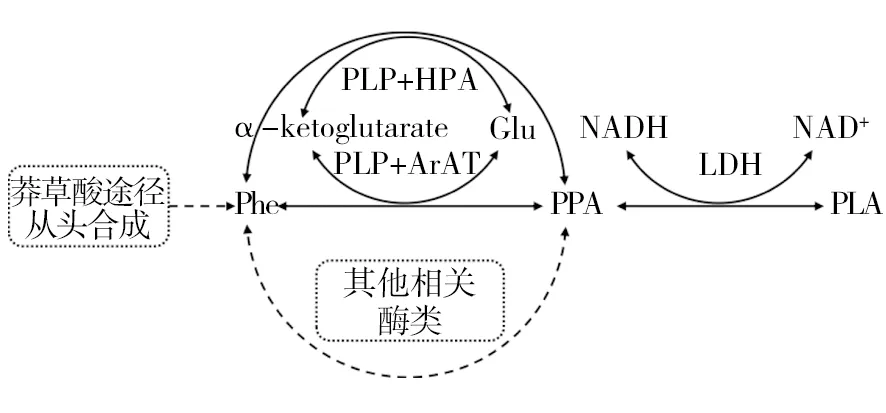
图5 干酪乳杆菌中PLA可能的代谢通路示意图Fig.5 Schematic diagram of possible PLA metabolic pathway in Lactobacillus casei图中虚线箭头表示有多个反应过程The dotted arrows in the diagram indicate that there are multiple reactions
——一道江苏高考题的奥秘解读和拓展