后基因组时代微生物天然产物高效发掘体系设计与展望
潘华奇
(中国科学院 沈阳应用生态研究所,辽宁 沈阳 110016)
长期的实践表明,不论是新型医药还是农药的创制,天然产物(natural products,NPs)及其启发合成的小分子药物均占据着极其重要的地位。Newman等统计了过去39年的药物,发现小分子药物占所有1 881种新批准药物的77.53%,其中以NPs为先导成药的有916种,占所有小分子药物65.71%[1]。同样在植物保护领域,Sparks和Bryant总结了过去 70 年超过800种植物保护化合物,发现NPs及其启发合成的农药占总数的近一半[2]。可见,化学多样性极其丰富的NPs拥有独特的化学空间和药效优势成为创新医药和农药的重要来源与启迪。微生物因具有极其庞大的类群和丰富的代谢产物成为新型医药和农药的重要来源,并且贡献了最多的抗生素[3-4]。自1928年 Fleming 发现青霉素开始,微生物来源的药物进入快速发展的黄金时期,并长期成为国际社会的研究热点,尤其是2015年著名杀虫剂阿维菌素的发明者卡贝尔和大村智获得诺贝尔奖,又掀起了微生物先导药物的研究热潮[5]。然而随着微生物(尤其土壤微生物)的不断开发和重复筛选(包括菌种和筛选方法的重复),发现新颖药用先导化合物的几率不断下降[6-7](图1A)。因此,科学家将目光投向包括植物内生菌[8]、海洋微生物[9]、极端环境微生物等尚未充分发掘的特境微生物新资源。进入21世纪以来,海洋微生物来源的新NPs进入快速增长期(图1B),尤其是2008年以后海洋新NPs的增量几乎都是源于海洋微生物。目前,海洋微生物每年贡献约1 000个新NPs,占海洋来源新NPs总数的2/3以上(图1B)。可见,特境微生物来源的新NPs已成为发现新型活性先导化合物的重要增长点和研究热点[8-10]。
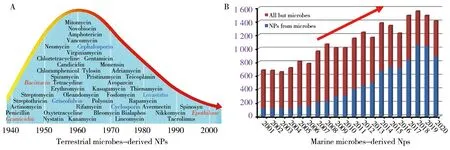
图1 不同生境微生物来源新NPs的发现趋势Fig.1 Trend in the discovery of new NPs from microbes in different eco-environment
近十多年来,随着基因组学的迅猛发展,人们认识到微生物具有丰富的编码次级代谢产物的生物合成基因簇(biosynthetic gene clusters,BGCs),估计高产的放线菌具有20~50个BGCs[11],真菌则具有30~70个BGCs[12-13],这赋予其生产新结构药用先导化合物的巨大潜能。但是目前发现的NPs仅是其冰山一角,绝大部分(大约80%~90%)的BGCs在常规条件下是沉默的或低表达的,因此许多基因组挖掘的激活策略应运而生[14]。特别是当前合成生物学和机器学习等新技术层出不穷,为微生物NPs的发掘带来了新的机遇。在后基因组时代如何实现微生物源药用先导化合物高效发掘?本文将从富含BGCs 的“潜力菌种”选择、生物信息学预测、“隐性”BGCs激活以及激活产物的高效识别等方面进行论述,并提出了在后基因组时代微生物NPs高效发掘的集成新线路,泛生物合成基因簇(pan-BGCs)的概念,以及对发掘NPs药用先导化合物过程中一些问题的思考。
1 富含BGCs“潜力菌种”的选择
从可培养微生物中发掘NPs依然是先导药物分子发现的主流,选择菌株则是最为关键的第一步。越来越多的研究发现,产生生物活性代谢产物的微生物在分类学分布上是很不均衡的,放线菌门(Actinobacteria)、厚壁菌门(Firmicutes)、变形菌门(Proteobacteria)、蓝细菌门(Cyanobacteria)和子囊菌门(Ascomycota)等类群的基因组中含有丰富的BGCs(表1)[15],特别是这些类群中的链霉菌属、芽孢杆菌属、假单胞菌属和曲霉属等,其成员是过去大多数生物活性物质的贡献者[16]。最新的宏基因组研究发现,在微生物的分类等级中,链霉菌目、分枝杆菌目、伯克氏菌目、根瘤菌目、假单胞菌目、肠杆菌目、杆菌目、放线菌目、蓝藻目和乳酸杆菌目依次是已报道基因组数据中蕴含BGCs最为丰富的Top10类群(表1)[17]。因此,利用分类学为导向优先从富含BGCs的微生物类群中挖掘药用先导分子是一种行之有效的策略。
根据泛基因组学的定义,编码次级代谢途径等菌株生长非必需的相关基因组成了特定微生物种属的非必需基因组[18],它们反映了物种的多样性和进化优势。因此作者认为,富含BGCs的微生物类群应具有高度开放的泛基因组,进化速度快,基因得失、倍增、水平转移或基因组重排事件频繁,同时也造就了更加丰富的种及以下分类单元的多样性。这一论断也得到链霉菌属和曲霉属等具有庞大种群数量的支持。截至2022 年 5 月 28 日,LPSN数据库公布链霉菌属的有效种为696个,也是MIBiG 数据库中BGCs条目最多的属,达到638个;曲霉属超过750个种(NCBI中数据),是MIBiG 数据库中BGCs条目第二多的属,为88个;芽孢杆菌属和假单胞菌属的有效种分别为103和299个,对应MIBiG 数据库中条目数分别为56个和68个。总之,上述富含BGCs类群往往具有较强的开放型泛基因组,且拥有较大数量的物种组成和多样性[19-20]。
“潜力菌株”所在类群具有高度开放的泛基因组,这是它们适应环境多样性的结果。比如评估链霉菌属泛基因组的流动性值揭示了它们暴露于不同生活方式和栖息地而形成的巨大多样性,它们容易通过水平转移获得遗传物质,从而更好地适应环境[19]。毫无疑问,这也导致了链霉菌较大的基因组和丰富多样的蛋白质编码基因。同样,弗兰克菌属泛基因组的流动性值高达0.9,说明该群体的个体拥有更多独特的基因[21],鉴于该属是富含BGCs的类群,提示该属的BGCs分布具有亚种甚至菌株的特异性。
特殊生境即特殊生态环境(special eco-environment),指在结构和功能上具有明显的异质性或特殊性,并导致生态元的数量或品质明显不同的生态环境。因此特殊生态环境决定了微生物的物种多样性,也塑造了它们独特的基因组。一个有趣的发现是链霉菌属的两株菌CNQ-509 和WAC 06738属于同一进化分支,但它们分别来自海洋和土壤,这种不同生境来源造就的主要区别是它们基因组中NRPS(non-ribosomal peptide synthetase)和I型PKS(polyketide synthase)基因簇的不同数量[19]。可见,特殊环境对微生物群体中独特BGCs的塑造是特境微生物作为新NPs重要源泉的根本。
什么样的特殊生境更容易塑造出“天才”或“潜力”微生物呢?作者认为不应该是极端的生态环境,而应该是生物多样性丰富、化学信号交流频繁,且具有一定空间独立性的微生境。其中植物内生的真菌具有成为高产NPs潜力菌株的巨大天然优势。主要原因如下:①地球上庞大的植物王国种类超过35万种,甚至不同地理环境和气候下的同种植物、同种植物的不同组织器官对其内生微生物都是一个独特的微生境,必然塑造出具有个体特异性的内生菌种群。可见,植物内生菌(包括真菌)是一个极其丰富多样的资源,同样它们也蕴含着数量庞大的NPs。②在长期的进化过程中内生真菌与其植物宿主逐渐形成了复杂的共生关系,它们参与了植物免疫系统的防御反应[22]。还有一些药用植物内生真菌,它们在生长过程中会产生与宿主相同或相似的生物活性成分[23],提示内生真菌相比其他微生物产生药用NPs的比例更高。③随着基因组学的发展,人们认识到,微生物NPs的丰富度与其基因组大小成正相关[11],而真菌作为基因组最大的微生物类群之一,蕴含着更加丰富多样的BGCs[12]。可以预见,植物内生真菌作为自然选择潜力菌株的大本营,将会成为未来药用NPs发掘的重要贡献者。
总之,作者建议富含BGCs“潜力菌株”的选择应优先遵循以下原则:①根据分类学导向,优先选择公认富含BGCs的类群(以属及属内特异分支为佳);②优选微生物的所在类群应广泛分布于不同生境且物种数量较多,具有开放的泛基因组;③依据系统进化树,利用与目标菌株处于同一或相近分支参考菌株或类群的基因组数据,参照预测目标菌株BGCs的潜能(以有可利用的种及种内菌株的参考基因组为佳),通过预测结果选择潜力菌株;④对于同一进化分支,微生物的去重复化非常必要,但根据相应类群的泛基因组学特征应有不同标准,如流动值高的要重视不同来源、不同表型的菌株;⑤除了表1提到的主要类群,富含BGCs的小众微生物类群也有不少值得系统开发(如异壁放线菌属、疣孢菌属等)。尽管本文更关注可培养的微生物类群,但相信随着宏基组学研究的深入,将有更多尚未被培养的蕴含丰富BGCs的类群被不断发现。
2 生物信息学预测
对于潜力菌株BGCs多样性、新颖性和合成产物结构的精准预测是微生物NPs发掘的基础与重点。研究初期,基于关键合成基因序列导向的分析方法,逐渐拉开了微生物NPs新结构预测的帷幕。如Hornung等通过对含有新型FADH2依赖型卤化酶的系统发育分析,推定相应卤化酶催化产生新型卤代化合物,并使用质谱验证了次级代谢物类别和卤化酶基因序列之间的显著相关性,从而快速发现了新的多环型xanthone类NPs[24]。随后美国Scripps海洋研究所Jensen研究小组基于PKS或NRPS缩合结构域KS(ketosynthase)或C(condenstion)的系统发育树而建立的在线分析方法NaPDoS (http://napdos.ucsd.edu/),可以预测聚酮或非核糖体肽类化合物的类别与结构[25]。尽管NaPDoS已被新的工具替代,但以关键合成基因序列导向的预测方法,在其他新颖同工酶及其催化合成产物的预测中仍持续发挥着重要作用[26]。
近十年来,随着基因组、宏基因组的测序成本不断降低,公共数据库中积累了大量可参考的基因组序列。同时越来越多NPs生物合成途径的阐明与新颖酶催化机制的解析,使得利用现有数据库预测BGCs成为可能。因此,催生了许多优秀的生物信息学程序包,如能快速鉴定整个微生物基因组中BGCs的antiSMASH(antibiotics & secondary metabolite analysis shell)[27];采用隐马尔科夫模型(hidden markov model,HMM)搜索策略,识别真菌基因组中 PKS、NRPS、hybrid-PKS/NRPS 和萜类BGCs中保守结构域的SMURF (secondary metabolite unknown regions finder)[28]等。目前,由诺和诺德基金会生物可持续性中心与丹麦技术大学维护的次级代谢产物生物信息门户网站SMBP为大多数NPs预测工具提供了一站式目录和链接,为个性化使用挖掘工具与访问数据库提供了便利[29]。SMBP网站包括了对PKS/NRPS结构域进行保守分析和功能预测的SBSPKS、预测腺苷酰化结构域活性位点结合底物的NRPSpredictor2、将PKS/NRPS基因簇数据与LC-MS/MS 数据关联的集成平台PRISM/GNP,以及使用系统发育基因组学来识别BGCs的EvoMining等。其中,antiSMASH能够快速鉴定和分析微生物基因组中的BGCs,并保持其功能的持续扩展与更新[27]。它利用最小信息和存储库MIBiG中已知功能BGCs,通过KnownClusterBlast模块比较分析已知BGCs来实现预测,并集成与交互连接了NCBI BLAST+、HMMer 3、Muscle 3、FastTree、PySVG和JQuery SVG等开源工具,成为当前最受青睐和使用最广泛的BGCs预测集成工具[30-31]。
值得注意的是,作者在使用antiSMASH挖掘真菌的NPs时,发现其对预测真菌BCGs中功能基因注释的准确率并不高,建议与SMURF预测结果比较确认,必要时还需结合2ndFind进行人工注释。2ndFind作为FramePlot 4.0beta的替代工具由日本国立传染病研究所维护,是利用已出现次级代谢相关蛋白的Pfam(protein family)结构域来搜索次级代谢生物合成蛋白的在线注释工具(https://biosyn.nih.go.jp/2ndfind/)。尤其它通过参考已知同源物种的次级代谢相关蛋白数据,并关联了BLASTP功能,可以实现基因编码蛋白与SwissProt数据库中蛋白序列的快速比对,从而获得更加严谨的功能注释,是人工注释基因功能的有力工具。
为了进一步挖掘微生物中蕴含BGCs的巨大潜能,Cimermancic等提出了基于已知BGCs中出现的蛋白质家族Pfam结构域频率的新算法ClusterFinder,使用HMM模型可在全基因组范围内预测潜在的BGCs,为非连锁BGCs协同合成未知类型化合物的预测提供可能[32]。他们从1 154个原核生物基因组预测得到33 351个可能的BGCs(评估的假阳性率为5%),高置信度的BGCs为10 724个,其中7 377 BGCs是在antiSMASH未被预测到的,并发现了尚未被表征的合成芳香多烯羧酸这类最大的BGC家族,这极大地提升了基因组挖掘的潜能[32]。而当antiSMASH选择加载ClusterFinder算法后,Swift等从肠道新美鞭菌纲的厌氧真菌基因组中相比SMURF预测出更多的生物合成基因和NPs类别,包括细菌素和脂肪酸类及糖类衍生物等[33]。可以预见,这些新算法的出现将为混合培养协同产生新颖NPs的预测提供分析工具。
当前,随着对先导分子药用功能的日趋重视,基因组挖掘技术从追求NPs的化学多样性向高效药理活性转变。2019年,Yan等使用抗性基因为导向的基因组挖掘策略从微生物中发现了一种新型天然产物除草剂aspterric acid,其通过靶向植物支链氨基酸合成途径中二羟酸脱水酶而显著抑制植物的生长[34]。最新的人工智能与基因组挖掘技术相结合,更是为NPs结构多样性高效挖掘与功能利用提供了典范。我国王军和陈义华团队整合了多种人工智能领域中自然语言分析(natural language processing)的神经网络方法,通过深度机器学习构建了准确率超过90%的抗菌肽预测模型,利用该模型对1万多个微生物组进行了小蛋白预测和抗菌肽挖掘,最后确定240余种为潜在新型的抗菌肽候选分子,并合成了其中216种;经实验验证,其中181种新型抗菌肽具有抗菌活性,发掘准确率达83.8%,部分活性接近迄今NPs中真核抗菌肽最强活性的表现,并对革兰阴性多重耐药菌具有较强的抑菌能力,在感染肺炎克雷伯菌的动物模型中有3个抗菌肽展现出良好的体内治疗作用和安全性[35]。综上可见,利用人工智能赋能微生物基因组挖掘深度拓展了自然界潜在跨物种BGCs和NPs总库容的认知,为NPs结构与生物活性关联提供线索,显著提高了微生物药物发掘的效率,是未来BGCs精准预测及其NPs定向发掘的发展方向。
3 沉默生物合成基因簇的激活
微生物具有丰富的编码次级代谢产物的BGCs,这赋予其生产新结构药用先导化合物的巨大潜能[36-37],但是广泛存在“隐性”BGCs在常规条件下是不表达或低表达的[38]。当前,高效基因编辑技术和合成生物学的兴起,给基因组挖掘带来了巨大的机遇。沉默BGCs激活的策略很多,且被研究者反复总结[13-14,39]。按照它们对激活BGCs的靶向性,可以分为靶向性激活和非靶向/多效性激活两大类[39]。这里按照激活作用的层次和位点,将内源性激活策略归纳为细胞水平(菌间的混合培养和菌内的培养调控)、细胞器/基因簇水平(表观遗传学、核糖体工程和代谢分流等)、基因水平(全局性调控基因、途径特异性调控基因、生物合成基因和前体供应相关基因等)、转录元件(启动子工程和转录因子诱饵)、蛋白水平(突变生物合成和翻译后修饰),外源性激活归纳为直接克隆、改造BGCs调控及优化宿主表达体系的异源表达,以及无需宿主细胞的体外重构(无细胞代谢工程系统和从头合成的无细胞合成系统[40])(表2)。
上述激活方法均有良好的激活效果且各有特点。比如,内源的靶向激活策略,能特异性激活,但需要全面认识目标BGCs,以及具备可行的遗传操作体系。表观遗传学和核糖体工程既可采用化学诱导剂,也可采用基因编辑的方式实现,但往往会出现“效应热点”,导致不同菌株的激活效应趋同。培养调控和混合培养随机性较强,但简单易行、使用广泛,且不受BGCs序列信息未知的影响,甚至可激活无法通过生物信息学预测的BGCs;当然它存在激活的“报酬递减效应”,当培养激活条件达到一定数量后,出现新激活效应将越来越难。BGCs激活时强化基因功能的策略有基因水平的基因倍增(过表达)和高活性基因置换(组合生物合成)、转录水平的强启动子置换、蛋白水平有利于提高酶活性的突变生物合成。在实际应用中这些激活方法经常联合交互使用,如基于遗传改造的激活方式与培养调控策略联合使用[41],以尽可能多地激活BGCs,提高微生物NPs的多样性,增加发现结构新颖药用先导化合物的几率。
尽管许多BGCs激活策略已被大家熟知并广泛应用,但近几年也提出了一些有启发意义的激活策略。如BGCs中的关键生物合成蛋白需要翻译后修饰才能发挥活性,在PKS和NRPS中的酰基或肽基载体蛋白(carrier protein,CP)初始为无活性的脱辅基apo-CP形式,需要通过磷酸泛酰巯基乙胺基转移酶(phosphopantetheinyl transferase,PPTase)修饰,将供体辅酶A 上的4-磷酸泛酰巯基乙胺转移到CP保守的丝氨酸残基侧链羟基上,才能成为有活性的全酶holo-CP形式。利用这一原理,我国瞿旭东团队首次构建了基于CP活性调控的翻译后修饰激活策略,通过在放线菌中过表达PPTase激活元件,实现约70%研究菌株中代谢物产量的增加[42]。转录因子诱饵(transcription factor decoy,TFD)是含有转录因子结合位点的一段核酸序列,其能与抑制型转录因子结合,使阻遏蛋白调控的沉默BGCs实现正常转录表达。基于此,研究人员构建了具有TFDs的载体,经结合转移,稳定游离的TFDs载体在链霉菌体内能引诱抑制转录的阻遏蛋白分子离开,成功激活了8个PKS和NRPS类型的沉默BGCs[43]。该研究为大型沉默BGCs的转录激活提供了新策略。此外,先进的CRISPR/Cas9工具盒,不仅在启动子工程激活等高效基因编辑中广泛使用,而且利用催化活性丧失的Cas9突变体(dCas9),将dCas9蛋白与转录激活因子和抑制因子融合,发展了激活基因转录的CRISPRa(CRISPR/dCas-mediated transcriptional activation)和干扰基因转录的CRISPRi(CRISPR interference)等系统,可调控微生物中次级代谢物生产的转录效率,实现沉默BGCs的可控表达[44-45]。
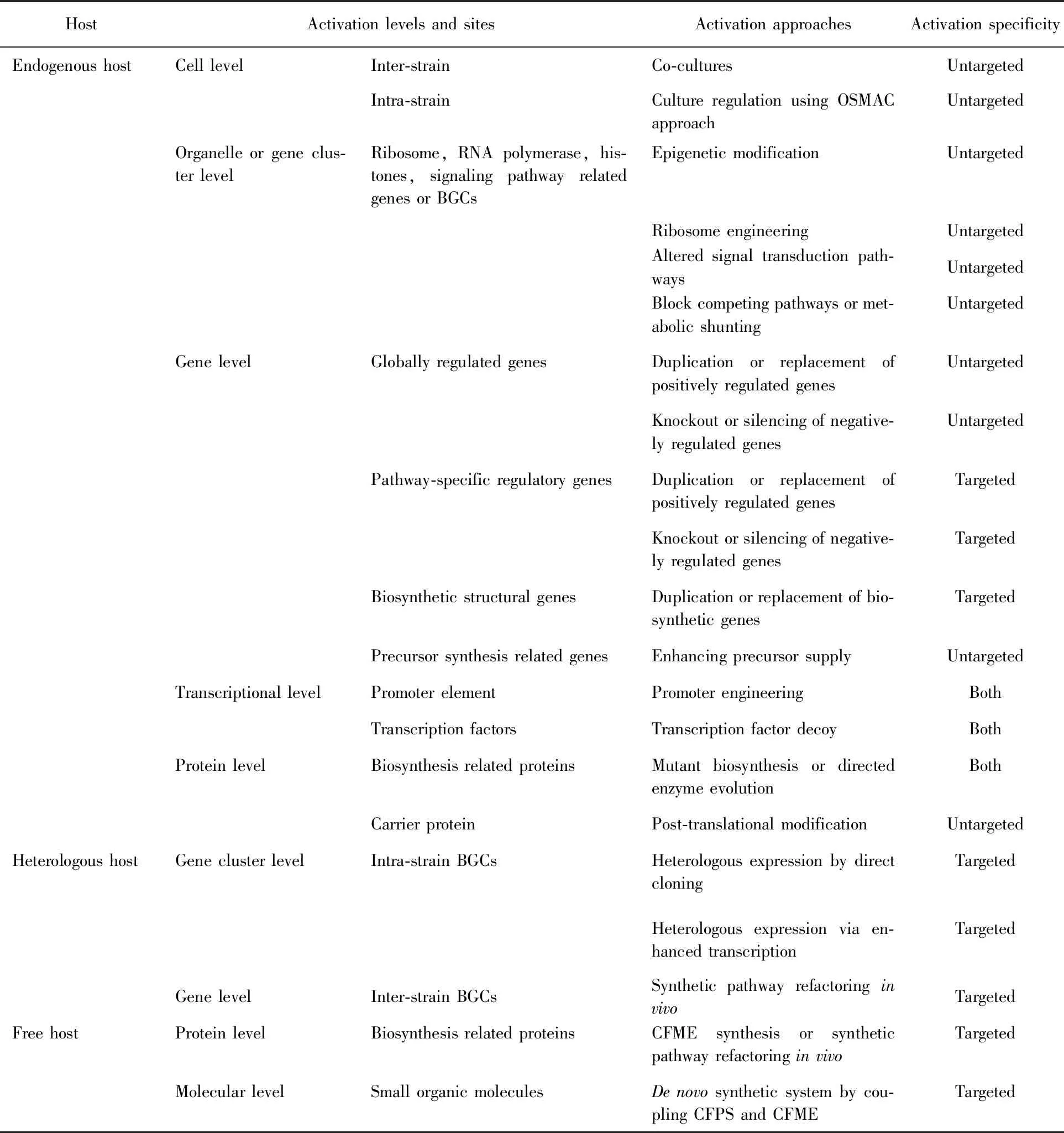
表2 沉默BGCs激活的策略与方法
事实上,次级代谢产物BGCs沉默或低表达的原因具有复杂性和多样性。正常的BGCs受到细胞内不同层级调控系统的严格控制,只有在特定的环境或培养条件下表达;同时其表达产物的产量水平(也可理解为产物对宿主细胞的反馈效应)可能取决于宿主细胞的稀有tRNAs、密码子偏好性、DNA修饰及各代谢途径的通量,也可能取决于产物稳定性以及其对宿主的毒性等因素。同时,BGCs中任何关键合成基因沉默或编码蛋白功能失活,也能导致BGCs沉默。因此,BGCs的激活策略也多种多样,不局限于这里提及的方法,凡是能避开现有宿主不同层级的调控系统,强化BGCs合成基因和前体供应相关基因的表达,以及编码蛋白活性提高的策略均可应用于沉默或低表达BGCs的激活。相信随着人们对BGCs沉默机制的认识不断加深,基因高效克隆、组装和编辑等新兴技术的不断推出,在合成生物学和人工智能的助推下,深入挖掘沉默BGCs蕴含的“暗物质”将使新NPs的发现更上一层楼。
4 激活产物的识别
尽管许多基因组挖掘策略在激活微生物BGCs中展现出良好的效果,但是在新颖先导化合物的高效发掘中,仅仅依靠激活目标BGCs或增加次级代谢产物的多样性是远远不够的,更重要的是激活BGCs及其产生化合物的高效识别。众所周知,从BGCs到活性物质的产生,要经历基因的转录、翻译和酶催化生物合成等阶段,因此在转录水平、蛋白水平和代谢物水平的检测,均能表征BGCs的激活情况。尤其是转录组、蛋白组、代谢组的发展,加速了BGCs表达的识别及其合成NPs的发现。比如,通过转录水平的挖掘能高效筛选出BGCs激活的方式以及相应的培养条件[46]。在蛋白水平,Gubbens等基于定量蛋白质组学技术建立了天然产物蛋白质组学发掘策略,该方法将生物合成蛋白水平的变化与次级代谢产物的产生联系起来,还能利用生物活性和蛋白质表达谱之间的相关性,快速发现负责生成目标NPs的BGCs[47]。
编码次级代谢产物的BGCs是否表达受到转录、翻译、翻译后修饰、功能蛋白质折叠组装等复杂机制的控制,所以转录或蛋白水平的检测,可以明确沉默BGCs激活时在每个阶段的表达状况,但并不能完全体现BGCs终端代谢产物的分泌情况。因此,基于代谢水平的高效识别方法对于微生物NPs的发现至关重要。目前没有一种化合物的分析方法可以无偏向性地涵盖所有代谢物的识别,基于色谱(LC和GC)、紫外吸收光谱(UV或DAD)、荧光光谱、红外光谱、质谱(MS)、核磁共振(NMR),甚至特定基团的显色反应都能作为激活产物识别的手段。包括Thin layer chromatography (TLC)、HPLC-UV、LC-MS、GC-MS、LC-NMR、NMR-PCA[48]等,但它们在分辨率、灵敏度、通量等方面各有特点。
基于质谱的分析具有灵敏度高、分析速度快、动态范围宽、结构信息丰富、样品用量少和通量高等优势,成为代谢物识别的主流分析平台,也在代谢组学研究中发挥着重要作用。质谱分析发展非常迅速,可根据化合物的理化性质、结构类型和检测目的,选择适宜的离子源(如EI、ESI、CI、APCI、FI、FAB、MALDI)和质量分析器(如Magnetic sector、Quadrupole、Ion traps、Time of flight、FTICR)。目前在微生物NPs领域使用最广泛、识别激活产物最高效的是色谱-紫外光谱-质谱联用技术(HPLC-DAD-MS),它通过分离粗提物、根据色谱峰的紫外吸收谱特征进行化合物分类、并结合质谱数据识别和发现目标激活化合物。如作者通过基因组挖掘发现芽孢杆菌SCSIO 05746具有产生4种NRPS类物质和3种PKS I型产物的潜能,然后使用OSMAC策略的培养调控进行这些BGCs的激活,采用HPLC-DAD-HRESIMS成功筛选到同时激活其中六种类型化合物的培养条件,并在正负离子扫描模式下对不同离子峰进行表征,识别了一系列新的bacillibactin、fengycin、bacillaene和macrolactin类型的衍生物,也为激活目标化合物的跟踪分离与结构鉴定提供指导[49]。
尽管上述色谱-质谱联用技术在产物识别上取得了很大成功,但一方面仅基于一级质谱数据很难获得可靠的化学注释,另一方面对原始数据采用人工识别和分析的方式,非常耗时费力。特别是对某一类群微生物NPs进行系统发掘时,不可避免地出现许多NPs的重复发现,不同的微生物类群在相同激活条件可能存在趋同效应,那么如何高效地排除不同菌株、不同激活条件下重复的NPs,发现新的目标分子?如何高效评价众多激活策略的效果,筛选更佳的激活方法?美国加州大学圣地亚哥分校的Dorrestein和Bandeira 团队开发了以HPLC-HRMS/MS为基础,结合了质谱碎裂数据分析和数据库搜索功能的分子网络技术(molecular networking,MN),较好地解决了上述问题。该方法根据MS/MS 质谱碎片的相似性建立了可视化分子网络图,直观显示了未知样品中各组成成分之间的关系和化学分类,解读了微生物表型和基因型之间的复杂关联[50]。随后他们倡导建立了GNPS(global natural products social molecular networking)开放网络数据库(http://gnps.ucsd.edu),供人们利用已处理或已识别的串联质谱数据(MS/MS),快速识别质谱图中已知成分和相关成分,从而促进目标分子的识别和追踪[51]。目前该技术应用十分广泛,已从微生物药用先导化合物发现扩展到整个药物发现和药物代谢等领域[52]。作者应用MN技术,从海绵来源链霉菌18A01中高效识别并靶向分离一系列具有显著己糖激酶II抑制活性的新颖α-吡喃酮类衍生物[53]。
尽管如此,MN也很难完整、准确地注释二级质谱中的庞大数据信息,而人工分析每个节点的碎片信息不能满足大数据集进行化学注释的需求。因此,面对如何更好注释代谢物的挑战,科学家开发了新的计算方法和碎片预测方法,极大提高了质谱数据的解析效率,实现了代谢产物的高效识别与解析[54]。如MS2LDA一种受文本挖掘启发的方法,将碎片光谱分解为一组保守的碎片和中性丢失特征(称为Mass2Motifs),它能识别同时出现与结构基序相关的离子,并将其分解成一组特征子结构Mass2Motifs,表示结构家族。目前还提供了用户可以Web访问的应用程序ms2lda.org[55]。DEREPLICATOR通过比较基于特定计算机碎片规则生成的理论光谱,系统地将肽类天然产物数据库中的结构与质量碎片光谱联系起来,能够注释具有相似氨基酸组成的肽类NPs。最近推出的 DEREPLICATOR+,将这种注释策略扩展到聚酮、黄酮、萜烯和其他类别的NPs[56]。网络注释传播 (network annotation propagation,NAP)基于对重叠结构指纹最合理的候选结构进行重新排序来改进类似物的注释准确性,通过光谱网络(spectral network)传播谱库匹配,进行计算机化学注释,实现NPs的高效识别与靶向分离[54]。
上述工具增强了从数据集中获取化学结构注释的能力,但是它们都有其自己的输出格式,阻碍了有效数据的匹配组合。因此,MolNetEnhancer整合MN、MS2LDA、NAP等的数据,通过ClassyFire对数据集中存在的类别进行自动化学分类,为代谢组学的数据提供更全面的化学概览,同时还能显示每个子结构模式的分子细节[57]。目前,这些先进的计算机注释工具已经被整合到GNPS平台,极大地弥补了现有公共存储库参考碎片谱不足的缺陷。使用增强的分子网络技术,Maimone等研究了立枯丝核菌共同培养诱导放线菌StreptomyceslunalinharesiiA54A产生具有抗真菌活性的次级代谢物,发现了仅在共培养时产生的158离子,并注释了去铁胺铁载体和茴香霉素衍生物两类物质,自动化化学分类发现最丰富的离子来自羧酸和衍生物(n=31)、异戊二烯醇脂质(n=15)等,以及共培养时产生相对数量更多的六种新结构化合物[58]。这一研究也为微生物微量激活产物的高效识别提供了有益启发。
以上详细论述了普适性最强、使用最广泛的非靶向代谢物检测与注释的最新进展,但还有适用于不同场景和目标的其他化学识别技术。比如被称为“分子显微镜”的质谱成像技术(imaging mass spectrometry,IMS)允许直接从生物样品中二维可视化代谢物、表面脂质、肽和蛋白质的分布,主要包括基质辅助激光解吸电离(matrix assisted laser desorption ionization,MALDI)质谱成像、解吸电喷雾电离(desorption electrospray ionization,DESI)质谱成像以及二次离子质谱(secondary ion MS,SIMS)成像等[59]。其中MALDI-IMS由于空间分辨率高和检测分子量范围广,在微生物肽类分子识别中广泛应用[60]。还有基于NMR精准预测结构的技术,如小分子精确识别技术SMART(small molecule accurate recognition technology)采用卷积神经网络将HSQC(heteronuclear multiple-quantum coherence)光谱与实验光谱库进行比较,识别结构相似的分子[61];MADByTE(metabolomics and dereplication by two dimensional experiments)则是基于TOCSY(total correlation spectroscopy)和 HSQC 光谱来识别复杂混合物中的独立自旋体系,通过匹配样品间的自旋系统特征创建各样品的化学相似性网络,通过匹配已知化合物光谱特征实现化合物去重复,优先识别有生物活性的成分[62]。值得一提的是TLC-bioautography和BioMAP(antibiotic mode of action profile)筛选模式是将化学特性与生物活性关联的识别方法,有助于活性NPs的快速发现。例如,BioMAP高通量平台通过匹配15种临床病原菌的抑制活性,依据抗菌谱类别实现了复杂混合物中潜在抗菌NPs的分类与识别,并高效和低成本地发现了结构独特的萘醌抗生素[63]。
总之,基于MS的化合物识别技术在不断发展,尤其当前机器学习赋能非靶向代谢组学的化学注释技术,实现了代谢物质谱数据集的高通量处理与化合物精准注释,将在微生物新NPs的发掘中发挥越来越重要的作用。当然,其他类型的化合物识别技术也各有特点,研究者应该根据实际需求选择使用。
5 系统学导向微生物产物发掘线路设计与展望
当前,癌症和感染性疾病严重威胁人类健康。据统计,全球每年有近 1 000 万癌症死亡病例[64],我国更是全球癌症高发率和高死亡率的“重灾区”[65]。在全球十大健康威胁中,和感染密切相关的多种疾病和行为占据了前10位中的6个[66]。同样,在农业领域,作物病害给农业生产造成了巨大的损失。尤其是植物真菌病害,按最低发病水平计算,可造成世界五大粮食作物每年减少1.94亿t[67]。可见,研制治疗癌症、感染性疾病的新型药物以及防治植物病害的新型绿色农药的需求巨大而迫切。另一方面,独特NPs的多样性是新药创制的源泉和基础,尤其在现代基因编辑技术、各类组学技术和人工智能的支持下NPs药物展现出巨大的发展潜能。因此,在后基因组时代NPs药物必将在需求和新技术的驱动下迎来又一个发展的黄金时期。
那么在新时代如何基于NPs的化学多样性,从中高效发掘微生物药用先导化合物是一个重要的基础性科学问题。为此,作者提出了基于潜力菌株优选和多组学导向的高效发掘新颖药用先导化合物的集成研究体系SPLSD(Systematic Pipeline for efficient Lead Structure Discovery from microbial natural products by promising strain selection and multi-omics mining)(图2)。首先选择动植物共附生微生物为优选的研究对象,测定其16S RNA或ITS(internal transcribed spacer)基因序列并构建系统发育树,初步认识其分类学地位以及基因型的独特性;然后文献调研或采用NCBI基因组数据分析其所在类群的泛基因组特征,依此进行分支菌株的选择和去重复(流动值低的可在种或亚种层次去重复,流动值高的要重视每一个独特来源的基因型菌株),并利用相近类群泛基因组数据参考分析选择菌株的BGCs丰富度、主要类别和新颖性,优选泛基因组开放程度高、流动值大、参考基因组富含新颖BGCs、且该进化分支或类群NPs研究尚不充分的菌株作为目标潜力菌株“promising strains”;对于所在类群泛基因组流动值较低且与参考菌株高度同源的,可以使用参考菌株基因组进行目标菌株BGCs的预测,对于相应流动值高的则还需要对选择的潜力菌株进行全基因组测序和生物信息学分析,精准认识其合成新颖次级代谢产物的潜能和特异性;随后对目标潜力菌株采用多种靶向或随机激活的策略(不局限于图2中展示的激活策略),改变其化学多样性或激活目标产物,并通过不同表达水平检测(转录和代谢水平)和效应物功能筛选,识别目标药用次级代谢产物的激活条件;最后使用活性导向和化学追踪的手段对目标产物完成分离、鉴定和活性评价,并通过结构优化不断改善成药性,实现新颖药用先导化合物的高效发掘。
针对SPLSD挖掘线路还有几点补充说明。①对于无需菌株本身基因组信息的激活策略,根据实际情况可不测定潜力菌株的基因组序列,但毫无疑问,基因组测序有助于全面了解选择菌株的潜能和特异性,也有助于利用靶向激活策略系统地发掘其蕴含的目标NPs。②antiSMASH等基于规则的预测工具对于含有尚未被阐明的生物合成新机制以及非线性催化合成的BGCs很难实现准确预测;同时在实践中往往由于化学结构多样性与其生物合成机制阐明的滞后性,导致生物信息学预测的新颖BGCs并不保证能产生新NPs。因此,基因组序列信息的挖掘与激活产物的化学识别相结合,不仅能筛选激活培养条件,还能互相印证、促进新NPs的发掘及其生物合成途径的阐明。③多样性导向的挖掘方法对于发现新颖NPs非常有效,但NPs的功能也应被优先考虑,因此针对特定功能和结构类型的NPs进行靶向发掘是非常必要的,也是SPLSD体系所提倡的。④非靶向NPs挖掘或靶向NPs识别困难时,对激活或多样性提高的提取物进行充分的活性筛选有助于提早发现NPs的功能和潜在应用方向。但对于为数不多的提取物进行多模型活性筛选是不经济的,因此非常有必要建立化合物差异的提取物库,并对其持续进行不同活性模型的高通量筛选,实现高值NPs的高效发现。
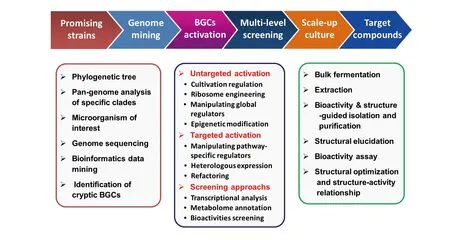
图2 基于潜力菌株优选和多组学挖掘的高效发现微生物天然产物先导结构的研究系统(SPLSD)Fig.2 The systematic pipeline for efficient lead structure discovery from microbial natural products by promising strain selection and multi-omics mining (SPLSD)体系中的激活策略不仅仅局限于图中列举的方法,任何能避开宿主的调控系统,强化BGCs合成基因和前体供应相关基因的表达,以及编码蛋白活性提高的策略均可应用于隐性BGCs的激活The activation approaches are not limited to these methods listed in the figure. Any strategy that can avoid the host′s regulatory system, enhance the expression of biosynthesis genes and precursor supply-related genes, and improve the catalytic activity of encoded proteins can be used to activate cryptic BGCs
在过去的实践中,结构多样性导向的新NPs发掘,会产生大量“无活性”NPs。据统计,过去五年海洋来源的7 500多种新NPs中绝大部分进行了细胞毒性、抗菌、抗真菌或抗炎活性的筛选,但占总数68%的新NPs不具有上述活性[68]。当前NPs 生物活性发掘侧重于癌症、微生物感染和炎症,这严重限制了NPs化学多样性在新出现代谢、免疫和神经退行性等疾病治疗,以及增强植物免疫、抗逆性和促生等方面的应用。作者倡导的NPs系统发掘线路SPLSD也强调扩展化学多样性与活性筛选相结合的重要性。小分子NPs与生物在自然界中长期共同进化,其生物学功能影响着宿主细胞特定的生理代谢过程,NPs作为“天生我才必有用”的自然进化产物,应充分进行各类生物活性模型筛选,相信随着通过更多细胞、酶/蛋白等不同功能和水平模型的高通量筛选,甚至虚拟药物筛选,NPs将会在生命科学研究、医药健康与现代农业生产中发挥越来越重要的作用。
还有,尽管NPs因为其结构的多样性和复杂性长期以来都是创制新药的重要来源和启迪,但是NPs直接成药却少之又少。据统计,去除9个植物源的提取药物,在1 202个小分子药物中,NPs直接成药的67个,仅占5.57%,而NPs衍生物药物为320个,达到26.62%[69]。特别在抗感染药(抗菌药、抗真菌药、抗寄生虫药和抗病毒药)中,NPs衍生物成药和其直接成药的比例高达83∶13,提高了约6.4倍[70]。农药领域NPs直接成药的仅占市场总量的4%,而以NPs为模板或启迪的占到50%[71]。药用NPs表现出“先导强、成药弱”的显著特点。这主要是因为NPs来源的药用先导化合物往往存在生物利用度差、代谢不稳定、副作用明显等缺陷。因此,以新颖的药用NPs骨架为先导,在保持其结构复杂性和药理活性的基础上,通过持续的结构优化,改善其药学性质,是创制新药的有效途径。而我国作为新NPs发现的大国,应加强自主发现NPs的化学修饰,通过持续改善其ADMET特性(absorption、 distribution、 metabolism、 excretion、 toxicity),引导实现创新药物的成功开发。
此外,尽管作者强调了可培养微生物中NPs的挖掘方法,但是随着宏基因组学、代谢组学、合成生物学和人工智能等的飞速发展,让非培养依赖的NPs挖掘技术限制因素越来越少。尤其适合大数据集处理的ClusterFinder等新算法和MolNetEnhancer等化合物注释工具,揭示了自然界蕴含BGCs的无限潜力,使得包含潜在跨物种的“Pan-BGCs”合成的“非自然”NPs挖掘变成了现实,极大地扩展了自然界总的NPs库。同时,基于合成生物学理念RetroPath、optStoic、RxnFinder等工具的建立[72],也为Pan-BGCs预测产物的人工合成提供了便利。可以相信,在新技术的推动下非培养依赖的挖掘策略有望成为未来新NPs产生的重要途径。
毫无疑问,特境微生物是巨大的资源宝库,其丰富的内涵已表现出了无限潜力。海洋微生物NPs经过20年的高速发展,现已进入平台期,相信未来动植物共附生微生物,特别是植物内生真菌是挖掘新NPs的优良资源和增长点。同时多组学技术和机器学习的不断发展增加了人们对NPs产生途径的理解和新NPs识别,当前高效的基因克隆与编辑技术、基于合成生物学理念的合成途径重构使得非培养依赖NPs的获取变得容易。此外,持续加强NPs成药性和可及性研究是更多NPs从实验室走向市场的关键。总之,充分利用特境微生物的物种多样性、遗传多样性和化学多样性,整合先进的生物技术、化学技术和计算技术,提高特境微生物先导化合物的发现速度和效率,持续推进NPs成药性和可及性的中下游研究,微生物NPs必将创造更大的社会效益和经济效益。