利用NGS技术同时检测缺失和点突变型地中海贫血
黄杰 杨旭 孙楠 孙彬裕 曲守方★
α-地中海贫血和β-地中海贫血是常见的常染色体隐性疾病。α地中海贫血主要是由于α-珠蛋白基因缺失导致的。--SEA缺失型α-地中海贫血是中国最常见的遗传性疾病之一,--SEA缺失型携带者无明显症状或显示轻度贫血,然而夫妻双方都是--SEA携带者怀有的胎儿就有25%机会患Hb Bart's胎儿水肿综合征[1]。除此之外,-α3.7和-α4.2也是最常见的α-地中海贫血。α-地中海贫血除了主要由缺失导致外,也有少部分是由于α-珠蛋白基因点突变引起,中国常见的有3种突变型分别是αWS、αCS、αQS。世界上目前为止至少发现了35种缺失型[2]和68 种突变型[3]。β-地中海贫血主要是由于 β-珠蛋白基因突变引起的,包括单碱基突变,小的插入或缺失,世界上已发现200多种β-珠蛋白基因突变型[4],中国已报道了 46 种[3]。除了基因突变之外,也有少部分β-地中海贫血是由于β-珠蛋白基因缺失导致的,目前至少发现12种缺失型[3]。
本研究使用下一代测序技术(next-generation sequencing technology,NGS)对来自中国食品药品检定研究院提供的地中海贫血核酸国家参考品进行检测,通过生物信息学,分析基因点突变(mutations)、小的插入缺失突变(indels)和缺失型(deletions)等信息,从而对地中海贫血基因变异作出准确的判断,为后期地中海贫血患者的变异检测提供基础,也为其他遗传性疾病的诊断和预防提供参考和研究方向。
1 材料与方法
1.1 材料
中国食品药品检定研究院提供的32例地中海贫血核酸国家参考品的DNA,具体突变型别见表1。
1.2 仪器与试剂
Applied Biosystems 2720 Thermal Cycler仪器和QubitTM3 Fluorometer仪器购自美国Thermo Fisher Scientific公司,DA8600测序仪购自中山大学达安基因股份有限公司;Hi-Fi Hotstart Ready Mix购自美国KAPA BIOSYSTEMS公司,Ion Xpress™ Plus Fragment Library Kit、Qubit dsDNA HS Assay Kit、Ion PITMHi-QTMOT2 200 Kit和 Ion PITMHi-QTMSequencing 200 Kit均购自Thermo Fisher Scientific公司。
1.3 引物
引物:具体引物序列如表2,在生工生物工程(上海)股份有限公司合成。
1.4 检测方法
1.4.1 引物Mix的配制
将1.3中15条引物分别稀释至100 μmol/L,各取1 μL进行混合。
1.4.2 PCR扩增
反应体系:10 μL Hi-Fi Hotstart Ready Mix,4 μL引物 Mix,60 ng DNA模板,双蒸水补齐到20 μL。
反应条件:94℃预变性3 min;94℃变性45 s,62℃复性 1 min 30 s,72℃延伸 2 min,5个循环;94℃变性 30 s,59℃复性 1 min,72℃延伸 2 min,30个循环;72℃最后延伸7 min。

表1 样本信息Table 1 Sample information
1.4.3 文库构建和定量
使用Ion Xpress™ Plus Fragment Library Kit进行文库构建,按照说明书进行操作。使用QubitTM3 Fluorometer进行文库定量。将文库稀释至200pmol/L,32个不同特异性接头文库等量混合。

表2 引物序列Table 2 The primer sequences
1.4.4 模板制备与富集
取1.3.3中的文库混合液12 μL,使用Ion PITMHi-QTMOT2 200 Kit进行模板制备与富集。
1.4.5 上机测序
使用Ion PITMHi-QTMSequencing 200 Kit对1.3.4的阳性模板进行测序,按照说明书操作。
1.4.6 结果分析
突变型别分析:使用测序仪配套服务器和软件进行序列比对和分析,运行Variant Caller插件,得出的xsl文件中“Allele Call”选项中筛选“Homozygous”和“Heterozygous”分别为杂合型和纯合型。
缺失型别分析:针对chr16:198728-235908和chr11:5190831-5275157范围内的每个位点depth进行分析,比对质量值为Q10,以染色体位置为横坐标,相应depth为纵坐标分别作图。
2 结果
2.1 点突变型和小的插入缺失型检测结果
本研究对32例国家参考品检测情况分析结果显示,22例携带基因突变。携带α珠蛋白基因突变3例,分别为13(QS杂合),21(CS杂合),22(WS杂合),其余19例携带β珠蛋白基因突变,样本26的检测结果为chr11:5247993delAAAG突变频率为100%,为Codons 41/42(-TTCT)纯合突变,30检测结果为 chr11:5247153G>A,突变频率为100%,为IVS-II-654(C>T)纯合突变。其他样本检测的突变频率为37.2%~54.2%,为杂合突变。具体结果见表3。
2.2 缺失型检测结果
对32例样本进行分析,对chr16和chr11的目标区域位置测序深度作图,如图1至图3。其中图1为野生型样本chr16和chr11的目标区域的测序深度分布图,只在chr16:221913-221936&chr16:223631-223612和chr11:5246996-5248448内有峰,其他位置无检测峰。图2显示的是α缺失型样本的chr16目标区域测序深度分布图。样本19、29在chr16:219303-219327和chr16:225173-225148范围内有峰为-α4.2;样本7、8在chr16:221913-221936和chr16:227726-227702 范围内有峰为-α3.7;样本5、6、7、30 在 chr16:235908-235888 和 chr16:215284-215308范围内有峰为--SEA缺失型;样本32 在 chr16:198728-199881 范围内有峰为--THAI。图3显示的是β缺失型样本的chr11目标区域测序分布图。样本25、26在chr11:5190831-5191339范围内有峰为Gγ+(Aγδβ)0型,样本 31 在 chr11:5222804-5223180和 chr11:5250214-5250590范围内有峰为SEA-HPFH型。
本研究的32例国家参考品检测情况分析,其中11例携带基因缺失。携带α珠蛋白基因缺失 8 例,样本5、6、30为αα/--SEA,7为-α3.7/--SEA,8为αα/-α3.7,19 为 αα/-α4.2,29 为-α4.2/-α4.2,32 为 αα/--THAI。3例携带β珠蛋白基因缺失,25、26为Gγ+(Aγδβ)0杂合,31为SEA-HPFH 杂合,见表 4。
3 讨论
地中海贫血主要是由珠蛋白基因异常引起的。目前,检测珠蛋白基因异常的方法包括特异性突变检测和整体基因组扫描。特异性突变检测利用常见的患者α-珠蛋白和β-珠蛋白突变信息(缺失、点突变或基因重组)形成的检测方法,使用基于PCR方法来识别这些已知突变,如跨越断裂点 PCR(Gap-PCR)[5]、反向点杂交(reverse dot blot,RDB)[6]等方法。针对整体基因组扫描,大量报道关于应用NGS检测α-珠蛋白基因(HBA1、HBA2)和β-珠蛋白基因(HBB),能检测到常规方法不能检测到的罕见突变及新发突变,降低漏检风险[7-8]。我们的研究旨在捕捉α-和β-珠蛋白基因簇关键区域,进行深度测序。考虑到地中海贫血跨种族的广泛突变谱和高患病率[9-10],将本研究的方法用于孕前的携带者筛查可以提供高危夫妇的检出率,用于疾病的辅助诊断可以提高疾病的检出率。
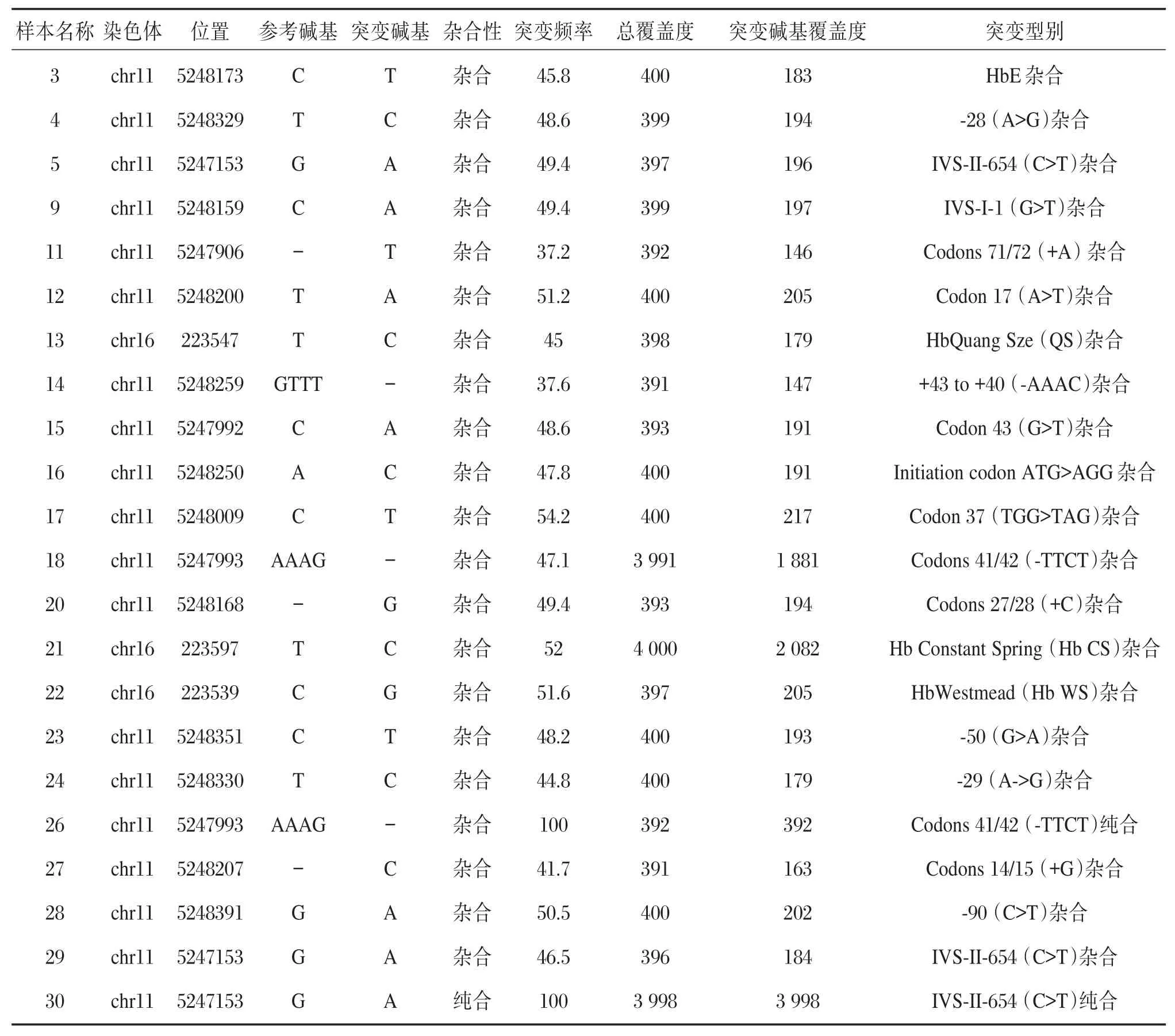
表3 样本点突变类型和小的插入缺失型检测结果Table 3 Results of samples'mutation and indel types
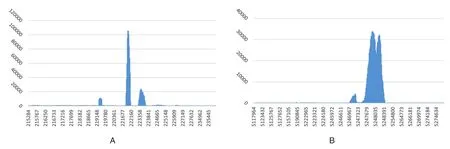
图1 野生型样本2目标区域的测序深度分布图Figure 1 Sequence depth map of target regions of sample 2 with wild type
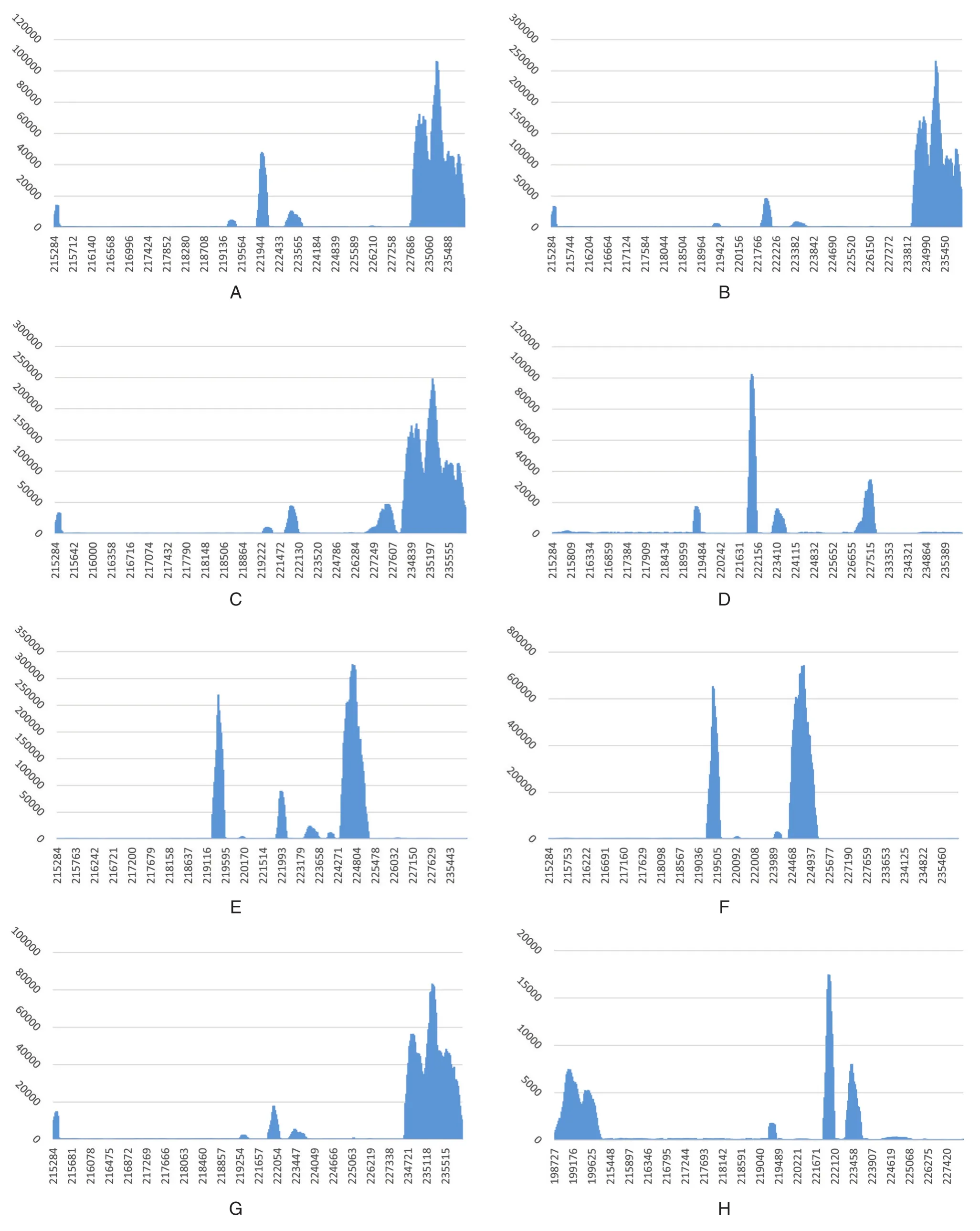
图2 α缺失型样本的chr16目标区域测序深度分布图Figure 2 Sequence depth map of chr16 target region sequence of samples with alpha deletions
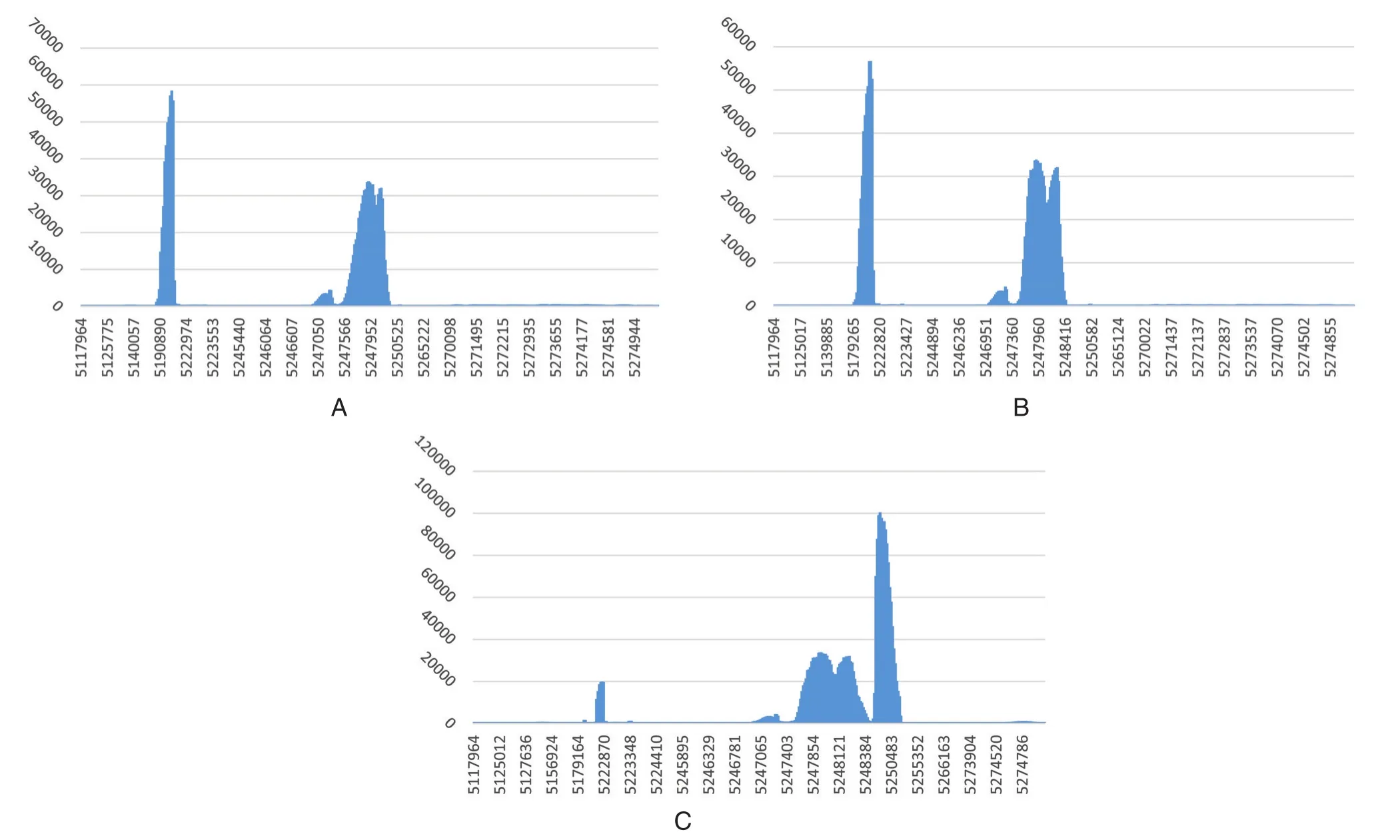
图3 β缺失型样本的chr11目标区域测序深度分布图Figure 3 Sequence depth map of chr11 target region sequence of samples with beta deletions
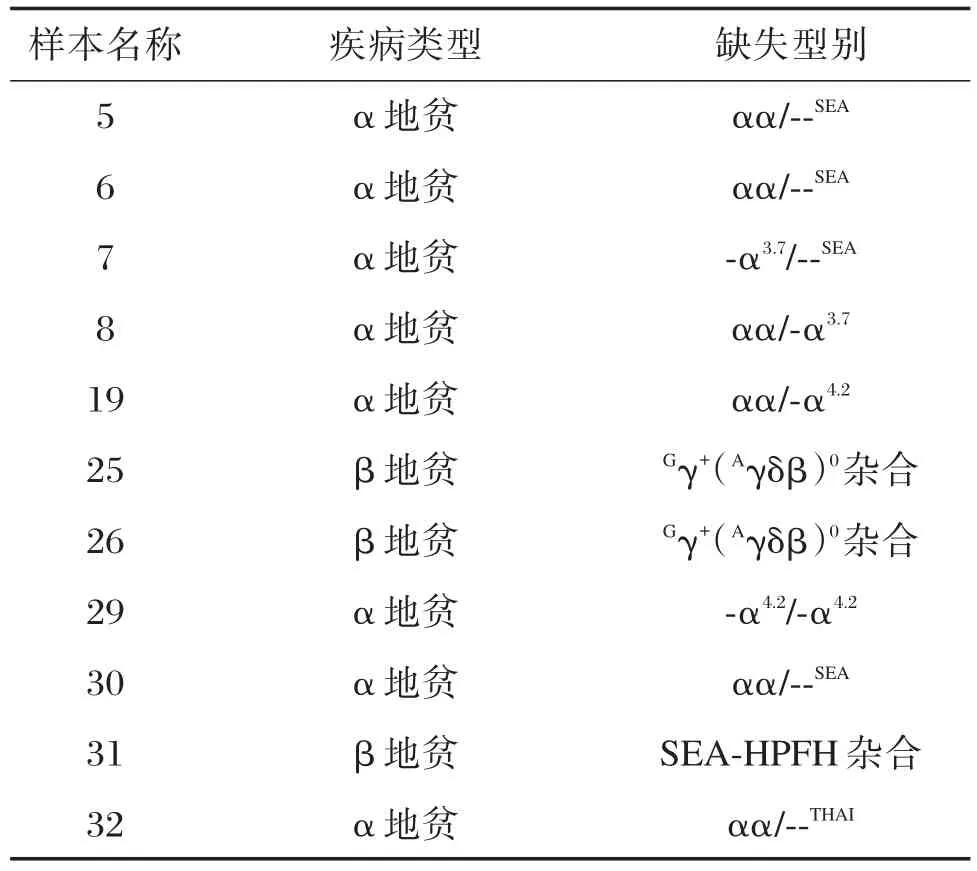
表4 样本缺失类型检测结果Table 4 Results of samples'deletion types
在大多数基因检测实验室,NGS实验操作已经流程化,对于基因突变,Ion torrent平台自带的插件能够进行分析,但对于缺失或者拷贝数变异(copy number variation,CNV)等,则需要重新建立生物信息学分析算法。本研究首先利用Gap-PCR原理对 α 地贫的--SEA、-α3.7、-α4.2及--THAI型缺失和β 地贫的 HPEH-SEA 和Gγ+(Aγδβ)0缺失的两端序列进行引物设计,同时分别设计一对用于扩增正常α珠蛋白和β珠蛋白基因的引物对照,对样本DNA进行一次性多重PCR扩增,扩增产物进行高通量测序和分析。这与谭梅等[11]报道的使用Gap-PCR产物同时检测地中海贫血缺失型及突变型的思路比较相似,不同的是本研究建立了基于CNV分析的生物信息学分析算法,无需琼脂糖凝胶电泳来检测Gap-PCR产物的长度。由于α珠蛋白2个基因HBA2和HBA1有着高度同源性,本研究将比对质量值提高到Q10,结果显示7号样本,8号样本,19号样本的结果均符合要求。
除此之外,与传统Gap-PCR相比,该技术在检测缺失型时增加了α-地中海贫血的--THAI型缺失型和β-地中海贫血的SEA-HPEH和Gγ+(Aγδβ)0缺失型。Clark等[13]通过探针捕获目标序列结合NGS技术,建立的生物信息分析算法也发现新的地中海贫血缺失型。Shooter等[14]通过该分析方法发现了一个59.0 kb长度序列(覆盖了HBG2、HBG1、HBD、HBB和OR51V1基因)的倒位和一个122.6 kb长度序列的缺失(覆盖了HBG2和HBE基因)。在检测基因突变型方面,测序区域包含了α珠蛋白和β珠蛋白基因整个区域,因此可以判断该基因内发生的任一基因突变,如本研究涉及了HBB基因的新突变类型-50(G>A)和-90(C>T)(见表2)。
在准确性方面,由于是地中海贫血国家参考品DNA原有型别已知,本研究的方法检测结果均与原有型别一致。而Shang等[12]利用NGS检测β地中海贫血突变,检测10 111对夫妇发现4 180人携带4 840个突变等位基因,通过一代测序验证,大部分一致。谭梅等[11]也使用NGS方法检测206例新生儿足底血发现6种α-地中海贫血和14种β-地中海贫血突变型,经一代测序验证准确性和特异性均为100%。说明NGS方法对于基因突变型检测具有较高的准确性。在灵敏度方面,本研究的方法仅需60 ng DNA进行6种缺失型和α-珠蛋白基因和β-珠蛋白基因检测,与其他对照方法检测这几种相比,样本量要求的更少。成本方面,由于地中海贫血的文库构建只需要普通的PCR扩增酶及连接酶等,测序数据量要求低,因此成本较低,适合大规模推广。
同时本研究也存在一定的局限性,即只能检测4种α-地中海贫血和2种β-地中海贫血缺失型别,对于新发现的缺失型别暂时还无法检出,还需要进一步的研究。对于基因突变方面,由于HBA1与HBA2基因具有较高的同源性,生物信息学分析需要首先去掉同源序列,导致部分基因信息丢失或者2个基因互相比对错误的情况。而对于HBB基因,可以达到全基因的100%覆盖。
除了地中海贫血之外,利用NGS技术对其他的几种常见单基因疾病如囊性纤维化[15]、肝豆状核变性病[16]及上百种遗传病 panel[17]等检测方法也已经建立。检测的方法也已经建立。由于NGS通量较大,因此可以结合笔者开发的地中海贫血检测方法,将多种孟德尔遗传性疾病综合检测,应用在婚前孕前的携带者筛查,实现多种疾病的快速、高通量检测。综上所述,使用NGS技术能够有效检出地中海贫血已知的缺失型别及未知的突变型别,本研究的方法的建立丰富了地中海贫血基因检测方法,同时为其他类似发病机制的研究提供了理论基础和技术支持。

