基于DNAzyme的d(GGA)重复序列折叠结构的可视检测
游 昕,贺荣荣,闫兴贺,南银杏,常天俊*,邴 涛
(1.河南理工大学 资源环境学院,河南 焦作 454003;2.中国科学院杭州医学研究所,浙江 杭州 310022)
G-四链体(G4)是由富含鸟嘌呤的核酸形成的特殊高级结构,可形成G4 的核酸序列在生物基因组中大量存在,与很多生理或病理过程密切相关。目前,G4的结构和生物功能研究聚焦于由三重G 串联形成的“常规”G4。然而,越来越多“非常规”G4,如两层四分体形成的G4[1]、空穴G4[2-3]等,被证实具有重要生物功能。d(GGA)三核苷酸重复序列是原癌基因c-myb启动子区的核心序列,已被证实在其基因转录中起关键调控作用[4];该序列也经常被发现存在于不同物种的其他基因中,被认为具有重要的生物学意义[5]。其中,4 组d(GGA)可形成由一个3’端G-四分体(T)和一个5’端G-A 七分体(H)形成的两层“非常规”G4,两个“非常规”G4通过5’端的七分体以“头碰头”堆叠形成T∶H∶H∶T二聚体(图1);8 组d(GGA)重复也能形成类似的分子内T∶H∶H∶T 堆积[4-5]。笔者所在研究组在水稻响应逆境胁迫重要基因的启动子区也发现具有d(GGA)三核苷酸重复的序列(OsHC-1 与flo2),但前者在体外生理条件下能形成G4,而后者只在G4 配体或高浓度盐离子存在下才发生折叠[6-7]。以上d(GGA)重复虽然具有相同的核心序列,但在不同序列环境中形成的结构却差异很大。因此,对实际序列环境中所形成结构的准确预测与检测成为研究其生物功能的前提。
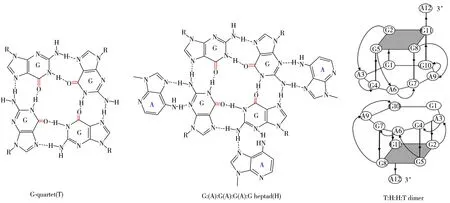
图1 G-四分体(T)、G-A七分体(H)和T∶H∶H∶T型G4二聚体结构示意图[4-5]Fig.1 Structure diagrams of G-quartet(T),G-A heptad(H) and T∶H∶H∶T G4 dimmer[4-5]
1996年,Li等[8]通过体外筛选技术获得了N-甲基卟啉二丙酸 Ⅸ(N-Methylmesoporphyrin Ⅸ,NMM)的核酸适体,并发现该序列可与血红素(Hemin)形成具有过氧化物酶活性的DNAzyme[9-10],而形成G4结构是其活性的基础。G4拓扑结构受其序列特征如四分体层数、loop 长度以及侧翼序列的影响,也受溶液条件如一价阳离子浓度、温度等的影响[11-14]。形成分子内平行的G4的DNAzyme活性一般要高于反平行、混合平行以及分子间G4[15],侧翼和loop 中的腺嘌呤(A)和胞嘧啶(C)可显著增强其DNAzyme 活性以及pH 耐受性[16-18]。对G4 的修饰也可提升其DNAzyme 活性[19-22]。这些研究阐释了G4 结构与其DNAzyme 活性的关系,目前基于DNAzyme 活性的分析方法已在G4 结构检测和配体发现、生物传感以及分子器件等领域得到应用[7,23-24]。
DNAzyme 活性分析在常规G4检测中有较多应用,但能否准确检测d(GGA)重复序列所形成的“非常规”G4 尚不清楚。鉴于d(GGA)三核苷酸重复序列形成的结构的特殊性以及其重要生物学意义,结合DNAzyme 分析的可视化与易操作性[7],本文尝试应用DNAzyme 活性分析对不同序列环境中d(GGA)的折叠结构进行快速可视检测。首先研究了d(GGA)4和d(GGA)8以及包含侧翼的实际序列在Hemin 存在下的DNAzyme 活性,并通过凝胶电泳及紫外-熔链(UV-Melting)分析了这些序列的二级结构和热稳定性;接着研究了不同侧翼、loop以及不同d(GGA)重复频次的序列所形成的二级结构,并与凝胶电泳结果相比较。通过以上研究,系统评估DNAzyme 活性分析用于d(GGA)重复序列所形成的结构的可靠性与适用性。
1 实验部分
1.1 试剂与仪器
所有DNA 均由生工(上海)股份有限公司合成纯化(序列见表1)。三羟甲基氨基甲烷(Tris)、N,N,N,N-四甲基乙二胺(TEMED)、丙烯酰胺以及甲叉双丙烯酰胺为分析纯,购自北京百灵威公司;氯化高铁血红素(Hemin)、2,2-联氮-二(3-乙基-苯并噻唑-6-磺酸)二铵盐(ABTS)购于Sigma-Aldrich 公司。硝酸银及其他化学试剂均为分析纯,购自国药集团化学试剂北京有限公司。Hemin用二甲亚砜(DMSO)溶解配制成10 mmol/L 母液,-20 ℃保存备用;ABTS用超纯水配成50 mmol/L的储存液,4 ℃遮光保存。
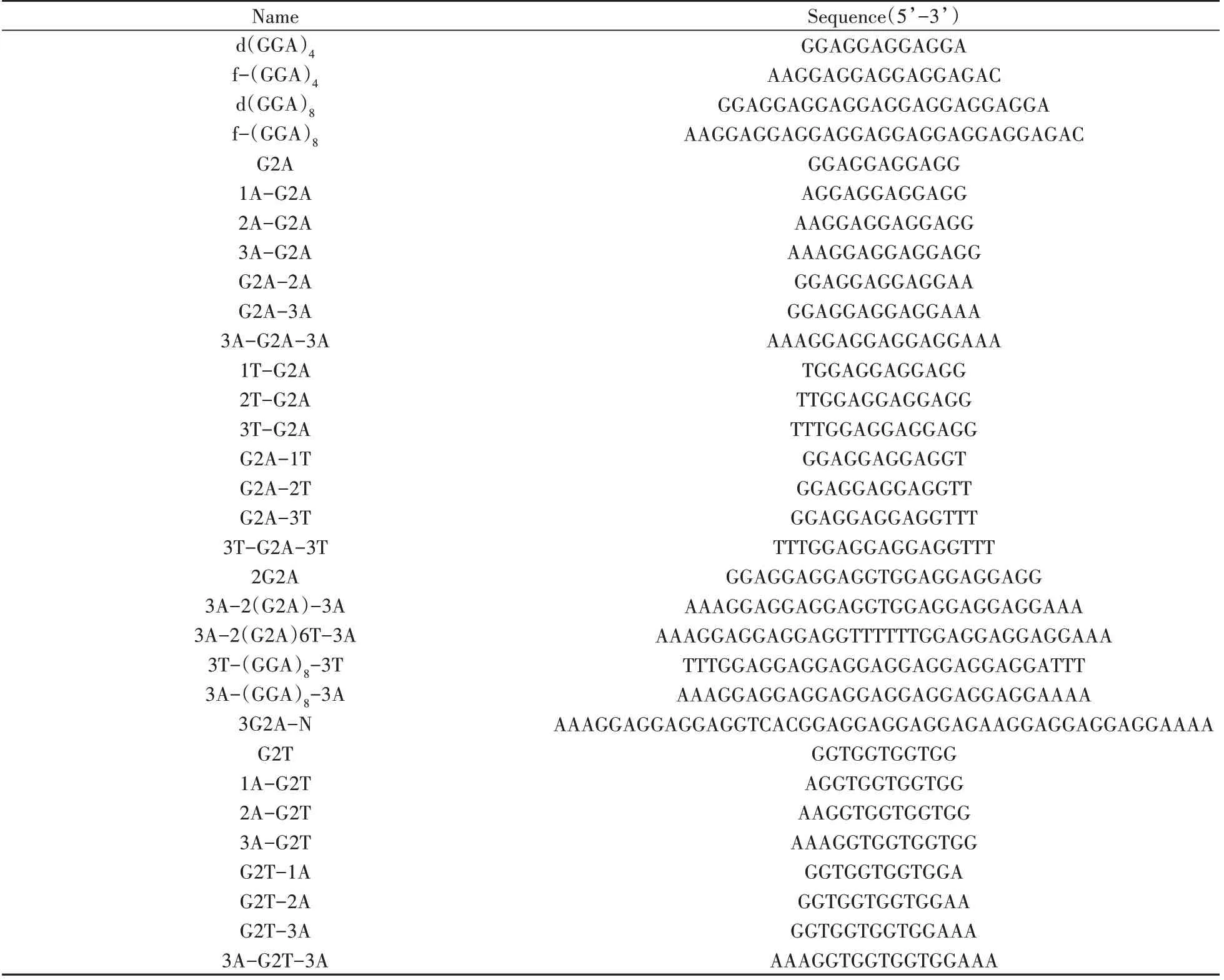
表1 本文所研究的核酸序列Table 1 The nucleotide sequences used in this work
UV-2550 紫外-可见分光光度计(日本Shimadzu),S-1700 温控设备(日本Shimadzu),垂直凝胶电泳仪(DYCZ-MINI2,北京六一),凝胶成像系统(美国Alpha Innotech),J-1500CD 光谱仪(日本JASCO),Milli-Q超纯水系统(德国Merck)。
1.2 DNA样品准备
DNA 样品以25 mmol/L Tris-HCl 缓冲液(pH 7.65)溶解,通过260 nm 处的吸光度值和摩尔消光系数计算浓度。为使其形成稳定的G4 结构,将DNA 溶液在含150 mmol/L KCl 的Tris-HCl 缓冲液中95 ℃变性10 min,缓慢复性至室温。
1.3 G4/hemin DNAzyme催化的ABTS氧化反应
G4/hemin DNAzyme 催化H2O2与ABTS 反应生成蓝绿色的阴离子自由基ABTS•-,该产物在415 nm 处有最大吸收峰[16]。将新准备的G4 与hemin 室温下遮光孵育1 h,然后加入2 mmol/L ABTS,最后以0.4 mmol/L H2O2引发反应。反应体系包含1 μmol/L DNA 与1 μmol/L hemin,0.025%(体积分数) Triton X-100、0.5%(体积分数) DMSO 和150 mmol/L KCl。采用紫外-可见分光光度计监测反应产物在415 nm 处的吸光度变化。利用吸光度与时间线性相关(最初20 s)的斜率以及ABTS 氧化产物的摩尔消光系数36 000 L•mol-1•cm-1计算氧化产物随时间的变化,获得DNAzyme 催化底物氧化的速率[16]。所有动力学测试至少重复3次。
1.4 聚丙烯酰胺凝胶电泳(PAGE)
变性聚丙烯酰胺凝胶电泳(Denaturing-PAGE):在1 μmol/L DNA 样品中加入等体积的去离子甲酰胺(95%),于 95 ℃变性10 min,冰浴冷却后及时上样,在含7 mol/L 尿素的20%聚丙烯酰胺凝胶(丙烯酰胺与甲叉双丙烯酰胺的体积比为19∶1)中于120 V 下恒压电泳,电泳缓冲液为1×TBE(含89 mmol/L Tris、2 mmol/L Na2EDTA、89 mmol/L硼酸,pH 8.3)。凝胶经银染显色,使用凝胶成像系统成像。
非变性聚丙烯酰胺凝胶电泳(Native-PAGE):在DNA 样品(含有1 μmol/L DNA/hemin、150 mmol/L K+)中加入等体积甘油(30%,体积分数),于20%聚丙烯酰胺凝胶中室温(25 ℃)60 V 下恒压电泳,银染显色。为了维持G4的结构,凝胶和电泳缓冲液中均加入20 mmol/L K+[11]。
1.5 Tm值测定
DNA(10 μmol/L)在含150 mmol/L KCl 的缓冲液中形成稳定结构。取400 μL置于石英比色皿中,在精密控温设备稳定10 min 后,以0.2 ℃/min 的速率降温或升温到一定温度(295 nm 处的吸光度达到稳定),温度每变化1 ℃记录一次样品在295 nm 处的吸光度;同时选择325 nm 处的吸光度作为参比进行校正以扣除基线漂移。采用已发表的方法计算Tm值[6],即以G4折叠分数(θ)对温度作图,Tm值为θ=0.5时对应的温度。θ由下式计算:θT=[A(T)-AU(T)]/[AF(T)-AU(T)]。其中θT为某一温度下的G4折叠分数,A(T)为测定温度下DNA在295 nm处的吸光度,AU(T)和AF(T)分别对应未折叠和完全折叠样品在295 nm处的吸光度。
受限于温控条件,对于在最低或最高温度下吸光度仍未达到稳定平台期的DNA 样品,以此温度下的吸光度作为未折叠或折叠基准来估算Tm值,因此实际Tm值低于或高于此估算值。
1.6 圆二色光谱(CD)
DNA 样品(5 μmol/L,含150 mmol/L KCl)经95 ℃变性10 min,室温缓慢复性处理后,于25 ℃测试其CD 光谱。在1 cm 光程的石英比色皿中加入400 μL样品,以100 nm/s的速度扫描3次,收集220~320 nm 的CD 信号。实验时预先扣除缓冲液的背景吸收,CD 光谱数据为扫描3 次的平均值,并经OriginPro 8.5软件进行平滑处理。
2 结果与讨论
2.1 DNAzyme活性分析
鉴于很多生物基因组中都发现了d(GGA)三核苷酸重复序列,并认为该序列具有结构相关的生物功能。为了快速检测该三核苷酸重复序列能否在体外折叠,合成了可形成T∶H∶H∶T型G4的典型序列d(GGA)4和d(GGA)8,以含有2~3个侧翼序列的f-(GGA)4与f-(GGA)8作对比,测试这些序列在血红素存在下的DNAzyme 活性(图2A)。结果发现d(GGA)4结合血红素催化H2O2氧化ABTS 的速率达到200 nmol/L•s-1,但f-(GGA)4的催化反应速率不到20 nmol/L•s-1,接近血红素自身的活性。d(GGA)8和f-(GGA)8的DNAzyme 活性分别为~300 nmol/L•s-1和400 nmol/L•s-1,是d(GGA)4的1.5~2 倍。反应20 min时,血红素对照和f-(GGA)4接近无色,d(GGA)4、d(GGA)8和f-(GGA)8样品为绿色到深绿色,这些样品裸眼可见的颜色与其反应速率一致。
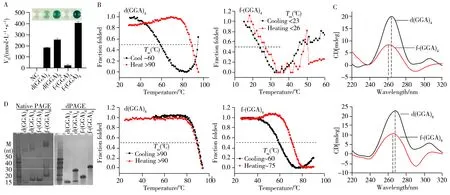
图2 d(GGA)三核苷酸重复序列的DNAzyme活性(A)、紫外-熔链曲线(B)、CD光谱(C)以及非变性和变性聚丙烯酰胺凝胶电泳图像(D)Fig.2 DNAzyme activity(A),UV-melting curvels(B),CD spectra(C),and the native and denaturing PAGE images(D) of d(GGA) trinucleotide repeats
2.2 结构和热稳定性分析
通过紫外-熔链曲线、CD 光谱、非变性和变性凝胶电泳评估这些序列的结构和热稳定性(图2B、C、D),发现d(GGA)4降温过程的Tm值约为60 ℃,而升温过程的Tm>90 ℃;f-(GGA)4升降温过程中的低温均未达到平台,不能准确测定其Tm值,估计的Tm值比d(GGA)4低40~60 ℃,表明常温下(25 ℃)难以形成G4。d(GGA)8和f-(GGA)8的热稳定性均较高(前者的Tm>90 ℃;后者的Tm值在降温过程中约60 ℃,升温过程约75 ℃),且它们的Tm值差异远小于d(GGA)4与f-(GGA)4。
CD光谱(图2C)显示d(GGA)4和d(GGA)8的正峰位于264 nm和267 nm,但f-(GGA)4和f-(GGA)8的正峰均发生了紫移,且CD 信号均大幅度下降;相对于其母序列,f-(GGA)4的正峰移动至260 nm,f-(GGA)8的正峰移动至265 nm。非变性凝胶电泳(Native PAGE,图2D)显示d(GGA)4(12-nt)、d(GGA)8(24-nt)以及f-(GGA)8(29-nt)的主要条带位于15~20-nt 或略大于20-nt,f-(GGA)4(17-nt)在非变性凝胶电泳中的迁移条带在15~20-nt 之间。结合紫外-熔链曲线、CD 光谱、凝胶电泳以及文献报道[4],认为d(GGA)4、d(GGA)8以及f-(GGA)8主要形成了T∶H∶H∶T型二聚体,而f-(GGA)4处于未折叠状态。变性凝胶电泳(dPAGE,图2D)还显示d(GGA)8存在接近15-nt 的快速迁移条带,提示其在变性条件仍保持部分折叠结构,进一步说明其分子内T∶H∶H∶T型二聚体高度稳定。
总的来说,d(GGA)三核苷酸重复的DNAzyme 活性与其形成T∶H∶H∶T 型二聚体的能力有关,说明DNAzyme 活性在其结构的可视检测中有应用潜能。此外,d(GGA)4和d(GGA)8形成的“非常规”G4二聚体的DNAzyme活性甚至高于形成三层四分体的G4[16],该结果扩大了高活性DNAzyme的范围。
2.3 讨 论
d(GGA)4和f-(GGA)8的升降温曲线存在较大的迟滞现象,与之前报道的可形成多种构型的不规则G4 序列类似[6]。一般来说分子内折叠优先于分子间的相互作用,因此d(GGA)4在降温过程中可快速形成分子内G4,而分子间G4的形成则相对较慢,从而滞后于温度的梯度变化;升温曲线中,d(GGA)4事先经历热变性/复性过程,已形成了稳定的分子间G4,Tm值表现为分子间与分子内G4 总体的解聚热力学,这可能是其升降温曲线具有较大迟滞现象的原因。而f-(GGA)8旁侧增加的碱基对其形成分子内聚集有一定阻碍作用,迟滞了其形成T∶H∶H∶T 聚集体的速度,因此相对于d(GGA)8显示明显的升降温曲线迟滞现象。以上现象也说明形成聚集可显著提高这些序列折叠的热稳定性,这为其执行生物功能提供了基础。
2.4 不同旁侧及不同重复频次序列分析
为了进一步评估DNAzyme 活性用于可视检测d(GGA)重复序列形成结构的适用性,以d(GGA)4为对照,首先研究了14 条具有不同侧翼和不同重复频次的d(GGA)序列。发现G2A(5’-GGAGGAGGAGG-3’)的活性接近250 nmol/L•s-1,略高于d(GGA)4;在其5’端增加A碱基,将导致活性明显下降,如增加3个A导致DNAzyme活性下降80%(图3A)。在两侧同时增加A或T,活性降低90%以上。然而,3’端增加A 或在5’端和3’端分别增加T 并未明显降低其活性。上述序列测得的催化反应速率与其反应20 min的显色结果均一一对应。
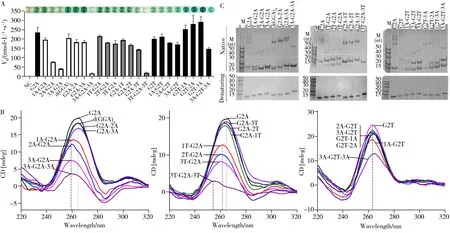
图3 含有不同旁侧序列的G2A的DNAzyme活性(A)、CD光谱(B)和非变性和变性聚丙烯酰胺图像(C)Fig.3 DNAzyme activity of G2A in the presence of different flanking sequences(A),CD spectra(B),and the native and denaturing PAGE images of these sequences(C)
CD 光谱(图3B)显示在G2A 的3’端增加A 或者T,其正峰波长均在265 nm 附近,且CD 信号未见明显降低,表明形成了类似d(GGA)4的聚集结构[6]。但是随5’端碱基个数增多,CD 信号下降加剧,表明5’端碱基不利于其形成G4结构。其中,增加A碱基的序列,CD 正峰紫移到略低于260 nm,提示G4结构成分很少;增加T 碱基的序列,CD 正峰移动到262 nm 附近,提示存在部分G4。另外,3A-G2A-3A的CD 正峰移至259 nm,负峰移至约246 nm;3T-G2A-3T 的CD 正峰移至254 nm;二者在相应组内的CD峰值最低,表明均难以形成G4。
在变性凝胶电泳中,以上序列均为单一条带且位置对应其实际序列长度;非变性凝胶电泳中,大部分d(GGA)重复序列存在多个迁移条带(图3C)。其中,G2A 的5’端加2~3 个A(2A-G2A 和3A-G2A)或两侧分别加3 个A 或T 的序列(3A-G2A-3A 和3T-G2A-3T) 的主要电泳条带在15-nt 附近,与其实际序列长度接近,表明这几个序列主要以未折叠形式存在;其他d(GGA)重复序列在15-nt附近都存在两个迁移条带:高于其实际序列长度的主要条带,提示为类似d(GGA)4的分子间聚集,另一个与其实际长度相当的含量较小的条带为单分子状态。凝胶电泳图像印证了CD 光谱对G2A 及其衍生序列折叠的判定结果。
以上数据显示,聚集状态占比多的序列的DNAzyme 活性普遍较高,而以未折叠为主的序列活性很低,进一步说明DNAzyme 活性与d(GGA)重复序列的折叠和聚集相关。此外,笔者还发现4组d(GGA)重复序列受侧翼的影响复杂:d(GGA)4可形成非常稳定的T∶H∶H∶T 型G4 二聚体(Tm>90oC),但5’端A 强烈抑制其形成G4 及二聚体,5’端T 的影响弱于A,3’端的A 或 T 则很少抑制其结构的形成;另外,3’或5’侧翼同时含有多个A 或T 碱基可强烈抑制其形成G4。考虑到实际序列一般有长的侧翼序列,4组d(GGA)重复在实际序列环境中将很难形成G4。
为了探讨d(GGA)重复序列受侧翼影响的现象是否具有普遍性,将G2A中的A替换成T(G2T:5’-GGTGGTGGTGG-3’),发现其DNAzyme 活性与G2A 相当(图3A),但其侧翼增加A 未导致明显的活性下降,甚至3’端增加的A 碱基可增强活性(如G2T-3A 的活性相对于G2T 增加了约50%)。CD 光谱(图3B)和凝胶电泳(图3C)都提示G2T重复序列以平行G4及平行G4聚集为主。以上结果表明d(GGA)重复的折叠受侧翼序列的影响具有特殊性,这可能与A碱基插入到G-四分体形成特殊的G-A七分体有关。
接着研究了6 条具有不同侧翼的8 组及12 组d(GGA)重复序列(图4),与G2A 和d(GGA)8对比,发现所有序列普遍有高DNAzyme 活性(DNAzyme 催化H2O2氧化ABTS 的初速度在200~400 nmol/L•s-1之间),对应样品反应后均具有明显可辨的深绿色(图4A)。非变性凝胶电泳表明这些序列与d(GGA)8类似,均形成多种聚集结构(图4B)。具有不同侧翼或连接子的8组d(GGA)重复序列,其CD 信号相对于2G2A 显著降低,但正峰并未明显移动。12 组d(GGA)重复序列(3G2A-N)的CD 正峰移动到274 nm 左右,但在265 nm 处仍具有较高的正信号(图4C)。这些证据表明所研究的6 条序列均能折叠成G4。然而,3A-2(G2A)6T-3A 与3G2A-N 的d(GGA)重复之间具有较长的连接子,其CD 信号较弱,相应的DNAzyme活性也较低,提示更长的连接子可能对d(GGA)重复序列的折叠或聚集不利。
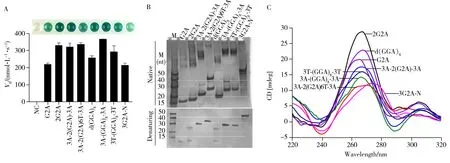
图4 8或12组d(GGA)三核苷酸重复序列的DNAzyme活性(A)、非变性和变性聚丙烯酰胺凝胶电泳图像(B)与CD光谱(C)Fig.4 DNAzyme activity(A),native and denaturing PAGE images(B),and CD spectra(C) of 8 or 12 d(GGA) repeats
上述多组d(GGA)重复包含了不同的侧翼序列以及连接子序列,其中3G2A-N 来自于人类c-myb原癌基因启动子区的一段具有多组d(GGA)重复的实际序列[4],因此实际序列环境中此类高频率重复的d(GGA)序列将更可能形成T∶H∶H∶T型G4而具有生物功能。结果表明DNAzyme活性分析对于检测真实序列环境中的多组d(GGA)重复所折叠的结构具有很好的适用性。
3 结 论
本研究发现T∶H∶H∶T 型“非常规”G4 的d(GGA)三核苷酸重复序列在血红素存在下具有高的DNAzyme 活性,其裸眼可视的颜色与形成结构有关。基于DNAzyme 活性可视检测d(GGA)重复序列的形成结构具有很高的可靠性和通用性,通过酶标仪进行高通量测试,可在20 min 之内完成反应,因此该类特殊序列的形成结构在实际序列检测环境中具有应用前景。另外,本文还发现d(GGA)重复序列的形成结构受到重复频次以及旁侧序列的特殊影响,丰富了对G4结构多态性的认识,启示重复频率高的d(GGA)在实际序列环境中更有可能形成稳定的G4聚集体,从而对基因的表达调控起作用。

