纤维素降解菌Raoultella ornithinolytica LL1的筛选及基因组测序
唐昊 孙灿 李沅秋 罗朝兵
(乐山师范学院生命科学学院,乐山 614000)
植物生物质主要由木质纤维素组成,是地球上最丰富的生物质组成部分。由于木质纤维素可作为生物燃料制备的可持续资源,因此其降解过程受到广泛关注[1]。纤维素是葡萄糖单体的聚合物,通过β-1,4-糖苷键连接,是木质纤维素生物质资源中最常见的成分之一[2]。大量文献报道,纤维素聚合物的降解需要多种碳水化合物活性酶(carbohydrate-active enzymes,CAZymes)家族成员的协同作用,这些酶家族之间通过多重互补和协调氧化、水解和非水解活性共同作用实现碳水化合物的降解[3]。关于纤维素水解成葡萄糖单体的过程,其需要3种酶的协同作用,包括内葡聚糖酶、外葡聚糖酶和β-葡萄糖苷酶[4]。在微生物中,如木霉属(Trichoderma)[5]、曲霉属(Aspergillus)[6]和青霉属(Penicillium)[7]等真菌分泌的纤维素降解酶已被广泛的研究,但细菌较真菌而言,具有生长速度更快、多酶复合物表达和极端环境的耐受性等优势,已成为目前纤维素酶生产研究的重点[8-10]。许多归属于芽孢杆菌属(Bacillus)[11]、类芽胞杆菌属(Paenibacillus)[12]、纤 维 菌 属(Cellulomonas)[13]、噬纤维菌属(Cytophaga)[14]等纤维素分解微生物都能产生纤维素降解酶。
研究表明昆虫共生微生物可降解由纤维素、半纤维素和木质素组成的植物细胞壁[15]。这些微生物包括细菌和真菌,帮助宿主昆虫利用植物细胞壁材料中的单糖。目前已有大量研究从昆虫肠道中筛离得到纤维素降解细菌,如黄胜威等[16]从暗黑鳃金龟幼虫肠道中分离得到假单胞菌属Pseudomonas,微杆菌属Microbacterium,德沃斯氏菌属Devosia等属中能够降解纤维素的细菌,其中Pseudomonass属中的消化还原假单胞菌Pseudomonas nitroreducens是暗黑鳃金龟幼虫肠道内最主要的纤维素降解菌;胡霞等和傅慧静等[17]探讨了松墨天牛(Monochamus alternatus)幼虫肠道细菌的多样性,并从中分离鉴定了以噬纤维细菌科细菌 Siphonobacter aquaeclarae 为优势菌等10个可降解纤维素的菌株;胡霞[18]从华山松大小蠹(Dendroctonus armandi)幼虫肠道中分离出 91 株纤维素降解菌,其中沙雷氏菌属为重要的纤维素降解菌等。
长足大竹象(Cyrtotrachelus buqueti)作为一种专食性害虫,严重危害竹产业的发展。Luo等[19-20]通过转录组测序等手段揭示了长足大竹象可降解慈竹木质纤维素,进一步通过微生物测序等手段发现长足大竹象肠道内存在大量的共生菌,并参与木质纤维素的降解,包括纤维素降解[21]。并且一些纤维素降解菌已被成功分离鉴定并表现出纤维素降解能力,如贝莱斯芽胞杆菌(Bacillus velezensis LC1)[22]。本研究以长足大竹象肠道为样本,利用CMC培养基和刚果红染色法筛选分离出具有较高纤维素能力的细菌Raoultella ornithinolytica LL1,通过基因组测序及分析揭示其降解纤维素的遗传基础,并且通过纤维素酶活的测定研究其纤维素降解能力。
1 材料与方法
1.1 材料
1.1.1 培养基配方 LB液体培养基:胰蛋白胨10 g/L,酵母提取物5 g/L,NaCl 10 g/L,pH调至7.0;CMC培养基:羧甲基纤维素钠5 g/L,NaNO33 g/L,KH2PO41 g/L,MgSO40.5g/L,KCl 0.5 g/L,FeSO40.01 g/L,琼脂15 g/L,pH调至7.0。
1.1.2 试剂配制 DNS试剂:200 g酒石酸钾钠加热溶于一定水中,再加入10.0 g 3,5-二硝基水杨酸、10.0 g NaOH、2 g苯酚、0.5 g无水亚硫酸钠加热溶解,冷却至室温后,定容至1 000 mL,常温保存;1% CMC缓冲溶液:200 mL蒸馏水中加入2 g羧甲基纤维素钠,用纱布过滤水浴加热后的溶液。取100 mL滤液、40 mL蒸馏水和20 mL的0.2 mol/L醋酸缓冲液(pH4.8)振荡混匀,4℃低温保存;1%水杨苷溶液:取1 g D-水杨苷和适量0.2 mol/L的醋酸-醋酸钠缓冲液(pH 4.8)于烧杯加热溶解,定容至100 mL,4℃低温保存;1%微晶纤维素:取1 g微晶纤维素和适量0.2 mol/L的醋酸-醋酸钠缓冲液(pH4.8)于烧杯中加热溶解,定容至100 mL,4℃低温保存;pH 4.8柠檬酸缓冲液:蒸馏水溶解稀释8.4 g柠檬酸和17.6 g柠檬酸钠至2 000 mL。
1.2 方法
1.2.1 菌株的分离筛选 长足大竹象样品采集于四川乐山市沐川县(东经103°98′,北纬28°96′)。在无菌条件下,使用无菌解剖工具解剖长足大竹象,挑出其完整肠道,于装有少量无菌水的灭菌研钵中研磨均匀。将肠道研磨液按浓度梯度稀释为 10-1-10-9,分别取0.1 mL的10-7-10-9的稀释液涂布于CMC培养基,30℃恒温培养。待培养基上长出单菌落后,反复在CMC培养基上划线接菌,对菌进行分离、纯化培养。CMC培养基用0.2%刚果红染液染色后用于筛选纤维素降解菌,水解能力比率 (HCR)用透明圈直径与菌落直径比值的大小表示[23]。通过镜检确保筛选的菌落为单菌落,并接种于斜面培养基,4℃保存。
1.2.2 细菌的分子生物学鉴定 以16S rRNA V3-V4 区为扩增区,采用27F(5′-AGAGTTTGATCMTGGCTCAG-3′)和1492R(5′-TACGGYTACCTTGTACGACTT-3′)分别作为正向和反向引物进行扩增。扩增子的检验使用1%琼脂糖凝胶电泳。通过MEGA7软件邻接法构建NCBI GenBank中检索到的序列和参考序列的系统发育树。PCR反应条件如下:94℃预变性2 min;94℃变性30 s,55℃退火30 s,72℃延伸100 s,循环30次;72℃修复延伸2 min。
1.2.3 葡萄糖标准曲线的绘制 配制0.1 mg/mL、0.2 mg/mL、0.3 mg/mL、0.5 mg/mL、0.6 mg/mL和0.7 mg/mL葡萄糖溶液。分别取上述浓度葡萄糖溶液0.1 mL、0.3 mL柠檬酸缓冲液(pH4.8)与0.6 mL DNS试剂于EP管中,振荡混匀后沸水浴5 min。4 mL蒸馏水在冷却后加入,混匀后用移液枪加0.2 mL反应液于酶标板,波长540 nm处测定OD值。以横坐标为葡萄糖浓度,纵坐标为吸光值做出标准曲线图,求得标准曲线方程为:y=0.977 9x-0.025 2,R2=0.992 4。
1.2.4 纤维素酶活的测定 粗酶液的制备:将1 mL菌液加入100 mL CMC培养基,30℃、200 r/min 下培养,分别取培养1、2、3、4、5和6 d后的培养液1 mL,4℃、4 000 r/min离心10 min,上清液经0.22 μm滤膜过滤灭菌后作为粗酶液。分别向0.3 mL的1% CMC、1%水杨苷或1%微晶纤维素溶液中加入0.1 mL粗酶液,50℃恒温反应60 min后加入0.6 mL DNS显色剂,经过5 min沸水浴令反应终止。酶标板中加入0.2 mL反应液,OD值于540 nm波长下测定。每组实验设置3个重复,空白对照为高温灭活的粗酶液。
1.2.5 基因组DNA提取与测序 将单菌落接种到LB液体培养基中,菌株在30℃、170 r/min摇床中生长2 d后,使用离心机8 000 r/min转速离心3 min收集菌体。菌株基因组DNA的提取采用CTAB法[24],纯化用中国南京诺唯赞生物科技有限公司生产的Wizard 基因组DNA纯化试剂盒。全基因组测序由上海美吉生物医药科技有限公司完成。利用HGAP 2.0过滤并进行基因组组装[25]。
1.2.6 基因预测和功能注释 用Glimmer 3.0预测编码基因的编码区域。将预测到的基因采用BLAST比对到NCBI非冗余蛋白库(NR)、蛋白质直系通源簇(COGs)数据库、Swiss-Pro、GO和KEGG数据库中进行基因功能注释。在生物信息学中,GO是一个重要的工具,并且统一表达了所有物种中的基因和基因产物[26]。KEGG是一个了解高级功能的数据资源库,也可以分析代谢途径。此外,AZy数据库,利用HMMER[27]软件与数据库比对,取e<1e-10的注释。
2 结果
2.1 纤维素降解菌的分离及鉴定
利用羧甲基纤维素钠(CMC)选择培养,从长足大竹象成虫肠道中分离出PX19、PX20、PX21 3株纤维素降解菌。随后将这3株菌分别涂布于刚果红培养基,观察降解圈并进行测量。结果表明此3株菌菌落周围均有明显降解圈(图1)。通过测定降解圈的大小并计算各菌株HCR值。菌株PX19(HCR:4.83)显著高于PX20(HCR:1.59)和PX21(HCR:1.51)(图1),表明菌株PX19纤维素降解能力最强,因此对PX19进行进一步研究。
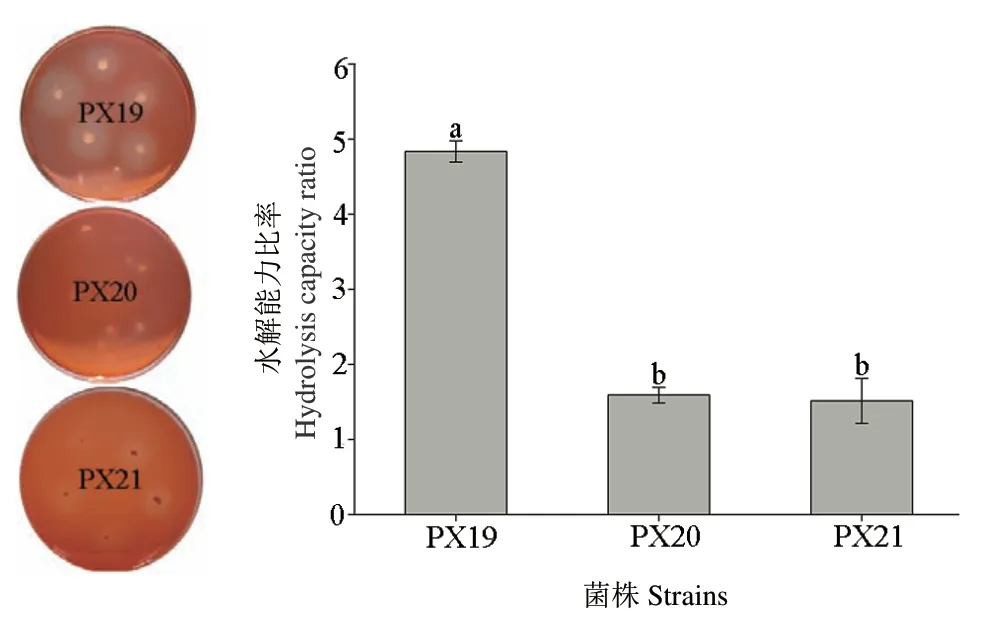
图1 纤维素降解菌PX19、PX20和PX21刚果红染色对比Fig.1 Comparison of Congo red staining of cellulose degrading bacterium PX19, PX20 and PX21
利用引物27F和1492R扩增16S rRNA基因序列并测序用于PX19的鉴定。PX19的16S rRNA基因序列长度为1 500 bp(登录号:CP061719-CP061720)。将16S rRNA基因序列提交NCBI,并进行序列blast分析并构建系统进化树。结果显 示PX19与Raoultella ornithinolytica B6(NZ_CP012555.1)菌株相似性最高,相似度达到98%,表明PX19与R. ornithinolytica B6亲缘性最高(图 2)。因此,PX19被鉴定为R. ornithinolytica,并命名为R. ornithinolytica LL1。
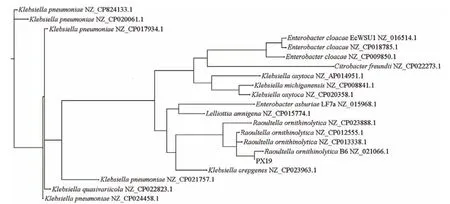
图2 纤维素降解菌PX19的鉴定Fig.2 Identification of cellulose degrading bacterium PX19
2.2 R. ornithinolytica LL1纤维素酶活
为了探究R. ornithinolytica LL1纤维素降解能力,测定了该菌株在羧甲基纤维素(CMC)培养中的纤维素酶活,包括内切葡聚糖酶、外切葡聚糖酶和β-葡萄糖苷酶。结果表明3种纤维素酶中,β-葡萄糖苷酶活性最高,其次是内切葡聚糖酶,外切葡聚糖酶活性最低。在处理过程中,内切葡聚糖酶处理第1天酶活为0.356 ± 0.008 U/mL,第4天达到最大酶活(1.232 ± 0.017 U/mL),随后降低;外切葡聚糖酶酶活从0.148 ± 0.006 U/mL升高至0.478 ± 0.003 U/mL(第3天),随后降低;β-葡萄糖苷酶酶活从0.682 ± 0.015 U/mL上升到1.443 ± 0.014 U/mL(第4天),随后降低(图3)。
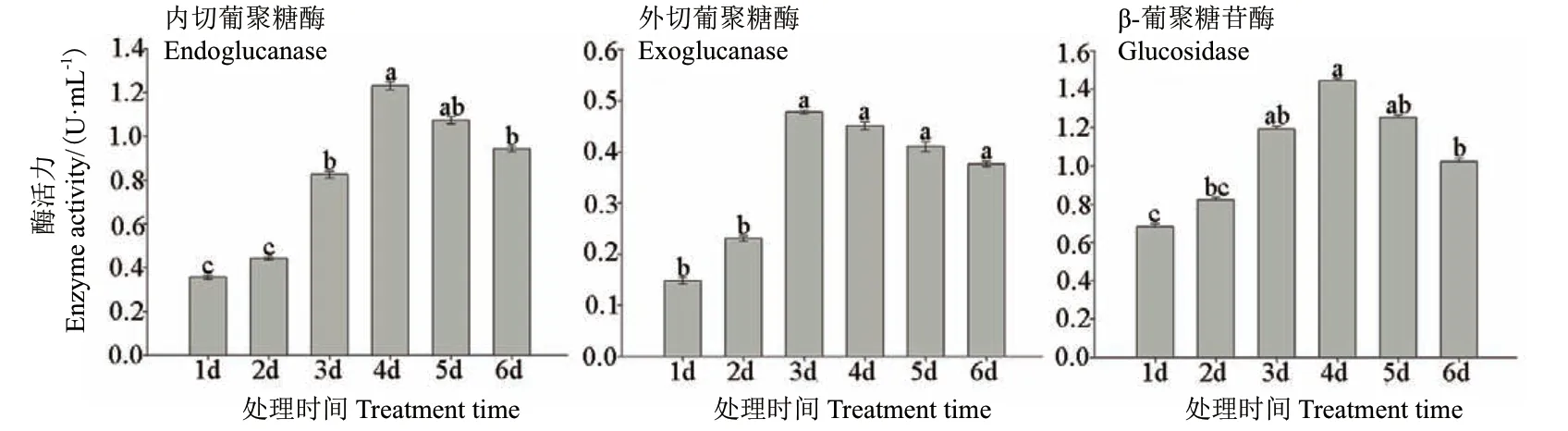
图3 R. ornithinolytica LL1纤维素酶活Fig.3 Determination of cellulase activity in R. ornithinolytica LL1
2.3 R. ornithinolytica LL1基因组测序与组装
本研究利用三代Pacbio Sequl和二代Illumina Novaseq平台对R. ornithinolytica LL1进行完整基因组测序。如表1所示,R. ornithinolytica LL1基因组大小为5 584 354 bp,其中包括大小为5 440 254 bp的环状染色体和144 100 bp的环状质粒。染色体GC含量为55.23%,其中基因和基因间区的GC含量分别为56.39%和48.63%;质粒GC含量为52.06%,其中基因和基因间区的GC含量分别为56.16%和55.13%。基因组共有5 512个基因,其中染色体含有5 396个基因,而质粒含有116个基因,基因总长度为4 789 311 bp;基因组内包含重复序列153个;5S rRNA、16S rRNA和23S rRNA的数量分别为9、8和8个;使用NR、Swiss-Prot、Pfam、COG、GO和KEGG数据库中分别注释到5 511、4 419、4 584、4 745、3 610和3 336个基因。进一步根据基因组相关信息,使用TBtools软件[28]绘制了R. ornithinolytica LL1基因组圈图(图4)。
图4 R. ornithinolytica LL1的全基因组圈图Fig. 4 Whole genome circle of R. ornithinolytica LL1
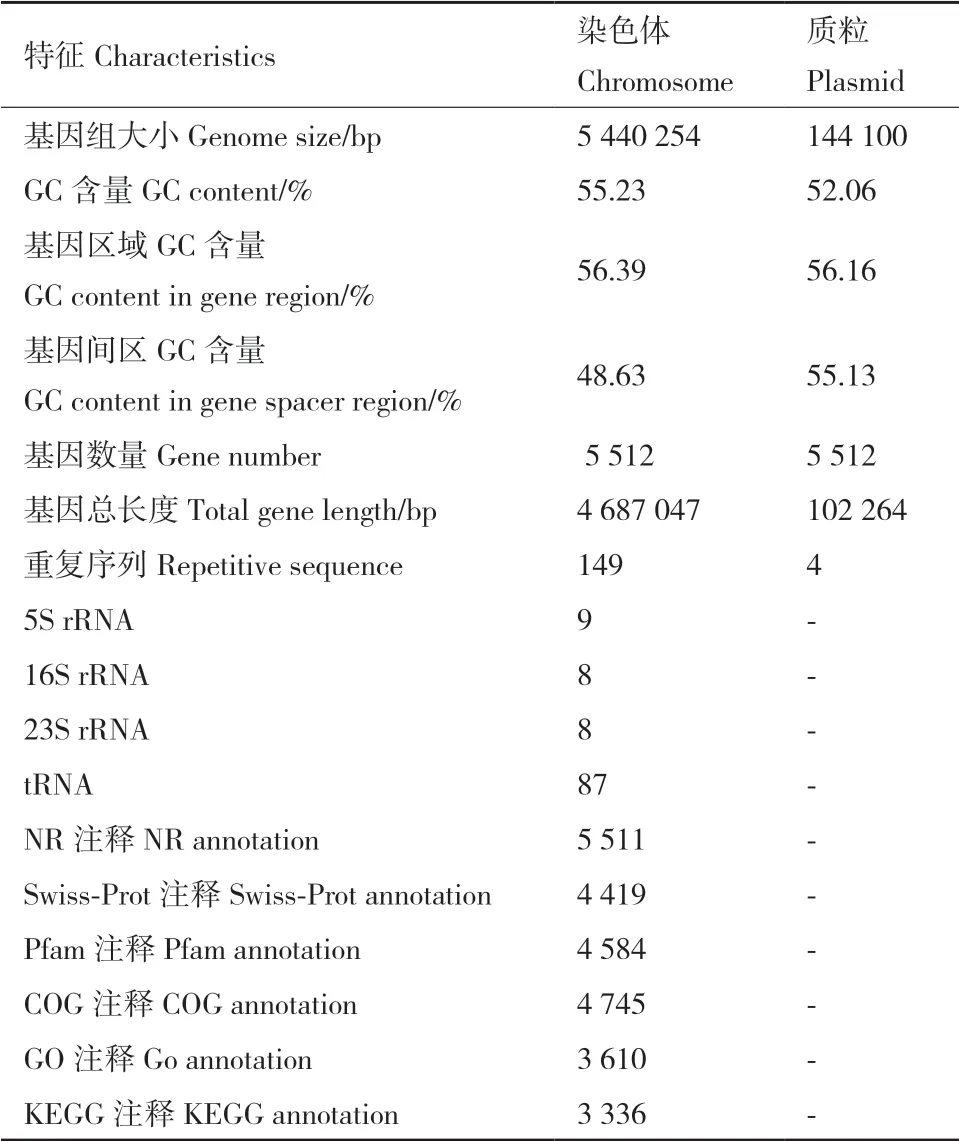
表1 R. ornithinolytica LL1的基因组特征Table1 Genome characteristics of R. ornithinolytica LL1
2.4 基因组注释中参与糖代谢的COGs
共有4 745个基因被注释到2 847个COGs中,其中碳水化合物转运与代谢(Carbohydrate transport and metabolism,9.57%)、氨基酸转运与代谢(Amino acid transport and metabolism,9.78%)和转录(Transcription,8.83%)三类最为丰富(表2)。为了从遗传水平阐明R. ornithinolytica LL1在纤维素降解中的潜能,本研究重点分析了参与碳水化合物代谢的COGs。共有454个基因被注释到碳水化合物代谢中,包括192个COGs,其中最丰富的COGs是COG0524(pfkb结构域蛋白)、COG2814(主要促进剂)、COG1263(PTS系统),COG0477(主要促进者超家族)、COG1940(ROK家族)和COG2723(β-葡萄糖苷酶)。功能注释的高度多样性表明R. ornithinolytica LL1具有降解纤维素的遗传基础。

表2 R. ornithinolytica LL1 COG注释Table 2 COG annotation of R. ornithinolytica LL1
2.5 GO注释
GO注释表明,分子功能包含最多的基因数(5 333),其次是生物过程(基因数:4 418)和细胞成分(基因数:2 300)(图5)。在分子功能方面,最主要的5个途径是DNA结合(DNA binding,GO0003677,422个基因)、ATP结合(ATP binding,GO0005524,411个基因)、转录因子活性(transcription factor activity,GO0003700,230个基因)、金属离子 结 合(metal ion binding,GO0046872;171个 基因)和载体活性(transporter activity,GO0005215;135个基因)。氧化还原过程(oxidation-reductionprocess,GO0055114,366个基因)和转录调控(regulation of transcription,GO0006355,336个 基因)是生物过程中最主要的途径,而膜的组成部分(integral component of membrane,GO0016021,906个基因)和细胞质(cytoplasm,GO0005737,433个基因)是细胞成分最主要的途径。此外,通过分析与碳水化合物代谢相关的GOs,确定了114个与碳水化合物代谢相关的GO项,包括GO0004553(水解O-糖基化合物的水解酶活性)、GO0005975(碳水化合物代谢过程)和GO0016787(水解酶活性)。
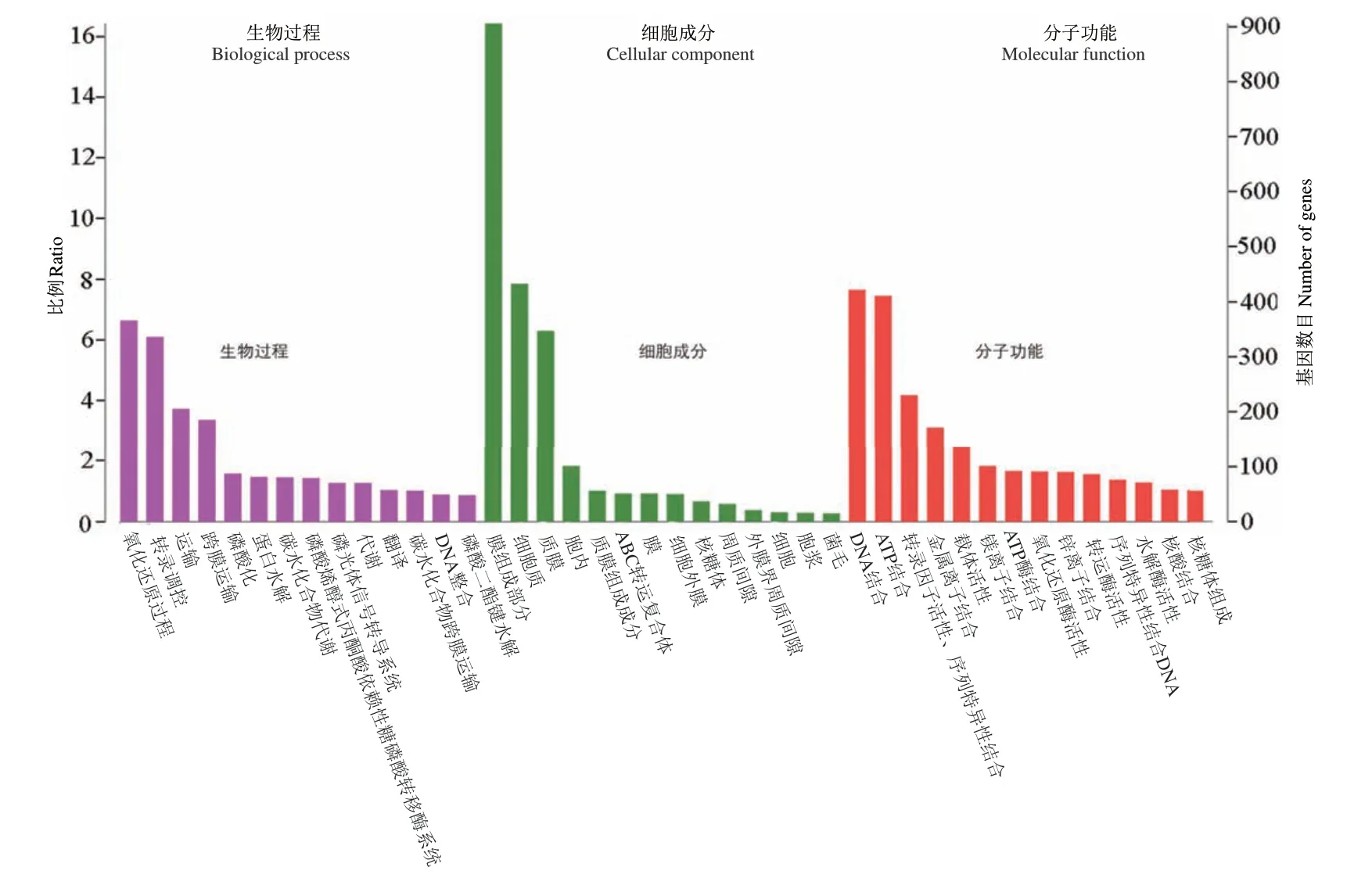
图5 R. ornithinolytica LL1 GO注释Fig.5 GO annotation of R. ornithinolytica LL1
2.6 KEGG注释
在KEGG途径的6个分类中,基因数量最多的是与代谢相关的途径,其次是环境信息处理(图6)。KEGG注释的代谢中碳水化合物代谢包含393个基因,其中蔗糖和淀粉代谢(starch and sucrose metabolism,ko00500,67个基因)、糖酵解/糖异生(glycolysis/gluconeogenesis,ko00010,62个基因)丙酮酸代谢(pyruvate metabolism,ko00620,58个基因)、戊糖磷酸途径(pentose phosphate pathway,ko00030,48个基因)和氨基糖和核苷酸糖代谢(amino sugar and nucleotide sugar metabolism,ko0052047个基因)等途径占主导地位(表3)。
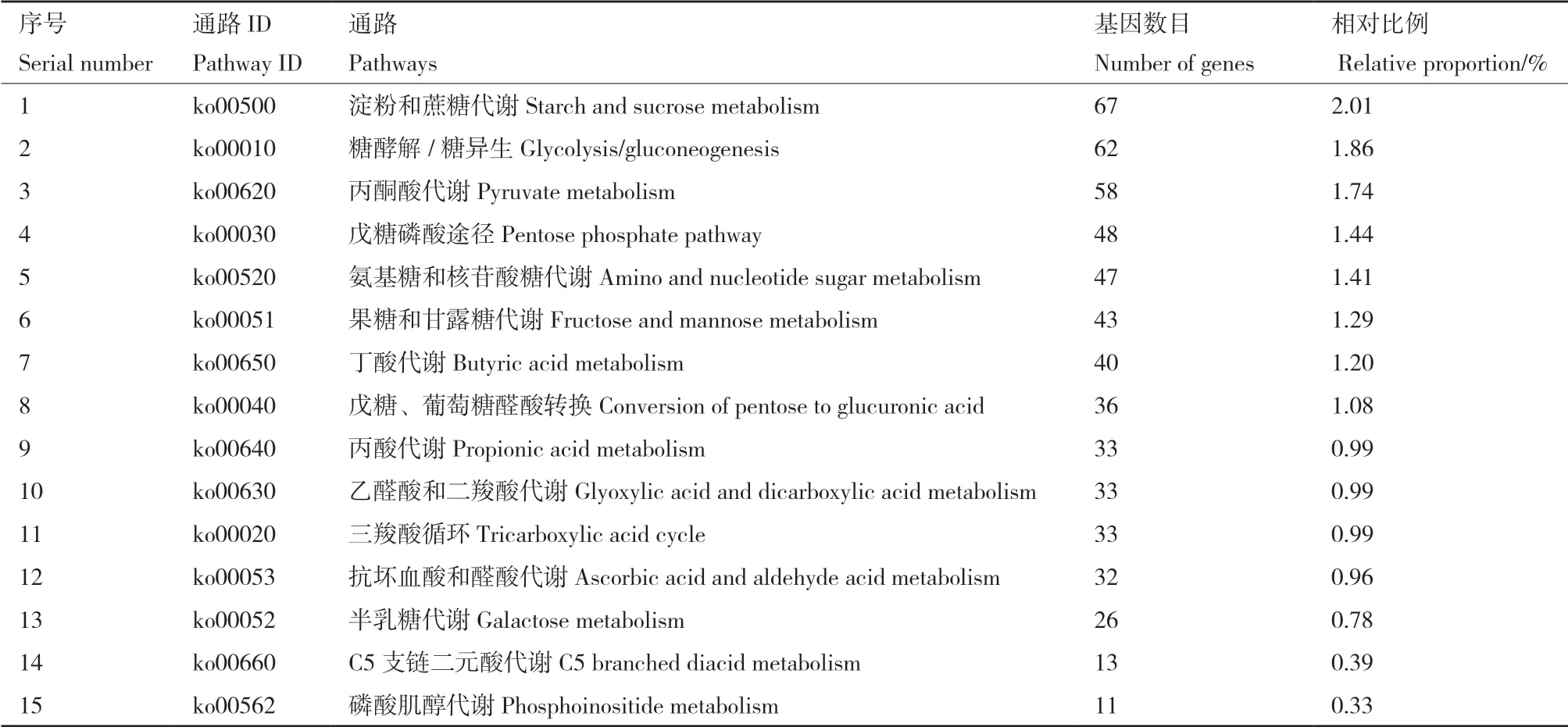
表3 碳水化合物代谢相关KEGG通路Table 3 KEGG pathways related to carbohydrate metabolism
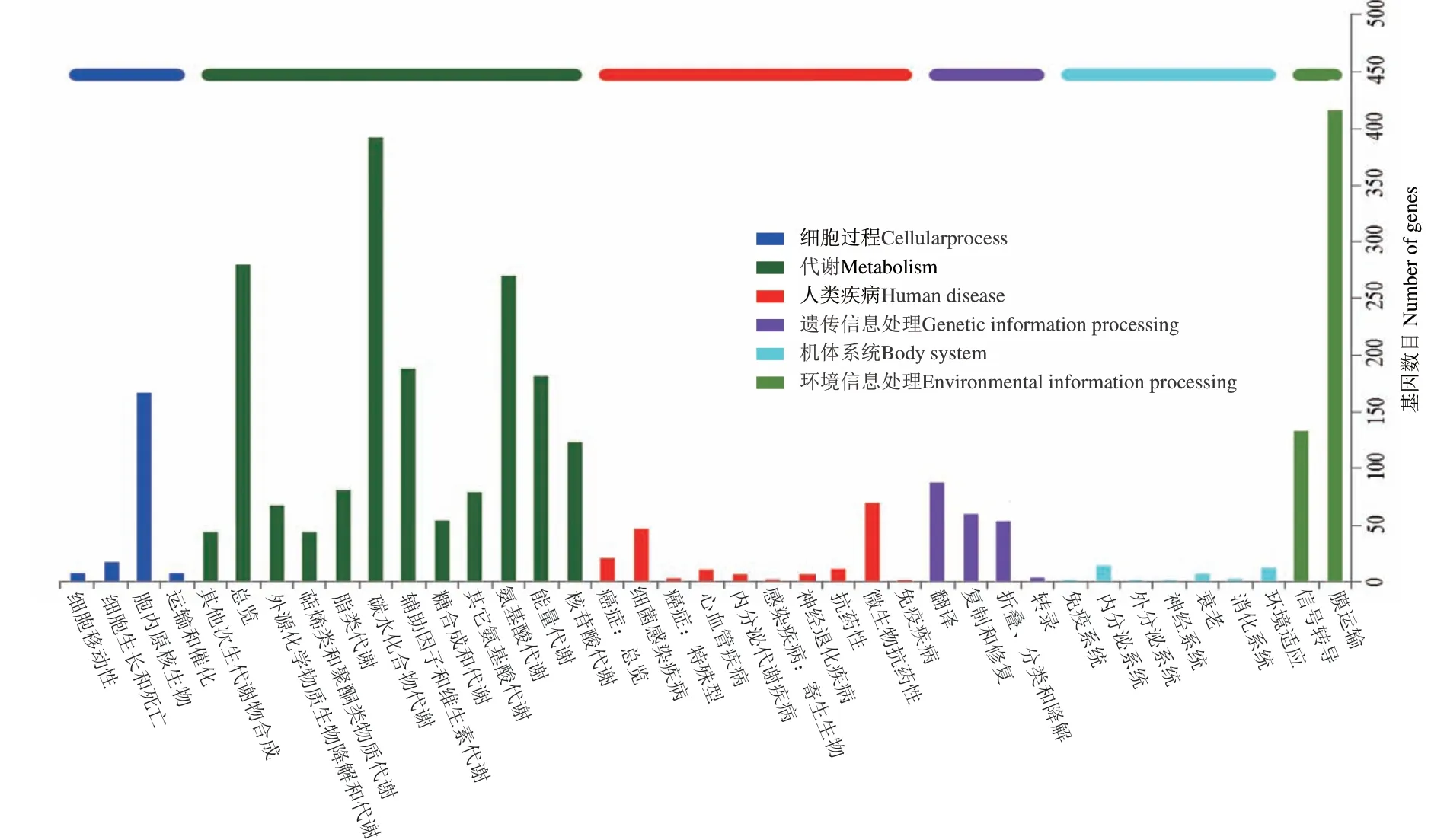
图6 R. ornithinolytica LL1 KEGG注释Fig.6 KEGG annotation of R. ornithinolytica LL1
2.7 碳水化合物活性酶(CAZymes)注释
基因组碳水化合物活性酶注释结果表明,R. ornithinolytica LL1基因组注释到128个CAZyme基因,占全部编码基因的2.32%,其中糖苷水解酶(GH)对碳水化合物的降解具有关键作用,在基因组中注释到55个GH基因(图7)。此外,还鉴定了32个糖基转移酶(GT)基因,23个糖酯酶(CE)基因,2个多糖裂解酶(PL)基因和16个具有辅助活性的酶(AA)基因(图7)。一共有23个GH家族被注释到,其中数量最多的为GH1、GH13、GH23和GH109,分别有9、8、7和5个成员;23个CE基因分配到8个CE家族,包括,CE1、CE3、CE4等,其中CE1成员数量最多,有9个;AA中共有7个家族被注释,其中成员数量最多的是AA3;2个PL家族被注释,包括PL9和PL22。
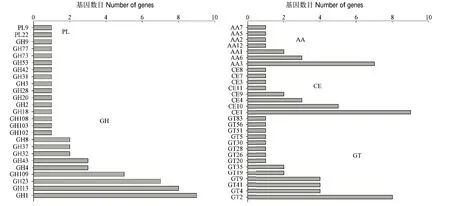
图7 R. ornithinolytica HZ1的碳水化合物活性酶注释Fig.7 Annotation of the carbohydrate active enzyme of R. ornithinolytica LL1
2.8 木质纤维素酶基因
R. ornithinolytica LL1基因组中共注释到25个纤维素降解相关基因,其中包括3个内切葡聚糖酶(endoglucanase,EC 3.2.1.4)基因、10个β-葡萄糖苷酶(beta-glucosidase,EC 3.2.1.21)基因、2个6-磷酸-β-葡萄糖苷酶(6-phospho-beta-glucosidase,EC 3.2.1.86)基因、9个α-葡萄糖苷酶(α-glucosidase,EC 3.2.1.20)基因和1个蔗糖-6-磷酸水解酶(sucrose-6-phosphate hydrolase,EC 2.4.1.-)(表4)。内切葡聚糖酶基因编码的蛋白质属于GH8和GH9家族;β-葡萄糖苷酶基因编码的蛋白质属于GH1和GH3;α-葡萄糖苷酶基因编码的蛋白质主要属于GH13和GH4家族;6-磷酸-β-葡萄糖苷酶属于GH4家族(表4)。丰富的纤维素酶基因表明R. ornithinolytica LL1具有较强的纤维素降解潜力。
R. ornithinolytica LL1基因组中共有6个基因编码半纤维素酶,包括3个木聚糖1,4-β-木糖苷酶(xylan 1,4-beta-xylosidase,EC:3.2.1.37)基因、2个β-半乳糖苷酶(beta-galactosidase,EC:3.2.1.23)基因和1个阿拉伯半乳聚糖内-1,4-β-半乳糖苷酶(arabinogalactan endo-1,4-beta-galactosidase,EC:3.2.1.89)基因(表4)。木聚糖1,4-β-木糖苷酶属于GH43家族;β-半乳糖苷酶属于GH2和GH42家族;阿拉伯半乳聚糖内-1,4-β-半乳糖苷酶属于GH53家族(表4)。
R. ornithinolytica LL1基因组中共有11个木质素降解相关基因,包括2个漆酶(laccase,EC 1.10.3.2)基因、1个锰过氧化物酶(manganese peroxidase,
EC 1.11.1.13)基 因、1个FAD结 合 蛋 白(FADbinding protein)和7个纤维素二糖脱氢酶(cellobiose dehydrogenase,EC 1.1.99.18)基因。其中漆酶基因编码的蛋白质属于AA1家族,锰过氧化物酶基因编码的蛋白质属于AA2家族,纤维素二糖脱氢酶基因编码的蛋白质属于AA3家族(表4)。该结果表明R. ornithinolytica LL1同时具有木质素降解能力。

表4 编码R. ornithinolytica LL1木质纤维素降解酶基因Table 4 Genes encoding lignocellulose degradation in R. ornithinolytica LL1
3 讨论
Raoultella ornithinolytica作为一类具有纤维素降解细菌,其基因组包含丰富的CAZymes基因[29]。本研究从长足大竹象肠道内筛选到一株纤维素降解细菌R. ornithinolytica LL1,基因组大小为 5 584 354 bp,与一种木质素降解相关的R. ornithinolytica S12基因组大小接近[29],但略大于序列相似度极高的R. ornithinolytica B6基因组[30]。R. ornithinolytica B6分离自石油污染的土壤,其基因组中包含4 909个基因,其可利用己糖、戊糖、二糖及甘油等作为碳源或在过表达budABC基因情况下合成2,3-丁二醇[30-32]。
R. ornithinolytica LL1基因组内共有128个编码CAZyme基因,分别归属于GH家族、GT家族、CE家族、PL家族和AA家族。GH家族含有作用于糖苷键的各种水解酶[33],而R. ornithinolytica LL1基因组中包含23个GH家族成员,其中GH1、GH8和GH9等家族编码纤维素酶,GH5、GH7、GH12等家族主要编码内切葡聚糖酶,GH1、GH3家族主要与β-葡萄糖苷酶相关,它们共同参与了纤维素的降解[34]。与半纤维素降解相关的CAZyme家族主要包括GH43、GH2、GH42和GH53。其中,GH43是木聚糖降解的重要成员[35],GH53被报道为内切-1,4-β-半乳糖苷酶且具有水解1,4-β-D-半乳糖苷键的能力[36]。R. ornithinolytica LL1基因组中还鉴定出促进木聚糖分解相关的CEs。CE3、CE4和CE7是乙酰木聚糖酯酶,可以促进木聚糖的溶解[37-38],此外CE4还含有参与甲壳素降解的肽聚糖N-脱乙酰基[39]。CE10鉴定出羧酸酯酶和木聚糖酶活性并与半纤维素降解相关[40]。AAs包括AA1、AA2、AA4及AA7等。其中,AA4含有香草醇氧化酶,可以转化某些酚类[41],AA7酶参与木质纤维素的生物转化[42]。这些结果表明R. ornithinolytica LL1具有降解纤维素、半纤维素和木质素的潜力。
本研究首次从长足大竹象肠道内筛选出R. ornithinolytica,解析了R. ornithinolytica LL1基因组,探讨了其纤维素降解的遗传基础,并通过酶活验证了其纤维素降解潜力,为该菌的应用提供了理论基础,可进一步将其作为开发木质纤维素酶的资源。
4 结论
本研究从长足大竹象肠道筛选到一株高效纤维素降解细菌R. ornithinolytica LL1,具有较高的纤维素酶活,其中纤维素酶β-葡萄糖苷酶活性最高,其次是内切葡聚糖酶,外切葡聚糖酶活性最低。R. ornithinolytica LL1基因组大小为5 584 354 bp,包含5 512个基因,其中NR、Swiss-Prot、Pfam、COG、GO和KEGG数据库中分别注释有5 511、4 419、4 584、4 745、3 610和3 336个基因。基因组内共有128个编码CAZyme基因,包括55个GH基因、32个GT基因、23个CE基因、2个PL基因和16个AA基因,同时鉴定了木质纤维素降解相关基因。本研究通过基因组测序及分析揭示了该菌其降解木质纤维素的遗传基础,为木质纤维素的降解提供新的微生物资源。

