转录因子KLF4与miR-106a的调控关系及其对人胃癌细胞迁移的影响
朱 萌 翟华亮 赵婉莹张 宁 * 和水祥*
(1.宁夏医科大学总医院消化内科,宁夏银川 750004;2. 宁夏医科大学总医院病理科,宁夏 银川 750004;3. 解放军第三二三医院消化内科,陕西 西安 710061;4. 西安交通大学医学部第一附属医院消化内科,陕西西安 710061)
RNA是生命活动的中心。其中,非编码RNA分子已被越来越多的证据证明其在生命活动中起着不可替代的作用。微小RNA(microRNA,miRNA)是一类主要的内源性非编码单链RNA分子,仅17~22个核苷酸长度,通过识别1个或多个mRNA分子的3´-非翻译区(3´-untranslated region,3´-UTR)而 在 转 录 后 水 平 抑 制mRNA翻译或降解mRNA[1-2]。这一经典理论明确了miRNA下游的调控机制,集中于miRNA与靶基因的相互作用,对于miRNA自身如何被调控的机制却鲜见报道。我们前期研究发现miR-106a靶向TIMP2调控胃癌细胞的侵袭转移,是肿瘤特异性RNA分子,可能成为预防和治疗进展期胃癌的关键靶点[3]。然而,正如现有大多数研究报道一样,关于miR-106a的研究多局限于对其靶基因的鉴定和细胞表型的检测上,而对其自身转录调控机制的认识仍知之甚少。因此,本研究拟从miRNA上游水平入手,构建miR-106a启动子的荧光素酶报告质粒,筛选并验证能够与启动子结合并调控启动子转录的转录因子Krüppel样因子4(Krüppel-like factor 4,KLF4);构建KLF4过表达质粒,探索KLF4在上游转录水平对miR-106a成熟体的表达调控及对下游事件胃癌细胞迁移能力的影响,以期为深入阐明miR-106a在胃癌中的作用机制奠定基础。
1 材料与方法
1.1 材料
PrimeSTAR®HS DNA Polymerase、DL2000 DNA Marker、Trizol购自TaKaRa公司,T4连接酶、 K pn I、XhoI购自Thermo公司,Biospin DNA提取试剂盒购自杭州博日科技有限公司,Plasmid Mini Kit I质粒提取试剂盒购自Omega公司,胰蛋白栋、酵母粉购自Oxiod公司,逆转录试剂盒、Bestar qPCR RT Kit购自DBI®Bioscience公司,胎牛血清购自Gibco公司,DMEM、RPMI-1640购自Hyclone公司,Dual-Luciferase Reporter Assay System购自Promega公司,LipofectamineTM2000购自Invitrogen公司。人永生化胃黏膜上皮细胞株GES、人胃中分化腺癌细胞株SGC-7901及HEK293T购自上海科学院典型培养物保藏委员会细胞库,置于含10%胎牛血清的RPMI-1640培养基中传代培养。
1.2 方法
1.2.1 miR-106a启动子野生型和突变型报告质粒的构建 根据miR-106a启动子和载体pGL3-basic的序列设计引物,见表1。200 μL GES细胞,采用Biospin DNA提取试剂盒提取模板DNA。目的基因PCR扩增体系:PrimeSTAR®HS DNA聚合酶0.25 μL,5×PrimeSTAR缓冲液5 μL,dNTP Mix 2 μL,F/R引物各0.5 μL,DNA 1 μL,ddH2O 15.75 μL ,共25 μL。反应条件:94 ℃、5 min,94 ℃、30 s,61 ℃、30 s,72 ℃、30 s,72 ℃、5 min。miR-106a启动子PCR 产物回收后与pGL3-basic载体双酶切,酶切体系:PCR回收产物/载体10 μL,Kpn I 1.5 μL, X ho I 1.5 μL,10× 缓 冲液5 μL,ddH2O 32 μL ,共50 μL,37 ℃水浴2 h。使用T4 DNA 连接酶连接目的片段与载体。连接产物转化大肠杆菌D H5α感受态细胞,挑选菌落,接种于装有800 μL含氨苄青霉素(100 μg/mL)的培养基中,取1 μL菌液为模板,行菌落PCR鉴定,PCR体系:rTaq 0.5 μL,2×Taq Master Mix 10 μL,F/R 引 物各0.4 μL,ddH2O 8.1 μL,共20 μL。选取阳性克隆的菌液按1∶100的比例接种至5 mL含氨苄青霉素(100 μg/mL)LB培养基中,洗脱质粒。重组质粒酶切鉴定:质粒3 μL, K pn I 0.4 μL,XhoI 0.4 μL,10× 缓 冲液1 μL,ddH2O 5.2 μL ,共10 μL,37℃、2 h。酶切产物行1%琼脂糖凝胶电泳检测、送测序。
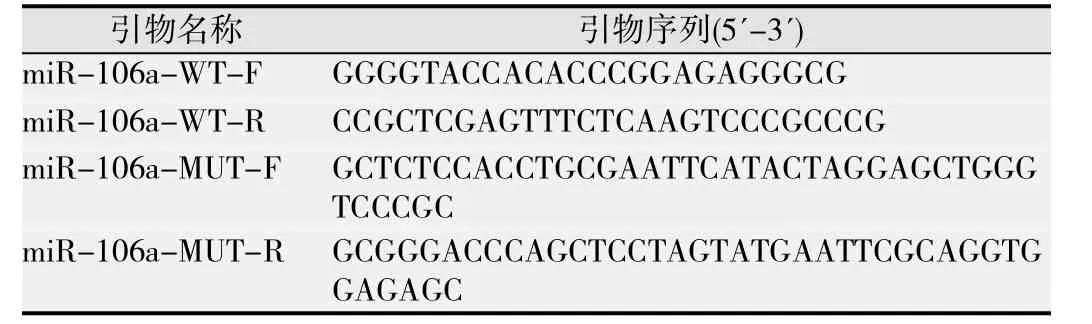
表1 miR-106a 启动子野生型和突变型引物序列
1.2.2 KLF4过表达质粒的构建 根据 K LF4基因和载体pcDNA3.0的序列设计引物。KLF4-F:GGGGTACCA TGAGGCAGCCACCTGGC;KLF4-R:CCGCTCGAGTT AAAAATGCCTCTTCATGTGTAAGG。Trizol 法提取GES细胞 模 板RNA。逆转录体系:5×RT 缓 冲液4 μL,RT Enzyme Mix 1 μL,Primer Mix 1 μL,RNase Free ddH2O 4 μL,RNA 1 μg 10 μL ,共20 μL。反应条件:37 ℃、60 min,98 ℃ 、 10 min。PCR扩增体系:PrimeSTAR®HS DNA聚合酶0.25 μL,5×PrimeSTAR 缓冲液5 μL,dNTP Mix 2 μL,F/R引物各0.5 μL,模板 1 μL,ddH2O 15.75 μL ,共25 μL。反应条件:94 ℃、5 min,94℃、30 s,62 ℃、30 s,72 ℃、 2 min,72 ℃、5 min。后续步骤同上。
1.2.3 双荧光素酶系统验证miR-106a转录因子KLF4 HEK293T细胞以8×105/mL接种于24孔培养板,分4组:pmiR-106a-WT+KLF4+-pcDNA3.0,pmiR-106a-WT+KLF4--pcDNA3.0,pmiR-106a-MUT+KLF4+-pcDNA3.0,pmiR-106a-MUT+KLF4--pcDNA3.0。使用LipofectamineTM2000转染,KLF4过表达质粒/miR-106a启动子质粒浓度0.8 μg,转染后48 h测定荧光值。每孔细胞加入100 μL Passive裂解液,室温轻微振摇15 min,收集细胞裂解液,GloMax生物发光检测仪读取背景值2 s,每样品加入100 μL LAR Ⅱ工作液,读值2 s,再加入100 μL Stop&Glo®Reagent,读值2 s,计算相对荧光活性值。
1.2.4 qPCR检测胃癌组织中miR-106a、KLF4 mRNA表达水平 收集2017年1月—2017年11月于宁夏医科大学总医院行胃癌根治术切除的胃组织标本,癌灶和对应癌旁组织(距癌灶中心≥5 cm)各30例。所有患者术前均已经胃镜活检确诊为胃腺癌,并通过医学伦理委员会批准并签署知情同意书。使用Trizol法提取RNA,RNA质量管控采用美国Bioteck公司Epoch超微量微孔板分光光度计检测核酸纯度, D ( 2 60)/D(280)比值在1.8~2.0认为符合要求。KLF4-F:CGTTGAACTCCTC GGTCTC;KLF4-R:GACGCCTTCAGCACGAACT。miR-106a逆转录引物:TCAACTGGTGTCGTGGAGTCGGCA ATTCAGTTGAGCTACCTGC;miR-106a-F:ACACTCC AGCTGGGAAAAGTGCTTACAGTGCA。统一的反向引物CTCAACTGGTGTCGTGGA。内参分别为GAPDH、U6。逆转录条件:37 ℃、15 min;98 ℃、5 min。PCR反应条件:95 ℃、2 min,94 ℃、20 s,58 ℃、20 s,72 ℃、20 s,40个循环。熔融解曲线分析:94 ℃、30 s,65 ℃、30 s,94 ℃、30 s。重复3次。以2-∆∆CT计算基因相 对表达量。
1.2.5 Transwell法检测人胃癌细胞迁移能力 人胃癌细胞株SGC-7901按2×105/mL接种于6孔培养板,分4组:miR-106a mimic,mimic NC,miR-106a mimic+KLF4-pcDNA3.0,miR-106a mimic+pcDNA3.0 vector。使用LipofectamineTM2000转染,收集细胞,安装小室,继续培养24 h后固定染色,显微镜下选取10个互不重叠高倍视野计数。
1.3 统计学分析
采用SPSS 17.-0统 计软件进行数据分析。计量资料的统计描述采用x± s 表 示,两样本均数的比较采用t检验(t-test),多个样本均数的比较采用方差分析(One-Way AVONA),变量之间关系的比较采用直条图表示,采用箱式图表示变量原始数据的分布特征,以 α=0.05为检验水准。
2 结 果
2.1 生物信息学预测miR-106a上游启动子和转录因子
使用美国加州大学圣克鲁兹分校生物信息数据库(University of California Santa Cruz,UCSC genome browser)检索hsa-miR-106a启动子序列,获得miR-106a上游启动子区域2 000 bp序列。miR-106a启动子序列和突变位点见图1。使用转录因子数据库JASPAR,选择脊椎动物JASPAR CORE Vertebrata,筛选调控miR-106a的转录因子,考虑结合位点与起始位点近、理论值达90%、CDS区片段长度等因素确定KLF4作为miR-106a转录因子的研究对象,并获得转录因子与启动子结合的位点。
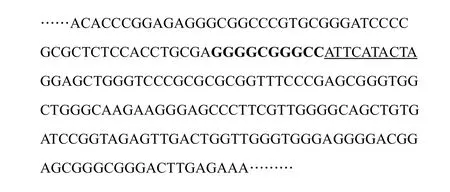
图1 miR-106a 上 游 2 0 00 b p 启 动子序列(http: //genome.ucsc.edu/)
2.2 miR-106a启动子报告质粒的鉴定
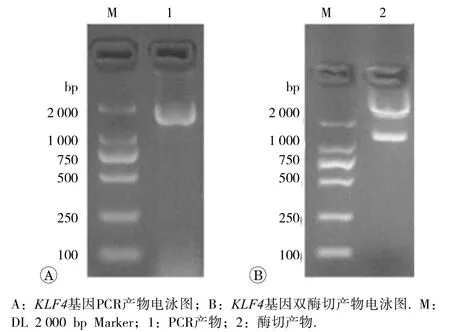
如图2所示,miR-106a启动子重组质粒PCR产物电泳图谱显示目的基因条带位于100~250 bp之间,理论值191 bp,符合预期片段大小,初步证明miR-106a启动子扩增成功。双酶切电泳图显示pmiR-106a-WT位于200 bp左右,pGL3 basic位于加样孔处(4 000 bp左右,原本4 818 bp),提示miR-106a启动子野生型重组质粒构建符合预期。miR-106a启动子定点突变重组质粒送测序后显示已成功在目的突变位点实现突变。
2.3 KLF4过表达质粒的鉴定

图2 miR-106a启动子报告质粒的鉴定
如图3所示,KLF4过表达质粒PCR产物电泳图显示电泳条带位于1 000~2 000 bp之间,理论值1 440 bp,提示 K LF4基因扩增顺利,双酶切电泳图显示KLF4基因酶切产物1 500 bp左右,大片段pcDNA3.0位于加样孔下方,原本 5 427 bp,提示KLF4重组质粒构建符合预期。
2.4 miR-106a转录因子KLF4的鉴定
双荧光素酶系统检测结果如图4所示,pmiR-106a-WT+KLF4+-pcDNA3.0和pmiR-106a-WT+KLF4--pcDNA3.0,在应用KLF4过表达质粒处理后,miR-106a野生型报告质粒荧光素酶活性下降,t检验得出,与空载体相比,差异具有统计学意义(t=-24.378,P=0.000),说明过表达的KLF4对报告载体上miR-106a启动子下游的萤火虫荧光素酶基因的表达有抑制作用。pmiR-106a-MUT+KLF4+-pcDNA3.0和pmiR-106a-MUT+ KLF4--pcDNA3.0,KLF4过表达处理后,miR-106a启 动子突变型报告基因活性下降不明显,差异无统计学意义(t= - 0.647,P =0.553),说明过表达KLF4对miR-106a突变型报告基因的荧光素酶活性无明显影响。

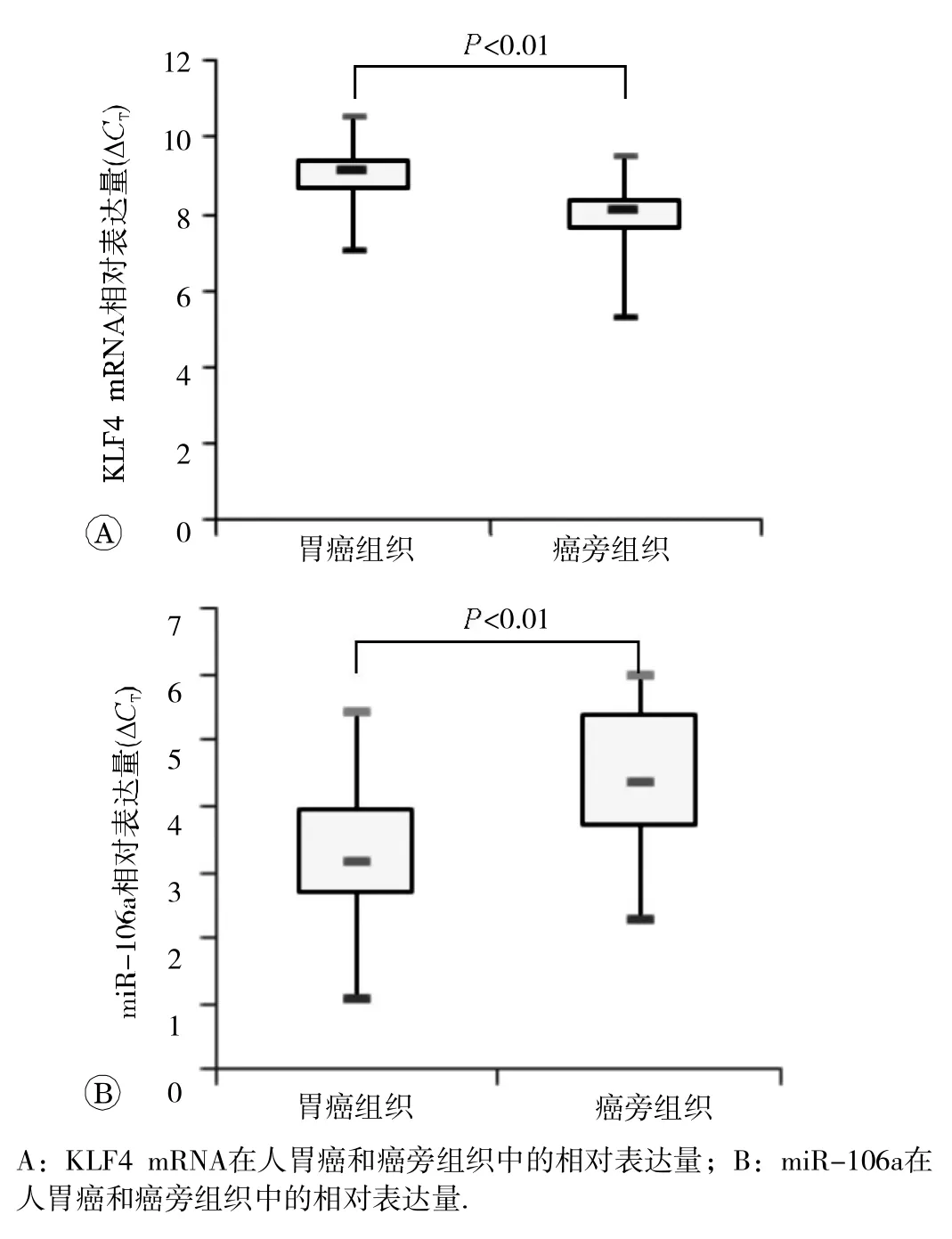
2.5 KLF4 mRNA和miR-106a在人胃癌组织中的表达水平
qPCR检测结果如图5所示,KLF4 mRNA在胃癌组织中的相对表达量为0.716±0.624,配对t检验得出,二者 差 异具有统计学意义(t= 4.228, P=0.000),说明KLF4 mRNA在胃癌组织中呈低水平表达。miR-106a的相对表达量为3.367±2.165,t检验得出两者 差 异具有统计学 意 义(t= -6.259, P=0.000),说明miR-106a在胃癌组织中高表达。提示miR-106a与转录因子KLF4在人胃癌中可能具有负调控关系。
2.6 miR-106a的表达调控对人胃癌细胞迁移能力的影响
人胃癌SGC-7901细胞行transwell小室实验,结果如图6所示,miR-106a mimic组穿膜细胞数量为100.50± 53.56, mimic NC组 为 49.90± 12.53, miR-106a mimic+KLF4-pcDNA3.0组为67.30±18.26,miR-106a mimic+pcDNA3.0 vector组为99.50±37.28。方差分析得出 F= 5.239, P=0.004,说明不同处理的 4组细胞迁移能力不全相同。与mimic NC组相比,miR-106a mimic组细胞数量显著增多(P=0.002);但当合并KLF4处理后,miR-106a mimic+KLF4-pcDNA3.0 组细胞数量减少,与miR-106a mimic组相比,差异具有统计学意义(P=0.038),而KLF4空载体对照组细胞数量无明显变化(P=0.949)。提示miR-106a mimic对胃癌细胞迁移有促进作用,但该促进作用可被 KLF4部分阻遏。

3 讨 论
miRNA的表达失控与多种人类疾病尤其是恶性肿瘤密切相关[4]。miR-106a是由约70个核苷酸长度的前体miR-106a~363经Dicer酶剪切形成约22个核苷酸长度的产物,构成miR-17家族成员之一,该家族成员在大多数的恶性肿瘤中被认为具有癌基因样的作用[5]。本文研究miR-106a的自身表达调控机制。之前有学者报道通过构建pre-miR-122启动子报告质粒转染小鼠肝星状细胞HSCs,以荧光素酶活性值可以初步判断miRNA的表达调控机制[6]。本研究选择转录因子KLF4与miR-106a启动子的相关关系开展研究,探讨miR-106a的表达调控对人胃癌细胞生物学功能的影响。
通常,启动子区域位于转录起始位点(transcription start site,TSS)附近,主要是在TSS上游1 kb的范围内。已知,miRNA的转录主要是由Ⅱ类RNA聚合酶(RNA-pol)介导[7]。RNA-polⅠ和Ⅱ多位于TSS上游,RNA-polⅢ位于TSS下游[8]。本研究于miR-106a上游2 000 bp序列预测启动子区域,序列分析发现,其广泛富含TATA盒 (TATA box)、 CAAT盒 (CAAT box)和 GC(GC box)等真核生物基因上游顺式作用元件,据此推测miR-106a转录可能由RNA-polⅠ或RNA-polⅡ介导。根据真核生物的转录起始是由能直接或间接辨认结合转录上游区段的反式作用因子启动、控制基因转录。而反式作用因子中,转录因子是与RNA聚合酶直接或间接结合的。故针对转录因子,筛选在胃癌中异常表达并能够与miR-106a启动子结合的转录因子KLF4作为研究对象,使用荧光素酶报告基因技术考察KLF4对miR-106a启动子的调控。结果显示KLF4的过表达载体对miR-106a启动子的野生型报告基因的荧光素酶活性具有明显的抑制作用,但对突变型质粒的作用不明显,提示miR-106a启动子区含有可能与转录因子KLF4结合的位点。KLF4是一种锌指结构蛋白,隶属于转录因子家族[9-10]。很多研究认为KLF4在多种类型实体肿瘤中是作为一种抑癌基因的角色而存在。KLF4的敲低具有促进肿瘤细胞生长、迁移和黏附的作用[11],激活KLF4则减少转移灶周遭环境的形成悖于转移,且是一种KLF依赖性的过程[12]。有研究报道KLF4可与miR-544的启动子序列互补结合调节miR-544在宫颈癌中的表达[13]。但KLF4与miR-106a在胃癌中的调控关系尚未见报道。我们此次在人胃癌组织中对比研究KLF4的表达水平,发现KLF4在胃癌组织中低表达,结合miR-106a在胃癌组织中高表达,提示在胃癌中,KLF4表达降低,KLF4结合于miR-106a启动子区域,低表达的KLF4对miR-106a的抑制作用弱,miR-106a表达升高;当应用KLF4过表达载体使其表达升高后,KLF4发挥抑癌基因样作用,在转录水平减少miR-106a野生型报告基因的荧光素酶活性,使miR-106a成熟体表达减少;再结合我们前期的研究结果,高表达的miR-106a在胃癌细胞中因具有显著地促使癌细胞获得更高水平的迁移能力的作用,使其在一定程度上体现促癌基因样的表型特征,此次KLF4在上游水平对其启动子的结合,负性调节其转录活性,致使在表观现象上我们看到miR-106a对胃癌细胞的促癌基因样作用得以削弱[3],细胞的迁移能力下降,部分阻遏或一定程度逆转胃癌细胞的侵袭性。
总之,本研究一定程度上揭示了转录因子KLF4与miR-106a启动子的结合和调控关系,KLF4和miR-106a结合于上游转录位点可能在胃癌的侵袭转移事件中发挥重要的调控作用,这将为进一步研究miRNA表达调控机制打开新窗口。
——一道江苏高考题的奥秘解读和拓展

