芘二聚体发夹探针用于细胞DNA单链断裂修复能力的评估
李印, 刘劲松, 王禹, 祁婷婷, 孟乐园, 张晶, 吕冬梅, 焦虎平
芘二聚体发夹探针用于细胞DNA单链断裂修复能力的评估
李印, 刘劲松, 王禹, 祁婷婷, 孟乐园, 张晶, 吕冬梅, 焦虎平
(吉林大学动物科学学院, 长春 130062)
基于芘分子二聚体的荧光特性设计了一种新型的发夹探针, 用于评估不同细胞的单链断裂修复(SSBR)能力. 首先利用芘荧光分子设计一个茎上有缺口的DNA发夹探针, 该探针在Bst DNA聚合酶作用下可以水解, 使荧光信号“关闭”. 如果探针茎上的缺口被DNA修复酶修复, 可以阻止Bst DNA聚合酶对探针的消化作用, 从而阻止荧光信号的“关闭”. 该设计可以用于评估细胞的SSBR能力, 并通过提取细胞的核蛋白考察了不同细胞的SSBR能力. 研究结果表明, 肿瘤细胞及细胞系不具有原代细胞的SSBR能力. 利用SSBR的关键酶进一步探索了肿瘤细胞SSBR能力缺失的原因, 证明是由于肿瘤细胞中含有可以抑制SSBR过程的活性物质. 此外, 该方法能够同时进行500个细胞的SSBR能力检测, 并用于抗衰老药物筛选.
单链断裂修复; 芘二聚体; 发夹探针; 原代细胞; 肿瘤细胞
单链断裂(SSBs)可以由直接氧化攻击产生或者作为DNA代谢过程的中间产物产生[1]. SSBs是各种DNA损伤类型中最常见的, 据估计每个细胞每天发生SSBs的频率达数万次[2~4]. 哺乳动物细胞已经进化出单链断裂修复(SSBR)途径, 以维持基因组完整性并防止SSBs引起的有害突变[5~7]. 然而, 在某些极端情况或病理条件下, SSBR能力必然会受到影响. 最近有研究表明, SSBR能力下降直接导致上皮细胞的衰老和肿瘤逃逸, 意味着衰老相关的上皮细胞源性肿瘤是SSBR缺陷的结果[8], 并且SSBR在心脏压力超负荷诱发心力衰竭的发病机制中也起着关键作用[9]. 增殖细胞中未修复的SSBs极有可能使DNA复制叉崩溃, 诱导DNA双链断裂, 导致基因组不稳定和大量遗传信息丢失, 进而导致细胞功能障碍或细胞死亡[10~13]. 鉴于SSBR的重要性及其在基因组稳定性中的作用, 需要可行的方法来评估SSBR的能力.
检测SSBR的传统方法是彗星电泳实验, 根据断裂DNA在蔗糖梯度[14]或琼脂糖[15~17]中迁移速率的差异来判断SSBR能力; 近年来, 下一代测序技术也被应用于SSBR能力评估[18]. 但上述方法耗时长, 需要专门的技术技能, 且成本高昂. 直接评估细胞SSBR能力的策略是基于一个带缺口的寡核苷酸双链, 在短的寡核苷酸5'端用放射性同位素32P标记, 与核蛋白复合物孵育; 经过核蛋白中的DNA修复酶作用, 修复后的寡核苷酸条带由磷屏成像仪显示[19]. 事实上, 复杂的过程和潜在的放射性污染问题极大地限制了该方法的应用. 因此, 迫切需要准确、 操作简单、 改进的直接评价细胞SSBR能力的方法.
荧光法以其操作简便、 结果可视化、 设计灵活等优点, 成为许多酶活性检测的常规策略[20~24]. 然而, 与放射性标记相比, 灵敏度限制了其应用. 因此, 很难用荧光团标记寡核苷酸以直接检测细胞的SSBR能力. 增加核蛋白的量是对荧光灵敏度的补偿, 但核蛋白复合物中的外切酶会降解整个双链, 从而掩盖SSBR的能力. 为了解决这一矛盾, 降低核蛋白的外切酶活性是必不可少的; 同时, 荧光的灵敏度也需要提高.
为了满足上述需求, 本文提出了一种新的荧光传感系统, 可以直接评估不同细胞的SSBR能力. 首先, 合成3′端芘修饰的寡核苷酸A(Oligo A)和5′端芘修饰的寡核苷酸B(Oligo B), 两条序列在一端互补; 退火后, Oligo A和Oligo B会形成一个带有缺口的茎环结构, 两个芘单体距离靠近, 形成一个芘分子二聚体发夹探针(Oligo mix), 缺口的修复与否决定了芘分子的荧光强度. 因此, SSBR可以直观地转化为芘分子二聚体和单体之间的荧光变化(Scheme 1). 通过实验观察到原代细胞的修复系统具有明显的二聚体荧光值; 而癌细胞和细胞系的修复系统中则几乎没有荧光信号. 该方法能够直接评估SSBR能力, 而不需要昂贵的设备和复杂的步骤. 此外, 该荧光报告系统对抗衰老药物的筛选也具有一定的应用价值.
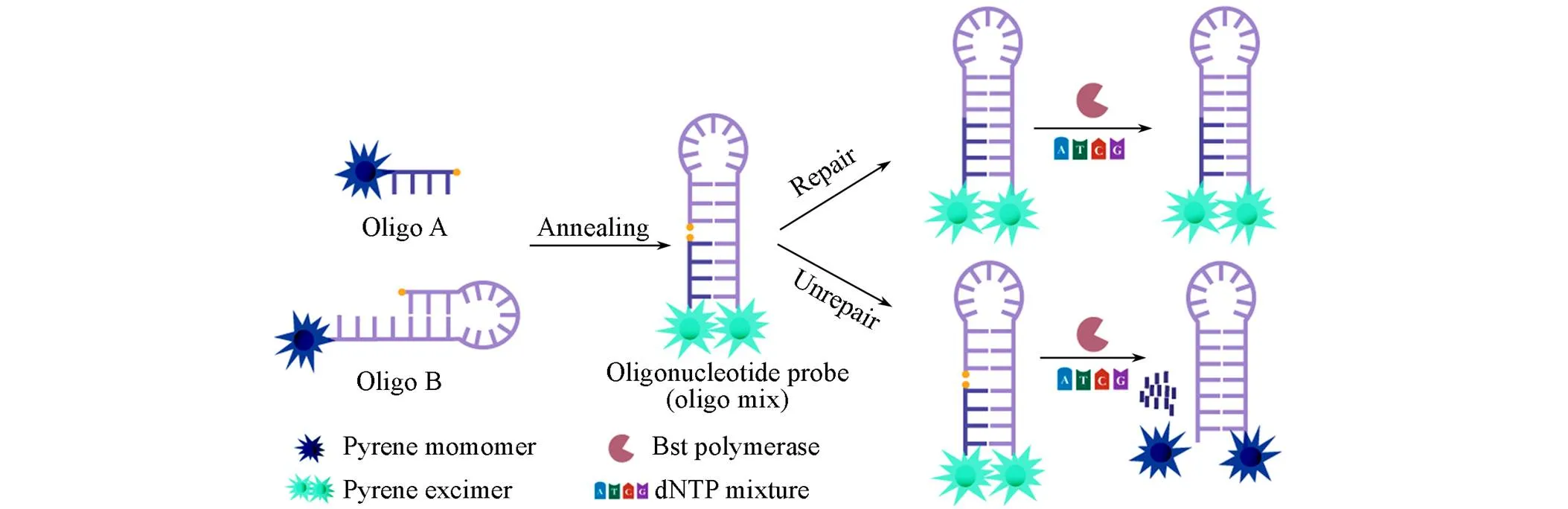
Scheme 1Schematic illustration of direct assessment of SSBR capacity based on pyrene excimer probe
1 实验部分
1.1 试剂与仪器
T4 DNA连接酶、 Bst DNA聚合酶(全长)、 T4多聚核苷酸激酶(T4 PNK)、 dNTP混合物和T4 DNA连接酶缓冲液(10×), 美国New England Biolabs公司; 三羟甲基氨基甲烷(Tris)、 乙二胺四乙酸二钠(EDTA二钠)、 甘氨酸和蛋白上样缓冲液(5×), 北京索莱宝科技有限公司; 硼酸, 上海麦克林生化科技有限公司; 十二烷基磺酸钠(SDS)、 丙烯酰胺/甲叉双丙烯酰胺(质量比29∶1, 40% Acr-bis)、 Tris盐酸盐缓冲液(Tris-HCl)和过硫酸铵, 生工生物工程(上海)股份有限公司; 四甲基乙二胺(TEMED)、 细胞培养基Dulbecco's Modified Eagle Medium(DMEM)、 胰蛋白酶- EDTA(0.25%)、 胎牛血清、 GlutaMAX™添加剂(100×)、 MEM非必需氨基酸(100×)、 Orange DNA上样染料(6×)和SYBR™ Gold核酸凝胶染料, 赛默飞世尔科技(中国)有限公司; 青霉素/链霉素, 大连美伦生物科技有限公司; 磷酸缓冲盐溶液(PBS), 武汉博士德生物工程有限公司; 样本释放剂, 润方(长春)生物技术有限公司;-环糊精(-CD)、 烟酰胺单核苷酸(NMN)和亚精胺, Sigma-Aldrich公司; 维生素C, 长沙鼎国生物科技有限公司; CCK-8(Cell Counting Kit-8™), 上海碧云天生物科技有限公司; 1×T4 DNA连接酶缓冲液[含50 mmol/L Tris-HCl(pH=7.5)、 10 mmol/L MgCl2、 10 mmol/L DTT和1 mmol/L ATP]用于Oligo退火和后续反应缓冲液.
DNA序列由Takara生物科技(大连)有限公司合成, 碱基序列为Oligo A: GCGCTCCATGTCCAG-3' Pyrene; Oligo B: 5' Pyrene-CTGGACATGGAGCGCCCTCTCAGTGACAAGAACAATGTCACTGAGAGG; Oligo full-length: CTGGACATGGAGCGCCCTCTCAGTGACAAGAACAATGTCACTGAGAGGGCGCTCCATGTCCAG.
F-4500型荧光分光光度计, 日本Hitachi公司; 1300型生物安全柜、 Sorvall Legend Micro 17R型和Sorvall ST8R型离心机、 iBright CL750型凝胶成像系统, 赛默飞世尔科技(中国)有限公司; ThermoCell型恒温金属浴, 杭州博日科技股份有限公司; ELx800型全自动酶标仪, 美国BioTek公司.
1.2 实验过程
1.2.1荧光检测在室温(25 ℃)下, 通过F-4500型荧光分光光度计进行稳定状态的荧光检测. 在含有8 mmol/L-CD的100 μL 1×T4 DNA连接酶缓冲液中, 将样品孵育10 min, 将激发波长设为344 nm, 在450 ~ 600 nm范围内记录样品的荧光发射光谱, 激发和发射狭缝宽度分别设为5 nm和10 nm, 扫描速度为240 nm/min, 光电倍增管电压设置为950 V.
1.2.2荧光报告系统的构建Oligo A和Oligo B用1×T4 DNA连接酶缓冲液稀释至50 μmol/L. 将两种存储液以等体积混合, 在95 ℃水中变性5 min, 随后在400 mL水中冷却至室温淬火. 经过淬火后, Oligo A和Oligo B将形成一个带有缺口的茎环结构, 并且两个芘分子相互靠近, 形成一个芘分子二聚体发夹探针(Oligo mix, 25 μmol/L). 在修复体系中, 将1.2 μL Oligo mix, 5 U T4 PNK和40 U T4 DNA连接酶加入到1×T4 DNA连接酶缓冲液中, 使最终体积为25 μL, 于37 ℃孵育90 min, 随后于65 ℃孵育 30 min, 使T4 PNK和T4 DNA连接酶失活; 再加入1 μL Bst DNA聚合酶(200 U/mL)和0.5 μL dNTP mix(200 μmol/L), 在65 ℃下反应30 min, 最后记录荧光光谱. 与修复系统相比, 未修复系统中未添加T4 PNK和T4 DNA连接酶.
1.2.3非变性DNA-聚丙烯酰胺凝胶(PAGE)按照DNA-PAGE配方制备厚度为1 mm的15%凝胶. 从反应体系中取出5 μL反应物与1 μL Orange DNA上样染料(6×) Loading Dye混合, 加入到凝胶孔中, 在1×Tris-硼酸-EDTA缓冲溶液(89 mmol/L Tris-硼酸, 2 mmol/L EDTA)中, 于40 V电压下电泳5 h; 电泳结束后, 用SYBR gold染料染色30 min; 最后, 用iBright CL750型凝胶成像系统拍摄凝胶图片.
1.2.4细胞培养和核蛋白获取所有细胞在37 ℃及5%(体积分数)CO2的湿润气体环境中培养. 培养基为添加10%胎牛血清(FBS)、 1%青霉素/链霉素、 1% MEM非必需氨基酸溶液和1% GlutaMAX™的DMEM(完全培养基). 根据生产商的使用说明, 用样本释放剂裂解细胞, 分离核蛋白, 收集细胞沉淀, 在细胞中加入20 μL样本释放剂, 室温孵育15 min, 在16000下离心10 min后将上清转移到干净的预冷离心管中, 于‒80 ℃保存待用.
1.2.5SDS聚丙烯酰胺凝胶(SDS-PAGE)根据SDS-PAGE配方表, 加入40% Acr-bis、 Tris-HCl、 10%过硫酸铵和TEMED, 依次制备10%下层分离胶和4%上层浓缩胶. 将5 μg蛋白与2.5 μL 5×蛋白上样 缓冲液混合, 补加去离子水至12.5 μL, 将样品加入到凝胶孔中, 在1×电泳缓冲液[25 mmol/L Tris, 192 mmol/L甘氨酸, 0.1%(质量体积分数)SDS]中, 于100 V电压下进行电泳; 最后, 使用iBright CL750型凝胶成像系统拍摄凝胶图片.
1.2.6不同细胞SSBR能力评估和系统灵敏度检测对不同数量的细胞用样本释放剂裂解获得核蛋白, 将1.2 μL Oligo mix和0.5 μL核蛋白复合物加入到1×T4 DNA连接酶缓冲液中, 使最终体积为 25 μL, 于37 ℃孵育90 min, 随后65 ℃孵育30 min, 使T4 PNK和T4 DNA连接酶失活, 终止反应. 随后的步骤与荧光报告系统的构建相同.
1.2.7细胞毒性检测在完全培养基中加入终浓度为20 μmol/L的亚精胺、 100 μmol/L的NMN和 100 μmol/L的维生素C, 用0.22 μm滤器过滤备用. 将1×104个小鼠胎儿成纤维细胞加入到96孔板中, 每孔加入100 μL上述不同的培养基(实验组,=3), 在培养箱中培养24 h; 然后, 加入100 μL PBS冲洗细胞2次, 将10 μL CCK-8溶液与90 μL DMEM混合, 共计100 μL加入每个培养孔中, 孵育4 h; 加了相应量完全培养液、 细胞和CCK-8溶液但没有加入药物的孔作为阴性对照组(=3), 加了相应量完全培养液和CCK-8溶液但没有加入细胞的孔作为空白对照(=3); 当CCK-8被活细胞还原成甲瓒后, 用酶标仪测定450 nm处的吸光度.
1.2.8抗衰老药物筛选细胞分别在含20 µmol/L亚精胺、 100 µmol/L NMN和100 µmol/L维生素C的完全培养基中传代培养, 提取第5代(P5)至第9代(P9)1×106个小鼠胎儿成纤维细胞的核蛋白复合物, 随后的步骤与不同细胞的SSBR评估一致.
2 结果与讨论
2.1 SSBR能力评估系统的可行性分析
Scheme 1示出了荧光报告系统评价SSBR的原理. 芘分子单体被标记在寡核苷酸末端, 通过核酸杂交形成芘二聚体[25~27]. 与典型的单体荧光发射峰在378和397 nm处相比, 二聚体荧光光谱发射峰(488 nm)发生红移(图S1, 见本文支持信息). 退火后的Oligo mix, Bst DNA聚合酶和dNTP在T4 PNK和T4 DNA连接酶存在或不存在的情况下进行反应. 由图1(A)可以看出, 在T4 PNK和T4 DNA连接酶存在的情况下, 缺口被修复以抵抗Bst DNA聚合酶的消化, 体系保持芘分子二聚体的绿色荧光; 在没有T4 PNK或T4 DNA连接酶存在的情况下, 缺口仍然存在, Oligo A被Bst DNA聚合酶通过5′-3′核酸外切酶活性水解, 导致二聚体的绿色荧光消失, 从而出现芘单体蓝色荧光. 实验观察到修复体系和未修复体系的荧光比值为780%(谱线和, 488 nm处).-CD具有特殊的环状中空圆筒形结构, 空腔内侧的非极性基团形成一个疏水环境, 环糊精外部的多羟基与极性分子亲和力强, 具有亲水性. 由于环糊精分子“环内疏水、 环外亲水”的特殊结构, 可通过范德华力将一些大小合适的分子嵌入环状空腔中, 络合成包合物, 为寡核苷酸探针两端标记的两个芘分子的空间接近提供了可能, 因此,-CD能够放大芘二聚体的荧光发射强度, 从而增强检测系统的灵敏度[28,29]. 体系中引入-CD后, 与背景二聚体荧光强度的微小增加相比, 修复体系中二聚体荧光信号显著增强, 可获得1003%的荧光比值(谱线和, 488 nm处). 此外, 本文还利用非变性DNA-PAGE来验证实验可行性. 由图1(B)可见, 当Oligo mix未进行任何处理时, 可以看到明显的带有两个芘分子的电泳条带(泳道c). 在修复体系中, T4 PNK和T4 DNA连接酶同时存在, 使缺口修复, 修复的磷酸二酯键可以抵抗Bst DNA聚合酶的外切酶活性, 从而保持完整的序列结构, 由于Oligo mix与修复产物仅相差一个磷酸二酯键, 而具有相同的碱基数, 因此在非变性DNA-PAGE中可以在Oligo mix相同的位置观察到条带(泳道d). 然而, 在未修复体系中, 不存在T4 PNK或T4 DNA连接酶时, 缺口为Bst DNA聚合酶提供了一个结合位点, Oligo A被外切酶活性完全水解, 然后通过聚合酶活性产生新的替代链, 因此, 可以检测到一条含有一个芘分子的未修复条带, 而没有出现修复的电泳条带(泳道e). 未修饰芘的Oligo B和Oligo full-length作为分子量标准, 序列最短的Oligo B位于底部(泳道a), 全长片段比未修复产物少1个芘分子, 比修复产物少2个芘分子, 因此位于两条带的底部(泳道b), 3条条带从下至上依次分布. 以上结果表明, 基于芘分子二聚体的寡核苷酸探针方法检测SSBR是完全可行的. 芘二聚体这种特征的大范围斯托克斯位移(约100 nm)和-CD的荧光信号放大作用增强荧光信号与背景比值差异, 使检测系统更加灵敏, 使芘分子代替传统荧光团标记核酸探针成为可能[29~32].
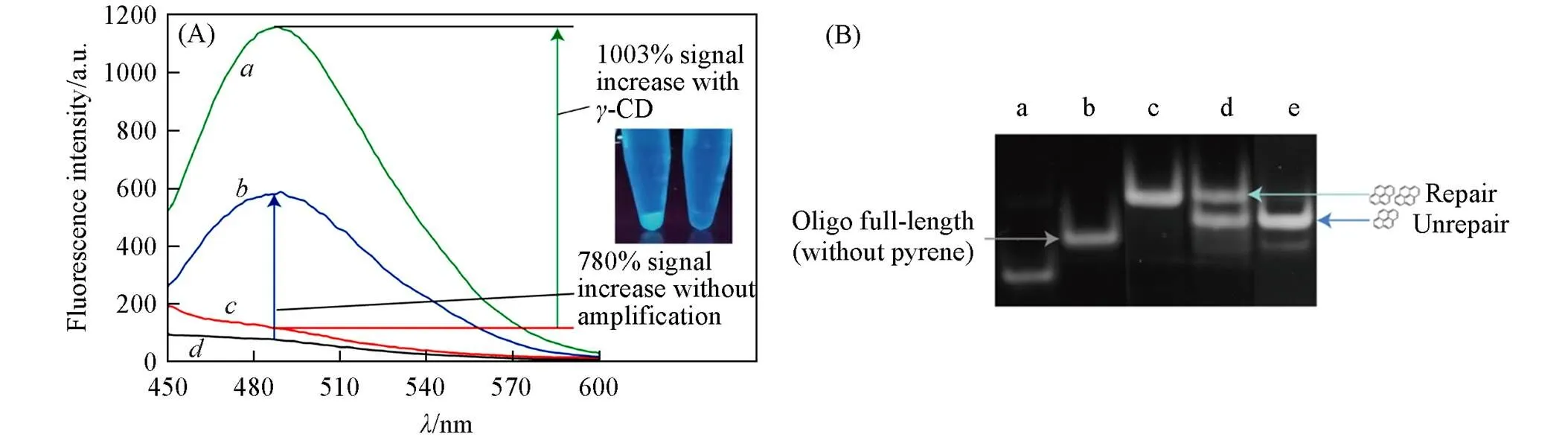
Fig.1 Practicability analysis of the pyrene excimer⁃based oligonucleotide probe system
(A) Fluorescence spectra change between repair and unrepair sensing system in the presence or absence of-CD. Repair:. Oligo mix+T4 PNK+T4 DNA ligase+Bst DNA polymerase+-CD,. Oligo mix+T4 PNK+T4 DNA ligase+Bst DNA polymerase; Unrepair:. Oligo mix+Bst DNA polymerase+-CD,. Oligo mix+Bst DNA polymerase. Inset: fluorescence photos of repair system(left) and unrepair system(right). (B) DNA-PAGE analysis of the different products. a. Oligo B; b. Oligo full⁃length(Oligo A+Oligo B without pyrene); c. Oligo mix; d. repair; Oligo mix+T4 PNK+T4 DNA ligase+Bst DNA polymerase; e. unrepair, Oligo mix+Bst DNA polymerase.
2.2 实验条件优化
为了得到最优的结果, 从Bst DNA聚合酶活性、 反应时间、 Oligo mix浓度、 反应温度等方面进行了一系列优化实验. Bst DNA聚合酶活性是影响荧光强度的关键因素, 直接决定了背景荧光. 首先, 研究了Bst DNA聚合酶活性对反应体系的影响. 向体系中加入100 U/mL或200 U/mL Bst DNA聚合酶, 由于不存在T4 PNK和T4 DNA连接酶, 水解后的芘二聚体荧光值代表背景荧光值. 反应体系中200 U/mL Bst DNA聚合酶的效果明显优于100 U/mL, 且在30 min时达到最小荧光值[图2(A)], 即在65 ℃条件下, 200 U/mL Bst DNA聚合酶需要30 min达到最佳效果, 背景值最低. 为了分析最佳反应时间, 使T4 PNK和T4 DNA连接酶与Oligo mix孵育不同时间(0~120 min), 由图2(B)和图S2(见本文支持信息)可见, 随着修复时间的延长, 检测系统的荧光强度先上升, 90 min后达到停滞期, 说明T4 PNK和T4 DNA连接酶在90 min时可以达到最高效率, 因此后续实验选择孵育90 min. Oligo mix浓度和反应温度的优化结果如图S3和图S4(见本文支持信息)所示.
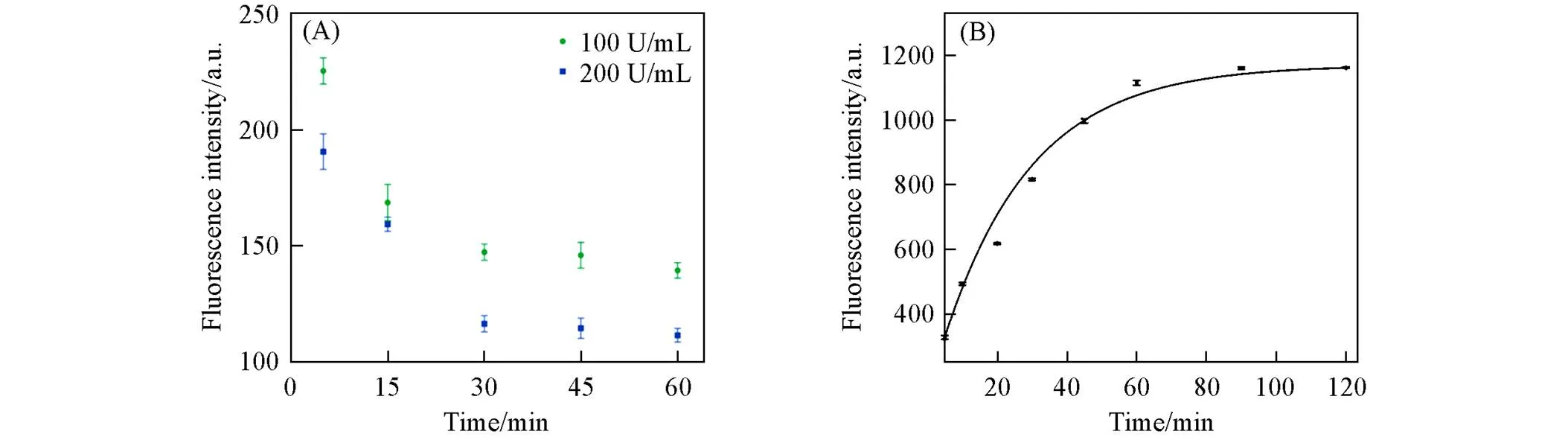
Fig.2 Condition optimization of the pyrene excimer⁃based fluorescence system
(A) Fluorescence intensity changes of solutions at 488 nm under different Bst DNA polymerase incubating at different times; (B) fluorescence intensity as a function of the reaction time.
2.3 关键修复酶活性的检测
T4 PNK和T4 DNA连接酶在3′-5′磷酸二酯链的重建中起着重要的磷酸化和连接作用. 通过不同酶浓度下的荧光发射光谱, 检测了T4 DNA连接酶和T4 PNK[图3(A)和(C)]. 首先, 准备了5种浓度T4 DNA连接酶, 验证了在T4 PNK浓度固定的情况下, 该报告系统对T4 DNA连接酶的检测能力, 并记录了488nm处的荧光强度. 由图3(B)可见, 芘二聚体的荧光强度随着T4 DNA连接酶浓度的升高而增加, 在T4连接酶浓度为1.6 U/μL时, 荧光强度达到峰值. 随后, 在上述最优实验条件下, 通过荧光光谱探索该系统对T4 PNK的检测能力. 由图3(D)可见, 随着T4 PNK浓度的增加, 488 nm处的荧光强度逐渐升高, 在T4 PNK浓度为100 U/mL时, 荧光强度达到最大值; 并且该报告系统对T4 PNK的最低检测限为0.1 U/mL. 此外, 建立了T4 PNK浓度与荧光强度的拟合曲线, 在0.1~2 U/mL浓度范围内存在良好的线性相关性(2=0.9901). 在适当的T4 DNA连接酶和T4 PNK存在时, 可以观察到明显的荧光, 表明系统完成了修复过程.
(A) Fluorescence emission spectra with different concentrations of T4 DNA ligase;(B) fluorescence intensity changes of solutions at 488 nm under different concentrations of T4 DNA ligase, T4 PNK concentration was fixed at 200 U/mL;(C) fluorescence emission spectra with different T4 PNK concentrations;(D) the relationship between fluorescence ratio at 488 nm and T4 PNK concentration. Ligase concentration was fixed at 1.6 U/μL.
2.4 不同细胞SSBR能力的评估
由于核蛋白中含有丰富的DNA修复酶, 并且据相关文献报道, 核蛋白被用于研究SSBR[19], 因此本部分实验不再加入T4 DNA连接酶和T4 PNK, 而将寡核苷酸探针与核蛋白孵育, 直接评估不同细胞的SSBR能力. 通过用20 μL样本释放剂裂解1×106个细胞, 获得核蛋白复合物, 以确保不同细胞之间具有可比性. 在Oligo mix中加入小鼠胎儿成纤维细胞核蛋白复合物后, 在488 nm处观察到显著的荧光值, 说明小鼠胎儿成纤维细胞具有SSBR能力. 然而, 并不是核蛋白浓度越高, 荧光强度越强. 核蛋白中的核酶水解寡核苷酸探针, 因此需要在增加荧光强度和核酶活性之间寻求平衡. 由图4(A)可见, 随着核蛋白含量的增加, 荧光值先升高后降低, 在0.5 μL处达到峰值; 基于芘二聚体的发夹探针设计具有更少的末端, 并非典型的三条寡核苷酸形成带有缺口或间隙的双链, 用于防止核蛋白中潜在的核酸外切酶活性. 在上述结果的基础上, 对肿瘤细胞和细胞系的SSBR能力进行了研究. 结果表明, 与原代胎儿成纤维细胞相比, 肿瘤细胞和细胞系的荧光强度与背景值几乎相同, 说明肿瘤细胞和细胞系几乎不具备SSBR能力[图4(B)和图S5, 见本文支持信息]. 此外, 简要研究了癌细胞不具备SSBR能力的原因. 向Hela细胞的反应系统中加入额外的T4 PNK或T4 DNA连接酶, 以确定肿瘤细胞SSBR缺失是否是关键修复酶活性降低的结果. 结果表明, 488nm处的荧光信号与原始强度相比没有明显变化, 相反, 在胎儿成纤维细胞中, 随着T4 PNK和T4 DNA连接酶的加入, 荧光明显增强[图4(C)], 由此推测肿瘤细胞核蛋白复合物中的某些物质抑制了SSBR, 而该物质在胎儿成纤维细胞中不表达. 随后对核蛋白进行SDS-PAGE检测, 结果表明不同细胞的核蛋白复合物中蛋白含量不同, 因此在SDS-PAGE上具有条带不同(图S6, 见本文支持信息), 蛋白质组成的差异决定了SSBR能力的差异. 此外, 还对该系统的适用性进行了研究. 分别从小鼠子宫细胞、 心脏细胞、 脾脏细胞、 肺细胞和肾细胞中获得核蛋白复合物, 将上述核蛋白复合物分别与Oligo mix孵育. 由图4(D)和图S7(见本文支持信息)可见, 在不同的体系中均可以观察到明显的二聚体荧光信号, 说明这些细胞都具有SSBR能力. 该系统不仅可用于评价小鼠胎儿成纤维细胞的SSBR能力, 也可用于评价其它原代细胞的SSBR能力. 因此, 该方法具有广泛的适用性, 并且SSBR可能作为鉴别原代细胞和肿瘤细胞系的新指标.
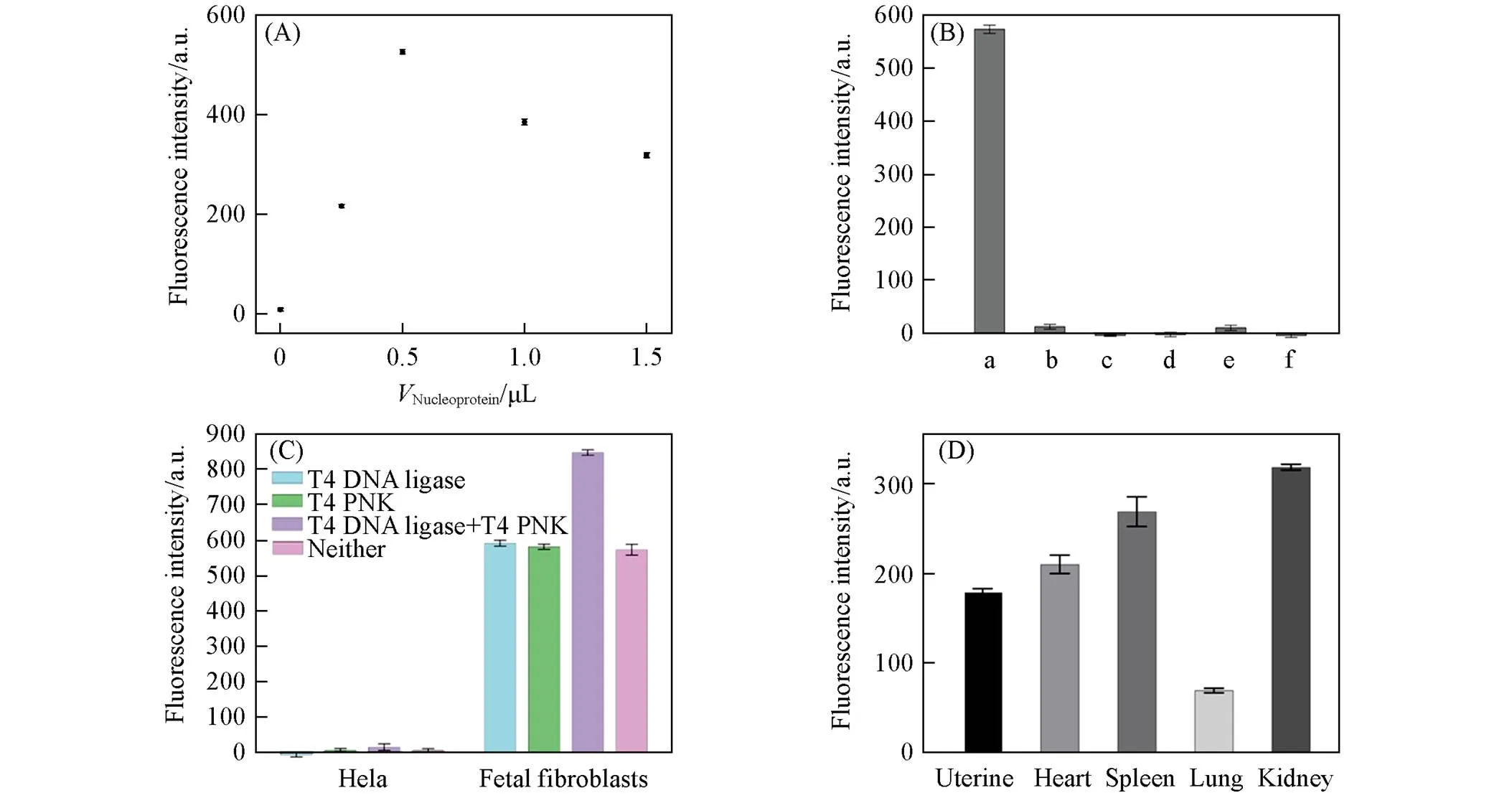
Fig.4 Study on the difference in SSBR ability between different cells
(A) Optimization of mouse fetal fibroblast nuclear protein complex; (B) fluorescence intensity of pyrene excimer in different cells’ nucleoprotein complexes. a. Fetal fibroblasts, b. HepG2, c. 296T, d. Hela, e. PK-15; f. A549; (C) fluorescence intensity of the Hela and fetal fibroblast detection system with extra ligase or T4 PNK; the amount of T4 DNA ligase and T4 PNK is 1600 U/mL and 200 U/mL, respectively; (D) fluorescence intensity of pyrene excimer in different primary cells.
2.5 报告系统对SSBR评估的灵敏度检测
为了探索基于芘分子的发夹探针灵敏度, 在最佳实验条件下, 利用该报告系统对小鼠胎儿成纤维细胞的SSBR检测限进行了研究. 用20 μL样本释放剂裂解不同数量的细胞, 在反应体系中加入0.5 μL核蛋白复合物. 由图5可见, 488 nm的处荧光强度随着细胞数量的减少而下降. 当细胞数量达到 500个时, 荧光强度达到最低点. 因此, 基于芘二聚体的荧光报告系统可检测到的最小细胞数为500, 即该报告系统可以直接评估500个细胞的SSBR能力. 或许, 在将来可以引入基于CRISPR-Cas12a/Cas13a的无扩增DNA生物传感器来提高检测灵敏度[33,34].
(A) Fluorescence emission spectrum with different numbers of cells; (B) fluorescence intensity of pyrene excimer at 488 nm with different numbers of cells.
2.6 抗衰老药物的筛选
在实验过程中, 发现二聚体荧光强度随着原代胎儿成纤维细胞的传代逐渐降低[图6(A), 对照组], 即在细胞衰老过程中, SSBR的能力下降, 但仍高于肿瘤细胞系, 这一现象与Nassour的研究 结果一致[9]. 因此, 本部分实验通过SSBR与衰老的关系, 利用报告系统评估SSBR从而筛选抗衰老 药物. 为了评价抗衰老药物, 首先进行了细胞毒性试验. 细胞毒性实验结果表明, 几种药物毒性较小[图6(B)]. 在细胞毒性试验后, 将该检测系统应用于抗衰老药物的筛选. 将NMN、 亚精胺和维生素C加入细胞培养基中, 随后进行细胞传代培养, 收集胎儿成纤维细胞, 获得核蛋白复合物, 评估P5~P9细胞的SSBR能力. 结果表明, 化合物NMN和亚精胺可以延缓胎儿成纤维细胞SSBR的下降, 从而延缓衰老; 而维生素C作用不显著[图6(A)]. 因此, 该报告系统可以通过评估SSBR应用于抗衰老药物筛选.

Fig.6 Detection of potential anti⁃aging drugs
(A) Cell viability test by CCK-8;(B) fluorescence intensity of the detection system treated with anti-aging drugs. The concentration of NMN, spermidine and Vitamine C is 100 μmol/L, 20 μmol/L, and 100 μmol/L, respectively.
3 结 论
利用芘二聚体发夹探针设计了一种评价细胞SSBR能力的方法. 该方法具有设计简单、 易于操作的优势, 可以使评价细胞SSBR能力如同PCR反应一样方便; 而且检测快速, 进行完整的检测工作仅需2.5 h, 大幅度缩短了测试时间; 该研究可用于评估细胞的SSBR能力, 在肿瘤细胞鉴别方面有巨大的应用潜力. 此外, 本文研究结果在抗衰老药物的筛选方面也具有很大的市场价值.
支持信息见http://www.cjcu.jlu.edu.cn/CN/10.7503/cjcu20230288.
[1] Abbotts R., Wilson D. M. Ⅲ.,, 2017,, 228—244
[2] Caldecott K. W.,, 2022,(9), 733—745
[3] Caldecott K. W.,, 2008,(8), 619—631
[4] Rettig T. A., Ward C., Bye B. A., Pecaut M. J., Chapes S. K.,, 2018,(1), e0190982
[5] Torchinsky D., Michaeli Y., Gassman N. R., Ebenstein Y.,, 2019,(76), 11414—11417
[6] Caldecott K. W.,, 2003,(1), 7—10
[7] Hirano M., Yamamoto A., Mori T., Lan L., Iwamoto T. A., Aoki M., Shimada K., Furiya Y., Kariya S., Asai H., Yasui A., Nishiwaki T., Imoto K., Kobayashi N., Kiriyama T., Nagata T., Konishi N., Itoyama Y., Ueno S.,, 2007,(2), 162—174
[8] Taylor R. M., Thistlethwaite A., Caldecott K. W.,, 2002,(8), 2556—2563
[9] Nassour J., Martien S., Martin N., Deruy E., Tomellini E., Malaquin N., Bouali F., Sabatier L., Wernert N., Pinte S., Gilson E., Pourtier A., Pluquet O., Abbadie C.,, 2016,, 10399
[10] Higo T., Naito A. T., Sumida T., Shibamoto M., Okada K., Nomura S., Nakagawa A., Yamaguchi T., Sakai T., Hashimoto A., Kuramoto Y., Ito M., Hikoso S., Akazawa H., Lee J. K., Shiojima I., McKinnon P. J., Sakata Y., Komuro I.,2017,(1), 15104
[11] Kuzminov A.,, 2001,(15), 8241—8246
[12] Lees⁃Miller S. P., Meek K.,, 2003,(11), 1161—1173
[13] Toulany M.,, 2019,(1), 25
[14] Tubbs A., Nussenzweig A.,, 2017,(4), 644—656
[15] Cook P. R., Brazell I. A.,, 1976,(5579), 679—682
[16] Speit G., Rothfuss A.,, 2012,, 79—90
[17] Olive P. L., Banáth J. P.,, 2006,(1), 23—29
[18] Wu W., Hill S. E., Nathan W. J., Paiano J., Callen E., Wang D. P., Shinoda K., van Wietmarschen N., Colón⁃Mercado J. M., Zong D. L., De Pace R., Shih H. Y., Coon S., Parsadanian M., Pavani R., Hanzlikova H., Park S., Jung S. K., McHugh P. J., Canela A., Chen C., Casellas R., Caldecott K. W., Ward M. E., Nussenzweig A.,, 2021,(7859), 440—444
[19] Whitehouse C. J., Taylor R. M., Thistlethwaite A., Zhang H., Karimi⁃Busheri F., Lasko D. D., Weinfeld M., Caldecott K. W.,, 2001,(1), 107—117
[20] Qing T. P., He X. X., He D. G., Ye X. S., Shangguan J. F., Liu J. Q., Yuan B. L., Wang K.,, 2017,, 456—463
[21] Jiao H. P., Wang B., Chen J., Liao D., Li W. Y., Yu C.,, 2012,(63), 7862—7864
[22] Zhu W. P., Li Z. Y., Dai L. Y., Yang W. J., Li Y. X.,, 2023,(3), 297—302
[23] Liu H. L., Zhu C. Q., Mou C. Y.,, 2022,(1), 26
[24] Furman J. L., Mok P. W., Shen S., Stains C. I., Ghosh I.,, 2011,(1), 397—399
[25] Ebata K., Masuko M., Ohtani H., Kashiwasake⁃Jibu M.,, 1995,(5), 836—839
[26] Kitamura M., Nimura A., Yamana K., Shimidzu T.,, 1991,, 67—68
[27] Hoche J., Schmitt H. C., Humeniuk A., Fischer I., Mitric R., Rohr M. I. S.,, 2017,(36), 25002—25015
[28] Wu C. C., Wang C. M., Yan L., Yang C. J.,, 2009,(5), 495—504
[29] Conlon P., Yang C. J., Wu Y., Chen Y., Martinez K., Kim Y., Stevens N., Marti A. A., Jockusch S., Turro N. J., Tan W.,, 2008,(1), 336—342
[30] Zhang Q. E., Deng T., Li J. S., Xu W. J., Shen G. L., Yu R. Q.,, 2015,, 253—258
[31] Masuko M., Ohtani H., Ebata K., Shimadzu A.,, 1998,(23), 5409—5416
[32] Ghosh P., Mandal S., Das T., Maity A., Gupta P., Purkayastha P.,, 2012,(32), 11500—11507
[33] Choi J. H., Lim J., Shin M., Paek S. H., Choi J. W.,, 2021,(1), 693—699
[34] Shinoda H., Taguchi Y., Nakagawa R., Makino A., Okazaki S., Nakano M., Muramoto Y., Takahashi C., Takahashi I., Ando J., Noda T., Nureki O., Nishimasu H., Watanabe R.,, 2021,(1), 476
Direct Evaluation of Single-strand Break Repair Capacity of Cells Using a Pyrene Excimer-based Hairpin Probe
LIYin, LIUJinsong, WANGYu, QITingting, MENGLeyuan, ZHANGJing,LYUDongmei, JIAOHuping*
(,,130062,)
A novel pyrene excimer-based hairpin probe with a nick at the stem was developed to directly evaluate the single-strand break repair(SSBR) ability. In the existence of appropriate activity of DNA repair-associated enzymes(DREs), the oligonucleotide probe could prevent the digestion of Bst DNA polymerase and keep the long-wavelength excimer signal. However, in the absence of DREs, the nicked probe could be digested by Bst DNA polymerase, bringing about a “turn-off” monomer fluorescence signal. Therefore, the fluorescence changes of the probe could be used to directly evaluate the SSBR capability. After feasibility verification and a series of conditions optimization, the assessment of SSBR capacity by nucleoprotein extracted from different cells was investigated and the results showed that, compared to the primary cell, the tumor cell lines do not have the ability of SSBR. We also explored the deficiency of tumor cells SSBR by compensation with extra SSBR key enzymes and the results indicated that something might inhibit the SSBR in tumor cell lines. Furthermore, the reporter system could detect SSBR of 500 cells and was successfully applied to anti-aging drug screening. The advantages of this method include simple procedure, less time, and good repeatability, and this method can be used for the rapid detection of SSBR capacity of different cells.
Single-strand break repair; Pyrene excimer; Hairpin oligonucleotide probe; Primary cell; Tumor cell
2023-06-19
焦虎平, 男, 博士, 教授, 主要从事细胞分化及胚胎发育过程中DNA修复方面的研究. E-mail: jiaohp@jlu.edu.cn
国家自然科学基金(批准号: 31501909)资助.
O657.39
A
10.7503/cjcu20230288
2023-07-06.
Supported by the National Natural Science Foundation of China(No.31501909).
(Ed.: W, K, M)