使用SPE软件筛选绿尾虹雉微卫星引物及其在亲缘关系鉴定上的应用
王立,李严,岳碧松,陈黎,沈富军,寇洁,王也,侯蓉,张亮*
(1.四川省濒危野生动物保护生物学重点实验室,成都大熊猫繁育研究基地,成都 610081;2.四川大学生命科学学院生物资源与生态环境教育部重点实验室,成都 610065;3.四川蜂桶寨国家级自然保护区管护中心,四川 雅安 625700)
绿尾虹雉是国家一级重点保护野生动物,仅分布在青海、四川、甘肃等省,是高山草甸生态系统中的伞护物种(雷富民等,2002;Wang.,2017)。我国于1955年建立了绿尾虹雉圈养种群:北京动物园、北京濒危动物驯养繁殖中心等多家单位曾在绿尾虹雉人工孵化、育雏等技术上取得了多项突破(程彩云等,1996;陈冬梅等,2015),然而最终大部分圈养种群都消失了。国内谱系上现存的绿尾虹雉繁育机构仅剩四川宝兴蜂桶寨国家级自然保护区1家。
目前圈养绿尾虹雉种群繁殖的雌雄配比为1只雄鸟配1~2只雌鸟(张敬等,2019)。由于配种过程中存在更换雄鸟的现象,1只雌鸟在一个繁殖期内产蛋的父本可能来自多只雄鸟。此外,由于繁殖期多只雌鸟混合饲养,雌鸟产蛋期交错,无个体特定产蛋位,管理者无法对同一间鸟舍的鸟蛋进行母本精准识别(杨本清等,2011),更无法推断其父本,由此导致了错误的亲缘关系记录,给未来种群内近交埋下隐患,因此开展绿尾虹雉的亲缘关系鉴定非常必要。
常见的用于亲缘关系鉴定的遗传标记有微卫星标记(Visscher.,2002)和单核苷酸多态性标记(周子文等,2021)等。微卫星标记的应用非常成熟,并在多种濒危动物的亲子鉴定中进行了广泛应用,如大熊猫(张志和等,2003)、小熊猫(陈玲等,2020)等。如何从大基因组中识别微卫星,并从庞大的结果数据集中筛选微卫星进行引物设计仍然是一个关键的技术问题。
早期微卫星位点筛选采用磁珠富集法(沈富军等,2005),但耗时长、检出率低。随着生物信息学的飞速发展,学者们从基因组中直接获取微卫星位点并设计引物,将设计出的上、下游引物分别比对到参考基因组上,利用大型数据库如NCBI、UCSC和ENSEMBL,基于BLAST功能查看引物特异性。引物合成后,经PCR扩增并经琼脂糖凝胶电泳检验。目前基于生物信息数据库比对的分析方法并不能解决微卫星引物3’端碱基唯一性以及预测PCR扩增产物丰富度的问题。成都大熊猫繁育研究基地开发的微卫星PCR引物特异性鉴定程序(specific PCR production prediction,SPE)(李严等,2021)基于上、下游引物序列在基因组中进行条件匹配,并根据比对结果进行引物特异性评估,不仅将极大地降低前期筛选工作量,也能很好地排除微卫星引物存在的特殊多位点扩增(多PCR产物片段长度接近)问题。
目前还没有绿尾虹雉特异性微卫星分子标记的公开发布。在缺乏物种特异性分子标记的情况下,鸟类保护遗传学研究常采用近缘物种的微卫星位点进行交叉扩增分析,如从来自雉科Phasianidae的多种属58个位点中筛选4种8个近源鸟类微卫星位点对黄腹角雉进行种群结构分析(刘园园,2012)等。但某些雉科鸟类间微卫星交叉扩增率不高(曹露莎等,2008;刘园园,2012)。考虑到亲缘关系鉴定对分子标记的高要求,本研究针对绿尾虹雉进行了物种特异性微卫星分子标记的开发与筛选。
本研究首次在微卫星筛选过程中,应用SPE软件进行特殊多位点扩增排查,同时对经典微卫星方法筛选的位点情况进行了验证分析,最后得到的12对稳定性高、多态信息含量丰富的引物,并在绿尾虹雉同圈舍鸟蛋亲缘关系的鉴定中进行了成功应用。
1 材料与方法
1.1 样品采集及DNA提取
采集2018—2019年四川蜂桶寨国家级自然保护区绿尾虹雉圈养种群6个圈舍蛋壳样品15份(孵化后蛋壳12份,死胎蛋壳3份)、野外救护个体的羽毛样品3份,合计18份样品(表1)。

表1 绿尾虹雉样品采集表Table 1 Sample collecting of Lophophorus lhuysii
DNA提取采用血液/组织提取试剂盒(Qiagen,Germany),详细步骤参考说明书。样品中的DNA浓度用NanoDrop 2000分光光度计(Thermo Scientific,USA)测定,用2%琼脂糖凝胶电泳进行质量检测。
1.2 微卫星引物设计与多态性验证
利用Krait(Du.,2017)在绿尾虹雉基因组(崔凯,岳碧松,2018)中进行微卫星位点筛选,随机选取大量微卫星位点,合并成上、下游普通引物。
随机选取3个DNA样品进行PCR扩增,PCR体系为10 μL,反应混合物含10~20 ng DNA,1 μL 10×Taq缓冲液,0.2 μL dNTP混合 0.1μL Taq DNA聚 合 酶 ,引 物 各 0.2 μL(10 μmol·L),0.8 μL 25 mmol·LMgCl,加水至终体积。PCR反应条件:95 ℃ 10 min;94 ℃ 15 s,56 ℃ 15 s,70 ℃ 30 s,35个循环;72℃10 min。扩增产物经2%琼脂糖凝胶电泳检测,筛选出条带清晰的微卫星位点,合成荧光引物,再进一步检测位点的多态性和稳定性。
将最后筛选出的引物,应用到18份绿尾虹雉DNA样品中进行基因分型。为了确保分型数据的准确性,所有PCR均进行3次重复,基因分型在生工生物工程(上海)股份有限公司进行。
1.3 引物特异性评估
SPE软件(李严等,2021)的参数设置为:3’端完全匹配碱基数目为10,模拟PCR产物的最大长度为3 000 bp,运行软件获得引物特异性评估文件:上游引物ID、上游引物所在染色体编号、上游引物起始基因组位置、上游引物终止基因组位置、上游引物匹配链的方向、下游引物ID、下游引物所在染色体编号、下游引物起始基因组位置、下游引物终止基因组位置、下游引物匹配链的方向和预测的PCR产物长度。
1.4 数据分析
利用 Cervus3.0(Kalinowski.,2007)对得到的分型数据微卫星位点联合非亲排除概率、联合同胞排除概率、各等位基因杂合度及多态信息含量(polymorphism information content,PIC)进行统计(0<PIC<0.25为低度多态,0.25<PIC<0.5为中度多态,0.5<PIC<1为高度多态;Botstein.,1980);利用ML-Relate(Kalinowski.,2006)计算15份蛋壳DNA样品的同胞关系。将分析结果与《2020年中国动物园协会绿尾虹雉谱系簿》(内部资料)进行比对。
2 结果
对绿尾虹雉基因组上微卫星序列的搜索和统计结果与崔凯和岳碧松(2018)的一致。随机选取三碱基及以上重复位点设计了274对普通引物序列(三碱基86对、四碱基72对、五碱基116对),退火温度统一设置为56℃。引物合成后对随机选取的3份绿尾虹雉DNA样品进行PCR,琼脂糖凝胶电泳进行引物初筛,得到93个微卫星位点,在此基础上合成93对荧光引物,在6份DNA样品中进行PCR扩增及基因分型,进一步筛选得到13对引物。其中,引物CM3227基因分型图上,stutter峰值显示偏高(图1)。
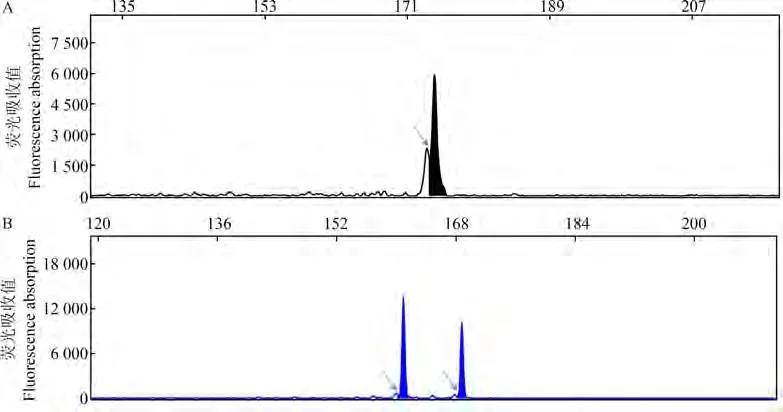
图1 2种基因分型的stutter峰Fig.1 Two types of stutter in genotype
排除了初步扩增失败的引物后,对212对微卫星引物序列利用SPE进行分析(表2)。除去11个碱基及以上匹配的多位点扩增标记,共筛选出的10个碱基匹配的分子标记为122个。

表2 212对微卫星引物的多位点扩增情况分析Table 2 Analyze of multiple amplification of 212 pairs of SSR primers
筛选出的13对引物对的SPE分析数据显示,CM3227在基因组上存在特殊多位点扩增(172 bp、173 bp)(表3),因此,分型图中异常的stutter峰其实反应的是多位点扩增的另一PCR产物。此外,用经典方法筛选出来的其他12对位点均不具有11个及以上碱基的错配。
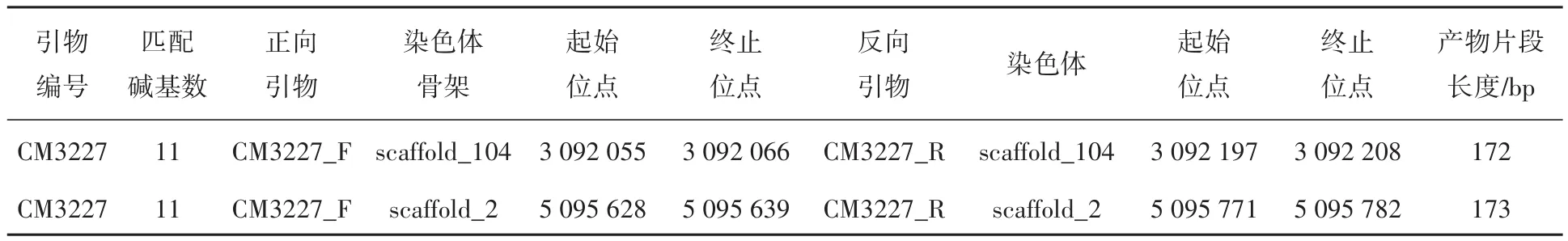
表3 引物CM3227特殊多位点扩增Table 3 Specific multiple amplification of CM3227
12个微卫星标记位点在18份绿尾虹雉DNA样品中的基因多态性分析结果显示,观察杂合度为0.278~0.941,期望杂合度为0.303~0.797,平均PIC为0.428 2;其中,8个位点(0.25<PIC<0.5)属于中度多态性位点,4个位点(PIC>0.5)属于高度多态性位点;标记组合的联合同胞排除概率为0.998 4(表4)。

表4 绿尾虹雉12 个微卫星位点的多态性特征Table 4 Polymorphic characteristics of 12 microsatellite loci in Lophophorus lhuysii
对蛋壳样品进行同胞关系(全胞、半同胞)计算(表5):5组为谱系记录的全胞关系,1组在谱系中无完整记录。经鉴定,3组同胞关系与谱系记录完全一致;1组(2份样品)内个体无同胞关系;1组(3份样品)内2只存在全胞关系,另一只与其余2只无亲缘关系;另鉴定出了1组(2份样品)内的全胞关系。

表5 15只绿尾虹雉的同胞关系鉴定Table 5 Kinship testing for 15 Lophophorus lhuysii from 6 groups
3 讨论
采用生物信息学的方法从基因组中开发微卫星位点的技术手段已经十分成熟(Warren.,2010;Zhan.,2013;Hou.,2018)。经典筛选流程在前期会设计大量引物,后期经PCR、琼脂糖凝胶电泳及基因分型等方式对引物进行逐步筛选。多位点扩增是微卫星引物在应用时最常见的问题之一,是导致设计出的引物高淘汰率的主要原因。以本研究为例,PCR扩增、琼脂糖凝胶电泳经典方法初期筛选率为33.9%(93/274),淘汰率近70%。本研究引入SPE软件进行引物特异性分析,旨在针对引物多位点扩增现象进行筛选。数据显示,SPE介入后的初期筛选率达到57.5%(122/212),接近经典方法的2倍,可以预见,在项目前期引入SPE软件进行特异性筛选,将节约近一半的时间及测试成本。
此外,SPE介入分析方法的特殊之处还在于:筛选剔除PCR产物长度接近的特殊多位点扩增。在微卫星标记的应用中,有一种特殊多位点扩增值得警惕,其扩增产物片段差异极小,无法用琼脂糖凝胶电泳或基因分型的方法进行鉴别。此类标记在应用中,容易形成分型数据的误读,从而降低标记体系整体的准确率。SPE软件分析可以对此风险进行规避,如本研究就从经典方法筛选后的13对引物中剔除了1对引物,该引物的2个PCR产物片段间仅有1个碱基的差异。
数据显示,设计出的大量绿尾虹雉微卫星引物在基因组上存在10个碱基及以上匹配的多位点扩增现象(78.8%),但11个碱基及以上位点匹配的多位点扩增现象锐减(21.2%)。实验中发现,严格控制PCR条件(如温度、Mg浓度等)可降低大量10碱基匹配的PCR扩增产物,因此,不具有11个碱基及以上多位点匹配可作为进行SPE多位点扩增筛选的基本条件。
濒危野生动物数量稀少,且动物管理者责任大,样品采集非常困难,其中血液采集的难度最大。绿尾虹雉应激反应强,特殊情况下,采血甚至可能诱发个体死亡,一般不进行个体采血。与血液相比,蛋壳与羽毛的DNA含量相对较少,且提取出的DNA常存在一定程度的降解,若以其为模板,则所应用的分子标记需要能在相对质量较低的基因组DNA中稳定扩增。本研究开发的12对引物稳定性高、多态信息含量丰富,在蛋壳及羽毛DNA中扩增效率高、稳定性好,解决了绿尾虹雉采样难、相关亲缘关系研究长期受制的局面。
应用以上12对引物,对采集到的18只绿尾虹雉样品进行多样性分析,3只野外救护个体的平均PIC为0.373,15只圈养个体的为0.406,整体为0.401。刘园园(2012)用8个微卫星位点对黄腹角雉圈养种群77只个体进行遗传多样性分析,其平均PIC为0.619。贝永建(2014)用12对微卫星引物对黑颈长尾雉天峨县种群16只个体进行遗传多样性分析,其平均PIC为0.234。本研究与其他雉类相比,PIC值偏低,这可能与样品数量较少,且多个样本间亲缘关系较近有关。
受采样限制,本研究未能获取到鉴定个体对应的父母本材料,因此只做了同胞关系分析。结果显示,目前绿尾虹雉记录中存在一定的错误,提示需要开展种群内亲缘关系鉴定。本研究开发的12个微卫星标记可进一步应用于绿尾虹雉亲子关系的鉴定。开展绿尾虹雉圈养种群遗传管理,对提高小种群的维持能力,尽可能保持种群遗传多样性具有重要意义。
感谢陈玲、吴里霞在实验部分所提供的帮助。感谢James Edward Ayala润色英文摘要。

