基于4个线粒体基因的不同海拔高原林蛙群体遗传多样性研究
郑海钱,徐志旺,徐康宁,李卉,李树然,张永普
(温州大学生命与环境科学学院,浙江 温州 325035)
受人类活动的影响,地球环境正面临前所未有的巨变,工业革命后剧烈的气候变化对全球生态系统产生了巨大的负面影响(Hoegh-Guldberg.,2019)。由于独特的生活史特征,两栖动物不仅是水生生态系统中重要的生物类群,还是联系水生和陆生环境的代表(徐士霞等,2004)。受环境变化的影响,全球超过2 000种两栖动物的生存正面临威胁,其中一半可能极度濒危或濒临灭绝(González-Del-Pliego.,2019)。群体灭绝率的升高与遗传多样性的丧失密切相关(Saccheri.,1998),低遗传多样性的群体通常表现出低适合度和高灭绝率(Markert.,2010)。研究两栖动物的遗传多样性有助于评估现有物种或群体的生存状态,从而为保护策略的制定提供科学的理论指导(崔朝霞等,2011)。
mtDNA以结构简单、进化速率较快、严格遵循母系遗传,以及几乎不发生重组的特点,被广泛应用于系统进化研究和生物遗传多样性评价(Avise.,1987;宋洁等,2008;Chen.,2009;杨文嘉,2015;杜民等,2021)。其中,部分mtDNA单个基因的分析已经得到普遍应用,但是不同基因进化速率往往存在差异,c是唯一由mtDNA编码的细胞色素,参与线粒体呼吸链电子传递,进化速率适中;基因是mtDNA中比较保守的部位,进化速率略低于基因;而线粒体基因和基因最保守(Hwang.,1999)。这导致同一物种采用不同基因分析可能会得出不同的结论,因此采用多基因联合分析物种系统发育与遗传多样性,以期得到更准确的结果(董依萌等,2020)。
青藏高原以其特殊的地理位置和气候条件(如温度、氧浓度、紫外线辐射等),孕育了一大批独特的生物资源,使青藏高原成为中国重要的生态安全屏障,以及研究生物对高原环境适应性进化的热点地区(鲁春霞等,2004;高伟等,2019)。高原林蛙是青藏高原常见的一种两栖动物,分布海拔为2 000~4 400 m,背面皮肤较粗糙,呈灰褐色、棕褐色、棕红色或灰棕色。对高原林蛙的研究集中于活动特征(齐银等,2007a,2007b)、种间竞争(张晋东等,2007;陈伟等,2018)、生活史(Zhao.,2014;Yu.,2018)和种间遗传关系(Yang.,2017;Wang.,2020)等方面,遗传多样性研究相对较少(Zhou.,2013),海拔梯度差异研究更少。本研究基于线粒体、、和基因对不同海拔(2 000 m、2 600 m、3 200 m和3 800 m)的高原林蛙群体进行遗传多样性研究,并通过对和2个蛋白编码基因的正选择分析,探讨高原林蛙的高原适应机制,以期为其保护提供科学依据。
1 材料与方法
1.1 样品采集
样品于2020年7—8月和2021年7月采自青海省民和县 巴 州 镇(100°45′E,36°12′N,海 拔2 000 m;=22)、海南藏族自治州共和县龙羊峡水库(100°41′E,36°4′N,海拔 2 600 m;=30)、共和县湖东种羊场(100°46′E,36°42′N,海拔 3 200 m;=24)和果 洛 藏 族 自 治 州 玛 沁 县(100°14′E,34°27′N,海拔3 800 m;=24),分别定义为2 000 m群体、2 600 m群体、3 200 m群体和3 800 m群体。剪取高原林蛙左后肢的第三趾(Perry.,2011),95%乙醇固定后-80℃保存。
1.2 DNA提取,PCR扩增与测序
采用EZUP柱式动物基因组DNA抽提试剂盒(上海生工生物工程股份有限公司),并参考说明书,提取样本总DNA,将DNA溶于CE Buffer中,用超微量核酸蛋白测定仪(Nano-600,上海嘉鹏科技有限公司)检测浓度,合格样品-80℃保存。
PCR 反应体系为 25 μL:2×Taq PCR StarMix(北京康润诚业生物科技有限公司)12.5 μL,去离子水9.5 μL,DNA模板1 μL,上、下游引物(表 1)各1 μL。PCR反应条件为:95℃预变性3 min;95℃变性30 s、退火30 s(退火温度分别为:57.1℃、:50℃、:50℃、:58℃)、72℃延伸30 s,35个循环;72℃延伸5 min,通过1%琼脂糖凝胶电泳(150 V,25 min)检测PCR产物,送北京擎科生物技术有限公司双向测序。
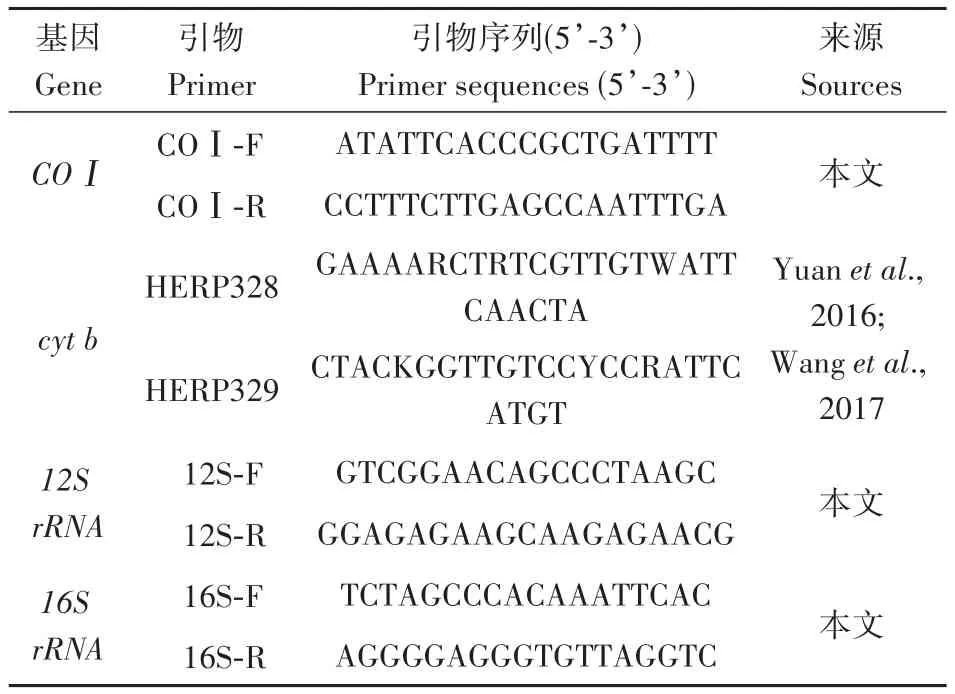
表1 本研究中使用的引物Table 1 The primers used in this study
1.3 数据处理
将所得双向序列经ContigExpress拼接后与NCBI数据库进行BLAST比对,排除假基因的可能,用DNAMAN(Ji.,2020)进行序列对比,剪去多余的片段,保存成fasta格式,使用Sequence Matrix将4个基因合并成联合数据集。
利用DNASP 5.0(Librado.,2009)分析多态位点数、单倍型多样性()、核苷酸多样性()、平均核苷酸差异数、错配分布;利用MEGA7.0(Kumar.,2016)分析碱基组成,计算变异位点数、简约信息位点数、单突变位点;以倭蛙和桓仁林蛙为外 群(NCBI),用 IQtree构 建 最 大 似 然 树(Nguyen.,2015)。
基于Easycodeml(Gao.,2019)分别对线粒体的2个蛋白编码基因(和)进行选择压力分析,在分支模型上选择位点模型,位点模型主要选用M0(单一比率)、M1a(近中性)、M2a(正选择)、M3(离散)、M7(beta)、M8(beta& ω)、M8a(beta& ω=1),利用似然比检验(LRT)统计评估成对模型:M0 vs.M3、M1a vs.M2a、M7 vs.M8、M8 vs.M8a,检测正选择压力。使用Arlequin3.5(Excoffier&Lischer,2010)计算群体遗传分化系数(),并利用分子变异分析(analysis of molecular variance,AMOVA)检测遗传变异的来源,通过1 000次重复抽样检验不同遗传结构水平上协方差的显著性,进行 Tajima’s和 Fu’s中性检验(Tajima,1989)评估高原林蛙群体动态,基因流()=(1-)/4(Slatkin.,1987)。通过 POPART(Leigh.,2015)绘制单倍型TCS网络图,推算各个单倍型间的进化关系。
2 结果与分析
2.1 线粒体基因序列特征遗传多样性分析
共得到100只高原林蛙的、、和基因序列片段,将每只个体的4条线粒体基因片段合并为1条长度为4 055 bp的线粒体序列,即4个线粒体基因联合数据集。检测到63个变异位点,其中单一信息位点和简约信息位点分别有31个和32个,A+T含量为55.4%。
4个群体线粒体基因联合数据集定义了34个单倍型,其中,2 000 m群体、2 600 m群体、3 200 m群体、3 800 m群体分别为10个、9个、5个、11个,共55个多态位点,、和平均核苷酸差异数分别为0.001 62±0.000 08、0.935±0.012和 6.443。3 200 m群体的遗传多样性最低,基于4个线粒体基因联合数据集的和分别为0.587±0.102和0.000 19±0.000 05;2 600 m群体的遗传多样性最高,基于4个线粒体基因联合数据集的和分别为0.860±0.036和0.001 21±0.000 21;2 000 m群体的遗传多样性高于3 800 m群体(表2)。
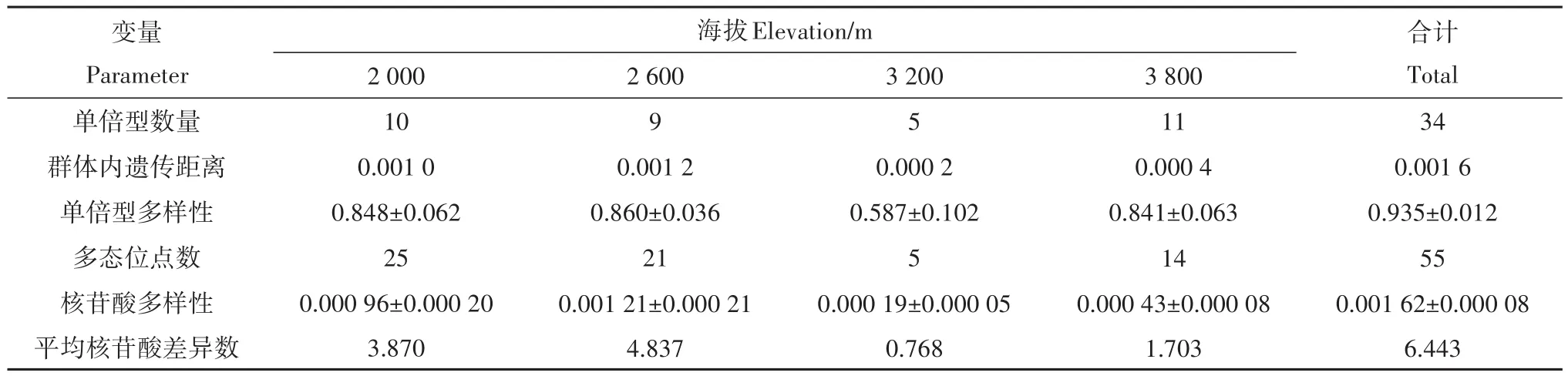
表2 基于线粒体基因分析青海省4个高原林蛙群体的遗传多样性Table 2 Genetic diversity of 4 Rana kukunoris populations in Qinghai Province based on mitochondrial genes
2.2 群体遗传结构与遗传分化
TCS单倍型网络图显示(图1),高原林蛙2 000 m群体和2 600 m群体组成一个进化单元。4个基因联合建树结果表明(图2),系统发育树分为2个支系,2 000 m群体多数样本(15/22)、2 600 m群体多数样本(24/30)和3 200 m群体全部样本为1个支系,2 000 m群体少数样本(7/22)、2 600 m群体少数样本(6/30)和3 800 m群体全部样本为另一支系。
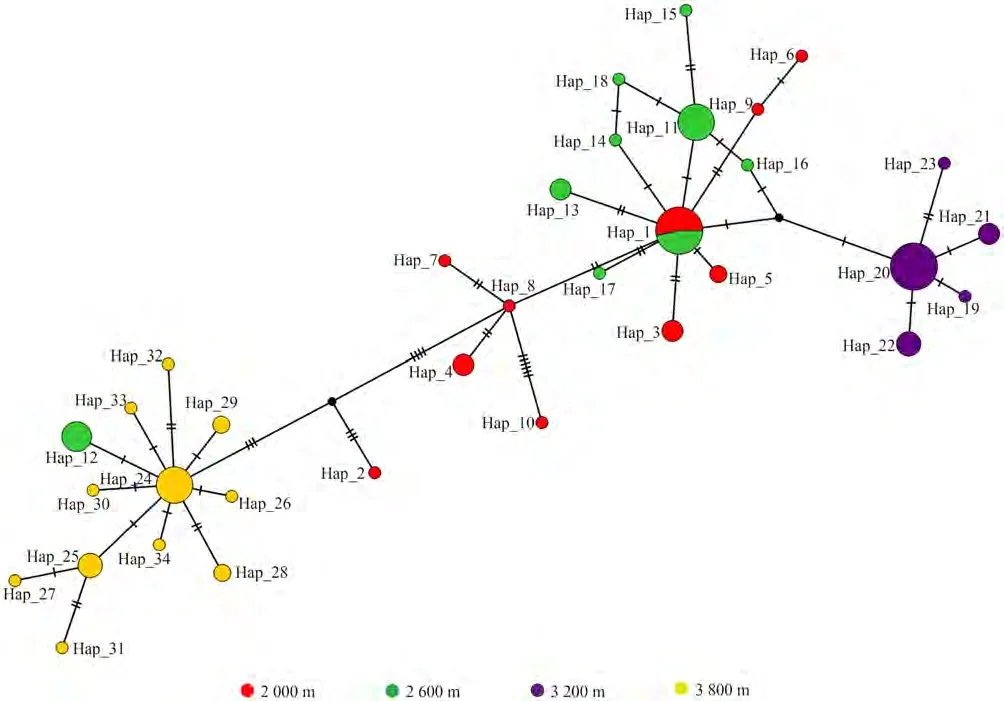
图1 青海省4个高原林蛙群体线粒体基因的TCS网络Fig.1 TCS network of mitochondrial gene of 4 Rana kukunoris populations in Qinghai Province
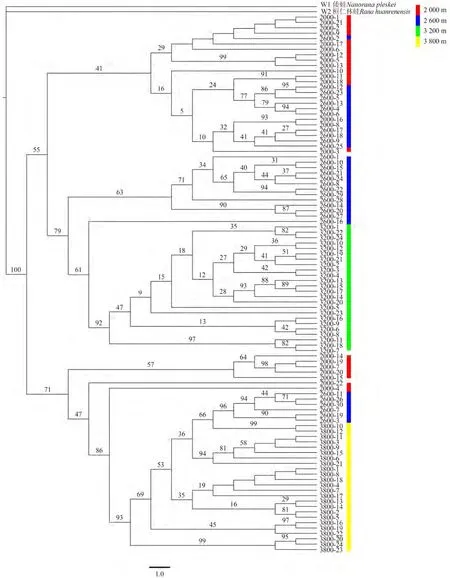
图2 基于线粒体基因构建的青海省4个高原林蛙群体最大似然法系统发育树Fig.2 Maximum-likelihood phylogenetic tree of 4 Rana kukunoris populations in Qinghai Province based on mitochondrial genes
群体间的为0.090~0.859,除了2 000 m群体和2 600 m群体外,其他两两群体间均发生了极显著的遗传分化(表3)。两两群体间的为0.09~2.53。AMOVA结果显示(表4),线粒体基因群体间分子变异和群体内分子变异分别占63.01%和36.99%,分子变异主要发生在群体间。总为0.630,且<0.001。群体间的是 2.52。基于和基因的选择压力分析结果表明(表5),M8模型更灵敏,M8模型中,基因检测到2个正选择位点,包括2 600 m群体检测到1个正选择位点(天冬氨酸D→天冬酰胺N),3 200 m和3 800 m群体检测到1个正选择位点(天冬酰胺N→赖氨酸K),基因没有检测到正选择位点。
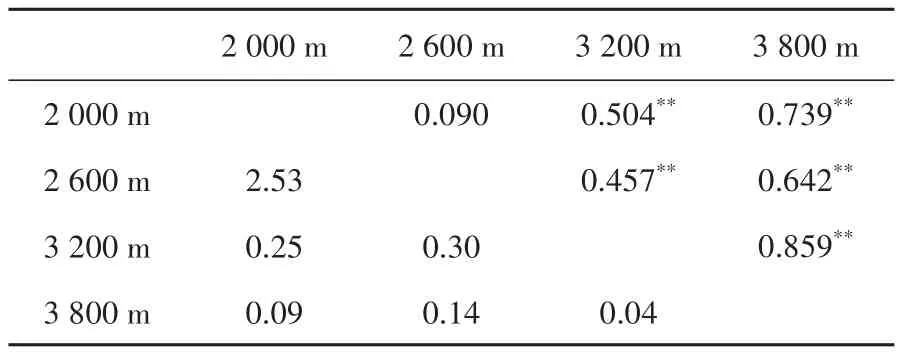
表3 基于线粒体基因的青海省4个高原林蛙群体间遗传分化系数(对角线上)和基因流(对角线下)Table 3 Genetic differentiation coefficient(above the diagonal)and gene flow(below the diagonal)among 4 Rana kukunoris populations in Qinghai Province based on mitochondrial genes
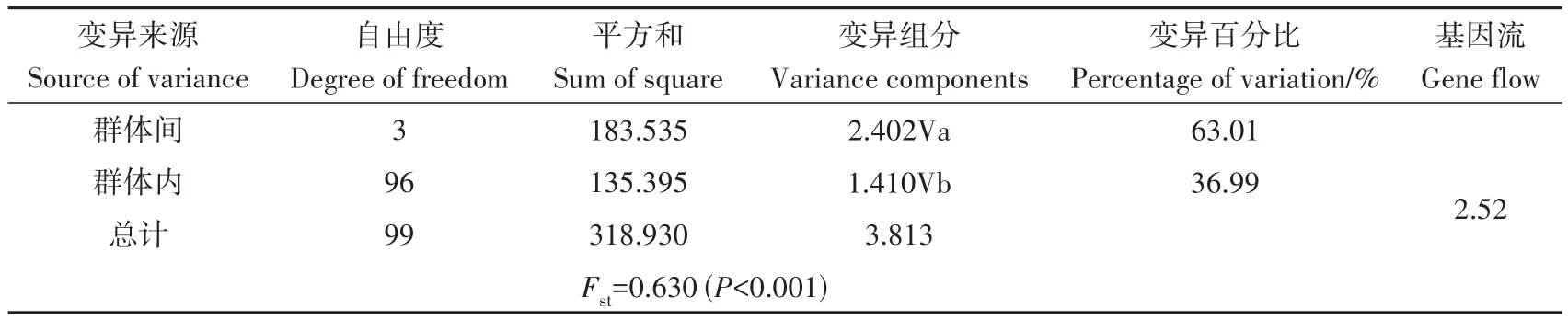
表4 基于线粒体基因的青海省高原林蛙4个群体分子方差分析结果Table 4 Results of molecular variance analysis of 4 Rana kukunoris populations in Qinghai Province based on mitochondrial genes
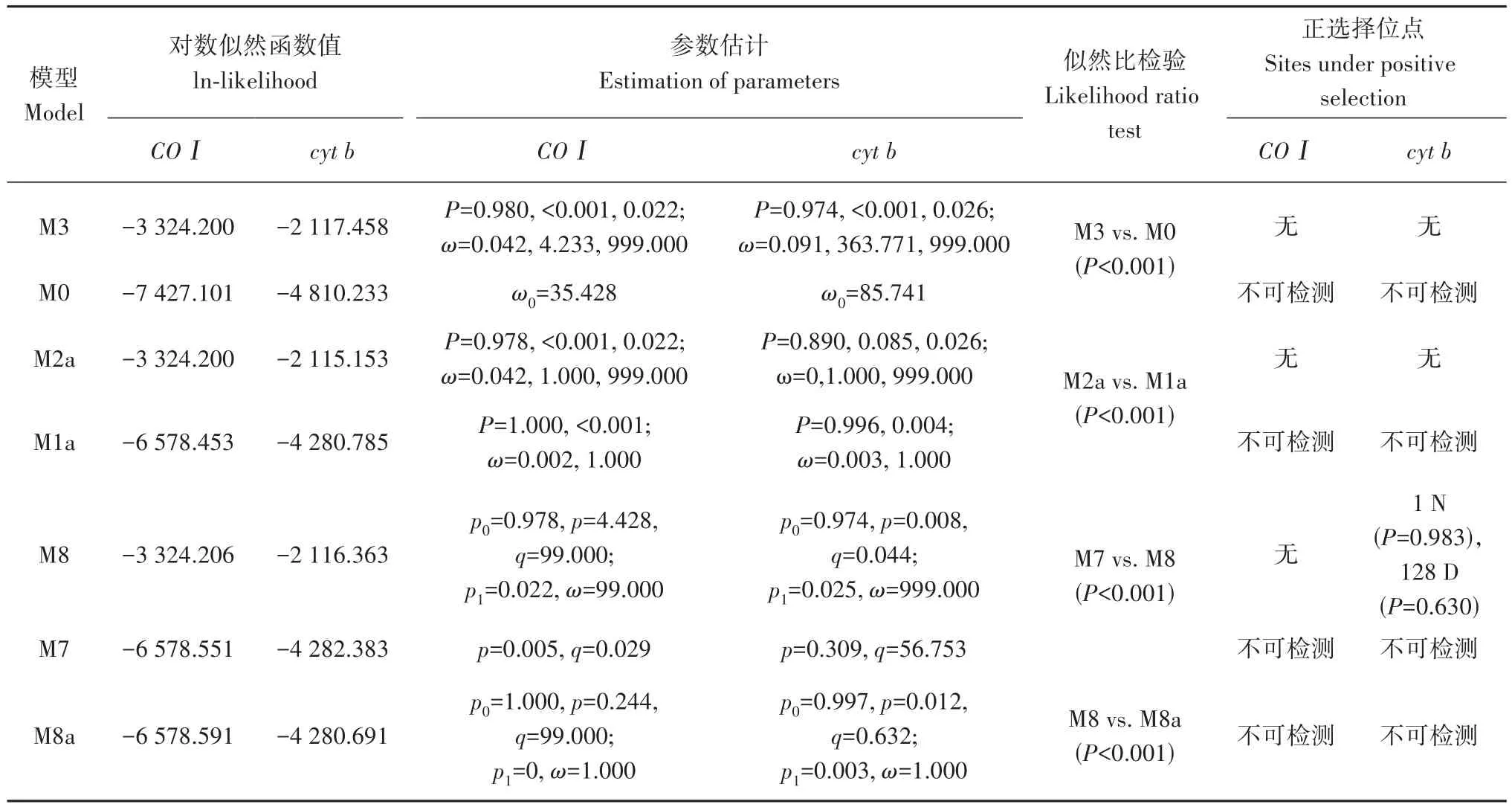
表5 青海省高原林蛙4个群体线粒体基因选择压力分析Table 5 Analysis of selection pressure of 4 Rana kukunoris populations in Qinghai Province based on mitochondrial genes
2.3 群体历史动态
4个群体中,除2 600 m群体的Fu’s检验值为正值(0.877),其余3个群体均为负值,3 800 m群体的Tajima’s和Fu’s绝对值最高、2 600 m群体的最低,Tajima’s统计显示2 000 m群体和3 800 m群体差异显著,Fu’s统计显示3 800 m群体差异显著(表6)。基于线粒体基因联合数据集的高原林蛙整体的Fu’s和Tajima’s均为负值,但差异都不显著,就单个群体而言,大多数群体的Tajima’s与Fu’s均为负值,且只有少数群体出现显著差异,另外,线粒体基因联合数据集结果显示(图3),4个群体的错配分布曲线呈双峰。
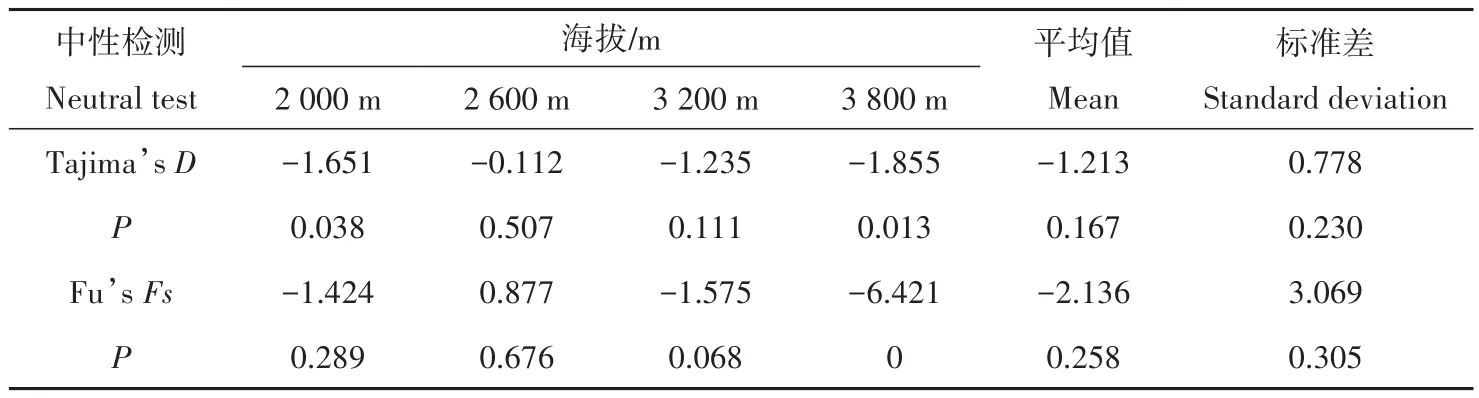
表6 青海省4个高原林蛙群体线粒体基因序列中性检测Table 6 Neutral test of 4 Rana kukunoris populations in Qinghai Province based on mitochondrial genes
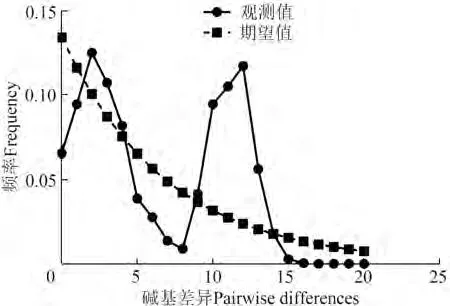
图3 基于线粒体基因的青海省4个高原林蛙群体的错配分布曲线Fig.3 Mismatch distribution curve of 4 Rana kukunoris populations in Qinghai Province based on mitochondrial genes
3 讨论
3.1 高原林蛙群体的遗传多样性
和是衡量遗传多样性的2个重要参数(刘绍平等,2010)。Grant和Bowen(1998)提出,和的标准临界值分别为0.5和0.005,将2个指数的搭配分为 4 种类型:<0.5,<0.005;>0.5,<0.005;<0.5,>0.005;>0.5,>0.005。基于线粒体4个基因联合数据集分析的遗传多样性结果属于第2种类型,这可能是高原林蛙群体曾经历过瓶颈效应后,伴随了迅速的群体扩张与变异的积累。这与北非绿蛙的遗传多样性相似(Farjallah.,2012)。此外,基于线粒体基因对不同地理群体的高原林蛙的遗传多样性研究表明,青藏高原北部地区适宜生境多于其他地区,其遗传多样性也高于其他地区(Zhou.,2013)。由此推测适宜生境的数量或面积影响遗传多样性,本研究中线粒体4个基因联合数据集对不同海拔的高原林蛙遗传多样性结果表明,2 000 m群体和2 600 m群体的和高于其他群体,推测低海拔生境适宜度整体上高于高海拔,但整体遗传多样性仍处于较低水平。而物种的遗传多样性降低将导致群体生存能力降低,甚至灭绝(Pauls.,2013)。目前高原林蛙被世界自然保护联盟(IUCN)列为无危(LC),但其较低的群体遗传多样性提示该物种同样值得进一步关注。
3.2 群体遗传结构和遗传分化
系统发育树的结果显示,2 000 m和2 600 m群体的多数样本以及3 200 m群体聚为一支,表明这3个群体的遗传关系较近。基于4个线粒体基因联合数据集的所有单倍型中,无4个群体共享单倍型,且大部分单倍型是群体独有,说明在进化过程中,每个群体为了适应外界环境形成了独特单倍型。3 800 m群体的单倍型数量高于其他群体,可能是高海拔地区人烟稀少、人为干扰较少,自然环境中高原林蛙的基因交流较频繁。AMOVA结果表明,群体间的差异大于群体内,遗传分化主要发生在群体间。Wright等(1978)建议:为0~0.05表示群体间遗传分化很小、0.05~0.15表示存在中等程度的遗传分化、0.15~0.25表示遗传分化较大、>0.25表示有很大的遗传分化。本研究中基于4个线粒体基因联合数据集的为0.630,表明高原林蛙群体遗传分化程度很大。
群体间的基因流与遗传分化负相关,当<1时,基因流弱,遗传分化处于较强水平;当1<<4时,基因流中等,遗传分化处于中等水平;当>4时,基因流强,遗传分化水平较弱(Slatkin.,1987;姚佩君,2020)。4个线粒体基因联合数据集的N为2.52,说明4个群体间的基因交流中等,但除了2 000 m群体和2 600 m群体间的N为2.53外,其他两两群体间的N均<1,和上述遗传分化结果相对应,青藏高原高原林蛙不同海拔群体间存在一定的地理隔离,这可能与两栖动物的迁移能力较弱,对所处生境较敏感有关(李郊,2014)。
检测正选择压力对于揭示生物的适应性进化机制具有重要意义(许瑜婷,高芳銮,2021)。本研究中,基因上没有检测到受正选择的氨基酸位点,说明基因未受到选择压力作用;而除了最低的2 000 m群体,其余3个群体中基因都检测到了正选择氨基酸位点。考虑到基因在有氧呼吸中的重要作用,其可能在高原林蛙适应高海拔低温、低氧环境的过程中起到了积极的作用。在高原林蛙的核基因中也发现了与能量代谢相关的基因受到了正选择(Yang.,2012)。
3.3 群体历史动态
应用Tajima’s与Fu’s中性检验推测群体历史时,如果均为负值,且达到显著差异(<0.05),则说明序列中含有比中性进化模型更多的核苷酸位点变化,预示着被研究群体可能经历过扩张(Tajima.,1989)。4个线粒体基因联合数据集的Tajima’s和Fu’s均为负值,但差异都不显著。单个群体中,大多数群体的Tajima’s与Fu’s也均为负值,且只有少数群体出现显著差异;从错配分布曲线来看,4个群体总体曲线为双峰。高原林蛙群体可能未经历过群体扩张事件。
2 000 m和2 600 m的高原林蛙群体遗传多样性高于3 200 m和3 800 m群体,4个海拔的群体遗传分化程度高,基因可能对其适应高海拔环境起到了积极作用,群体可能未经历扩张事件。本研究可为理解高原林蛙适应高原环境及其保护提供参考。

