11份蜜环菌种质rDNA-ITS序列分析
莫盛龙,夏宗元,李继,李园园,刘红昌,黄明进
(1.贵州大学农学院,贵州 贵阳 550025;2.贵州大学石斛研究院,贵州 贵阳 550025;3.福泉市华源生态农业发展有限公司,贵州 福泉 550506;4.贵州省药用植物繁育与种植重点实验室,贵州 贵阳 550025)
蜜环菌(Armillaria mellea)隶属于担子菌亚门(Basiaiomycotina)伞菌目(Agaricales)泡头菌科(Physalacriaceae)蜜环菌属[Armillaria(Fr.)Staude],是一类经济价值高并且为天麻生长所必须的共生真菌,为本身无法进行光合作用的天麻的生长繁殖提供营养物质,对天麻品质及产量的影响较大[1-4],同时它又是重要的药食兼用真菌,其产品制剂是治疗眩晕、头痛、神经衰弱的有效药物,目前已广泛应用于临床治疗[5]。蜜环菌在世界各大洲均有分布,广泛分布于北美洲、欧洲和亚洲等许多国家的热带及温带森林地区,前人通过互交不育性实验鉴定蜜环菌生物种多达40种,蜜环菌种类繁多,在我国主要有5个族17个种,其中高卢蜜环菌族有9个种,分别是Armillaria ingular、Armillaria gallica、Armillaria calvescens、Armillaria cepistipes、Armillaria sinapina、NABSX、Armillaria nabsnona、Armillaria luteopileata、Armillaria jozaensis;奥氏蜜环菌族有Armillaria ostayae、Armillaria gemina、Armillaria borealis 3个种;蜜环菌族有Armillaria mellea、CBSG 2个种;假蜜环菌族有Armillaria tabescens、Armillaria monadelpha 2个种;棘皮蜜环菌族只有Armillaria ectypa 1个种,主要分布在黑龙江、吉林、河南、山西、青海、云南、内蒙古、西藏及台湾等地区[6]。长期以来,由于鉴定手段的局限,用于鉴定真菌种的都是形态学特征,随着研究的不断深入,蜜环菌的鉴定方法从传统的形态学鉴定发展到如今的分子生物学鉴定。当前用于蜜环菌分子鉴定的技术手段主要包括扩增核糖体基因(Ribosomal DNA,rDNA)、限制性内切酶的酶切、限制性片段多态性(restriction fragment length polymorphism,RFLP)和随机扩增微卫星(random amplified microsatellite,RAMS)等[7]。其中核糖体基因被认为是最能反映物种间遗传关系的指标之一[8],真核生物核糖体基因是高度重复的串联序列单位,18S、5.8S和26S rDNA联结在一起,作为一个转录单位。18S~26S和rDNA之间的内转录间隔区(internal transcribed spacer,ITS)被5.8S分为ITS1和ITS2两部分,由于该区具有选择压力较小、DNA碱基替换速率较快、拷贝数较大、适中的长度以及快速的同步进化等特点被广泛应用于真菌类群的系统演化和发育研究[9-11]。不同蜜环菌菌株在ITS区存在丰富的变异,变异以点突变为主,且变异位点相互独立,通过扩增rDNA-ITS保守序列,可以对广泛的种类进行鉴定[12]。本文采用真菌通用引物ITS4/ITS5对11份蜜环菌种质的rDNAITS区段序列进行扩增和测序,利用MEGA-X软件对11份蜜环菌进行分类,并基于ITS序列进行核酸序列数据库GenBank同源性检索比对和系统发育树构建,使用DNAMAN软件对其特征序列进行比对分析,以期为蜜环菌种质资源的分类、鉴别及种质资源评价提供参考。
1 材料与方法
1.1 材料
供试菌株于2020年6月收集,其中M-01~M-07均来自贵州大学农学院、M-08购买于都匀市匀洞镇马场村峻康生态农业发展有限公司、M-09购买于黔东南兴昌菌业科技有限公司、M-10购买于宜昌市裕禾菌业有限公司、M-11在福泉市仙桥乡月塘村野生蜜环菌子实体分离纯化,共11份蜜环菌种质。
1.2 主要试剂及设备
真菌基因组DNA提取试剂盒(Solarbio,D2300);金牌Mix(Cat.TSE101,Lot.0BH21301);琼脂糖(Bio topped,Cat No.A6190,Lot No:190715);DNA Ladder(BIOMIGA,Cat.#:9156,Lot.#:Specified on product label);Marker(BIOMIGA,Cat.No:M2000,Lot.No:20200907);1 TBE速溶颗粒(擎科生物,Cat.TSG002,Lot.1GO20701);马铃薯葡萄糖琼脂培养基(上海博微生物科技有限公司,190710);立式高压蒸汽灭菌器(上海申安医疗器械厂,LDZF-50L);超净工作台(北京东联哈尔仪器制造有限公司,DL-CJ-1NDⅡ);电泳仪(BIORAD,Model No.PowerpacTMBasic);离心机(Thermo Fisher Scientific,Sorvall Legend 17R);PCR仪(成都锦世昌祥科技有限公司,C1000 TouchTMThermal Cycler);凝胶成像仪(BIORAD,Model No:Universal HoodⅡ,Serial No:721BR03885)等。
1.3 培养基配制
分离纯化培养基:马铃薯葡萄糖琼脂培养基(potato dextrose agar,PDA),按照说明书使用;活化培养基:每升培养基中加入马铃薯200.0 g、葡萄糖10.0 g、蔗糖10.0 g、蛋白胨2.0 g、磷酸二氢钾1.0 g、硫酸镁0.5 g及琼脂粉12.0 g,pH=6.0,马铃薯去皮,切成丁,加入适量的清水煮沸,持续沸腾期间用铁勺捣碎,单层纱布过滤,分3次进行,滤液加水至1 L后,加入其他原料,搅拌煮沸。
1.4 蜜环菌分离纯化与活化
蜜环菌的分离:将野外采集到的蜜环菌子实体在超净工作台中用75%的酒精棉球擦试菌盖表面,迅速用无菌水冲洗菌盖,再用无菌棉球擦干菌盖上的水,用镊子撕去菌盖表皮,在菌柄与菌盖交接处切取0.3~0.5 cm2的组织块,接种于PDA培养基上,置于25℃的恒温培养箱中避光培养30 d,与搜集的蜜环菌原种存放于4~8℃环境中保存备用。蜜环菌的活化:将分离纯化和收集的原种菌丝或菌块接种于活化培养基中,置于25℃的培养室内避光培养,根据褐化情况置于4~8℃环境中保存备用。
1.5 样品基因组DNA提取及测定
取活化后的新生蜜环菌菌丝,用液氮速冻,磨成粉状,按照真菌基因组DNA提取试剂盒操作。取2.5L提取的基因组DNA用0.9%琼脂糖凝胶进行电泳分析以便了解提取情况。
1.6 序列扩增
1.6.1 引物序列 本实验引物为核糖体基因序列的通用引物ITS4/ITS5[13]:ITS4碱基序列为5'-TCC TCC GCT TAT TGA TAT GC-3',ITS5碱基序列为5'-GGA AGT AAA AGT CGT AAC AAG G-3',由北京擎科生物科技有限公司重庆分公司合成。
1.6.4 扩增序列分析 通过DNAMAN6.0软件进行序列比对分析,根据初步分类结果,登录NCBI网(https://www.ncbi.nlm.nih.gov/)提交序列,BLAST搜索分析,条件为高相似度序列,下载与11份蜜环菌种质相似大的15份蜜环菌基因序列和1份茶树耐铝内生真菌序列作为外群(表1),利用MEGA-X软件进行系统发育分析。

表1 用于系统发育分析的类群的GenBank登录号Table 1 GenBank accession numbers of the taxa used for phylogenetic analysis
2 结果
2.1 PCR扩增产物检测
采用1%琼脂糖凝胶电泳在凝胶成像系统中拍照,对PCR扩增产物进行检测,结果表明,11份蜜环菌种质在ITS序列上的扩增成功率均为100%,每份蜜环菌种质在750~1 000 bp处只有1个条带且条带清晰(图1)。
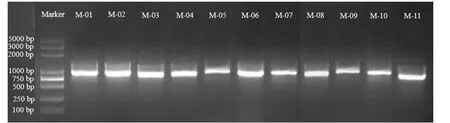
图1 11份蜜环菌种质rDNA-ITS序列PCR扩增产物检测Fig.1 Detection of PCR amplification products of the 11strains with rDNA-ITS sequences
2.2 11份蜜环菌种质聚类
11份蜜环菌种质样品测序峰图较好,序列拼接后,长度为851~895 bp,利用DNAMAN软件对所有序列进行多重对位排列,手动将对位排列结果中5'和3'端的非对位排列区去除,序列长度为848~866 bp,通过构建同源树对11份蜜环菌种质进行聚类,结果表明,11份蜜环菌种质划分为两个大类和4个亚类,第Ⅰ类包括M-01~M-05和M-08~M-10,相似度为99%,第Ⅰ-Ⅰ类包括M-01、M-02、M-04、M-05和M-08~M-10,相似度为100%,第Ⅰ-Ⅱ类为M-03;第Ⅱ类包括M-06、M-07和M-11,相似度为99%,第Ⅱ-Ⅰ类包括M-06和M-07,相似度为100%,第Ⅱ-Ⅱ类为M-11(图2)。

图2 基于rDNA-ITS序列的11份蜜环菌种聚类分析Fig.2 Clustering analysis ofstrains base d on rDNA-ITS sequence
2.3 特征序列分析
分别对这两个大类的蜜环菌种质进行多序列比对分析,同上(2.2)去除非对位排列区后,第Ⅰ类序列长度均为847 bp,第Ⅱ类序列长度均为873 bp,参考模式菌株蜜环菌(登录号:KT822289)对各区段进行大致划分,即18S区为1~34 bp,ITS1区为35~273 bp,5.8S区为274~431 bp,ITS2区为432~815 bp,28S为816~864 bp,其中第Ⅰ类蜜环菌种质M-03在ITS1区段发生5个碱基置换,分别在120 bp、121 bp、148 bp、164 bp和266 bp处由C→T,在ITS2区段发生3个碱基置换,分别在597 bp和693 bp处由A→G,在664 bp处由G→A,共有序列长度为840 bp,其中碱基A、C、G和T所占比例分别为33.0%、23.1%、21.1%和22.9%,C+G含量为44.2%(表2);第Ⅱ类蜜环菌种质M-11在ITS1区段发生4个碱基置换,分别在100 bp处由T→C,在125 bp和147 bp处由G→A,在373 bp处由A→G,在ITS2区段仅有1个碱基发生置换,在706 bp处由C→T,共有序列长度为869 bp,其中碱基A、C、G和T所占比例分别为33.7%、23.6%、19.0%和23.7%,C+G含量为42.6%(表3)。上述表明,11份蜜环菌种质在18S、5.8S和28S区较保守,变异的信息位点主要在ITS1和ITS2。
表2 第Ⅰ类蜜环菌种质特征位点Table 2 Characteristic loci of the classⅠstrains

表2 第Ⅰ类蜜环菌种质特征位点Table 2 Characteristic loci of the classⅠstrains
编号 Serial number特征位点 Characteristic loci ITS1 ITS2 120 121 148 164 266 597 693 664 M-01 C C C C C A A G M-02 C C C C C A A G M-03 T T T T T G G A M-04 C C C C C A A G M-05 C C C C C A A G M-08 C C C C C A A G M-09 C C C C C A A G M-10 C C C C C A A G
表3 第Ⅱ类蜜环菌种质特征位点Table 3 Characteristic loci of the classⅡ

表3 第Ⅱ类蜜环菌种质特征位点Table 3 Characteristic loci of the classⅡ
编号 Serial number特征位点 Characteristic loci ITS1 ITS2 100 125 147 373 706 M-06 T G G A C M-07 T G G G C M-11 C A A G T
对两个大类的共有序列进行两对序列对比分析,同上(2.2)去除非对位排列区后,两对共有序列相似度为88.71%,碱基变异位点丰富,且除碱基的置换外,还存在碱基的缺失,在18S区有1个位点碱基发生置换,在ITS1区有23个位点碱基缺失,22个位点碱基发生置换,在5.8S区有8个位点碱基发生置换,在ITS2区有21个位点碱基发生置换,12个位点碱基缺失,在28S区有4个位点碱基发生置换,1个位点碱基缺失(表4)。表明采用通用引物ITS4/ITS5扩增的序列可以对这两类蜜环菌种质进行区分。
表4 第Ⅰ类和第Ⅱ类蜜环菌种质共有序列特征位点Table 4 Characteristic loci of the classⅠandⅡcommon sequencesstrains

表4 第Ⅰ类和第Ⅱ类蜜环菌种质共有序列特征位点Table 4 Characteristic loci of the classⅠandⅡcommon sequencesstrains
编号 Serial number特征位点 Characteristic loci 18S ITS1 12 64 98 100 102 104 105 108ⅠG G G A T--AⅡA C A C G A C T编号 Serial number ITS1 109 120 121 122 123 124 125 126ⅠA G------ⅡT C C G C G C T编号 Serial number ITS1 127 128 133 144 148 162 164 165Ⅰ--G G G C C AⅡA A T C C A --编号 Serial number ITS1 196 199 235 238 240 241 242 243ⅠA T T G----Ⅱ-C G A A G T T编号 Serial number ITS1 244 255 246 249 254 257 258 262Ⅰ---C G T G CⅡC C T T C A A T编号 Serial number ITS1 263 271 275 282 365 374 379 380Ⅰ-G--A A C AⅡT A C C G C A T编号 Serial number 5.8S 381 391 392 399 400 405 409 414ⅠT G C C C G G GⅡA A T T G A C C

续表
2.4 种内种间遗传距离计算
利用MEGA-X软件,对11份蜜环菌种质进行多序列比对,同上(2.2)去除非对位排列区后,对其种内种间遗传距离进行计算,遗传距离的大小既反映了物种间的相似度大小,也反映了它们之间的遗传多样性,遗传距离越小,表明它们之间相似度越大,遗传多样性就越小。反之,遗传距离越大,它们之间相似度越小,遗传性多样就越大。结果表明:11份蜜环菌种质种内遗传距离均为0.000,第Ⅱ-Ⅰ类与第Ⅱ-Ⅱ类蜜环菌种质的种间遗传距离最大为0.035,第Ⅰ-Ⅰ类与第Ⅱ-Ⅱ类、第Ⅰ-Ⅱ类与第Ⅱ-Ⅰ类及第Ⅰ-Ⅰ类与第Ⅱ-Ⅰ类蜜环菌种质的种间遗传距离次之,分别为0.033、0.032和0.031,第Ⅰ-Ⅰ类与第Ⅰ-Ⅱ类和第Ⅰ-Ⅱ类与第Ⅱ-Ⅱ类蜜环菌种质的种间遗传距离最小,分别为0.004和0.002(表5)。因此,第Ⅱ-Ⅰ类与第Ⅱ-Ⅱ类蜜环菌种质的相似性小,具有较丰富的遗传多样性,而第Ⅰ-Ⅰ类与第Ⅰ-Ⅱ类和第Ⅰ-Ⅱ类与第Ⅱ-Ⅱ类蜜环菌种质,相似性大,遗传多样性程度较低。
表5 11份蜜环菌种质种内种间遗传距离计算Table 5 Genetic distance between species and within species of 11strains

表5 11份蜜环菌种质种内种间遗传距离计算Table 5 Genetic distance between species and within species of 11strains
编号Serial number M-01 M-02 M-03 M-04 M-05 M-06 M-07 M-08 M-09 M-10 M-01 M-02 0.000 M-03 0.004 0.004 M-04 0.000 0.000 0.004 M-05 0.000 0.000 0.004 0.000 M-06 0.031 0.031 0.032 0.031 0.031 M-07 0.031 0.031 0.032 0.031 0.031 0.000 M-08 0.000 0.000 0.004 0.000 0.000 0.031 0.031 M-09 0.000 0.000 0.004 0.000 0.000 0.031 0.031 0.000 M-10 0.000 0.000 0.004 0.000 0.000 0.031 0.031 0.000 0.000 M-11 0.033 0.033 0.035 0.033 0.033 0.002 0.002 0.033 0.033 0.033
2.5 系统发育树分析
从NCBI数据库中BLAST对比,搜索并下载与11份蜜环菌种质相似较高的15份同源的蜜环菌序列及1份茶树耐铝内生真菌序列(表1),利用MEGA-X软件进行多序列比对,采用邻接法(NJ)构建ITS序列系统发育树(图3)。结果显示11份蜜环菌种质与下载的同源蜜环菌序列均没有归在一类。因此,11份蜜环菌可能是一个较为庞大的复合群,采用通用引物ITS4/ITS5扩增的核糖体基因可以对此类蜜环菌进行分类但不适合作为鉴定的持家基因。

图3 基于rDNA-ITS序列的系统发育树状图Fig.3 Phylogenetic tree based on the rDNA-ITS sequence
3 讨论
本实验采用通用引物ITS4/ITS5扩增11份蜜环菌种质的核糖体基因,通过构建同源树聚类、特征序列对比、种内种间遗传距离计算及在NCBI数据库中下载15个相似度较大的蜜环菌序列进行系统发育分析,得出结论:11份密环菌种质可能是一个较为庞大的复合群,采用通用引物ITS4/ITS5扩增的核糖体基因可以对此类蜜环菌进行分类但不适合作为鉴定的持家基因。
杨海旭[14]对秦巴山区分离的18株蜜环菌利用通用引物ITS1/ITS4对ITS序列扩增,对序列进行两两相似比较,通过构建系统进化树对18株蜜环菌进行了鉴定及亲缘关系分类;彭述敏[15]采用通用引物ITS4/ITS5对供试3个蜜环菌菌株进行鉴定,初步确定各蜜环菌菌株的分类地位为:SNA01为芥黄蜜环菌Armillaria sinapina Berub&Dessur,SNA04为高卢蜜环菌Armillaria gallica Marxm&Romagn,京-234为蜜环菌Armillaria mellea(Vahl:Fr.)P.Kumm;陆春云[16]利用通用引物组合ITS4/ITS5和通用引物组合983F/2218R对7株蜜环菌进行鉴定,将菌株1~5号归为了一族,菌株6~7号分为另一族,且菌株6号形成了一个分离良好(70%)的姐妹系Armillaria mellea(AFTOL-ID449),7号菌株与其他近缘种分离良好(72%),表明菌株6号和菌株7号可能与其他蜜环菌为不同的种,甚至可能为不同的族。以上这些研究方法和研究结果对蜜环菌属的分子鉴定和分类提供了重要的参考资料。蜜环菌属种质资源丰富,外观鉴定较困难,为保证蜜环菌的优质稳定,种质溯源是关键,但在本次研究中,采用通用引物ITS4/ITS5扩增核糖体基因,仅将11份蜜环菌种质分为了两个大类4个亚类,不能进一步鉴定到种,因此,需继续找更加合适、稳定的持家基因或结合其他引物扩增的序列,利用NCBI数据库BLAST对比进一步对11份蜜环菌种质进行鉴定。

