1 株高产几丁质脱乙酰酶红球菌的基因组测序及其应用潜力分析
肖 宇,刘 洋,刘建军,卢海强,桑亚新,孙纪录,*
(1.河北农业大学食品科技学院,河北 保定 071000;2.山东省食品发酵工业研究设计院,山东 济南 250013)
壳聚糖是碱性多糖,可作为食品添加剂,在肉制品[1]、果蔬制品[2]和海产品[3]中起保鲜作用,此外,还具有澄清果汁[4]、延缓淀粉老化和增加面包持水性[5]等功能,从而改善食品风味和质感。虾蟹壳富含几丁质,是制备壳聚糖的优良原料。全球每年产生(6~8)×106t废弃虾蟹壳,这些废弃的虾蟹壳通常是被倾倒在垃圾填埋场或海洋中,不仅造成了严重的环境污染[6],也造成了巨大的浪费。我国是海洋大国,如何高效利用海洋资源,已成为我国重要的研究热点。
目前,工业生产中主要利用浓碱(40% NaOH)加热法从几丁质制备壳聚糖[7],此传统化学方法不仅会造成严重的环境污染,而且过程不易控制,生成的壳聚糖为脱乙酰度不同的混合物,质量不稳定,反应过程耗时长、耗能高[8],增加了生产成本。利用几丁质脱乙酰酶(chitin deacetylase,CDA,E.C.3.2.1.41)脱去几丁质的乙酰基是一种绿色、高效的方法。该方法具有高度特异性,可定向得到所需的降解产物,产物的聚合度、脱乙酰度、脱乙酰化模式单一[9]。该酶是目前已知的唯一一类可以使几丁质转化成壳聚糖的酶[10]。到目前为止,研究报道的CDA主要来源于真菌,关于细菌产CDA的报道较少[11]。并且,在众多微生物中产生的CDA基本为胞内酶。岳红霞等[12]筛选出1 株产CDA活力较高且性能稳定的菌株11-3,并鉴定为红球菌(Rhodococcussp.)。
目前报道的CDA产生菌主要为真菌,包括卷柄根霉(Rhizopus circinans)[13]、酿酒酵母(Saccharomyces cerevisiae)[14]、菜豆炭疽菌(Colletotrichum lindemuthianum)[15]等。与真菌相比,细菌所需培养时间更短,产酶速度更快,因此在生物法中产CDA细菌是比真菌更好的选择。但是,目前产CDA细菌报道较少,只有Rhodococcus qingshengii[16]、枯草芽孢杆菌(Bacillus subtilis)[17]等。红球菌11-3具有优良的几丁质脱乙酰能力,目前已开展了一些研究,如刘丽[18]通过优化红球菌11-3菌株的产酶培养基及培养条件,将其CDA活力由最初的58 U/mL提高到5 890 U/mL,提高了近100 倍。该菌株与已报道较部分菌株如卷柄根霉[13]和短杆菌(Brevibacterium)[19]等相比,其所产的CDA具有较好的热稳定性,45 ℃保温1 h仍能保持90%以上的活力,最适作用温度为50 ℃;最适作用pH 7.0,在pH 7.0~10.0之间有较高的活性。由此可见,红球菌11-3是一个具有开发潜力的CDA生产菌株。然而,迄今为止,关于该类菌产生CDA的研究报道还较少,缺乏基因组信息是进一步研究该菌的关键限制之一。为深入挖掘红球菌降解几丁质的酶类资源,亟需对红球菌的相关降解途径基因进行深入研究。
因此,本研究采用Illumina HiSeq第2代测序技术,对1 株高产CDA的红球菌11-3菌株进行全基因组测序,并对其基因组序列进行系统的生物信息学分析,以期为该菌株的功能基因组学研究提供基础。研究结果将为进一步挖掘红球菌降解几丁质的潜力及其遗传多样性提供理论依据,继而推动和扩大壳聚糖在食品工业方面的应用。
1 材料与方法
1.1 材料与试剂
红球菌菌株11-3,山东省食品发酵工业研究设计院刘建军教授惠赠。
DNA抽提试剂盒(细菌)Wizard®Genomic DNA Purification Kit、Wizard®基因组DNA纯化试剂盒 美国Promega公司;二代建库试剂盒NEXTflexTMRapid DNASeq试剂盒 美国Bioo Scientific公司;其他试剂均为分析纯或生化试剂。
1.2 仪器与设备
PHS-3DW型pH计 安徽合肥桥斯仪器设备有限公司;TG16-WS台式高速离心机 湖南湘仪实验室仪器开发有限公司;FA1004电子天平 上海越平科学仪器有限公司;XL-100型马弗炉 河南省鹤壁市亿欣仪器仪表有限公司;ZWY-2102C恒温培养振荡器 上海智城分析仪器制造有限公司;HH-4数显恒温水浴锅 金坛市良友仪器有限公司;WP25AB台式电热恒温培养箱天津市泰斯特仪器有限公司;GeneAmp®9700型聚合酶链式反应(polymerase chain reaction,PCR)仪 美国ABI公司;JY600 C电泳仪 北京市六一仪器厂;ABSON MIFLY-6小型离心机、5424R高速台式冷冻离心机德国Eppendorf公司;NanoDrop2000(纯度)分光光度计美国Thermo公司;TBS-380荧光仪、Illumina HiSeq测序仪 美国Illumina公司;Covaris M220粉碎仪 中国香港基因有限公司;高通量粉碎研磨仪 上海万柏生物科技有限公司。
1.3 方法
1.3.1 菌株总DNA的提取
从低温(4 ℃)保藏的斜面培养基上刮取适量待测菌株,接种于100 mL液体培养基中,于30 ℃培养24 h后,按照Wizard®基因组DNA纯化试剂盒说明书进行基因组DNA提取。纯化的基因组DNA采用TBS-380荧光仪进行定量。高质量的DNA(OD260nm/OD280nm=1.8~2.0,DNA总量≥1 μg,质量浓度≥20 ng/μL)被用于之后的建库测序。
1.3.2 Illumina文库构建
取至少1 μg基因组DNA,利用Covaris破碎仪进行基因组DNA片段化,将DNA样本剪切成约400 bp的片段,使用NEXTflexTMRapid DNA-Seq试剂盒进行文库制备。具体步骤如下:连接A&B接头;筛选去除接头自连片段;使用琼脂糖凝胶电泳进行片段筛选,保留一端是A接头、一端是B接头的片段;使用氢氧化钠变性,产生单链DNA片段;桥式PCR扩增。
1.3.3 全基因组测序及数据质控分析
制备的文库在Illumina HiSeq×10仪器上进行双端测序(2×150 bp)。具体步骤如下:加入改造过的DNA聚合酶和带有4 种荧光标记的dNTP,每次循环只掺入单种碱基;用激光扫描反应板表面,读取每条模板序列第1轮反应所聚合上去的核苷酸种类;将“荧光基团”和“终止基团”化学切割,恢复3′端黏性,继续聚合第2个核苷酸;统计每轮收集到的荧光信号结果,获知模板DNA片段的序列。
利用Illumina平台生成的数据进行生物信息学分析。所有分析均在上海美吉生物医药科技有限公司的I-Sanger云平台(www.i-sanger.com)上进行。具体程序如下:基因组组装,Illumina平台将测序图像信号经CASAVA碱基识别转换成文字信号,并将其以FASTQ格式储存作为原始数据。对原始数据进行质量剪切,具体步骤如下:去除reads中的adapter序列[20];剪切去除5’端非A、G、C、T的碱基;修剪测序质量较低的reads末端(测序质量值小于Q20);去除含N比例达到10%的reads;舍弃去adapter及质量修剪后长度小于25 bp的小片段。利用组装软件SOAPdenovo2对优化序列进行拼接[21],得到最优的组装结果。
1.3.4 基因预测及注释
利用Glimmer[22]对基因组中的编码序列(coding sequence,CDS)进行预测,获得功能基因的核酸序列和氨基酸序列,用于后续功能和系统进化分析。使用tRNAscan-SE进行tRNA预测,使用Barrnap进行rRNA预测。利用BLAST、Diamond、HMMER等序列比对工具,从非冗余蛋白库(Non-Redundant Protein Database,NR)、Swiss-Prot[23]、Pfam[24]、基因本体论(Gene Ontology,GO)、直系同源群集(Clusters of Orthologous Groups,COG)[25]、京都基因与基因组百科全书(Kyoto Encyclopedia of Genes and Genomes,KEGG)[26]数据库中对预测到的CDS进行蛋白功能注释。
1.3.5 碳水化合物活性酶(carbohydrate-active enzymes,CAZy)注释
红球菌11-3中的CAZy通过HMMER(version3.0)预测软件,在CAZy数据库[27]中比对得到。
1.3.6 几丁质降解相关酶基因的生物信息学分析
在NCBI数据库中对CDA、几丁质酶和壳聚糖酶的基因序列进行比对;使用ProtParam软件分别对目的蛋白基本性质(相对分子质量、等电点、不稳定系数)进行分析;使用ProtScale软件分别分析目的蛋白亲疏水性;使用TMHMM软件分别对目的蛋白的跨膜结构进行预测;使用SOPMA软件分别对目的蛋白二级结构进行分析。
1.4 数据统计
使用SOAPdenovo(Version 2.04)进行二代测序数据组装;使用CGView(Version 2)进行圈图绘制。使用Excel 2019处理GO、COG、KEGG注释,结果用条形统计图呈现;利用Prism 8处理CAZy注释基因,结果用条形统计图呈现。
2 结果与分析
2.1 红球菌菌株11-3的基因组测序和组装
红球菌11-3基因组测序结果见图1。最外圈为基因组大小的标识,红球菌11-3的完整基因组为包含6 089 866 bp的环状染色体,第2圈和第3圈为正链、负链上的CDS,不同的颜色表示CDS不同的COG的功能分类,编码基因的数量为5 904 个,其中COG注释的基因为4 866 个,占编码基因的82.42%。第4圈为所含rRNA和tRNA数量,红球菌11-3基因组中含有5 个rRNA操纵子,分别由2 个5S rRNA、1 个16S rRNA、2 个23S rRNA组成,含有55 个tRNA基因,分别转运Ala、Gly、Arg、Leu等20 种不同的氨基酸。第5圈为GC含量,向外的红色部分表示该区域GC含量高于全基因组平均GC含量,峰值越高表示与平均GC含量差值越大,向内的蓝色部分表示该区域GC含量低于全基因组平均GC含量,峰值越高表示与平均GC含量差值越大,基因组平均GC含量为70.514%。最内一圈为GC-Skew值,具体算法为(G-C)/(G+C),可以辅助判断前导链和后滞链,一般前导链GC-Skew大于0,后滞链GC-Skew小于0,也可以辅助判断复制起点(累计偏移最小值)和终点(累计偏移最大值),尤其对环状基因组最为重要。基因组圈图可以使研究者对菌株基因组的特征有更全面、更直观的认识。

图1 红球菌11-3基因组图Fig.1 Whole genome map of Rhodococcus sp.11-3
2.2 红球菌11-3的基因功能注释
2.2.1 基因的GO功能注释
红球菌11-3在GO数据库中注释到4 244 个基因,占基因总数的71.88%。菌株的GO注释结果见图2。

图2 红球菌11-3基因组GO功能注释分类Fig.2 Classification of GO functional annotations of genome of Rhodococcus sp.11-3
由图2可见,注释到与分子功能相关的基因最多,有3 510 个,表明该菌株的基因产物主要集中在分子功能方面。其次是与生物过程相关的基因,有3 185 个,而与细胞组成相关的基因有1 496 个。在分子功能上,最主要的途径是DNA结合(GO:0003677;489 个基因),ATP结合(GO:0005524;368 个基因),金属离子结合(GO:0046872;203 个基因),水解酶活性(GO:0016787;172 个基因),转录因子活性、序列特异性DNA结合(GO:0003700;158 个基因)。氧化还原过程(GO:0055114;902 个基因)和转录调控(GO:0006355;396 个基因)是生物过程中的主要途径。膜组分(GO:0016021;956 个基因)、细胞质(GO:0005737;252 个基因)和质膜(GO:0005886;159 个基因)是细胞组分中的主要通路。此外,分析确定了67 个与碳水化合物代谢有关的GO注释,可能与几丁质代谢有关,包括GO:0004553(水解O-糖基化合物的水解酶活性),GO:0005975(碳水化合物代谢过程)和GO:0016787(水解酶活性)。
2.2.2 基因的COG功能注释
通过COG数据库对该菌基因组进行BLAST比对分析(E-value<10-5),成功获得 COG功能注释的有4 866 个蛋白基因(图3)。

图3 红球菌11-3基因组COG数据库比对分析结果Fig.3 COG functional annotations of genome of Rhodococcus sp.11-3
由图3可见,具有未知功能的注释结果最为丰富,共1 713 个,占注释基因总数的35.20%。其次为具有转录功能和与能量产生与转化密切相关的注释结果,分别为451 个和365 个,分别占注释基因总数的9.27%和7.50%。与脂质转运与代谢、氨基酸转运与代谢、无机离子转运与代谢等功能相关的基因也得到较多的注释结果,分别为361、330 个和245 个。
为了在基因水平上阐明红球菌11-3在几丁质降解中的功能,分析了参与碳水化合物代谢的特定COG。总共有241 个基因被注释到碳水化合物的代谢中,包括125 个COG,其中最丰富的COG是ENOG410XP7I(转运蛋白)、COG0477(主要促进者超家族)、COG2301(柠檬酸裂解酶)、COG1940(ROK家族)、COG3839(ABC转运蛋白)。此外,该菌株还注释到COG366、COG2814。COG366编码一种作用于淀粉和糖原的α-淀粉酶,将多糖水解为葡萄糖和麦芽糖[28]。COG2814参与某些化合物(如碳水化合物和氨基酸)的细胞运输。辅助活性转运蛋白COG0477有助于催化各种底物的转运[29-30]。此外,注释了碳水化合物代谢中的其他重要COG,例如,COG0395参与了碳水化合物的吸收[31],而COG1109则催化了6-磷酸氨基葡萄糖的转化[32]。功能注释的高度多样性表明,红球菌11-3在几丁质降解方面可能具有强大的能力。
2.2.3 基因的KEGG功能注释
对该菌株的2 249 个基因进行了KEGG注释,结果见图4,占总基因的38.09%。
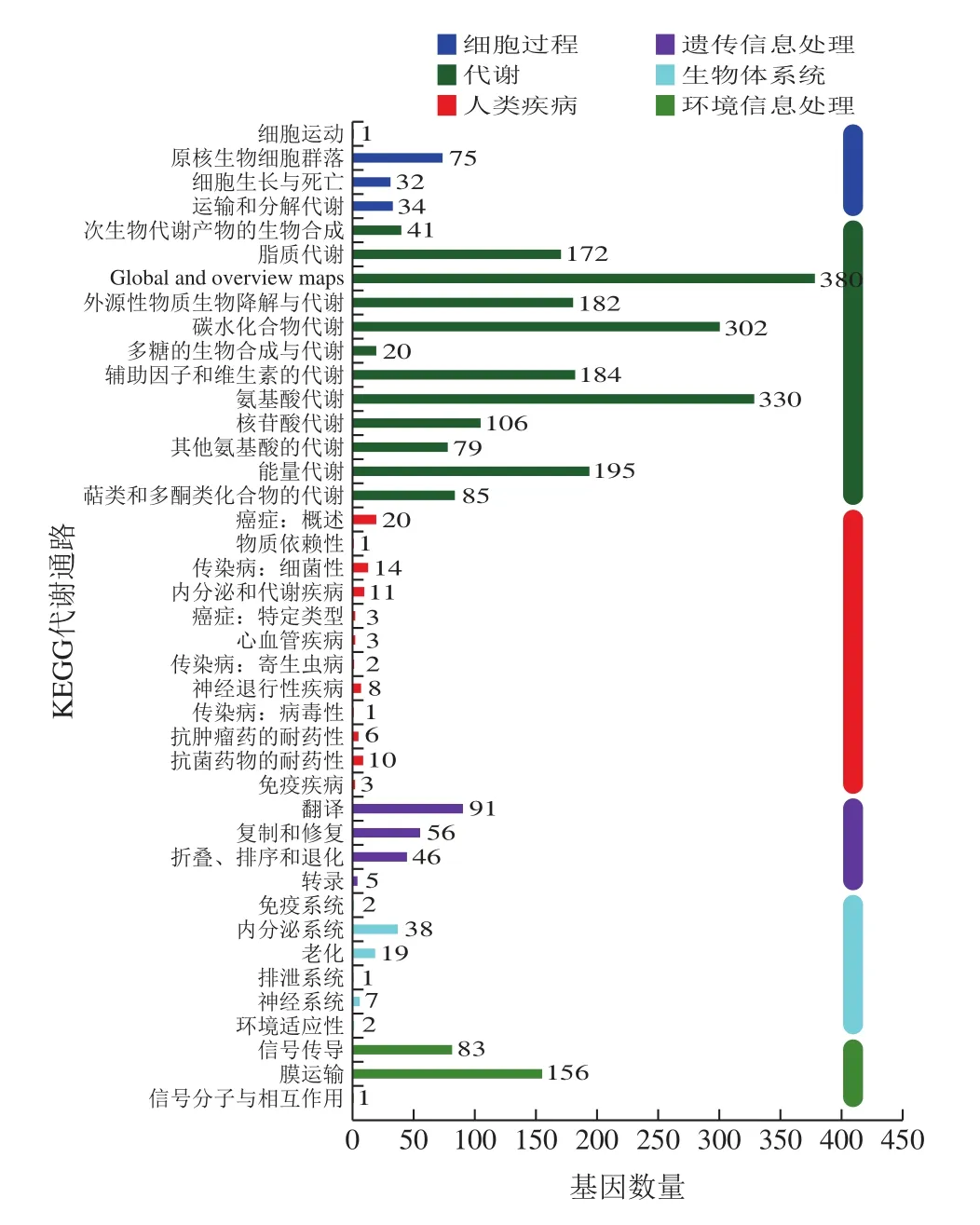
图4 红球菌11-3基因组KEGG功能分类Fig.4 KEGG function classification of genome of Rhodococcus sp.11-3
由图4可见,红球菌11-3的2 249 个KEGG注释基因分为六大类型:细胞过程(6.31%)、环境信息处理(10.67%)、遗传信息处理(8.80%)、人类疾病(3.65%)、代谢(92.31%)和生物体系统(3.07%)。其中每一类型又包含有各自的亚型。在菌株的KEGG代谢注释中,碳水化合物类代谢和氨基酸类代谢被认为是其主要功能,分别包含555 个和511 个基因。对于这些代谢,某些途径占主导地位,例如碳代谢(ko01200)、ABC转运蛋白(ko02010)以及氨基酸的生物合成(ko01230)。在代谢通路中,发现有1 个与几丁质代谢能力相关的基因,即gene5619,且只有1 个KO被注释到,K03791并未包含在代谢通路中。
2.3 红球菌11-3的CAZy基因注释分析
CAZy数据库[27]是关于合成或分解复杂碳水化合物和糖复合物的酶类的专业数据库。根据蛋白质结构域中氨基酸序列的相似性,可将不同物种来源的CAZy分成糖苷水解酶(glycoside hydrolases,GHs)[33]、糖基转移酶(glycosyl transferases,GTs)[34]、多糖裂合酶(polysaccharide lyases,PLs)[35]、碳水化合物酯酶(carbohydrate esterases,CEs)[35]、碳水化合物结合模块(carbohydrate-binding modules,CBMs)、辅助氧化还原酶(auxiliary activities,AAs)[36]六大类蛋白质家族。红球菌11-3共注释到165 个CAZy基因,如图5所示。
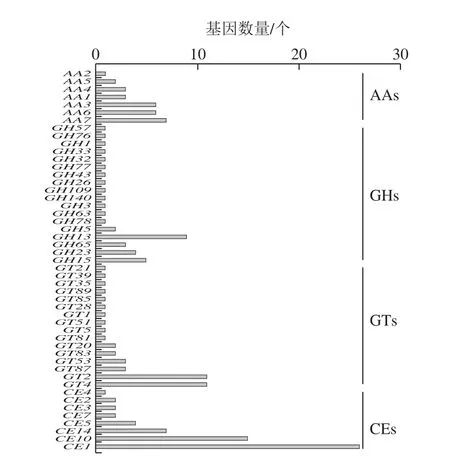
图5 红球菌11-3不同CAZy基因分布情况Fig.5 Distribution of different CAZy genes in Rhodococcus sp.11-3
由图5可见,红球菌11-3注释到的CAZy基因包括59 个CEs基因、42 个GTs基因、36 个GHs基因和28 个AAs基因。在基因组中鉴定出参与几丁质降解的1 个CDA基因(gene4907),其属于CE4家族;4 个几丁质酶(EC 3.2.1.14)基因(gene1286、gene1287、gene3810、gene4754),均属于GH23家族;2 个壳聚糖酶(EC 3.2.1.132)基因(gene4921、gene5362)。因此,该菌株具有高效降解几丁质和壳聚糖潜力。
2.4 CDA基因的生物信息学分析
红球菌11-3的CDA基因gene4907长度为894 bp,编码氨基酸数量为297 个。其与已报道的CDA氨基酸序列比对结果如图6所示。由图6可见,红球菌11-3的CDA与已报道的CDA的氨基酸序列一致性为26.60%~32.43%,其中,与来源于海洋的节细菌(Arthrobacter)的ArCE4A(GenBank LT630322)[37]的序列一致性最高,为32.43%,两者有相似的分子质量、理论等电点及二级结构。ArCE4A以几丁质为底物时,其脱乙酰度为0.003%~0.006%,以乙酰木聚糖为底物时,其脱乙酰度可高达18.9%[37]。与卷柄根霉的RcCDA(GenBank EU086737)[13]的序列一致性最低,为26.60%。因此,红球菌11-3的CDA为一种新型的CDA,这很可能是该菌株高效脱乙酰的关键。

图6 不同CDA的氨基酸序列对比分析Fig.6 Alignment of amino acid sequences of different CDAs
通过TMHMM在线工具预测红球菌11-3的CDA跨膜结构,发现该蛋白无跨膜结构域。通过SignalP 4.0在线工具对其信号肽序列进行分析,发现其含有29 个氨基酸长度的信号肽,成熟蛋白含有267 个氨基酸残基。通过ProtParam工具对其基本性质进行分析,发现其计算分子质量为30.57 kDa,等电点为5.22,含量最高的氨基酸为丙氨酸(15.5%)。通过ProtScale工具对其亲疏水性进行分析,发现在整条链中,最高分值为1.626,为排在24位的亮氨酸,代表疏水性最强;最低分值为-1.685,为排在167位的酪氨酸,代表亲水性最强。总平均亲水性(grand average of hydropathy,GRAVY)值被定义为序列中所有氨基酸亲水值的总和与氨基酸数量的比值,负值越大表示亲水性越好,正值越大表示疏水性越强。红球菌11-3的CDA的GRAVY值为0.005,表明该蛋白质是一种不溶性蛋白。利用SOPMA法对其二级结构进行分析,发现其二级结构由50.17%的无规卷曲、26.94%α-螺旋、16.84%延伸链和6.06%β-转角组成。
2.5 其他几丁质降解相关酶基因的生物信息学分析
与上述对红球菌11-3CDA基因gene4907的分析方法相同,对该菌株的几丁质酶基因(gene1286、gene1287、gene3810、gene4754)和壳聚糖酶基因(gene4921、gene5362)进行生物信息学分析,结果如表2所示。
分别将表2中各基因的核苷酸序列翻译成氨基酸序列,用NCBI中的BLASTp功能,与数据库中的蛋白序列进行比对。在数据库中,并没有相似的几丁质酶蛋白和壳聚糖酶蛋白。其原因可能是该基因来源的菌株比较新颖,其产生的酶有较大可能是新酶。
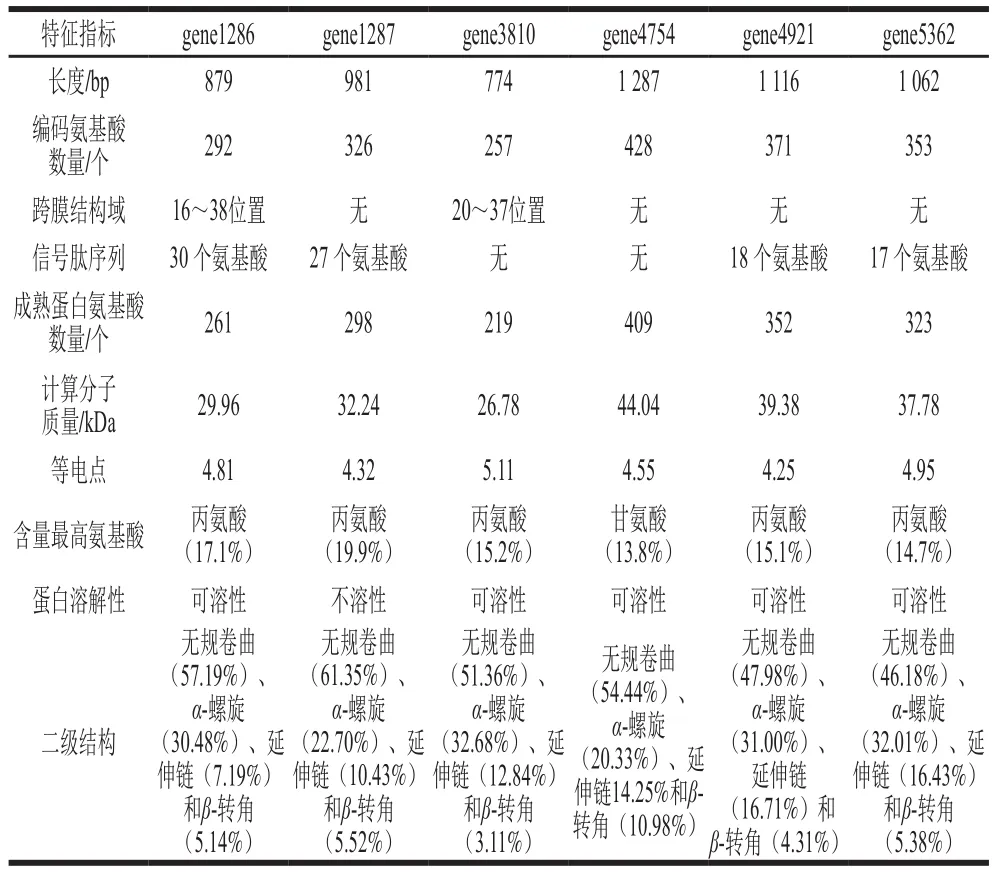
表2 红球菌11-3的几丁质酶和壳聚糖酶基因及其表达蛋白的特征预测Table 2 Predicted characteristics of chitinase and chitosanase genes and proteins of Rhodococcus 11-3
3 结 论
红球菌菌株11-3基因组大小为6 089 866 bp,是一种GC含量高达70.514%的微生物类群,预测到5 904 个编码基因,其中编码基因总长度为5 502 237 bp,平均长度为931.95 bp,平均密度为0.97 个/kb,基因中包含5 个rRNA操纵子和55 个tRNA。
从功能预测的角度看,红球菌11-3基因组中能够注释到GO信息的基因数目为4 244 个,包含了40多种功能特性,占所有编码基因的71.88%。能够注释到COG信息的基因数目为4 866 个,注释基因占比为82.42%。在KEGG数据库中共有2 249 个基因分别在代谢、遗传信息处理、环境信息处理、细胞过程、生物体系统、人类疾病6大功能41 个通路上得到功能注释,还发现1 个与几丁质代谢通路相关的基因。预测到可能的毒力基因360 个,耐药基因266 个。此外,该菌株在CAZy数据库中注释到1 个CDA基因、4 个几丁质酶基因和2个壳聚糖酶基因。3 种酶基因编码的氨基酸序列与数据库中的CDA氨基酸序列一致性普遍较低,其原因可能是这些基因来源的菌株比较新颖,其产生的酶有较大可能是一些新酶。
值得注意的是,该菌株还有相当多的未知功能基因,具有极大的研究价值。因此,本研究得到了红球菌11-3的大量基因组学信息,为该菌株的功能基因挖掘及改造提供了坚实依据。

