2种重叠延伸PCR技术构建5种立克次体融合基因的方法学比较 *
郑雨桐,闫美田,万楠
(1.大连医科大学研究生院,辽宁大连 116044;2.中国人民解放军北部战区总医院检验科,沈阳 110016)
重叠延伸PCR(overlaping extension PCR, SOE-PCR)技术最早于1989年由Horton等[1]创建,经过30多年的探索研究,现已是分子生物学及基因工程研究领域中较为常见的PCR技术。该方法是通过设计内部具有重叠部分的特异性引物,经PCR,将2个及2个以上的基因片段拼接起来的过程[2]。与传统的基因融合重组构建方法不同,SOE-PCR技术无需限制性内切酶和DNA连接酶的处理[3],是一种方便、快捷及有效的分子生物学方法。
随着研究的不断进展,基于SOE-PCR技术可以将多个DNA片段同时无缝融合在一起,目前已获得的最大融合基因片段长度超过2×104bp[4]。利用此技术可以在目的序列中引入突变、删除和替换位点等[5]。另外,此技术还可以将若干个短的相互重叠的引物拼接起来进行全基因合成[6]。但是该技术在实验过程中仍有许多不足之处,例如在实验开始时必须使用特异性引物将各基因片段进行单独扩增后才能进行基因融合,即需要进行2次PCR反应(两步SOE-PCR法),不仅耗时,而且增加了实验成本。基于繁琐的两步法,本实验设计出一步SOE-PCR法,将整个实验PCR反应减少至1次;并采用一步法及两步法SOE-PCR分别对普氏立克次体、嗜吞噬细胞无形体、莫氏立克次体、羌虫病东方体、贝氏柯克斯体5个基因片段进行拼接融合,并对2种方法进行比较性分析,为SOE-PCR技术及更多的基因融合提供实验依据。
1 材料与方法
1.1研究对象(靶基因选取与质粒) 从NCBI基因数据库(https://www.ncbi.nlm.nih.gov/)中下载普氏立克次体(AF123718)、嗜吞噬细胞无形体(AF153716)、莫氏立克次体(KF241858)、羌虫病东方体(RIRGM47G)、贝氏柯克斯体(AF146298)全基因序列,通过BLAST分析找到保守且特异的序列并由通用生物系统公司合成基因片段。
1.2仪器与试剂 pGMT-easy克隆载体、pGMT-easy连接试剂盒、2×EasyTaq PCR SuperMix、2×TransStart FastPfu PCR SuperMix、Trans2K Plus DNA Marker、6×DNA Loading Buffer、琼脂糖凝胶粉剂均购自北京全式金生物技术公司,50×TAE购自北京赛文创新生物科技公司。AG 5333型PCR扩增仪购自德国Eppendorf公司,琼脂糖凝胶电泳仪购自美国Bio-Rad公司,UVP凝胶成像仪购自美国UVP公司。
1.3方法
1.3.1引物设计及验证 5对特异性引物合成由上海生工公司完成,片段大小及引物设计序列见表1。将5个靶基因质粒及5对特异性引物分别构建50 μL PCR扩增体系进行PCR扩增,体系中各成分含量包括:靶基因质粒模板1 μL,10 μmol/L上、下游特异性引物各1 μL,2×EasyTaq PCR SuperMix 25 μL,ddH2O补足至50 μL。循环参数:96 ℃ 5 min;96 ℃ 22 s,56 ℃ 22 s,72 ℃ 25 s,共30个循环;72 ℃ 5 min,4 ℃保存。扩增结束后进行20 g/L琼脂糖凝胶电泳,并通过UVP凝胶成像仪观察是否扩增出相应的5个目的基因。

表1 5种立克次体靶基因的选取及重叠引物的设计
1.3.2一步法SOE-PCR 以5个靶基因质粒为模板,构建50 μL PCR扩增体系,体系中各成分含量如下:5个模板各1 μL,设计出的5对重叠引物各1 μL,2×TransStart FastPfu PCR SuperMix 25 μL,ddH2O补足至50 μL。循环参数:96 ℃ 5 min;96 ℃ 22 s,56 ℃ 22 s,72 ℃ 25 s,共5个循环;72 ℃ 5 min;反应结束后将循环参数调整为:96 ℃ 22 s,60 ℃ 1 min,72 ℃ 2 min,共5个循环;72 ℃ 5 min。待反应结束后取出产物,加入另外50 μL反应体系,体系中各成分含量如下:普氏立克次体F与贝氏立克次体R(10 μmol/L)各1 μL,2×TransStart FastPfu PCR SuperMix 25 μL,ddH2O补足至50 μL。循环参数:96 ℃ 22 s, 60 ℃ 1 min,72 ℃ 25 s,共30个循环;72 ℃ 5 min,4 ℃保存。为了能获得均一性较好的融合基因,本实验对反应过程中重叠引物的浓度进行优化,梯度如下:10 μmol/L、5 μmol/L、2.5 μmol/L、1 μmol/L、0.5 μmol/L、0.25 μmol/L、0.1 μmol/L、0.05 μmol/L、0.025 μmol/L、0.01 μmol/L。分别按上述浓度进行一步法SOE-PCR,获得的融合基因产物进行20 g/L琼脂糖凝胶电泳,并通过UVP凝胶成像仪观察结果。
1.3.3两步法SOE-PCR 以5个靶基因质粒为模板,构建50 μL PCR扩增体系,分别扩增出相应的目的基因产物(此过程中重叠引物浓度为10 μmol/L),获得的产物无需进行回收纯化处理。将5种目的基因产物进行等体积混合,构建50 μL PCR扩增体系,除等体积混合的产物外,体系中其他各成分含量如下:2×TransStart FastPfu PCR SuperMix 25 μL,ddH2O补足至50 μL(此步骤无需加入任何引物)。循环参数:96 ℃ 22 s,60 ℃ 1 min,72 ℃ 2 min,共5个循环;72 ℃ 5 min;待反应结束后取出产物,加入另外50 μL反应体系,体系中各成分含量如下:普氏立克次体F与贝氏立克次体R(10 μmol/L)各1 μL,2×TransStart FastPfu PCR SuperMix 25 μL,ddH2O补足至50 μL。循环参数:96 ℃ 22 s,60 ℃ 1 min,72 ℃ 25 s,共30个循环;72 ℃ 5 min,4 ℃保存。为了能获得均一性较好的融合基因,本实验对各靶基因产物加入量进行优化,梯度如下:0.1 μL、0.15 μL、0.2 μL、0.25 μL、0.5 μL、1 μL、1.5 μL、2 μL、2.5 μL、3 μL。分别按上述体积进行两步法SOE-PCR,获得的融合基因产物行20 g/L琼脂糖凝胶电泳,并通过UVP凝胶成像仪观察结果。
1.3.4测序验证 将2种不同方法获得的最佳融合基因产物送往上海生工公司进行测序。测序结果用Chromas及Seqman软件进行比对分析。
2 结果
2.1引物验证 将5个靶基因质粒分别进行PCR扩增,结果中出现5个相应片段长度的条带,表明本研究所设计的引物可以扩增出相应的目的基因。见图1。
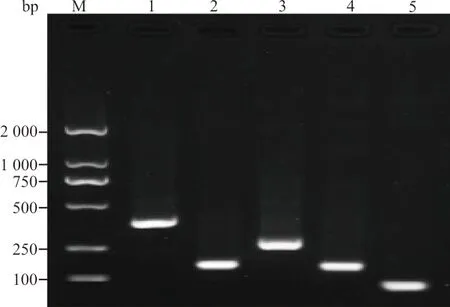
注:1~5分别为普氏立克次体(364 bp)、嗜吞噬细胞无形体(132 bp)、莫氏立克次体(213 bp)、羌虫病东方体(127 bp)、贝氏柯克斯体(76 bp);M,DNA Marker。
2.2一步法融合基因检测结果 利用方法1.3.2进行一步法SOE-PCR,电泳结果显示均可扩增出长度约为912 bp的目的基因条带,初步证明5条目的基因已融合成1条;另外,当重叠引物的浓度为0.1~0.01 μmol/L时,912 bp的融合基因条带亮度较高,且杂带较少。见图2。
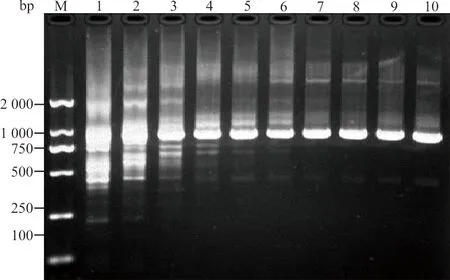
注:M, DNA Marker;1~10,重叠引物浓度梯度分别为10 μmol/L、5 μmol/L、2.5 μmol/L、1 μmol/L、0.5 μmol/L、0.25 μmol/L、0.1 μmol/L、0.05 μmol/L、0.025 μmol/L、0.01 μmol/L。
2.3两步法融合基因检测结果 利用方法1.3.3进行两步法SOE-PCR,电泳结果显示均可扩增出长度约为912 bp的目的基因条带,初步证明5条目的基因已融合成1条;另外,当各靶基因产物加入量为0.1~0.25 μL时,912 bp的融合基因条带亮度较高,且杂带较少。见图3。
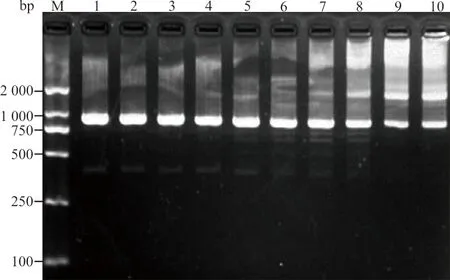
注:M, DNA Marker;1~10,各靶基因产物加入量梯度分别为0.1 μL、0.15 μL、0.2 μL、0.25 μL、0.5 μL、1 μL、1.5 μL、2 μL、2.5 μL、3 μL。
2.4测序验证结果 2种不同方法获得的最佳融合基因产物测序结果与融合基因序列进行比对,结果显示一步法和两步法PCR产物中均含有与融合基因全长序列完全相同的部分,因此,该2种方法构建融合基因均获得成功。见图4。
注:A为一步法融合基因产物部分测序结果比对结果;1,部分融合基因序列;2,一步法测序部分序列结果;3,一步法融合基因产物部分测序峰图;B为两步法融合基因产物部分测序结果比对结果;4,两步法测序部分序列结果;5,两步法融合基因产物部分测序峰图。
3 讨论
1989年SOE-PCR技术问世,实现了将不同或相同来源的不同基因片段“拼接”,与传统的基因重组构建方法相比,这种功能强大且技术简单的方法具备许多优势[7-9]。在实际科研工作中,我们发现多数研究人员会选择两步SOE-PCR方法,例如伊强等[10]通过两步SOE-PCR方法构建了EB病毒BMRF1-BZLF1N融合基因;龚咏晴等[11]通过两步法构建与子宫内膜癌相关的PTEN基因3位点的融合重组载体;董巍檑等[12]通过两步法将exon18和exon20片段进行融合,且成功构建了宫颈癌相关的EGFR基因重组表达载体。
本实验通过2种不同步骤的SOE-PCR方法,均成功将5种立克次体目的基因“拼接”在一起。应用一步法进行基因融合时,重叠引物的浓度需在0.1 μmol/L以下,可融合出单一程度较高的912 bp全长基因,即重叠引物的浓度与融合基因上、下游引物的浓度比例需在1∶100以下。与陈国梁等[13]运用一步法SOE-PCR进行的3种淀粉合成关键酶的融合结果相似。另外,运用传统两步法进行基因融合时,以第一步获得的5个目的基因产物为模板加入量需控制在0.1~0.25 μL之间,可融合出单一程度较高的912 bp全长基因。将2种方法进行对比发现,一步法融合基因操作简单,耗时短(所用时间约为两步法的二分之一),同时DNA聚合酶用量较少;但在反应体系中一次性加入所有模板及引物,退火过程中,模板的互补配对部位会进行拼接,同时引物也在退火,模板的拼接和扩增将会同时进行,这样就相对降低了扩增初始阶段的模板浓度,导致最终融合基因产物浓度较低。因此,一步法中重叠引物浓度、退火温度及退火时间的把握至关重要。两步法融合基因耗时较长,过程较为繁琐,但由于先分别扩增出5个目的基因产物,故而仅存在少量非特异性扩增,目的基因浓度较高。但由于在基因融合过程中并不存在扩增反应,只是单纯的互补延伸,所以在第二步反应的初始阶段需要加入较高浓度的目的基因产物,从而获得大量融合基因。因此,大量的耗材是两步法融合基因的另外一个缺点。从上述分析中发现一步法SOE-PCR可以更加经济、简单、高效地完成多个基因片段的融合。
无论进行何种基因融合方法时,为确保融合成功,应注意以下多个方面:(1)引物重叠部分的设计:徐芳等[14]认为SOE-PCR技术应注重重叠引物的设计,除应考虑引物设计的一般原则外,还应考虑重叠部分的长度,并建议重叠部分序列长度为15~20 bp。另外,还应注意重叠引物的Tm值,建议设计所有引物的Tm值应相同或相近,有助于提高融合效率。(2)DNA聚合酶的种类:由于普通Taq酶进行PCR扩增反应时,会在3′末端添加A,造成移码突变,因此选用高保真聚合酶是良好的选择,从而为后续实验的成功奠定基础。(3)退火温度的控制:一步法SOE-PCR在退火过程中,模板的拼接和扩增将会同时进行,因此适当提高退火温度及延长退火时间可以提高融合基因的效率。
综上所述,本研究成功通过SOE-PCR技术在体外将5种立克次体进行融合,但本实验仍然存在以下不足:如仅对2种方法的重叠引物的浓度及目的基因产物加入量进行优化,其他环境参数(例如最合适的引物长度、最佳连续延伸的循环数、最佳退火温度及时间等)主要是通过实验经验进行掌控,这些问题还需要进一步实验证实。另外,后续实验中我们将继续进行立克次体融合基因表达载体构建,同时为立克次体分子生物学多重检测提供实验模板。

