果梅NAC 基因家族的鉴定及组织表达分析
姚 丹 倪晓鹏 侍 婷 倪照君 渠慎春 高志红
(南京农业大学园艺学院,江苏 南京 210095)
NAC(NAM/ATAF/CUC)转录因子是近年来发现的一类植物特有的转录因子,其命名来源于矮牵牛NAM 基因、拟南芥ATAF1/2 和CUC1/2 基因首字母的缩写,NAC 结构中含有N-端高度保守的NAC 结构域和C-端高度变异的转录激活域[1]。 Souer 等[2]于1996年首次在矮牵牛(Petunia hybrida Vilm.)中克隆得到NAM 基因,随后越来越多的NAC 基因家族成员被鉴定出来, 目前已在拟南芥( Arabidopsis thaliana)[3]、水稻(Oryza sativa)[3]、毛果杨(Populus trichocarpa)[4]、 大 豆 ( Glycine max )[5]、 番 茄( Lycopersicon esculentum Mill.)[6]、 苹 果 ( Malus pumila)[7]、 葡 萄(Vitis vinifera)[8]、 甘 蓝(Brassica oleracea L.)[9]、 小 兰 屿 蝴 蝶 兰 ( Phalaenopsis equestris)[10]等多种植物中分别鉴定出117、151、163、152、104、180、74、188、86 个NAC 基因。 大量研究表明,NAC 转录因子具有多种重要的生物学功能,如广泛参与植物的分生组织发育[11]、花器官的发育[12]、侧根发育[13]、次生细胞壁的形成[14]以及植物器官衰老[15]等,此外,NAC 转录因子还能对一些生物或非生物胁迫作出应答,如病原菌[16-17]、干旱[18-19]、低温[20]、高盐[21-22]等。
果梅(Prunus mume Sieb.et Zucc)为蔷薇科李属植物,是我国重要的核果类树种之一,我国果梅栽培历史悠久,种质资源丰富[23]。 果梅果实中富含多种维生素、有机酸及碱性矿物质等,具有较高的保健作用及经济价值[24]。 随着分子生物学及生物信息学的迅猛发展,越来越多的研究者在基因组水平上对植物中编码重要功能的基因进行了预测和功能分析[3,6,8]。 目前关于NAC 基因的研究报道很多,但在果梅上尚鲜见报道。 本研究利用多种生物信息学方法对果梅NAC 基因家族进行鉴定及分析,并采用实时荧光定量PCR技术分析果梅NAC 基因的组织表达特异性,旨在为进一步研究果梅NAC 基因的生物学功能奠定基础。
1 材料与方法
1.1 材料
以南京市浦口区南京农业大学园艺实验站中国家果梅种质资源圃的果梅品种龙眼为试验材料,采集其花芽、幼叶、嫩茎、果皮、果肉和果胚,液氮速冻后,-70℃保存备用。
1.2 方法
1.2.1 果梅NAC 基因家族的鉴定 从拟南芥转录因子数据库RARTF(http:/ /rarge.gsc.riken.jp/rartf/)下载拟南芥NAC 蛋白序列,利用Hmmer 3.0 软件建立Profile HMM 模型, 并利用此模型检索从 NCBI(https:/ /www.ncbi.nlm.nih.gov/)下载的果梅蛋白数据库,去除冗余序列,即得到候选蛋白序列[25]。 利用SMATR(http:/ /smart. embl-heidelberg.de/)在线平台预测这些候选蛋白是否存在NAC 结构域,存在NAC结构域的属于果梅NAC 蛋白家族。
1.2.2 染色体定位及亚细胞定位分析 根据果梅NAC 蛋白的基因注释,得到每个PmNAC 基因的染色体位置及果梅各染色体长度,然后利用MapInspect 软件绘制PmNAC 基因在染色体上的定位分布。 利用在线工具Protcomp 9.0 和CELLO v2.5 进行亚细胞定位分析。
1.2.3 系统发育分析 利用ClustalX 2.0 软件将果梅NAC 家族蛋白序列与拟南芥NAC 家族蛋白序列进行多序列比对分析,利用MEGA 5 软件采用邻接(neighbor-joining,NJ)法构建NAC 蛋白家族的系统发育进化树,对构建的进化树进行自检,bootstrap 值设为1 000[26]。
1.2.4 保守基序的鉴定及结构预测 利用MEME 在线程序对果梅NAC 蛋白进行保守基序(motif)分析,motif 最大检索值设为10;利用ExPaSy 提供的在线平台Protparam 对果梅NAC 蛋白进行一级理化性质分析;利用SOPMA 程序对果梅NAC 蛋白进行二级结构预测;利用Swiss-Model 程序对果梅NAC 蛋白进行三维结构同源建模[27]。
1.2.5 实时荧光定量PCR 分析 采用改良的CTAB法[28]提取果梅不同组织的总RNA,并参照PrimeScriptⅡ1stStrand cDNA Synthesis Kit(TaKaRa,日本)操作说明书合成cDNA 第一链。 利用Oligo7 软件设计荧光定量引物,引物序列详见表1。 以6 个不同组织(花芽、幼叶、嫩茎、果皮、果肉和果胚)的cDNA 为模板,果梅actin-RP-Ⅱ基因为内参,采用7300 Real Time PCR System(ABI,美国)对12 个PmNAC 基因进行实时荧光定量PCR 分析。
实时荧光定量PCR 反应体系:1 μL 稀释10 倍的cDNA 模板,上下游引物各0.2 μL、SYBR Premix Ex TaqTM 荧光染料10 μL,ddH2O 8.6 μL。 反应程序:95℃预变性4 min;95℃变性20 s,60℃退火20 s,72℃延伸40 s,40 次循环。 循环结束后95℃1 min,60℃30 s,40℃2 min 进行溶解曲线分析。 每样品设3 次生物学重复,采用2-ΔΔCT法[29]分析基因表达量。

表1 PmNAC 基因实时荧光定量PCR 引物序列Table 1 Real time fluorescence quantitative primer sequences of PmNAC genes
2 结果与分析
2.1 果梅NAC 基因家族的鉴定与染色体分布分析
根据拟南芥NAC 基因的蛋白序列,通过对果梅蛋白数据库的比对搜索,初步从果梅基因组中获得129条蛋白序列。 去冗余后再用SMART 程序检测NAC 结构域,最终鉴定得到106 个果梅NAC 基因。 由表2 可知,NAC 蛋白在氨基酸数目、分子量、理论等电点等方面均存在差异,其中PmNAC12 和PmNAC30 这2 个基因由于序列中包含连续几个不明确的氨基酸,导致其分子量和理论等电点无法确定。 PmNAC4 的氨基酸数目最少有 129 个, 其分子量为 14 763.98 Da;PmNAC101 的氨基酸数目最多有832 个,其分子量为88 815.51 Da。 蛋白质理论等电点介于4.12 ~9.70 之间,但总体而言,果梅NAC 家族蛋白成员的理论等电点多在酸性范围内,说明蛋白质分子中富含酸性氨基酸。 蛋白质疏水性介于-0.378 ~-1.037 之间,疏水性负值越大表示越亲水,正值越大表示越疏水,疏水值介于-0.5~0.5 之间的为两性氨基酸[30]。 预测结果显示,果梅106 条NAC 蛋白序列中有101 条序列疏水值为负值且小于-0.5,表明大部分果梅NAC 蛋白是亲水蛋白,其中,仅有PmNAC4(-0.493 )、PmNAC101(-0.487 )、PmNAC20(-0.440)、PmNAC46(-0.388 )、PmNAC68(-0.378 )为两性氨基酸。
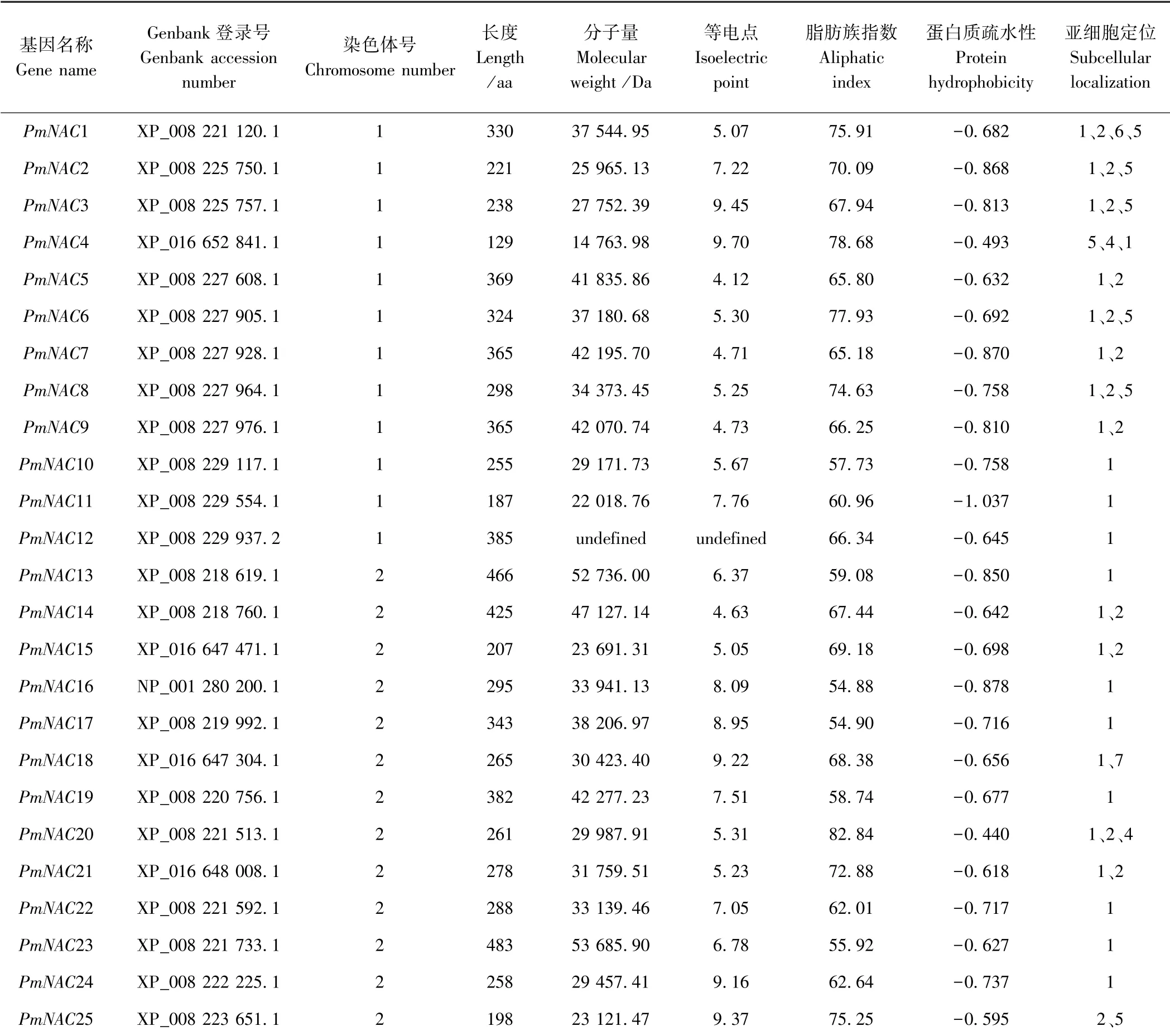
表2 果梅基因组中NAC 基因家族的鉴定Table 2 Identification of NAC gene family in the P. mume genome
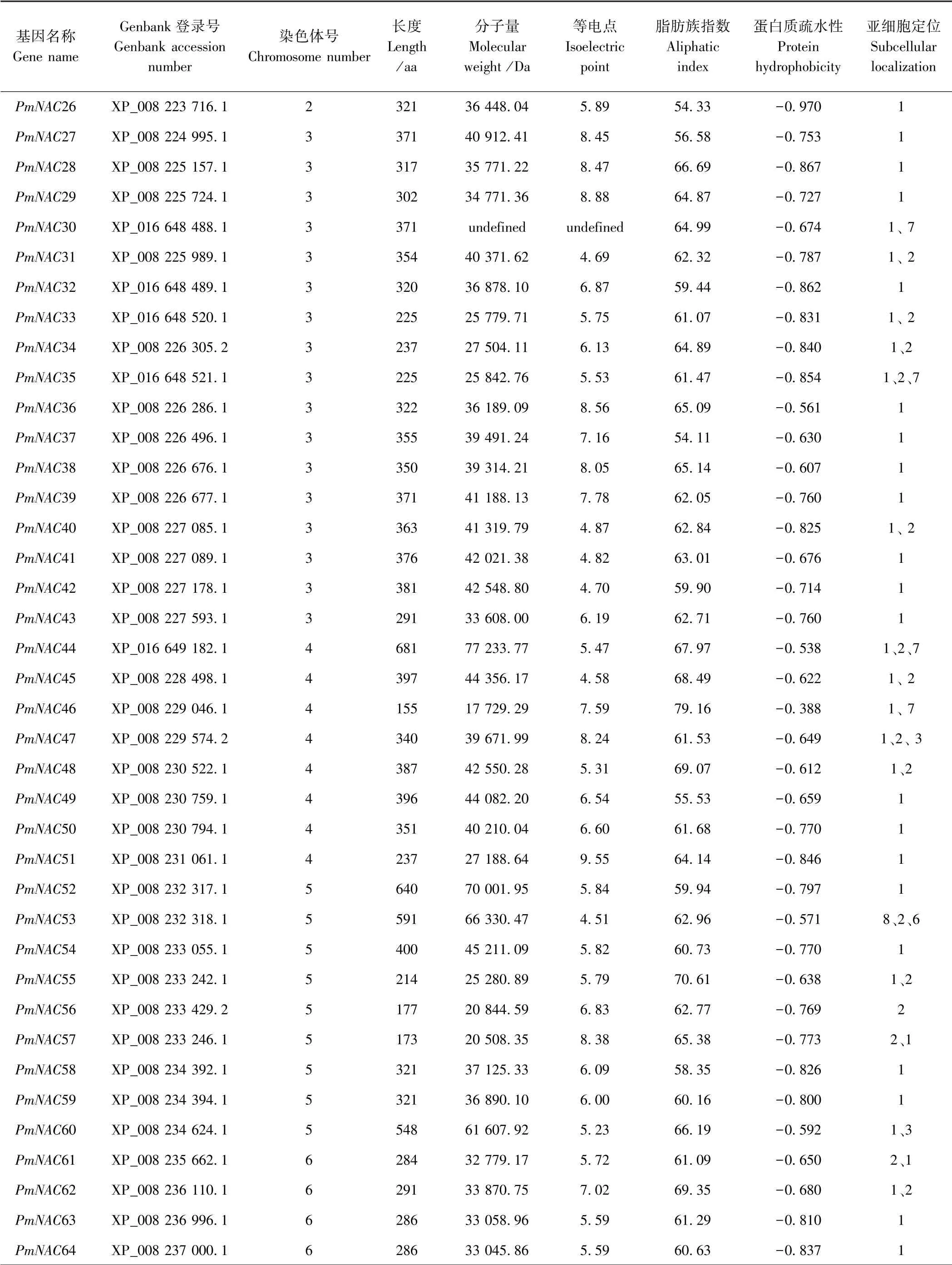
表2(续)
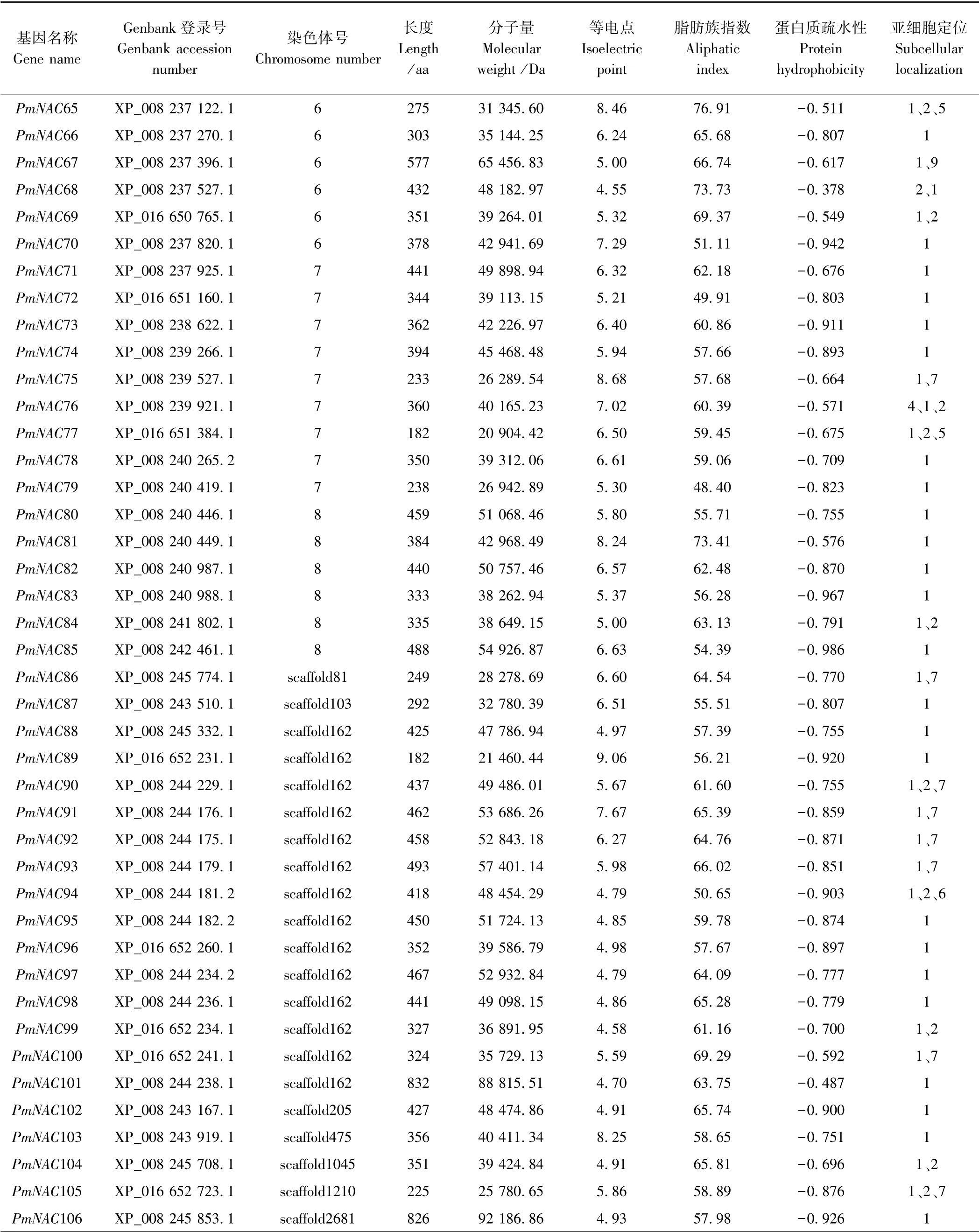
表2(续)
利用MapInspect 软件绘制果梅NAC 基因家族成员在染色体上的定位分布,并参照番茄NAC 基因的命名方法[31],按照染色体位置对所有PmNAC 基因进行系统编号。 由图1 可知,PmNAC 中有21 个基因(PmNAC86~PmNAC106)无匹配的染色体定位信息。染色体定位分析发现,NAC 基因在果梅的8 条染色体上呈现不均匀分布,其中第3 号染色体上分布最多,为17 个;其次是第2 号染色体,为14 个;第8 号染色体分布最少,为6 个。 此外,NAC 基因在果梅染色体上的分布存在明显的串联重复现象,共发现14 个串联重复的基因簇,包含33 个基因,占PmNAC 基因总数的31.13%。 亚细胞定位预测显示,大部分的果梅NAC基因都能定位于细胞核中,属于典型的核蛋白。
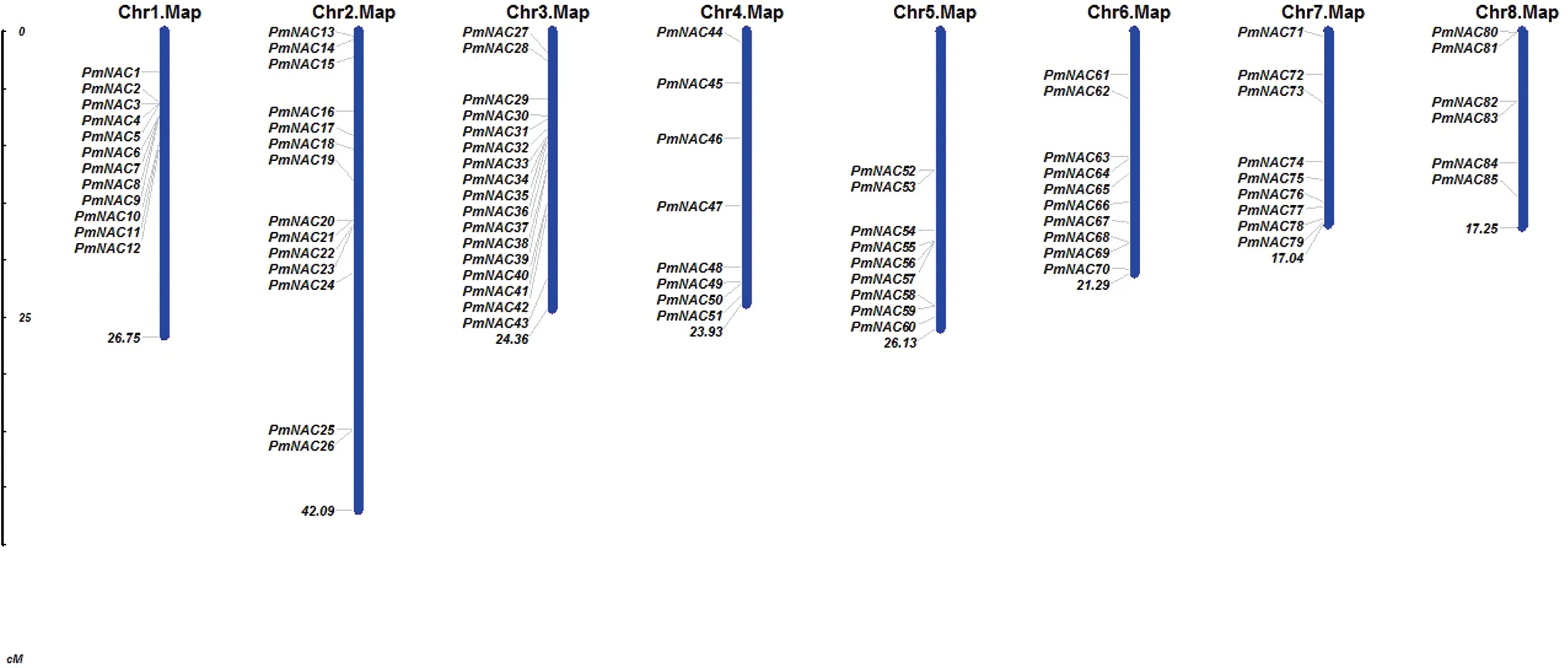
图1 PmNAC 基因在果梅基因组染色体上的分布位置Fig.1 Chromosomal locations of PmNAC genes on P. mume chromosomes
2.2 系统发育分析
由图2 可知,果梅和拟南芥NAC 基因共被分成12个亚族。 其中,第12 亚族中的果梅NAC 基因数量最多,为25 个;其次是第5 亚族,为21 个;第7 和第10亚族中成员数量最少,均只有1 个。 在第7 亚族中拟南芥NAC 基因较为密集,但果梅NAC 基因只有1 个(PmNAC62),说明PmNAC62 与其他果梅NAC 基因进化关系较远。 第9 亚族全部由PmNAC 基因组成,这可能是由于该亚族NAC 基因在果梅中独自进化,也可能是在拟南芥中发生了基因丢失的原因。 除第9 亚族外,每个亚族均同时含有果梅和拟南芥NAC 基因。
2.3 保守基序分析
利用MEME 在线程序分析发现,果梅NAC 蛋白具有10 个保守基序,主要分布于序列N 端(图3),且在大多数果梅NAC 转录因子中都有分布,属于高保守基序。 一般聚在同一亚族的蛋白会有相似的保守基序组成,第1~第9 亚族中果梅NAC 蛋白的保守基序大多相似,表明在这些亚族中NAC 基因家族成员的进化关系较近;而第10、第11、第12 亚族中果梅NAC 蛋白的保守基序相差较大,表明这些亚族中NAC 成员进化关系较远。 在InterPro 数据库中对这些基序进行注释,发现基序1~6 属于NAM 结构域,在分子功能上均具有DNA 结合活性,在生物过程中,主要参与转录调控过程,其他基序均无显著的数据库记录(表3)。
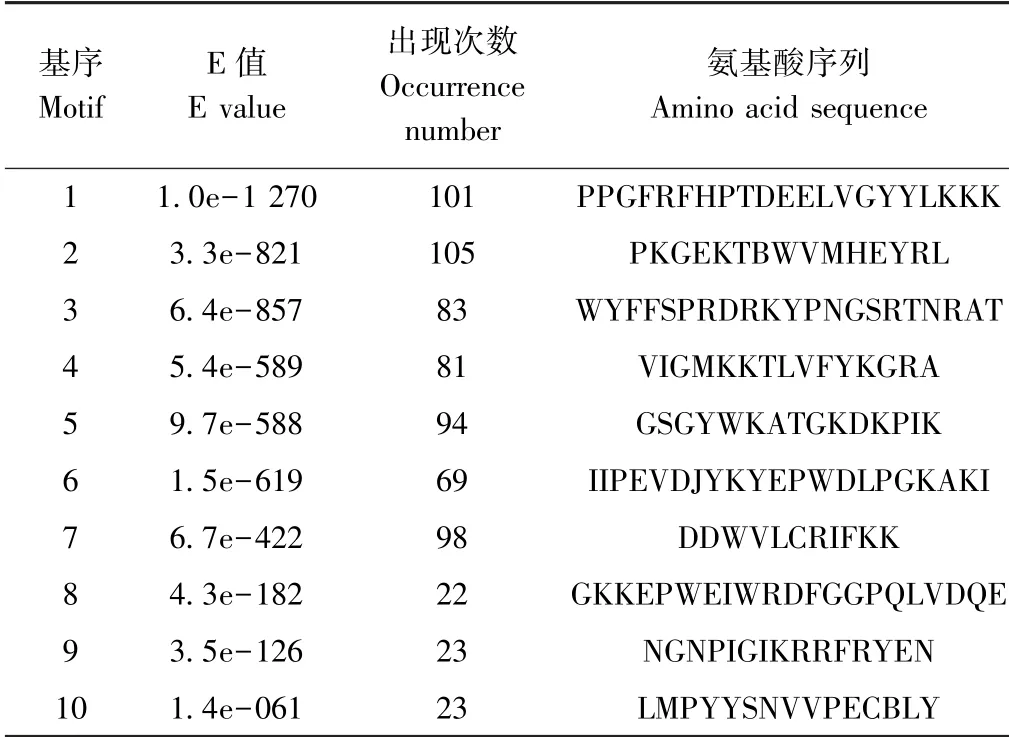
表3 果梅NAC 蛋白保守基序的氨基酸序列Table 3 Amino acid sequences of conserved motifs of NAC protein in P. mume
2.4 果梅NAC 蛋白结构分析
利用SOPMA 程序对106 个果梅NAC 蛋白进行二级结构预测,结果显示,果梅NAC 蛋白家族的二级结构大多都以无规则卷曲为主要构成元件,所占比例为27.1%~62.26%,而α-螺旋(10.09%-45.68%)、β-折叠(11.34% ~33.55%) 和 β - 转 角(3.09% ~13.87%)则分散于整个蛋白质中。
利用SWISS-MODEL 在线工具对106 个果梅NAC蛋白的氨基酸序列进行三级结构同源建模,发现它们的三维构象基本类似。 每个亚族选出一个能代表该亚族三级结构特征的NAC 蛋白,记录其组成元件(图4)。结果显示,其中6 个蛋白质(PmNAC54、PmNAC32、PmNAC62、PmNAC51、PmNAC48 和PmNAC2)的α-螺旋和β-折叠的个数都分别为5 和9;4 个蛋白质(PmNAC71、PmNAC31、PmNAC68 和PmNAC60)的α-螺旋和β-折叠的个数都分别为4 和8;2 个蛋白质(PmNAC61 和PmNAC95)的α-螺旋和β-折叠的个数都分别为6 和8。 虽然部分果梅NAC 蛋白的α-螺旋和β-折叠个数相同,但其三维构象存在差异,这可能与α-螺旋、β-折叠及无规则卷曲的长度、分布不同有关,从而导致其功能上的差异。
2.5 果梅NAC 基因的组织表达分析
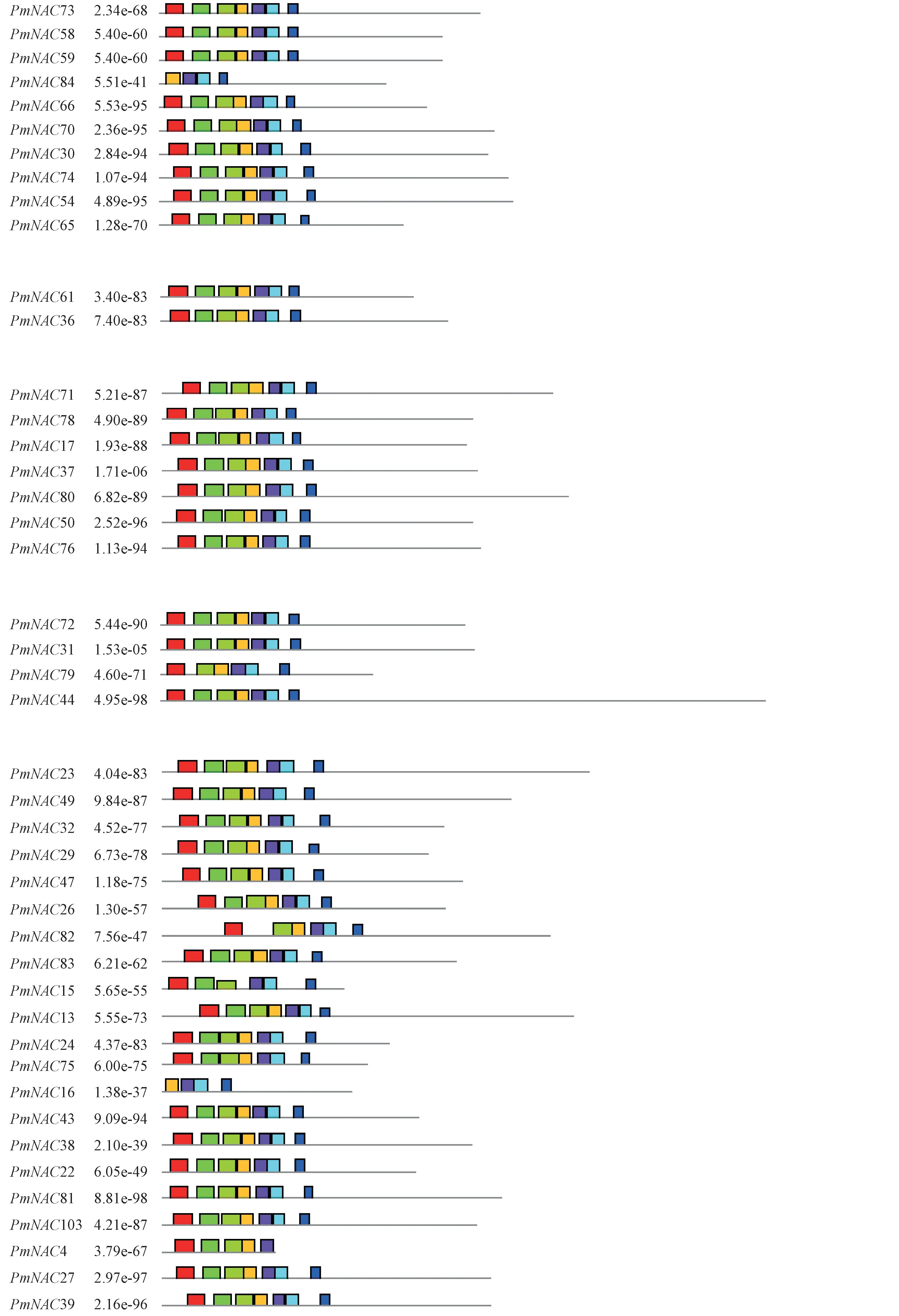

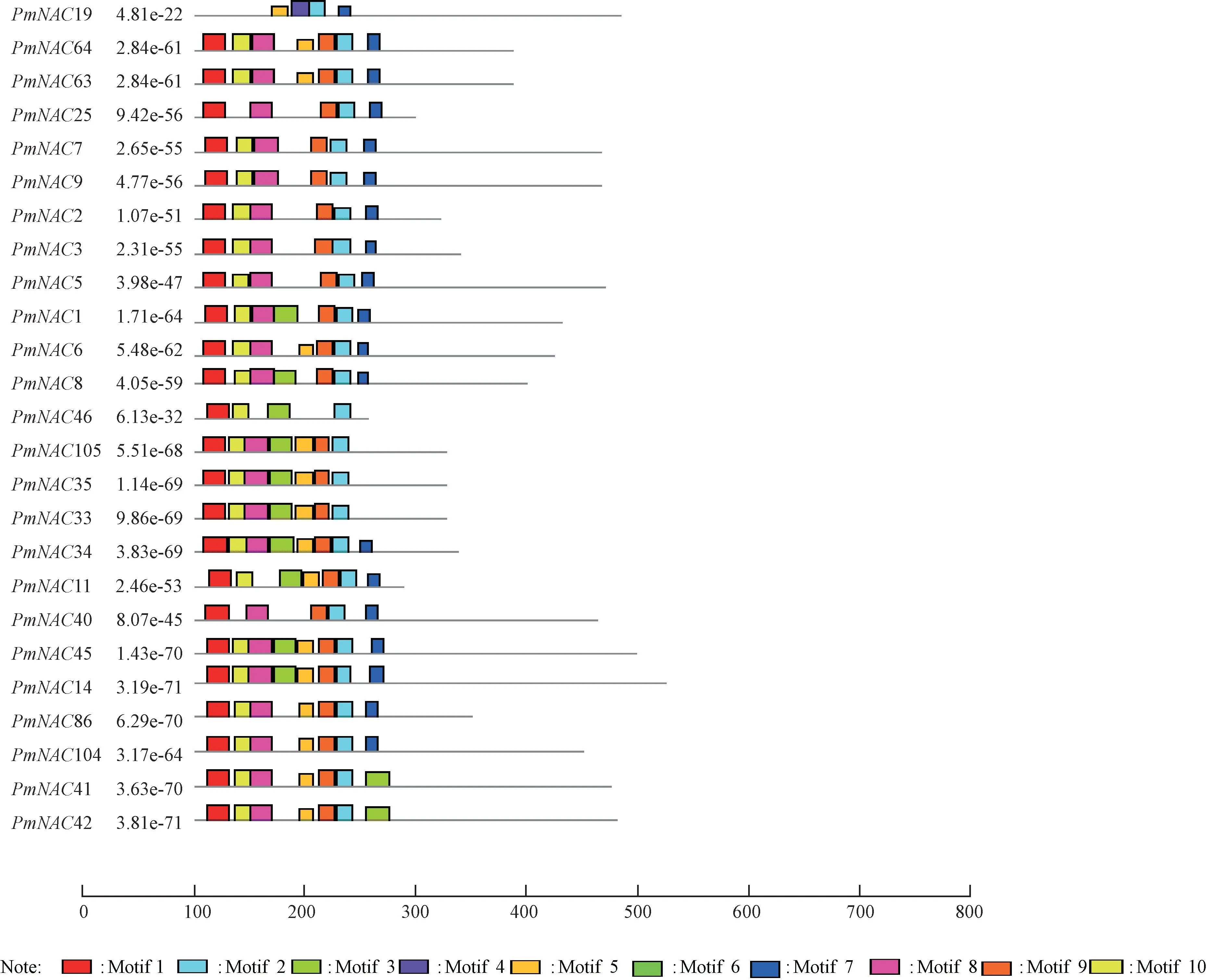
图3 果梅NAC 蛋白保守基序Fig.3 The NAC proteins conservative motif of P. mume
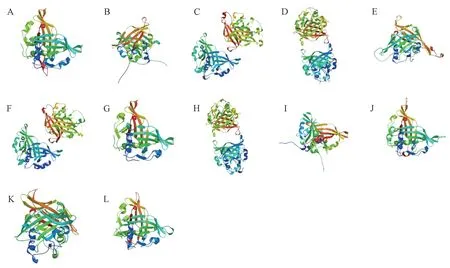
图4 12 个代表性的果梅NAC 蛋白质的三级结构Fig.4 The tertiary structure of 12 representative NAC proteins in P. mume
由图5 可知,12 个PmNAC 基因在不同组织中的表达差异明显,其中3 个基因(PmNAC54、PmNAC32 和PmNAC68) 在果皮中的表达量最高, 4 个基因(PmNAC60、PmNAC95、PmNAC51 和PmNAC2)在果肉中的表达量最高,3 个基因(PmNAC31、PmNAC62 和PmNAC48)在嫩茎中的表达量最高,其他2 个基因PmNAC61 和PmNAC71 分别在果胚和花芽中的表达量最高。 此外,PmNAC54 在果胚和嫩茎中的表达相对较高,在花芽和果肉中表达量极低,在幼叶中几乎不表达;PmNAC61 在花芽中的表达量相对较高,在嫩茎中也有表达;PmNAC71 在嫩茎、果肉、果胚中表达相对较高;PmNAC31 在花芽和果肉中的表达量也相对较高,其次在幼叶中也有表达;PmNAC32 在果肉中的表达量较高,在其他组织中几乎不表达;PmNAC68 在花芽中的表达量较高,其次在果肉和果胚中也有表达;PmNAC62 在果皮、果肉、果胚中的表达量较高,在幼叶中也有较高的表达;PmNAC60 在嫩茎和果皮中也有表达相对较高,其次在花芽、幼叶中也有表达;PmNAC95在果皮中表达相对较高,在其他组织中几乎不表达;PmNAC51 在果皮和嫩茎中的表达量较高;PmNAC48在嫩茎中表达量相对较高,其次在果肉、果胚、果皮也有表达;PmNAC2 在果皮和果胚中的表达也相对较高,其次在花芽和嫩茎也有表达。 PmNAC 基因不同的表达特性表明它们在进化过程中出现了功能分化。 总体来看, PmNAC32 几乎仅在果皮和果肉中表达,PmNAC68 在果皮中也具有很高的表达水平,推测它们可能在果梅的果实发育和果皮着色方面发挥重要作用,但其具体分子机理仍需进一步深入研究。
3 讨论
NAC 转录因子是植物基因组中最大的转录因子家族之一,且主要存在于陆生植物中[32]。 前人研究认为植物体某一特定基因家族的多成员现象,是由于其在生物进化过程中,基因组的广泛复制和多样化造成的[33]。 本研究利用生物信息学方法首次在果梅中鉴定得到106 个NAC 基因家族成员,低于拟南芥(117)、水稻(151)[3]、大豆(152)[5]等作物,可能是由于果梅基因组中没有大规模的片段复制,且相对保守等原因造成的。 基因可以通过多种方式进行扩增,包括全基因组复制、串连复制、片段复制和逆转座复制等[33]。通过染色体定位分析发现,PmNAC 在果梅基因组中广泛分布,且存在明显的串联重复现象,表明串联重复是果梅NAC 基因扩增的一种重要方式。 蛋白结构分析发现,果梅NAC 基因多编码酸性氨基酸,且多数NAC蛋白为亲水蛋白。 蛋白质二级结构中的α-螺旋、β-折叠、β-转角和无规则卷曲,以及三级结构中的蛋白质折叠的空间构象具有较高的保守性,与在其他植物的NAC 基因家族的研究结果一致[6,25,27]。 根据系统进化树将106 个果梅NAC 基因分为12 个亚族,除第9亚族外,每个亚族均同时含有果梅和拟南芥NAC 基因,在各个亚族中的数目不同可能是由基因复制或基因丢失导致的,且聚在同一亚族的基因,其亲缘关系较近,预示它们可能具有相似的功能[10]。
前人针对不同作物的NAC 基因家族进行了大量的研究,如韩芹芹[34]发现在番茄果实的绿熟期和破色期,SINAC3(JF701987)基因在果皮和果肉中的表达较高,在果实破色期达到最高值,干涉表达的转基因番茄果实成熟时间明显延迟,果实中番茄红素的合成受到明显抑制,说明该基因在果实的成熟转变过程中发挥重要作用;Kou 等[35]研究发现,番茄多个NAC 基因参与调控果实的发育成熟,其中SNAC5 基因在番茄果实粉红期表达量较高,SNAC8 和SNAC9 基因在番茄破色期和红熟期的外果皮大量表达,粉红期的表达量则较低。 本研究结果表明,PmNAC 基因在果梅不同组织中均有表达,推测NAC 基因可能参与果梅的整个生长发育过程,但不同亚族的NAC 基因表达差异明显,且大部分的PmNAC 基因在果皮、果肉上都具有较高的表达量,其中PmNAC32 和PmNAC68 表达量最高,推测NAC 基因可能在果梅的果皮着色和果实发育过程中发挥重要的调控作用。
有研究还发现,NAC 基因通过乙烯信号转导途径参与果实的发育成熟,表明NAC 协同乙烯共同调控果实的生长发育过程,且可以通过改变NAC 基因的表达水平,从而达到调控果实发育成熟,延缓衰老,延长采后贮藏保鲜的目的[36-37]。 本研究结果表明,PmNAC61基因在果胚的表达量最高,推测该基因可能与种子或胚的发育有关。 此外,PmNAC31 和PmNAC71 分别在茎和花芽中表达量最高,推测它们可能分别与茎和花器官的发育相关,与Christianson 等[38]降低拟南芥AtNAC102 的表达水平,其种子萌发率显著下降的研究结果一致。 本研究结果表明,果梅NAC 基因在不同组织中的多种表达模式可能是由于进化过程中为了适应环境而出现功能分化的结果,从而表现出功能的多效性。 随着生物技术的发展,利用转基因技术导入编码重要功能的基因,对于改善作物品质、提高抗逆性具有重要意义,因此,有关果梅NAC 蛋白的功能分析及其所参与的信号转导途径可采用该技术进一步深入研究。
4 结论
本研究首次在果梅基因组鉴定得到106 个NAC基因家族成员,分析表明,果梅NAC 基因多是编码酸性氨基酸,大部分NAC 蛋白为亲水蛋白,且为典型的核蛋白,根据系统发育特征可分为12 个亚族。 此外,通过实时荧光定量PCR 技术得到不同亚族的PmNAC基因在果梅各个组织的表达模式,NAC 基因在不同组织中均有表达,但表达差异明显,说明NAC 基因参与果梅的整个生长发育过程。 其中 PmNAC32 和PmNAC68 表达特征明显,在果皮和果肉中具有较高的表达量,可能对果梅的果实发育和果皮着色发挥重要的调控作用,但其分子机理仍需进一步研究。 本研究结果为果梅NAC 蛋白的功能分析奠定了一定的理论基础。
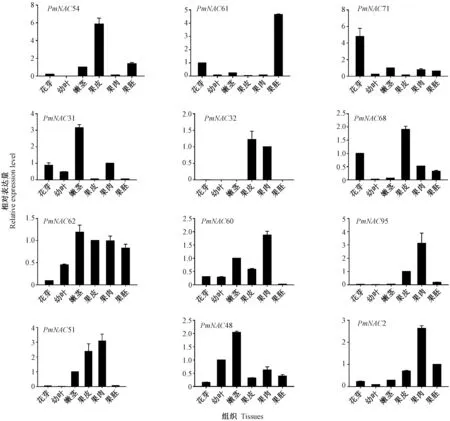
图5 PmNAC 基因在不同组织中的相对表达水平Fig.5 Relative expression levels of PmNAC genes in different tissues of P. mume

