高分辨率熔解曲线技术检测结直肠癌患者KRAS基因突变
毛旭华, 汤俊明, 乔国洪, 陈姝颖, 王海啸
论 著
高分辨率熔解曲线技术检测结直肠癌患者KRAS基因突变
毛旭华, 汤俊明, 乔国洪, 陈姝颖, 王海啸
目的探讨高分辨率熔解曲线技术(HRM)检测结直肠癌患者KRAS基因突变的可行性及其临床意义。方法采用HRM技术检测60例结直肠癌患者石蜡包埋组织标本KRAS基因2号外显子12和13位密码子突变,并与Sanger测序法检测结果进行对比分析。结果HRM法检测结直肠癌患者KRAS基因2号外显子12和13位密码子突变检出率为36.67%(22/60),Sanger测序法检测突变检出率为33.33%(20/60)。HRM法检测突变检出率高于Sanger测序法,HRM法检测敏感性为100%,特异性为95.24%。结论HRM法检测KRAS基因突变,灵敏度高,敏感性和特异性好,具有操作简便、节约时间、成本低的特点,方法可行。
结直肠肿瘤; 基因,KRAS; 突变; 高分辨率熔解曲线
结直肠癌是常见的恶性肿瘤之一,全球每年约有140万例患者诊断为结直肠癌,其中有近70 万人死于该病[1-2]。KRAS基因是常见的致癌基因之一,其中30%~50%结直肠癌患者伴有KRAS基因突变。KRAS基因是否突变是了解肿瘤发展、预后、疗效的关键指标。KRAS基因最常见的突变为2号外显子12和13位密码子点突变,约90%的突变发生于此,极少部分发生于61、63、117和146位密码子[3-4]。高分辨率熔解曲线(high resolution melting,HRM)是2003 年兴起的一种基因序列分析新技术,具有快速、准确、廉价、闭管操作等优点[5-6]。本研究采用HRM法针对结直肠癌患者KRAS基因2号外显子12和13位密码子突变检测进行研究,并探讨其临床意义。
1 资料和方法
1.1 标本收集
2015年1月至2016年12月,江苏大学附属宜兴市人民医院收治的经病理诊断为结直肠癌的石蜡包埋组织样本60例,其中男34例,女26例,年龄38~72岁,中位年龄62岁。本研究通过本院医学伦理委员会批准,患者均签署知情同意书。
1.2 实验方法
1.2.1 引物 设计适用于HRM技术检测KRAS基因2号外显子12和13位密码子突变的引物,引物序列如下:正向引物:5’-AAGGCCTGCTGAAAATGAC-3’,反向引物:5’-TGGTCCTGCACCAGTAATATG-3’。
1.2.2 HRM反应体系与反应条件 HRM技术检测KRAS基因2号外显子12和13位密码子突变反应体系为:DNA模板,200 nM primers,3 mM MgCl2和LightCycler HRM Master Reaction Mix (瑞士Roche公司),反应体积20 μl。采用仪器为Roche LightCycler 480 system。HRM技术检测KRAS基因2号外显子12和13位密码子突变扩增反应条件为:95 ℃预变性10 min,然后95 ℃变性10 s,65 ℃退火10 s,72 ℃延伸20 s,共45个循环(采用touchdown技术[7],前10个循环退火温度从65 ℃降到55 ℃,每个循环降1 ℃)。扩增结束进行熔解曲线分析,反应条件为95 ℃ 1 min,40 ℃ 1 min,熔解曲线数据收集从70 ℃到95 ℃,温度上升速率为1 ℃/s。40 ℃冷却。
1.2.3 HRM技术检测KRAS基因 2号外显子12和13位密码子突变结果均与Sanger测序法检测结果进行对比分析。分析其特异性和敏感性。
2 结果
2.1 HRM法和Sanger测序法检测KRAS基因2号外显子12和13位密码子突变结果统计与分析
在60例结直肠癌患者中,HRM法共检测出KRAS基因2号外显子12和13位密码子突变22例,检出率为36.67%。其中G12S检出6例,G12D检出7例,G12V检出3例,G12C检出2例,G13D检出4例。Sanger测序法检测到KRAS基因2号外显子12和13位密码子突变20例,检出率为33.33%。其中G12S检出6例,G12D检出6例,G12V检出2例,G12C检出2例,G13D检出4例。结果见表1。图1为实验中选取的HRM法检测KRAS基因2号外显子12和13位密码子突变示意图,图2为Sanger测序法检测KRAS基因2号外显子12和13位密码子突变示意图。

表1 HRM法和Sanger测序法检测KRAS基因2号外显子12和13位密码子突变结果
2.2 HRM法特异性和敏感性
以Sanger测序法结果作为金标准,HRM法检测KRAS基因2号外显子12和13位密码子突变,敏感性为100.00%,特异性为95.24%。
3 讨论
KRAS基因是一种重要的原癌基因,是RAS基因家族成员之一,其定位于12p12.1,编码相对分子质量为21kD,具有GTP 酶活性的p21 蛋白[8-9]。突变型KRAS基因,在无需活化EGFR的条件下,激活下游信号传导通路,导致细胞的生长、增殖和凋亡发生异常。细胞传导信号的紊乱以及增殖的失控会导致细胞癌变[7]。KRAS基因突变主要在2号外显子12和13位密码子上发生点突变为主。KRAS基因是否突变是了解肿瘤发展、预后、疗效的关键指标。目前,KRAS基因突变的检测已得到广泛的认同和使用,《美国国立癌症综合网络结直肠癌临床实践指南》指出,结直肠癌患者应该检测KRAS基因的状态,只有KRAS基因野生型的患者才建议使用表皮生长因子受体抑制剂的治疗。
检测KRAS基因突变的方法有很多,直接测序法、二代测序法、Taqman探针法、变性高效液相色谱法、突变扩增阻滞系统(amplification refractory mutation system,ARMS)法等。目前,国内临床检测KRAS 基因突变主要采用Sanger测序法和ARMS法。Sanger测序法被公认为是基因检测的“金标准”。但随着时代的发展,基因检测技术的不断进步,Sanger测序方法学的限制,其检测灵敏度已不能满足基因检测的需要。对于肿瘤组织DNA含量少,基因突变比例低的样本,Sanger测序法无法准确检测,易产生假阴性。ARMS法操作简便,灵敏度高,但其价格昂贵。因此寻找并建立一个成本低、操作简单、准确可靠的检测方法,并推广到临床应用,显得尤为重要。
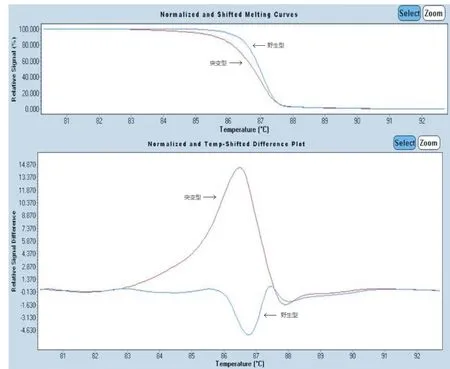
图1 HRM法检测KRAS基因2号外显子12和13位密码子突变示意图
HRM 技术是PCR 扩增技术与熔解曲线分析技术相结合,依靠高分辨温度检测PCR仪和新型饱和荧光染料实现基因序列分析的新技术[6]。HRM法的原理在扩增含突变位点基因的过程中,由于突变位点不匹配导致双链 DNA在升温过程中会先解开,荧光染料从局部解链的 DNA分子上释放,从荧光强度与时间曲线上就可以判断是否存在突变位点[7]。HRM技术具有快速、准确、廉价、闭管操作等优点[6],同时HRM技术具有较高的灵敏度。文献报道,HRM技术检测限为5%[6],有学者指出,HRM方法检测基因突变的检出限可达1%[10]。其唯一不足之处是无法得知具体突变类型,对于KRAS基因这类已广泛深入研究的基因检测而言,此不足在实际临床检测工作中影响甚小。
本研究中,HRM法检测KRAS基因2号外显子12和13位密码子突变,检出率为36.67%,结果与文献报道[11-12]的30%~40%检出率相似。本研究结果显示,HRM法突变检出率略高于Sanger测序法,已初步表现出HRM法检测灵敏度的优势。同时,敏感性和特异性均符合临床应用要求。本研究中HRM法比Sanger测序法多检出的突变是否存在假阳性,还需进一步的实验验证,以及临床疗效观察验证。
总之,HRM法检测KRAS基因2号外显子12和13位密码子突变,灵敏度高,敏感性和特异性好,操作简便,节约时间,成本低的特点,适合临床应用。

图2 Sanger测序法检测KRAS基因2号外显子12和13位密码子突变示意图
[1] Siegel RL, Miller KD, Jemal A. Cancer statistics, 2016[J]. CA Cancer J Clin, 2016,66(1):7-30.
[2] 潘雪峰, 范乃军, 高春芳. LncRNA与结直肠癌诊断治疗的研究进展[J].肿瘤学杂志,2017,23(2):111-115.
[3] 凌云, 应建明, 邱田, 等. 结直肠癌患者KRAS、BRAF及PIK3CA基因突变检测分析[J].中华病理学杂志,2012,41(9):590-594.
[4] Lea IA, Jackson MA, Li X, et al. Genetic pathways and mutation profiles of human cancers: site- and exposure-specific patterns[J]. Carcinogenesis, 2007,28(9):1851-1858.
[5] Gundry CN, Vandersteen JG, Reed GH, et al. Amplicon melting analysis with labeled primers: a closed-tube method for differentiating homozygotes and heterozygotes[J]. Clin Chem, 2003,49(3):396-406.
[6] 王姣, 尤崇革. 高分辨率熔解曲线技术及应用新进展[J].兰州大学学报(医学版),2016,42(5):55-61.
[7] 王卓, 井昶雯, 曹海霞, 等. 高分辨率熔解曲线分析技术检测 EGFR 基因突变方法的建立及其初步临床应用[J].中国肿瘤外科杂志,2014,(4):236-239.
[8] Tan C,Du X.KRAS mutation testing in metastatic colorectal cancer[J].World J Gastroenterol, 2012,18(37):5171-5180.
[9] 李丽娟, 罗婉珊, 张家彬, 等. 基于下一代测序平台的结直肠癌KRAS基因检测[J].分子诊断与治疗杂志,2016,8(6):401-406.
[10] 刘丽琴. 结直肠癌和肺癌KRAS和BRAF基因突变HRM检测方法的建立[D].广州: 南方医科大学,2012.
[11] 刘影, 郑细闰, 朱亚珍, 等. 252例结直肠癌组织中KRAS、NRAS、 BRAF、PIK3CA的基因突变分析[J].临床与实验病理学杂志,2016,32(8):851-855,859.
[12] 祁秀敏, 张熔熔, 唐威. Kras、Nras、Braf基因突变联合检测在转移性大肠癌中的意义[J].广东医学,2017,38(2):276-279.
DetectionofKRASmutationsincolorectalcancerpatientsbyhighresolutionmelting
MAOXuhua1,TANGJunming1,QIAOGuohong1,CHENShuying2,WANGHaixiao3.
(1.ClinicalLaboratory,AffiliatedYixingPeople’sHospital,JiangsuUniversity,Wuxi214200,China;2.DepartmentofPathology,AffiliatedYixing’sPeopleHospital,JiangsuUniversity,Wuxi214200,China;3.DepartmentofGastrointestinalSurgery,Huai’anFirstPeople’sHospital,NanjingMedicalUniversity,Huai’an223300,China)
WANGHaixiao,Email:whxnjmu@163.com
ObjectiveTo explore the clinical significance of detecting KRAS mutations in colorectal cancer (CRC) patients by high resolution melting (HRM).MethodsThe mutations of KRAS 12 and 13 codons in exon 2 were detected with HRM in 60 formalin-fixed paraffin-embedded samples,and compared with Sanger sequencing.ResultsThe KRAS mutation rate of 12 and 13 codons in exon 2 was 36.67% (22/60) detected by HRM and was 33.33% (20/60) detected by Sanger sequencing.The KRAS mutation rate detected by HRM was higher than that by Sanger sequencing.Compared with Sanger sequencing,the sensitivity of HRM was 100%, and the specificity of HRM was 95.24%.ConclusionsDetecting KRAS mutations by HRM possesses high specificity,good sensitivity,simple operation,saving time and low cost quality.It is feasible,and suitable for promotion.
Colorectal neoplasms; Genes, KRAS; Mutation; High resolution melting analysis
2016年宜兴市科技局民生项目(社会发展类)
214200 江苏 无锡,江苏大学附属宜兴市人民医院 检验科(毛旭华,汤俊明,乔国洪); 病理科(陈姝颖); 223300 江苏 淮安,南京医科大学附属淮安第一医院 胃肠外科(王海啸)
王海啸,Email:whxnjmu@163.com
10.3969/j.issn.1674-4136.2017.05.006
1674-4136(2017)05-0296-04
2017-07-05] [本文编辑:李庆]

