藜麦SOD 家族基因的鉴定及其对混合盐碱胁迫的响应
邓玉荣 韩联 王金龙 韦兴翰 王旭东 赵颖 魏小红 李朝周

摘要:超氧化物歧化酶(superoxide dismutase,SOD)是植物抗氧化系统的关键酶,在保护植物免受生物和非生物胁迫方面发挥重要作用。以拟南芥SOD为基础,通过序列比对在藜麦基因组中鉴定出12个SOD 基因,分别定位于細胞核、微体及线粒体,在11条染色体上不均匀分布,其编码蛋白质三级结构显示Cu/Zn-SODs与Fe-SODs为同源二聚体,Mn-SODs为同源四聚体。CqSOD 基因内含子/外显子分布模式不尽相同,内含子数介于4~7个,保守基序差异明显。系统发育关系显示,SOD蛋白可分为Cu/Zn-SODs、Fe-SODs及Mn-SODs 3个亚族。此外,所有的CqFe-SODs 及CqMn-SODs 启动子区都含有脱落酸激素反应顺式元件,CqSOD12 与11 个CqSOD蛋白及4个CqCAT蛋白存在相互作用。表达谱分析表明,12个CqSOD 基因对混合盐碱及硝普钠均有较强响应。研究结果为SOD 基因在植物发育和胁迫响应中的作用及分子机制研究奠定基础。
关键词:藜麦;SOD 家族基因;盐碱胁迫;生物信息学分析;表达分析
doi:10.13304/j.nykjdb.2022.0558
中图分类号:S519 文献标志码:A 文章编号:10080864(2024)01002812
在自然条件下,植物容易受到干旱、盐、极端温度、重金属及其他对植物生长和发育产生重大影响的非生物因素胁迫[1]。当植物受到胁迫时,会在其细胞中产生更多的活性氧(reactive oxygenspecies,ROS)来氧化蛋白质、破坏细胞膜并导致DNA损害。ROS主要在质外体、线粒体、质膜、叶绿体、过氧化物酶体、内质网和细胞壁中形成[2],其主要包括羟基自由基(·OH)、超氧阴离子自由基(O2· -)、过氧化氢(H2O2)和单线态氧(1O?),能引起细胞膜的损伤、大分子的过氧化和变质,最终导致细胞死亡。ROS也被认为是不同生物体中的信号分子,可以影响植物的各种生理过程。使用活性氧清除剂可以通过调节酶反应家族基因,如超氧化物歧化酶(superoxide dismutase,SOD)、过氧化氢酶(catalase,CAT)、过氧化物酶(peroxidase,POD)和谷胱甘肽过氧化物酶(glutathioneperoxidase,GPX)等编码基因的表达来抵抗环境胁迫[3]。因此,为了减轻ROS的毒性,植物建立了组织良好的复合抗氧化防御系统,包括许多非酶和酶抗氧化剂。
SOD是一种金属酶,首先在红细胞中发现,然后在细菌、脊椎动物和高等植物中进行了研究[4]。SODs可以催化O2· -进行歧化反应生成H2O2和O2,SOD可在植物的根、果实、叶子和种子中检测到,其为细胞抵御氧化应激提供了必要的保护[5]。根据SOD 与金属辅助因子的结合模式,可分为铁SOD(FeSOD)、铜/锌SOD(Cu/ZnSOD)、锰SOD(MnSOD)、和镍SOD(NiSOD)[6]4 个亚型,不同亚型具有不同的功能。此外,它们具有不同的氨基酸序列、体外亚细胞定位和晶体结构以及不同的过氧化氢敏感性[7],某些SOD单独分布在细胞隔室中,在氧化应激中发挥重要作用。Cu/ZnSODs主要分布在叶绿体、细胞外空间和细胞质中,也存在于某些细菌和所有真核生物中,而MnSODs主要存在于植物线粒体中[8]。植物基因组中的MnSOD 在消除线粒体ROS 中发挥作用[9]。FeSODs主要分布在原生动物、原核生物、细胞质和植物叶绿体中,而NiSODs存在于链霉菌和一些蓝细菌中,但在植物中不存在[2]。
最近的研究表明,SODs 可以保护植物免受热、干旱、寒冷、脱落酸、盐和乙烯等非生物胁迫的影响[10]。SOD 基因可以在不同的植物中受到热、干旱、冷、盐、渗透胁迫、氧化胁迫和激素信号转导等条件下诱导和转录。近年来,越来越多植物基因组信息的发布使SOD 基因家族研究成为热点,Su等[6]在油菜(Brassica campestris)中鉴定了31个SOD 基因,其中8个在不同激素和非生物胁迫条件下显著上调;丹参(Salvia miltiorrhiza)的8 个SOD 基因对寒冷、盐、干旱、重金属和植物激素表现出不同的响应特征[11];大麦(Hordeum vulgare)的SOD 基因家族共有7 个成员,其中HvSOD1、HvSOD4 和HvSOD5 在干旱和盐胁迫下表达量显著变化[12];在番茄(Solanum lycopersicum)中,SlSOD1 是9 个SlSOD 基因中唯一显著上调的基因,而SlSOD2、SlSOD5、SlSOD6 和SlSOD8 受盐胁迫的调节;Feng 等[13] 发现,SlSOD2、SlSOD5、SlSOD6 和SlSOD8 4个基因的表达水平在强干旱环境下明显提高。此外,同一类型的SOD 基因在不同物种中的表达模式也不尽相同,氧化胁迫下拟南芥中MnSODs 的表达没有变化,而盐胁迫下豌豆(Pisum sativum)和小麦(Triticum aestivum)中MnSODs 的表达有显著变化[14]。以上研究表明,不同的SOD 基因在不同环境胁迫下的表达模式不同。迄今为止,SOD 基因家族已在许多植物中得到解析鉴定,如拟南芥(Arabidopsis thaliana)、高粱(Sorghum bicolor)、菜豆(Phaseolus vulgaris)和杨树(Populus)[15]。藜麦起源于南美洲安第斯山脉,为藜科藜属一年生四倍体双子叶草本植物,是世界重要的经济和营养作物[16],具有较强的耐旱、耐盐、耐瘠薄特性。赵颖等[17]发现,藜麦种子能耐受较高水平的混合盐碱胁迫,有些品种抗盐性极强,是抗盐作物棉花的2~3 倍。刘文瑜等[18]发现,随NaCl含量的升高和处理时间的延长,藜麦幼苗叶片叶绿素含量先升高后下降,可溶性糖、脯氨酸和丙二醛含量逐渐升高,SOD、POD、CAT和抗坏血酸过氧化物酶(ascorbate peroxidase,APX)活性增强以提高藜麦应对盐胁迫的能力。但关于藜麦分子方面研究较少,有研究发现盐胁迫下藜麦大多数基因在转录水平发生变化[19],目前尚未见SOD 家族的报道。因此,本研究深入分析藜麦中SOD 基因在不同组织中的系统发育谱系、基因结构、基本基序及非生物胁迫下的表达差异等,为藜麦SOD 基因的功能鉴定奠定了基础。
1 材料与方法
1.1 植物材料和处理
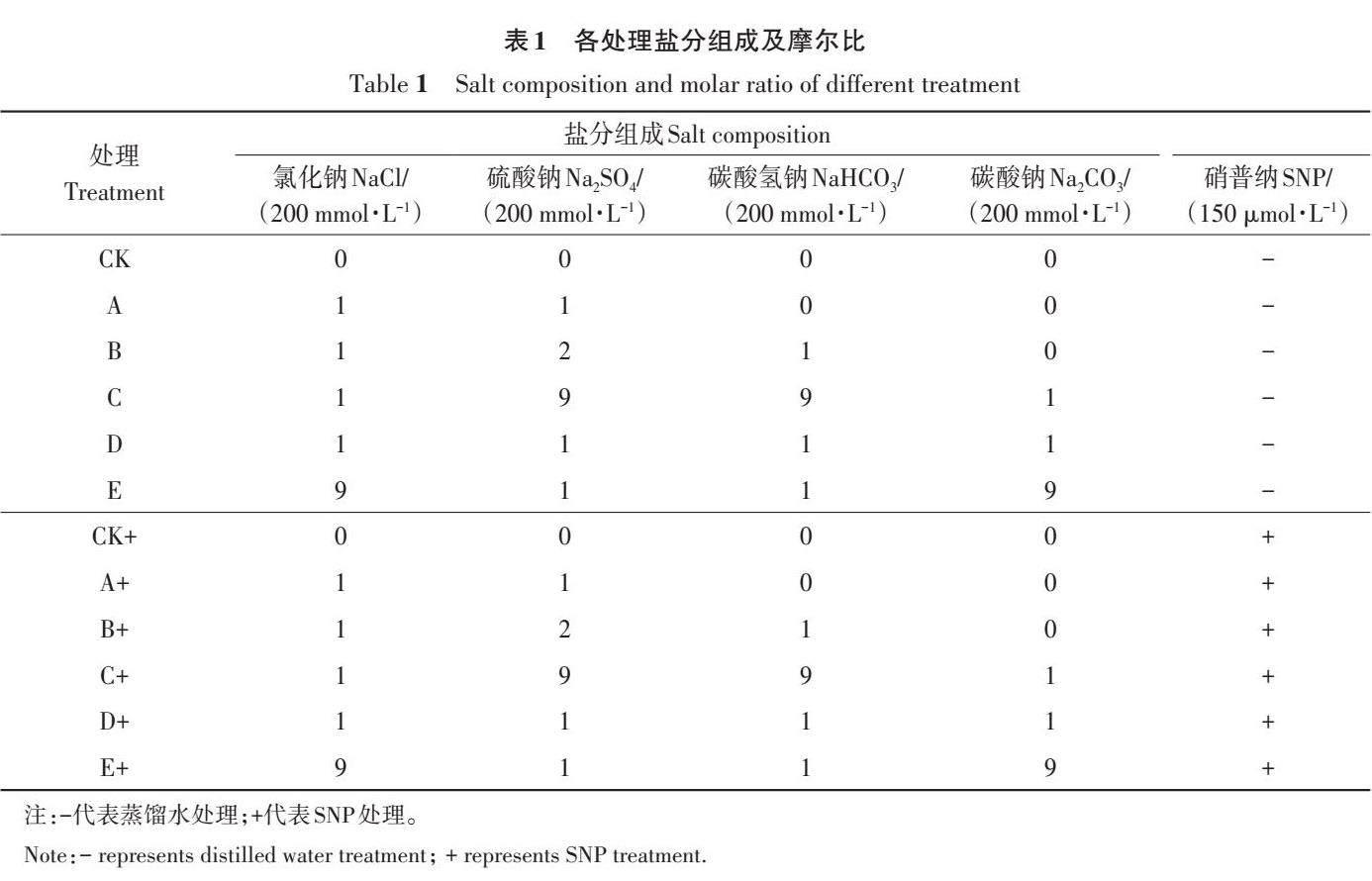
以‘陇藜2号(甘肃省农业科学院提供)为试验材料。藜麦种子用体积分数75%的乙醇灭菌30 s,无菌水洗涤2次,10%次氯酸钠灭菌15 min,用无菌水洗涤5次,然后接种在MS固体培养基上,种子在光照培养箱25 °C、光照16 h/黑暗8 h下培养48 h直至发芽,然后将发芽的种子种植在含有沙子、珍珠岩和泥炭(体积比1∶1∶1)的托盘中,并在生长室中培养,条件为:相对湿度60%~70%,光照强度4 000 lx,时间12 h,白28 ℃/昼18 ℃。幼苗生长2个月至6叶期,参照赵颖等[17]的方法配置混合盐碱处理液,分别进行混合盐碱(200 mmol·L?1,碱性盐比例递增)胁迫及混合盐碱+外源NO供体硝普钠(sodium nitroprusside,SNP,150 μmol·L?1)喷施处理,喷施100 mL SNP后24 h,每盆喷施100mL混合盐碱,均匀喷施于叶片上。共计12个处理(表1),每个处理3个重复。在处理后第9天收集叶片,于?80 ℃的冰箱中储存,用于后续的实时荧光定量试验。
1.2 藜麦SOD 基因家族的鉴定
以拟南芥的7个SOD 基因编码的氨基酸序列(CAA43270、AAD10208、AAC24833、AAA32791、CAA73188、AAC24834 和AAC24832)为比对序列,通过BLAST搜索藜麦氨基酸序列文件,藜麦的基因组、氨基酸序列以及GFF 注释文件来自Phytozome 数据库(https://phytozome.jgi.doe.gov/pz/portal.html)。使用Pfam(https://pfam.xfam.org/)[20]、SMART(http://smart.embl-heidelberg.de/)与NCBICDD(https://www.ncbi.nlm.nih.gov/cdd/)数据库获得和鉴定候选蛋白质序列,CqSOD 家族最终由SOD保守结构域的蛋白质序列确定。
1.3 理化特性及亚细胞定位
使用ProtParam 工具(http://web. expasy. org/protparam/)计算CqSOD蛋白的理化特性,包括氨基酸数、分子量、理论等电点(isoelectric point,pI)和亲水性的总平均值。用ProtComp 9.0(http://linux1.softberry.com/)预测CqSOD蛋白的亚细胞定位。
1.4 CqSODs 的二级和三级结构与染色体定位
采用ExPASy(https://www.expasy.org/)提供的SOPMA 和SWISS MODEL 在线服务器分析CqSODs的二级和三级结构。通过分子可视化软件VMD(https://www.ks.uiuc.edu/Research/vmd/)检测蛋白质的三级结构。使用NetPhos 3.1 Server分析CqSOD的磷酸化位点。通过NetOGlyc-4.0分析糖基化位点。根据藜麦GFF文件,预测CqSOD 基因在染色体上的位置。
1.5 CqSOD 的基因结构与保守基序预测
CqSOD 基因家族的cDNA和DNA序列取自基因组数据库。利用基因结构展示服务器GSDS2.0(http://gsds.cbi.pku.edu.cn/)对其基因结构进行分析,并解析各CqSOD 的内含子分布模式。使用默认设置的MEME Suite(http://meme-suite.org/)预测保守的蛋白质基序。
1.6 CqSOD 的系统发育树构建
为研究不同植物之间SODs 的系统发育关系,使用Clustal W 以默认参数比对多个SOD蛋白序列,并使用MEGA X.10.0.5 使用邻接(neighborjoining,NJ)方法和Poisson model 模型,引导程序设置1 000次重复,构建系统发育进化树。
1.7 CqSOD 基因的启动子序列分析及蛋白互作网络图的构建
分析CqSODs 启动子中的顺式元件有助于了解基因表达调控的信息。每个CqSOD 基因起始密码子的2.0 kb上游序列作为启动子区。然后,使用PlantCARE 服务器(http://bioinformatics.psb.ugent.be/webtools/plantcare/html/search_CARE.html)预测CqSOD 基因启动子中的顺式元件,通过TBtools(https://github.com/CJ-Chen/TBtools)中的启动子可视化工具绘制启动子的分布图。为预测藜麦中SOD家族的蛋白质-蛋白质相互作用网络,使用与藜麦SOD 基因直系同源的拟南芥SOD 基因通过STRING构建网络图。
1.8 混合盐碱及SNP 处理下CqSOD 基因家族的实时定量PCR
使用HiPure HP Plant RNA Mini Kit(Magen)提取总RNA。使用NanoDrop 2000C 分光光度计(Thermo)评估提取RNA的纯度和质量浓度,以无RNase水作为空白。用HiScript Ⅱ Q RT SuperMixⅡ(Vazyme Biotech Co., Ltd.)合成cDNA。将单次40 μL反应中约1 μL(1 000 ng·μL?1)的总RNA反转录合成cDNA,稀释50 倍备用。按照ChamQTMSYBR qPCR Master Mix(Vazyme Biotech Co., Ltd.)说明书中方法,在Roche LightCycler 96 PCR 系统中进行qRT-PCR分析。20 μL反应体系包含10 μLSYBR qPCR Master Mix、5 μL 稀释cDNA、0.5 μL正向和反向引物(10 μmol·L?1)(引物序列见表2)和4 μL ddH2O。反应程序如下:95 ℃变性30 s;95 ℃ 10 s,60 ℃ 30 s,45个循环。每个基因进行3次重复。CqActin (GenBank accession:LOC110715281)用作标准化CqSOD 基因表达的内部对照。使用2-ΔΔCt [21]方法计算基因的表达水平。
1.9 数据处理
使用Excel绘制图表,使用Dunnett检验分析统计显著性。
2 结果与分析
2.1 CqSOD 家族基因的鉴定及理化性质分析
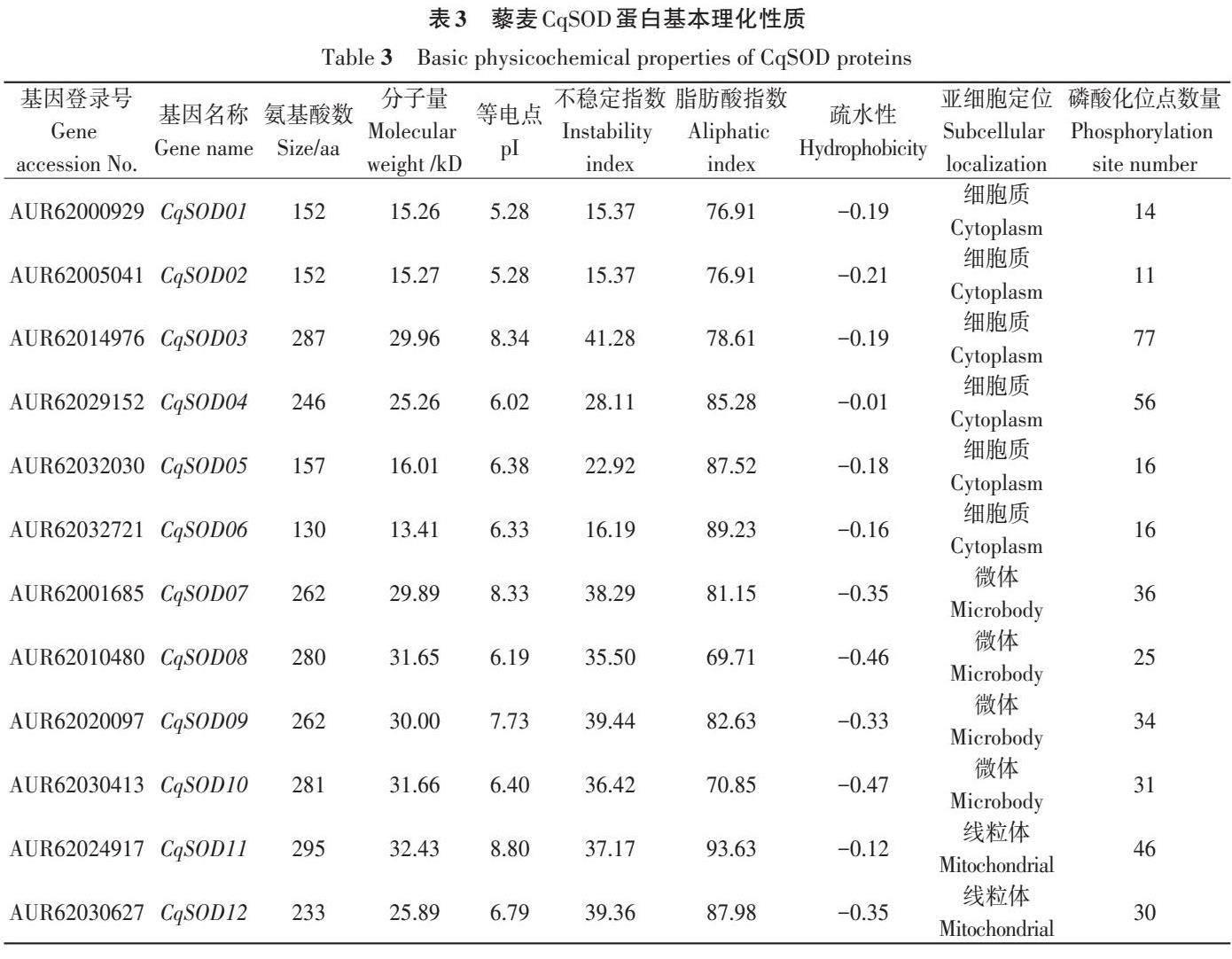
基于已知拟南芥SOD 蛋白序列进行BLAST搜索以及SMART和Pfam 数据库的鉴定,在藜麦中鉴定了12 个SOD 基因家族成员,将其命名为CqSOD01~CqSOD12。根据蛋白结构域分析,将12个CqSODs 分为3 类:Cu/Zn-SODs(CqSOD01~CqSOD06)、Fe-SODs(CqSOD07~CqSOD10)和Mn-SODs(CqSOD11~CqSOD12)。其中,CqSOD01~CqSOD06 都包含1 个保守的SOD_Cu 结构域(Pfam: PF00080),该结构域为Cu/Zn-SOD蛋白的典型特征。CqSOD07~CqSOD10 和CqSOD11~CqSOD12中都存在N端铁/锰SOD-α发夹结构域(Pfam:PF00081)和C 端铁/锰SOD 结构域(Pfam:PF02777),表明它们均属于Fe/Mn-SODs亚家族。理化分析(表3)表明,12个CqSOD蛋白的氨基酸序列长度、分子量、等电点(pI)、脂肪酸指数和疏水性不尽相同。CqSOD 蛋白的氨基酸数在130(CqSOD06)~295(CqSOD11),存在较大差别;分子量在13.41~32.43 kD,pI 在 5.28~8.80。脂肪酸指数介于69.71~93.63;所有 CqSOD蛋白的疏水性指数均小于0,都属于亲水性蛋白质;不稳定指数介于15.37~41.28,其中11个蛋白质的不稳定指数小于40,属于稳定蛋白。亚细胞定位显示,6个Cu/Zn-SODs亚家族成员定位于细胞质,4个Fe-SODs 定位于微体,2 个Mn-SODs 成员定位于线粒体基质中。此外,潜在磷酸化位点的数量介于11~77,其中CqSOD03磷酸化位点最多(77个),潜在的磷酸化位点越多,多功能的可能性就越大。
2.2 CqSOD 蛋白结构及染色体定位分析
蛋白质的结构分析对于了解其功能具有重要意义。二级结构预测分析(表4)表明,CqSOD蛋白主要由α螺旋、延伸链和无规卷曲组成,5个Cu/Zn-SODs中无规则卷曲>延伸链>α螺旋;所有的Fe-SODs与Mn-SODs的二级结构组成均为无规则卷曲>α 螺旋>延伸链。染色体定位显示,除CqSOD06、CqSOD07 定位于同一染色体外,其余10个基因均定位于不同的染色体上。
三级结构分析表明, 所有Cu/Zn-SODs 和Fe-SODs 都是同源二聚体,Mn-SODs 都是同源四聚体。除了CqSOD03与CqSOD04外,所有CqSODs都有不同数量的金属离子结合位点,其中CqSOD01、CqSOD02、CqSOD05 与CqSOD06 螯合2 个铜离子,CqSOD07~CqSOD10 螯合2 个铁离子,CqSOD11~CqSOD12 螯合4 个锰(Ⅱ)离子。以上3 种金属离子全部通过非共价键与肽链结合。
2.3 基因结构与基本基序分析
基于CqSODs 的cDNA和相应的DNA序列分析基因结构(图1),所有CqSOD基因均含有4~7个内含子,其中,4 个CqFe-SODs、1 个CqMn-SODs(CqSOD11)和1个CqCu/Zn-SODs(CqSOD11)均包含7个内含子,CqSOD06 包含最小数量的内含子(4个)。此外,在8个基因的两端发现了UTR非编码区。值得注意的是,CqSOD08 和CqSOD10,CqSOD07 和CqSOD09 在基因结构上表现出高度相似性,它们的cDNA和DNA序列高度一致,表明这2对Fe-SOD 基因很可能是重复的。
利用MEME 软件分析保守基序,共鉴定出10个保守基序(图2),在3个亚族间可以清晰观察到motif 的差异。其中,与Cu-Zn 结构域(PF00080)相关的motif 1 在6 个Cu/Zn-SOD 中都能观察到,motif 5 与motif 6 仅存在于Cu/Zn-SOD蛋白中。所有Fe-SOD和Mn-SOD都包含motif 2、motif 3与motif 4,这些基序涉及C端Fe/Mn SOD结构域(PF02777)。motif 2包含Mn-SODs和Fe-SODs的保守金属结合域“DVWEHAYY”,motif 7、motif 8与motif 9仅存在于Fe-SODs中。
2.4 藜麦SOD 蛋白系统发育分析
为更准确地预测CqSODs 的功能,进一步探讨SOD蛋白在不同物种间的进化关系,对拟南芥、水稻、小麦、陆地棉等5个物种的SOD蛋白氨基酸序列进行了系统发育分析(图3)。系统发育树显示,SOD蛋白可分为3个亚族,其中第Ⅰ亚族均为Cu/Zn-SODs,第Ⅱ亚族均为Fe-SODs,第Ⅲ亚族均为Mn-SODs。此外,在藜麦中存在6对旁系同源基因(CqSOD01/CqSOD02、CqSOD03/CqSOD04、CqSOD05/CqSOD06、CqSOD07/CqSOD09、CqSOD08/CqSOD10、CqSOD11/CqSOD12)。上述结果表明,SOD 基因在整个进化过程中可能发生了基因丢失或复制事件,特定SOD 基因成员的获得和损失导致其基因功能的差异。
2.5 CqSOD 蛋白互作分析
为探索藜麦SOD 蛋白及其调控功能之间的关系,使用STRING软件构建了拟南芥和藜麦之间的SOD家族蛋白互作网络(图4)。根据预测,CqSOD 蛋白出现在已知的拟南芥SOD蛋白互作网络中。CqSOD 和AtSOD 蛋白之间存在密切关系,暗示可以通过已知AtSOD 基因的功能预测CqSOD 基因的未知功能。结果显示,藜麦CqSOD12与11个CqSOD蛋白及4个CqCAT蛋白相互作用,而拟南芥中SOD 与CAT 基因在应对多种生物胁迫及非生物胁迫中起着关键作用,推测藜麦中SOD 基因可能具有类似的功能。
2.6 CqSOD 基因启动子元件分析
为进一步确定CqSOD 在各种胁迫下的作用,分析CqSOD 基因启动子序列中的顺式元件(表5),CqSOD 基因家族的启动子区域富含非生物胁迫顺式调节元件和植物激素反应元件。顺式元件分为激素反应性、应激反应性和组织特异表达3个亚族。激素响应方面,参与ABA反应的顺式调节元件(ABRE)广泛存在于所有的CqFe-SODs 与CqMn-SODs 的启动子区,参与MeJA反应的顺式调节元件(TGACG 基序)在大多数CqCu/Zn-SODs、CqFe-SODs 与CqMn-SODs 的启动子区均有发现。而参与SA反应的顺式调节元件(CCATCTTTTT基序)在所有的CqFe-SODs 的启动子区均存在。在压力响应方面,与低温胁迫相关的响应元件存在于CqSOD02~CqSOD04、CqSOD06, CqSOD10 与CqSOD11 的启动子区,CqSOD02、CqSOD07、CqSOD11 与CqSOD12 的启动子具有1个与干旱诱导相关的MYB结合位点。CqSOD06~CqSOD08、CqSOD11 与CqSOD12 的启动子区具有与应急反应相关的元件。在组织特异表达方面,ARE元件参与厌氧反应,存在于所有的CqSOD 中(除CqSOD07),且在CqSOD03 的启动子区具有7 个ARE元件。CAT-box元件是与分生组织表达有关的顺式作用调控元件,存在于CqSOD05 与CqSOD11 的启动子区。CqSOD08 的启动子具有MYB结合位点,参与黄酮类生物合成基因调控,暗示CqSOD08 可能与黄酮类生物合成有关。O2-site是玉米醇溶蛋白代谢调控顺式调控作用元件,以单拷贝的形式存在于CqSOD01、CqSOD02、CqSOD05、CqSOD08 与CqSOD12 的启动子区。
2.7 CqSODs 在混合盐碱胁迫下的qRT-PCR分析
为了解藜麦SOD 基因的作用,使用qRT-PCR分析SOD 基因在混合盐碱胁迫下的表达模式(图5)。与CK 相比,CqSOD 基因在不同处理下的表达水平存在显著差异,其表达模式具有复杂性。然而总体上可以观察到3个亚族基因间(6个Cu/Zn-SOD、4个Fe-SOD 和2个Mn-SOD 基因)具有相似的表达模式。本研究发现,不同比例的混合盐碱处理均显著诱导了SOD 基因的表达,对于不同组合的盐碱处理而言,E处理下11个CqSOD 基因(除CqSOD12)的表达量均达到最大值,A处理下12个CqSOD 基因的表达量均较CK增加最少。而在外源施加150 μmol·L?1 的SNP 后,6 个Cu/Zn-SOD (CqSOD01~CqSOD06) 与4 个Fe-SOD(CqSOD07~CqSOD10)基因的表达量在A+处理下达到最大值。
3 讨论
长期以来,逆境胁迫不断制约着作物的生产。超氧化物歧化酶(SOD)在植物应对盐、干旱和金属毒性等不同胁迫中发挥着重要作用,SOD 基因家族广泛分布于多种植物中,如拟南芥、谷子[22]、水稻[23]、高粱[24]、棉花、番茄[13]、杨树[15]和日本落叶松[25]等。本研究对藜麦SOD 家族基因进行了系统和全面的全基因组进化分析,结果显示,SOD 参与了大量氧化过程,是保护植物免受ROS侵害的关键酶,是所有抗氧化酶中的核心酶,是最早参与清除活性氧的物质之一。当植物应答逆境胁迫时,SODs 可清除生物在胁迫下产生的过量活性氧,从而有效减少氧化应激。SOD 基因在植物适应非生物胁迫中的重要作用已被大量研究所证实,例如植物的异质表达赋予了它们对多种非生物胁迫的耐受性[26]。本研究在藜麦基因组中鉴定了12个CqSOD基因,SOD蛋白的氨基酸分子量在13.41~32.43 kD,呈现出显著的变化。此外,进化枝之间的差异可能与外显子/内含子的不同功能和多样性以及保守的基序结构有关。根据结构域和基序将其分为3 种类型(6 个Cu/Zn-SOD、4 个Fe-SOD 和2 个Mn-SOD),其成员数量高于拟南芥(7 个)、蒺藜(7个)、番茄(8个)、水稻(8个)、谷子(8个)和黄瓜(9 个),与葡萄(10 个)、香蕉(13 个)相近[11]。然而,Cu/ZnSOD、Fe-SOD 和Mn-SOD 的基因数量在不同物种中出现差异,例如谷子和番茄中有4个Cu/Zn-SODs、3个Fe-SODs 和1个Mn-SOD,而本研究发现了6 个Cu/Zn-SODs、4 个Fe-SODs 和2 个Mn-SODs。SOD 家族成员数量的差异可能是由不同物种的基因组大小不同或基因重复造成的。亚细胞定位进一步研究了SOD 之间的多样性,12个SOD 基因分别定位于细胞质(50%)、微体(33%)、线粒体(17%),CqSOD基因的定位需要进一步研究证实。
研究表明,藻类和苔藓植物中仅有Fe-SOD 和Mn-SOD,而不存在Cu/Zn-SOD,Cu/Zn-SOD 仅出现于高等植物中,这意味着Fe-SOD 和Mn-SOD 进化时间较早[27]。随着地球环境的不断变化,植物面临的逆境挑战变得更加复杂。Fe-SOD 和Mn-SOD 不足以应答复杂的逆境,因此进化出了功能更复杂的Cu/Zn-SOD 以抵御不利的外部环境条件,保证植物的正常生长发育[28]。
基因结构分析表明,12个CqSOD 基因内含子数量不一。已有研究表明,植物SOD 基因的内含子模式高度保守,并且在大多数细胞质和叶绿体SOD 中存在7 个内含子[29]。同样,本研究中6 个CqSOD 基因包含7个内含子。SOD 基因结构的差异可能归因于外显子/内含子的插入/缺失机制[30]。值得注意的是,CqSOD08 和CqSOD10,CqSOD07和CqSOD09 表现出相似的内含子/外显子组织模式,它们编码的蛋白质高度一致,并在系统发育树中聚集在一起。但是,它们启动子中的顺式元件不同,表明它们可以对不同的逆境条件作出反应。内含子和外显子变异在不同基因的进化中起主要作用。内含子/外显子和基序结构的变化表明,不同物种之间SOD 基因存在高度的复杂性。
通过对SOD 基因启动子中的顺式元件分析发现,CqSOD 基因存在与非生物胁迫和激素反应相关的2种主要类型的顺式元件以及与发育过程和组织特异性表达相关的顺式元件。在SOD 基因启动子中鉴定出一系列与非生物胁迫反应相关的顺式元件,如MBS、LTR、富含TC 的重复序列,它们可能在多种胁迫下调节基因表达。拟南芥、香蕉、水稻、番茄、杨树、棉花和其他不同植物中的大多数SOD 基因可以被诱导响应各种非生物胁迫,如热、冷、干旱和盐度[13,27],与本研究结果一致。
非生物胁迫导致的过量ROS 会对藜麦幼苗及产量构成威胁。SOD在各种非生物胁迫引起的植物ROS清除响应中发挥着积极作用[31]。藜麦对混合盐分的具体响应尚不清楚,qRT-PCR使了解CqSOD 基因在藜麦应答混合盐碱胁迫中的功能成为可能。基于对CqSOD 基因启动子的顺式元件的评价,发现主要存在于光、非生物胁迫和激素反应相关的3种主要顺式元件类型。CqSOD 启动子中的顺式元件包括富含TC的基序、LTR基序、MBS 基序、ARE 基序和ABRE 基序,为参与非生物胁迫反应提供了可能。这些基序参与了植物对非生物胁迫的响应,例如香蕉、番茄、芸苔属植物、烟草和小米[32-34]。本研究中,12个CqSOD 基因在混合盐碱胁迫下的表达水平发生了显著变化,同时对混合盐碱处理的幼苗外源施加SNP也显著诱导了CqSOD 基因的表达,表明藜麦SOD 基因在盐碱胁迫反应中发挥着重要的调控作用。上述结果表明,CqSOD 在盐胁迫期间对ROS的解毒具有积极作用,而SNP则不同程度提高了在混合盐碱胁迫期间CqSOD 基因对ROS的解毒能力。
参 考 文 献
[1] MILLER A. Superoxide dismutases: ancient enzymes and newinsights [J]. FEBS Lett., 2012, 586(5): 585-595.
[2] MITTLER R. ROS are good [J]. Trends Plant Sci., 2017, 22(1):11-19.
[3] BAFANA A, DUTT S, KUMAR S, et al .. Superoxidedismutase: an industrial perspective [J]. Crit. Rev. Biotechnol.,2011, 31(1):65-76.
[4] ZELKO I N, MARIANI T J, FOLZ R J. Superoxide dismutasemultigene family: a comparison of the CuZn-SOD (SOD1), Mn-SOD (SOD2), and EC-SOD (SOD3) gene structures, evolution,and expression [J]. Free Radic. Biol. Med., 2002, 33(3):337-349.
[5] TEPPERMAN J M, DUNSMUIR P. Transformed plants withelevated levels of chloroplastic SOD are not more resistant tosuperoxide toxicity [J]. Plant Mol. Biol., 1990, 14(4): 501-511.
[6] SU W, RAZA A, GAO A, et al .. Genome-wide analysis andexpression profile of superoxide dismutase (SOD) gene familyin rapeseed (Brassica napus L.) under different hormones andabiotic stress conditions [J/OL]. Antioxidants (Basel), 2021, 10(8): 1182 [2022-06-03]. https://doi.org/10.3390/antiox10081182.
[7] ABREU I A, CABELLI D E. Superoxide dismutases-a reviewof the metal-associated mechanistic variations [J]. Biochim.Biophys. Acta, 2010, 1804(2): 263-274.
[8] SONG J, ZENG L, CHEN R, et al .. In silico identification andexpression analysis of superoxide dismutase (SOD) gene familyin Medicago truncatula [J/OL]. 3 Biotech., 2018, 8(8): 348[2022-06-03]. https://doi.org/10.1007/s13205-018-1373-1.
[9] 魏婧, 徐畅, 李可欣, 等. 超氧化物歧化酶的研究进展与植物抗逆性[J].植物生理学报, 2020, 56(12): 2571-2584.
WEI J, XU C, LI K X, et al .. Progress on superoxide dismutaseand plant stress resistance [J]. Plant Physiol. J., 2020, 56(12):2571-2584.
[10] ASENSIO A C, GIL-MONREAL M, PIRES L, et al .. Two Fesuperoxidedismutase families respond differently to stress andsenescence in legumes [J]. J. Plant Physiol., 2012, 169(13):1253-1260.
[11] HAN L M, HUA W P, CAO X Y, et al .. Genome-wideidentification and expression analysis of the superoxidedismutase (SOD) gene family in Salvia miltiorrhiza [J/OL].Gene, 2020, 742:144603[2022-06-03]. https://doi.org/10.1016/j.gene.2020.144603.
[12] ZHANG X, ZHANG L T, CHEN Y Y, et al .. Genome-wideidentification of the SOD gene family and expression analysisunder drought and salt stress in barley [J]. Plant GrowthRegul., 2021, 94(1): 49-60.
[13] FENG K, YU J H, CHENG Y, et al .. The SOD gene family intomato: identification, phylogenetic relationships, and expressionpatterns [J/OL]. Front. Plant Sci., 2016(7): 1279 [2022-06-03].https://doi.org/10.3389/fpls.2016.01279.
[14] ZHOU Y, HU L F, WU H, et al .. Genome-wide identificationand transcriptional expression analysis of cucumber superoxide dismutase (SOD) family in response to variousabiotic stresses [J/OL]. Int. J. Genomics, 2017, 2017: 7243973[2022-06-03]. https://doi.org/10.1155/2017/7243973.
[15] MOLINA-RUEDA J J, TSAI C J, KIRBY E G. The Populussuperoxide dismutase gene family and its responses to droughtstress in transgenic poplar overexpressing a pine cytosolicglutamine synthetase (GS1a) [J/OL]. PLoS One, 2013, 8(2): e56421[2022-06-03]. https://doi.org/10.1371/journal.pone.0056421.
[16] ZURITA-SILVA A, FUENTES F, ZAMORA P, et al .. Breedingquinoa (Chenopodium quinoa Willd.): potential and perspectives [J].Mol. Breed., 2014, 34(1): 13-30.
[17] 赵颖, 魏小红, 李桃桃. 外源NO对混合盐碱胁迫下藜麦种子萌发和幼苗生长的影响[J]. 草业学报, 2020, 29(4):92-101.
ZHAO Y, WEI X H, LI T T. Efferts of exogenous nitric oxideon seed Emination and seedling growth of Chenopodium quinoaunder complex saline-alkali stress [J]. Acta Pratac. Sin., 2020,29(4): 92-101.
[18] 刘文瑜, 杨发荣, 黄杰, 等. NaCl 胁迫对藜麦幼苗生长和抗氧化酶活性的影响[J]. 西北植物学报, 2017, 37(9): 1797-1804.
LIU W Y, YANG F R, HUANG J, et al .. Response of seedlinggrowth and the activities of antioxidant enzymes of Chenopodiumquinoa to salt stress [J]. Acta Bot. Bor-Occid. Sin., 2017, 37(9):1797-1804.
[19] RUIZ K B, BIONDI S, MART?NEZ E A, et al .. Quinoa-a modelcrop for understanding salt-tolerance mechanisms in halophytes [J].Plant Biosyst., 2016, 150(2): 357-371.
[20] 李美丽, 宿俊吉, 杨永林, 等. 陆地棉COI 家族基因鉴定及在干旱和盐胁迫下的表达分析[J].中国农业科技导报, 2022,24(4):63-74.
LI M L, SU J J, YANG Y L, et al .. Identification of COl familygenes and their expression in Gossypium hirsutum L. underdrought and salt stress [J]. J. Agric. Sci. Technol., 2022, 24(4):63-74.
[21] LIVAK K J, SCHMITTGEN T D. Analysis of relative geneexpression data using Real-time quantitative PCR [J]. Methods,2002, 25(4): 402-408.
[22] WANG T, SONG H, ZHANG B H, et al .. Genome-wideidentification, characterization, and expression analysis ofsuperoxide dismutase (SOD) genes in foxtail millet (Setariaitalica L.) [J/OL]. 3 Biotech., 2018, 8(12): 486 [2022-06-03].https://doi.org/10.1007/s13205-018-1502-x.
[23] DEHURY B, SARMA K, SARMAH R, et al .. In silico analysesof superoxide dismutases (SODs) of rice (Oryza sativa L.) [J]. J.Plant Biochem. Biotechnol., 2013, 22(1): 150-156.
[24] GOSAVI G U, JADHAV A S, KALE A A, et al .. Effect of heatstress on proline, chlorophyll content, heat shock proteins andantioxidant enzyme activity in sorghum (Sorghum bicolor) atseedlings stage [J]. Indian J. Biotechnol., 2014, 13(13):356-363.
[25] XMHA B, QXC B, QI Y B, et al .. Genome-wide analysis ofsuperoxide dismutase genes in Larix kaempferi [J]. Gene, 2019,686: 29-36.
[26] TANG Y H, BAO X X, ZHI Y L, et al .. Overexpression of aMYB family gene, OsMYB6, increases drought and salinity stresstolerance in transgenic rice [J/OL]. Front. Plant Sci., 2019(10):168 [2022-06-03]. https://doi.org/10.3389/fpls.2019.00168.
[27] LIN Y L, LAI Z X. Superoxide dismutase multigene family inlongan somatic embryos: a comparison of CuZn-SOD, Fe-SOD,and Mn-SOD gene structure, splicing, phylogeny, andexpression [J]. Mol. Breeding, 2013, 32(3): 595-615.
[28] WANG W, ZHANG X, DENG F, et al .. Genome-widecharacterization and expression analyses of superoxidedismutase (SOD) genes in Gossypium hirsutum [J/OL]. BMCGenomics, 2017, 18(1): 376 [2022-06-03]. https://doi. org/10.1186/s12864-017-3768-5.
[29] FINK R C, SCANDALIOS J G. Molecular evolution andstructure-function relationships of the superoxide dismutasegene families in angiosperms and their relationship to othereukaryotic and prokaryotic superoxide dismutases [J]. Arch.Biochem. Biophys., 2002, 399(1): 19-36.
[30] XU G X, GUO C C, SHAN H Y, et al .. Divergence of duplicategenes in exon-intron structure [J]. Proc. Natl. Acad. Sci. USA,2012, 109(4): 1187-1192.
[31] GILL S S, TUTEJA N. Reactive oxygen species and antioxidantmachinery in abiotic stress tolerance in crop plants [J]. PlantPhysiol. Biochem., 2010, 48(12): 909-930.
[32] FENG X, LAI Z X ,LIN Y L, et al .. Genome-wide identificationand characterization of the superoxide dismutase gene familyin Musa acuminata cv.Tianbaojiao (AAA group)[J/OL]. BCMGenomics, 2015, 16:823 [2022-06-03]. https://doi.org/10.1186/s12864-015-2046-7.
[33] HU X X, HAO C Y, CHENG Z M, et al .. Genome-wideidentification, characterization, and expression analysis of thegrapevine superoxide dismutase (SOD) family [J/OL]. Int. J.Genomics, 2019: 7350414 [2022-06-03]. https://doi. org/10.1155/2019/7350414.
[34] PILON M, RAVET K, TAPKEN W. The biogenesis andphysiological function of chloroplast superoxide dismutases [J].Biochim. Biophys. Acta, 2011, 1807(8): 989-998.
(责任编辑:胡立霞)

