基于SNP标记的小麦品种遗传相似度及其检测准确度分析
许乃银 金石桥 晋 芳 刘丽华 徐剑文 刘丰泽 任雪贞 孙 全 许 栩 庞斌双,*
基于SNP标记的小麦品种遗传相似度及其检测准确度分析
许乃银1金石桥2,*晋 芳2刘丽华3徐剑文1刘丰泽2任雪贞2孙 全2许 栩1庞斌双3,*
1江苏省农业科学院经济作物研究所, 江苏南京 210014;2全国农业技术推广服务中心, 北京 100125;3北京市农林科学院杂交小麦研究所, 北京 100097
遗传相似度检测的准确度估计是对SNP标记法在农作物品种检测体系中应用的必要补充和完善。本研究基于2021年小麦品种SNP标记法跨实验室协同验证实验数据, 分析了该方法的检测准确度及在品种间的遗传相似度。分析结果表明: (1) 10个实验室对55组小麦品种组合的标记位点相似度检测的总体准确度约为98%。(2) GGE双标图的品种遗传关系功能图显示, 7组小麦品种的组内遗传相似度在95%以上, 其余组合的遗传相似度较低。(3) 依据GGE双标图的“正确度-精确度”功能图和“准确度排序”功能图, 发现洛旱7号/洛旱11等品种组合的相似度检测准确度较高, 晋麦47/临抗11的检测准确度一般, 而济麦22/婴泊700的检测准确度较差。(4) 10个实验室的检测准确度存在显著差异, 其中2个实验室检测的正确度、精确度和准确度表现显著差于其余实验室。(5) 各实验室检测正确度的容许误差分布于1.3%~1.9%之间, 平均为1.5%; 准确度的容许误差分布于1.5%~2.0%之间, 平均为1.7%。其中, Lab2和Lab3的检测正确度和准确度的容许误差显著差于其余实验室。本研究构建了SNP标记法对品种相似性检测的准确度统计模型, 分析了品种组合和实验室的检测准确度及其容许误差, 采用GGE双标图方法对检测正确度、精确度和准确度进行可视化分析, 验证了各实验室对品种位点相似性检测的准确度和可靠性, 为SNP标记法在农作物品种遗传相似性检测中的准确度评价提供了理论支持和应用范例。
小麦(L.); GGE双标图; SNP标记; 遗传相似度; 位点相似度; 准确度
随着农作物种业市场化程度的不断提高, 品种创新对振兴国家种业、提高种业核心竞争力和实现种业高质量发展变得越来越重要[1]。保护品种创新、解决品种同质化以及打击种子假冒套牌侵权的核心是快速、高效和科学地进行品种真实性验证和品种身份鉴定。近年来, 单核苷酸多态性(single nucleotide polymorphism, SNP)标记检测技术在农作物种子的真实性检测、纯度检测和DNA指纹数据库构建等方面被广泛研究和应用, 在农作物种子质量检测中发挥着越来越重要的作用[1-2]。随着小麦(L.)[3]、玉米(L.)[4]和水稻(L.)[5]等主要农作物《品种真实性鉴定 SNP标记法》行业标准的颁布和实施, SNP标记法已经逐步成为我国品种管理、种子市场管理和品种权纠纷处理等领域最重要的新一代分子检测技术。SNP标记法主要通过比对品种间位点相似度或差异位点数确定品种间遗传相似性, 并依据预设的差异位点数或差异位点比例阈值判定品种间遗传关系和品种权属性[1,6]。理论上, SNP标记法的检测效率和可靠性主要决定于标记的数量及其代表性, 不受外界环境条件的影响, 不同实验室、不同平台对同样的检测样品应获得相同的检测结果[1]。然而, 每次检测所采用的标记数量有限, 相对于全基因组水平庞大的分子标记数量库仅为一小部分采样, 不可避免引入抽样误差。实际操作中, 无法有效控制所有可能影响分子标记检测结果的潜在因素, 在每次检测程序中都可能出现随机误差。因而在对检测结果进行分析和解释过程中, 应当考虑和阐明这种不确定性。2023年国际种子检验协会(international seed testing association, ISTA)报道了24个豌豆品种在8个实验室中的简单序列重复(simple sequence repeats, SSR)标记检测验证试验, 发现实验室间检测结果的一致性约为90%[7]。检测准确度包括正确度与精确度, 正确度指测试结果与真值或参照值之间的符合度, 精确度指测试结果之间的一致程度[8]。在多环境品种试验数据分析中广泛应用的基因型主效应加基因型与环境互作效应模型(genotype plus genotype by environment interaction model, GGE)双标图[9-14]可以通过视图直观表达正确度、精确度和准确度的关系。GGE双标图技术与现代育种计划中的基因组选择(genome-wide selection, GS)模型相结合, 还可以提高基因组育种的选择效率和预测精度[15]。关于如何基于SNP标记检测数据计算检测准确度, 以及如何利用GGE双标图技术对准确度进行可视化分析, 文献中尚未见报道。本研究以2021年全国农业技术推广服务中心(简称全国农技中心)组织实施的主要农作物品种SNP标记法检测标准的跨实验室协同验证试验中的小麦品种样本检测实验数据为例, 构建了SNP标记法检测准确度统计模型, 并采用GGE双标图方法对检测准确度进行可视化分析和展示, 验证了各实验室对小麦品种位点相似性检测的准确度和可靠性, 为SNP标记法检测农作物品种遗传相似性的准确度评价提供理论支持和应用范例。
1 材料与方法
1.1 数据来源
数据来源于2021年全国农技中心组织开展的小麦品种真实性鉴定的SNP标记法准确度验证实验的SNP标记数据集。实验由《小麦品种真实性鉴定 SNP标记法》行业标准(简称“小麦SNP标准”)[3]制定单位北京市农林科学院杂交小麦研究所在国家农作物品种标准样品库中抽取11个品种的种子样品, 依据标准的要求、位点及引物组合、程序和方法检测出各个种子样品的96个SNP位点的基因型信息, 作为各实验室SNP位点检测结果的标准参照值(reference value)[16]。全国农技中心将各抽检品种的0.5 g种子粉末统一分配给相关省市有代表性的种子质量检验机构, 采用与标准制定单位相同的样品、SNP位点及引物组合, 进行重复性实验, 以验证标准规定的方法及SNP位点组合在多单位、多平台技术条件下的可重复性、可行性、实用性及各实验室检测的准确性和可靠性。小麦96个SNP位点及竞争性等位基因特异性PCR (kompetitive allele- specific PCR, KASP)分型引物信息详见小麦SNP标准[3]。承担小麦SNP标准验证的10个实验室和11个抽样小麦品种信息详见表1。
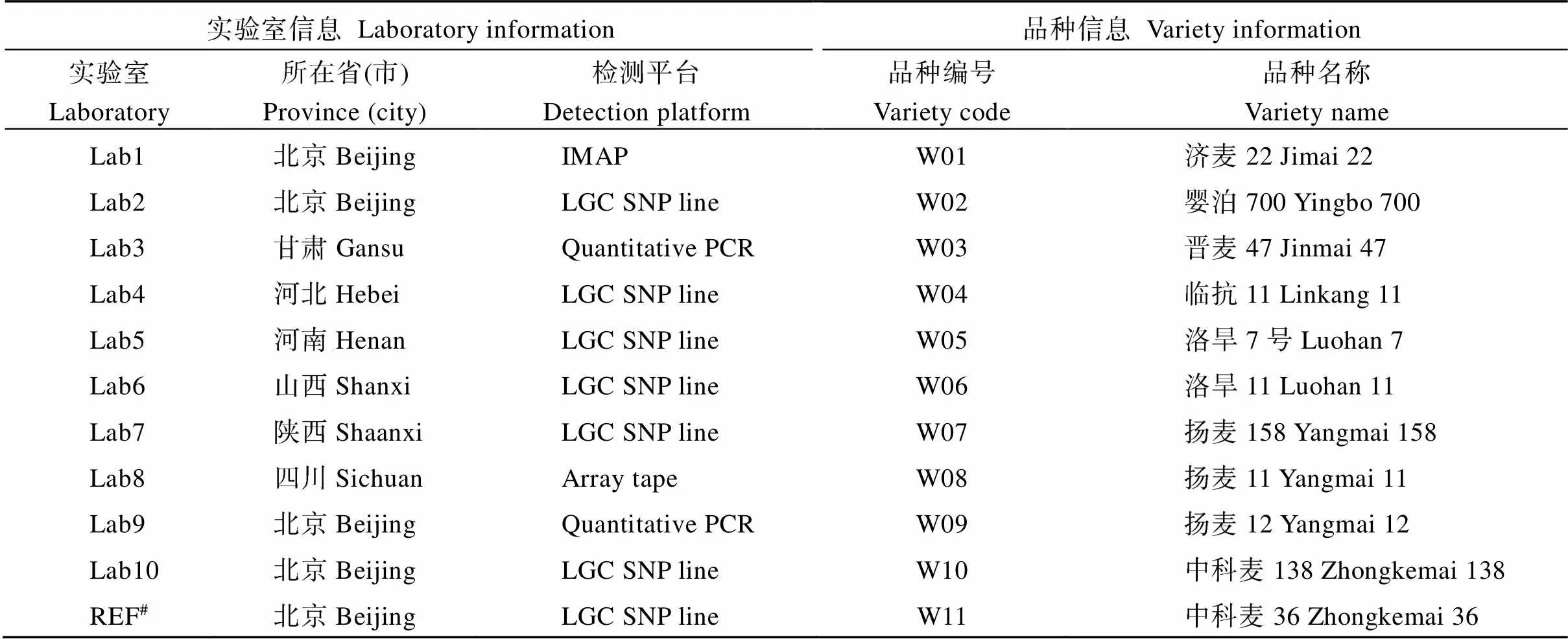
表1 基于SNP标记鉴定小麦品种遗传相似性的实验室和抽样品种信息表
REF#为提供标准检测结果作为参照的制标单位。
REF#is a standard unit that provides standard test results as a reference.
1.2 统计分析方法
1.2.1 品种间位点相似度计算 采用“小麦品种真实性鉴定SNP标记法”行业标准[3]推荐的SNP位点相似度(locus similarity, LS)为品种间遗传相似度参数[6]。基于96个SNP位点的基因型对各实验室检测的品种间相互比对, 基因型相同用“1”表示, 不同用“0”表示, 数值总和即2个样品间的相同位点数, 据此计算品种间的位点相似度。任意品种间的平均位点相似度用该品种组合在各试验室的位点相似度的平均值表示。基于标准参照值或各实验室平均值计算的品种间位点相似度矩阵可作为品种位点相似度的参照值。计算公式如下:
实验室内品种间位点相似度,
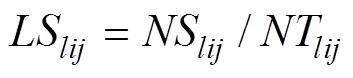
基于参照品种间位点相似度,
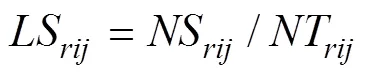
式中,LS、NS和NT分别表示实验室内品种和的位点相似度、相同位点数和比对总位点数;LS、NS和NT分别表示基于标准参照(r)的品种和的位点相似度、相同位点数和总位点数。
1.2.2 品种间位点相似度的检测准确度计算 检测准确度包括正确度与精确度。正确度指检测结果与参照值之间的一致程度, 精确度指检测结果之间的一致程度[8,16]。某实验室检测的任意2个品种间的位点相似度, 与基于标准参照值得出的相应品种组合的位点相似度相比较的一致程度, 就是该品种组合相似度在该实验室检测的正确度。任意2个品种位点相似度在各实验室检测正确度的平均值表示该品种组合的平均检测正确度, 而在实验室内不同测试次数间或不同实验室间测试结果的一致性为精确度指标。实验室内所有品种组合位点相似度检测正确度的平均值表示该实验室的总体检测正确度, 各品种组合位点相似度检测正确度间的平均标准差表示该实验室的检测精确度。准确度为正确度和精确度平方根[8]。计算公式如下:
实验室内2个品种位点相似度的正确度,
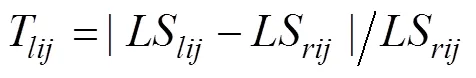
2个品种位点相似度的平均正确度,
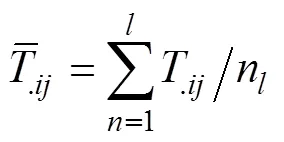
实验室对品种位点相似度检测的平均正确度,

2个品种位点相似度的准确度,

实验室对品种位点相似度检测的准确度,
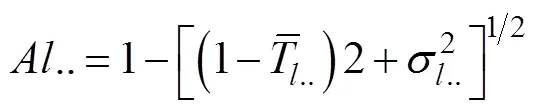

1.2.3 基于二项分布的品种遗传相似度检测准确度的容许误差估计 各实验室检测的品种间基于96个SNP位点的基因型相互比对, 或与SNP标准参照值比对, 结果以“1, 0”表示, 从而形成具有二项分布(Binary distribution)特征的数据集[17]。由于实验室检测的不确定度为小概率事件, 故采用Wilson算法[18]计算准确度的容许误差。计算公式如下:
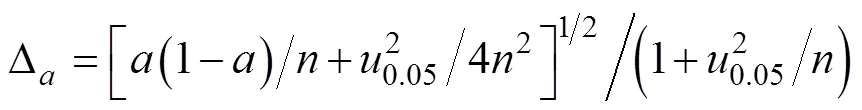
式中, Δ、、分别表示准确度容许误差、样本容量、准确度,0.05表示5%显著水平的临界值。
1.2.4 基于GGE双标图的品种遗传相似度和检测准确度可视化分析方法 依据各实验室SNP检测结果的相互比对分析形成的品种间位点相似度矩阵,借助GGE双标图“性状间关系”功能图[13,19]构建的“品种遗传关系”功能图, 对SNP检测结果中的品种间遗传相关模式进行可视化分析。SNP检测结果的品种间遗传相关程度通过“品种遗传关系”功能图中各品种向量间的夹角大小反映, 夹角越小, 则相关性越强[13,19]。基于各实验室检测的品种位点相似度与基于参照值相比较得出“实验室-品种组合”的正确度矩阵, 利用GGE双标图“均值-稳定性”功能图[20]构建SNP检测结果的“正确度-精确度”功能图, 其中,平均环境轴(average environment axis, AEA)指向检测正确度大的方向; 通过原点垂直于AEA轴的纵轴(average environment coordinate, AEC)指向检测精确度差的方向, 越接近于AEA轴, 则精确度越好。在“正确度-精确度”功能图基础上, 以标准参照或理想品种坐标为圆心, 作同心圆构建“准确度排序”功能图[20], 各品种组合或实验室图标到同心圆圆心的距离表示检测准确度, 距离越小, 则检测准确度越好。采用统计软件GGEbiplot[12](http://www.ggebiplot. com/)进行双标图分析。
2 结果与分析
2.1 小麦品种间遗传相似度及其检测准确度分析
11个抽检小麦品种在10个实验室检测, 基于96个SNP位点进行基因型两两比对, 品种间平均位点相似度及其检测准确度分析结果表明: (1) 在55对品种组合比对中(表2), 济麦22/婴泊700 (编号W01/02)、晋麦47/临抗11 (W03/04)、洛旱7号/洛旱11 (W05/06)、扬麦158/扬麦11 (W07/08)、扬麦158/扬麦12 (W07/09)、扬麦11/扬麦12 (W08/09)和中科麦138/中科麦36 (W10/11)等7对品种组合的位点相似度在95%以上, 其中扬麦158/扬麦12 (W07/09)的位点相似度在98%以上; 其余品种组合间的位点相似度较低, 介于42%~62%之间。(2) GGE双标图的“品种遗传关系”功能图(图1-a)直观表达了品种间的遗传相关模式, 11个抽检品种可划分为5个相关性强的品种组合, 即济麦22/婴泊700 (编号W01/02)、晋麦47/临抗11 (W03/04)、洛旱7号/洛旱11 (W05/06)、扬麦158/扬麦11/扬麦12 (W07/ 08/W09)和中科麦138/中科麦36 (W10/11)。(3) 55对品种组合间位点相似度检测的总体准确度约为98% (表2), 其中准确度在99%以上的组合只有洛旱7号/洛旱11 (W05/06)和中科麦138/中科麦36 (W10/11), 准确度在98%以上的组合数约占总比对组合数的45%, 准确度在97%以上的组合数约占总比对组合数的85%, 准确度在96%以上的组合数约占总比对组合数的96%, 其余组合的检测准确度均在95%以上。(4) 基于图1-a和表2分析的品种间遗传关系, 将遗传相似度最高的品种组合及其在各实验室检测结果在图1-b中直观表达, 对品种组合间的遗传相关性及检测误差可视化分析表明, 不同实验室对各品种组合的遗传相似度检测结果均不同程度地存在误差, 但相对于品种组合间关系, 误差较小, 表明实验室检测的准确度总体较高。(5) 图1表达的品种间遗传相关性相比于品种间位点相似度矩阵(表2)更加直观明确, 并可表达品种组合之间的相关性检测的误差模式。
2.2 小麦品种遗传相似度检测准确度的GGE双标图分析
各实验室检测的小麦品种间遗传相似度相对于标准参照值可算得相应的“实验室-品种组合”正确度矩阵, 利用GGE双标图可直观分析品种间遗传相似度检测的准确度。以上述7对位点相似度高的品种组合为例, 其“正确度-精确度”功能图(图2-a)和“准确度排序”功能图(图2-b)分析表明, (1) 图2-a表达了各品种组合间的位点相似度与标准参照值比较的检测正确度和精确度, 图中横坐标值与正确度正相关, 纵坐标的绝对值与精确度负相关, 单箭头的横轴指向正确度大的方向, 双箭头的纵轴指向精确度差的方向。各品种组合的位点相似度检测正确度由高到低依次排序为中科麦138/中科麦36 (W10/11)>洛旱7号/洛旱11 (W05/06)>扬麦158/扬麦11 (W07/08)>扬麦158/扬麦12 (W07/09)>扬麦11/扬麦12 (W08/09)>晋麦47/临抗11 (W03/04)>济麦22/婴泊700 (W01/02)。各品种组合的检测精确度由高到低依次排序为中科麦138/中科麦36 (W10/11)>洛旱7号/洛旱11 (W05/06)>扬麦11/扬麦12 (W08/09)>扬麦158/扬麦12 (W07/09)>扬麦158/扬麦11 (W07/08)>济麦22/婴泊700 (W01/02)>晋麦47/临抗11 (W03/04)。(2) 图2-b中各品种组合图标到同心圆圆心的欧氏距离表明各品种组合的准确度大小, 越接近圆心, 准确度越好。各品种组合的准确度由高到低依次排序为中科麦138/中科麦36 (W10/11)>洛旱7号/洛旱11 (W05/06)>扬麦158/扬麦12 (W07/09)> 扬麦158/扬麦11 (W07/08)>扬麦11/扬麦12 (W08/ 09)>晋麦47/临抗11 (W03/04)>济麦22/婴泊700 (W01/02)。
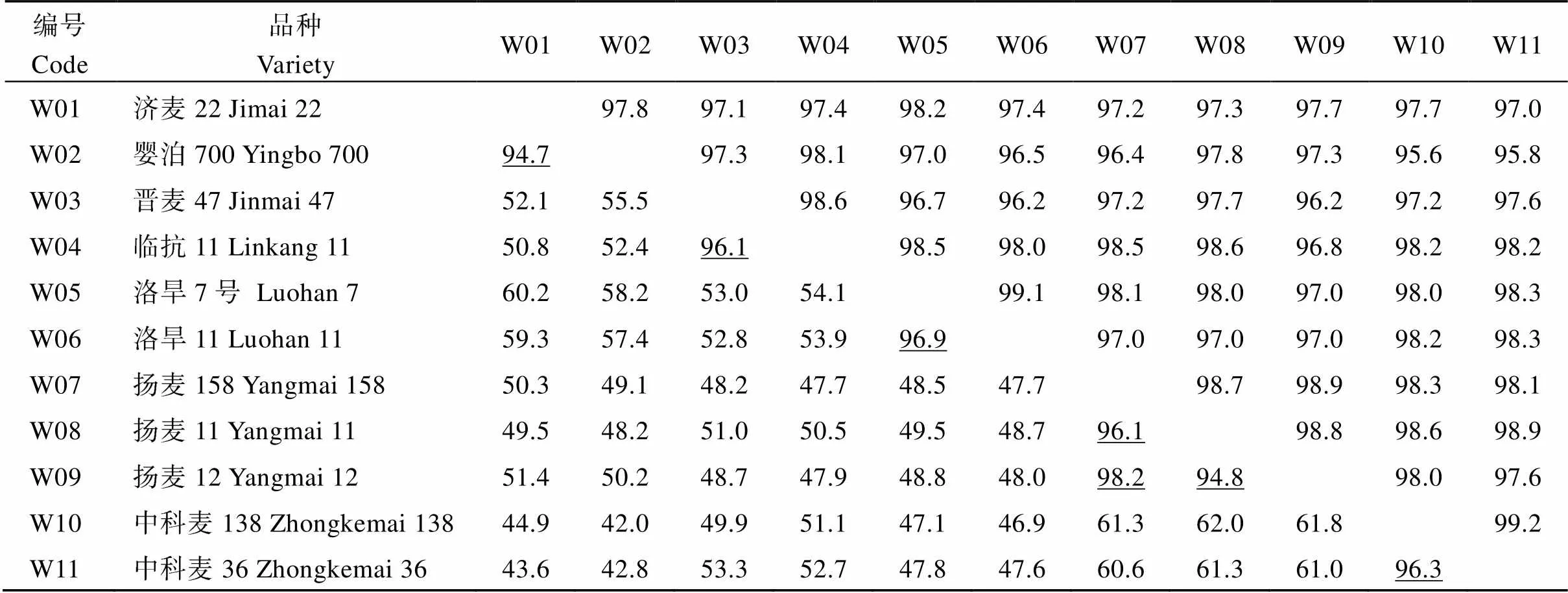
表2 小麦抽检品种SNP位点相似度与检测准确度平均值矩阵
左下三角为小麦品种间SNP位点相似度矩阵, 右上三角为相应的检测准确度矩阵。下画线表示位点相似度高的品种组合。
The lower left triangle is the SNP locus similarity matrix among wheat varieties, and the upper right triangle is the corresponding detection accuracy matrix. The data underlined indicate the variety combinations with high locus similarity values.

图1 基于小麦品种SNP位点相似度平均值的GGE双标图“品种遗传关系”功能图(a)和“品种组合关系+误差”功能图(b)
大写字母W后面的数字为品种编号, 具体品种名称详见表1。品种向量间的夹角表示品种间的遗传相关性, 夹角越小则相关性越强。图1-b中的蓝色小点表示各实验室检测的品种组合相似性图标, 其到品种组合图标的连线长短表示误差大小, 连线越长误差越大。
The uppercase W followed by the numbers represents variety codes and the specific name of the breeding is shown in Table 1. The angle between the cultivar vectors indicates the genetic correlation between the cultivars, and the smaller the angle, the stronger the correlation. The blue dot in Fig. 1-b represents the similarity mark of variety combination tested by each laboratory, and the length of the line from it to the variety combination mark represents the error size, and the longer the line, the larger the error.
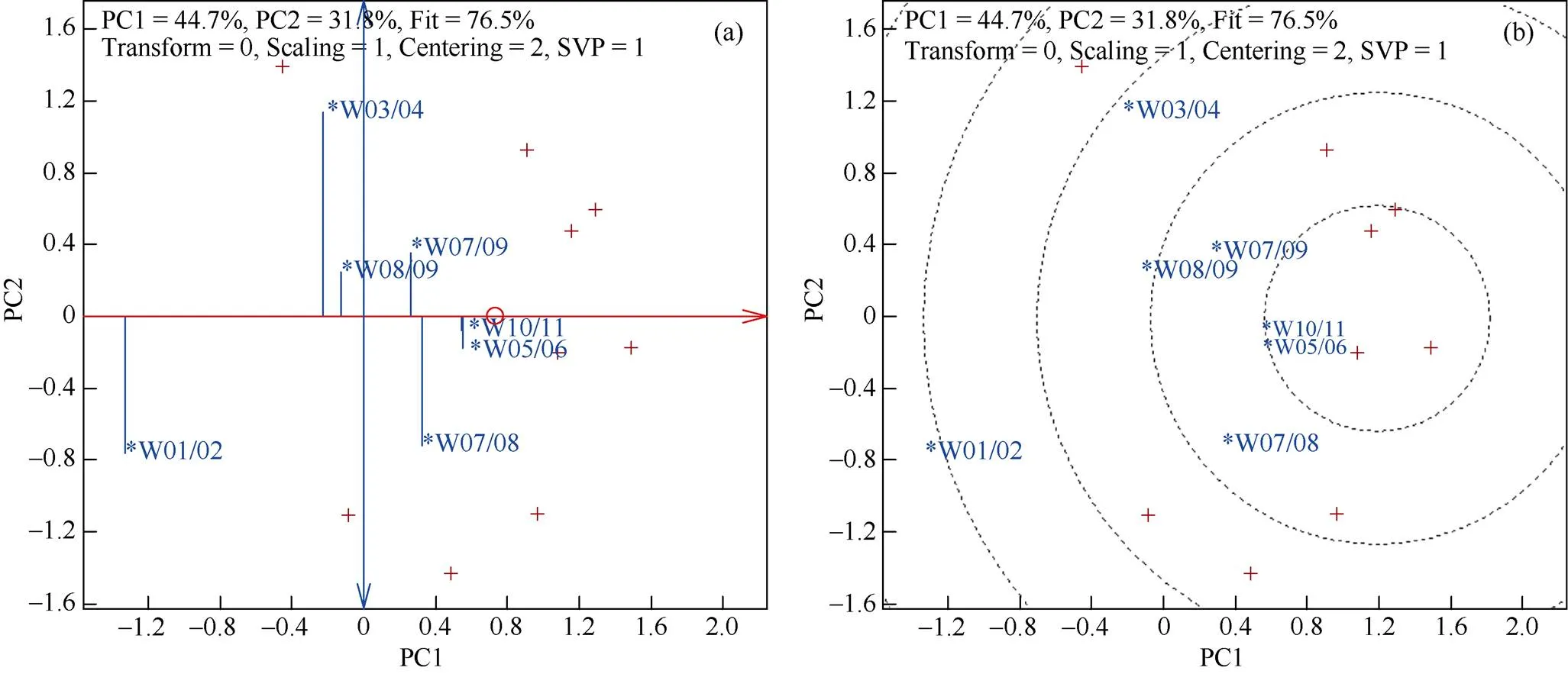
图2 基于标准参照的小麦品种遗传相似度检测准确度GGE双标图分析的“正确度-精确度”功能图(a)和“准确度排序”功能图(b)
大写字母W后面的数字为品种组合编号, 如W01/02表示品种组合W01和W02, 具体品种名称详见表1。PC1相当于品种位点相似性检测的正确度, PC2的绝对值相当于精确度。图2-a中, 单箭头的横轴指向正确度大的方向, 双箭头的纵轴指向精确度差的方向。图2-b中的同心圆圆心为理想品种组合坐标, 品种组合图标到圆心的欧氏距离表示准确度, 距离越小则准确度越好。“+”为实验室图标。
The uppercase W followed by numbers represents variety combination codes. For example, W01/02 indicates the wheat variety W01 compared to W02. See Table 1 for detail. PC1 corresponds to the trueness in variety locus similarity detection and the absolute value of PC2 corresponds to the precision. In Fig. 2-a, the single-arrowed horizontal axis points to the direction of higher accuracy, while the double-arrowed vertical axis points to the direction of lower precision. The origin of concentric circles in Figure 2-b is the ideal variety combination mark, and the Euclidean distance from the variety combination mark to the origin represents the detection accuracy. The smaller the distance, the better the accuracy. The plus sign “+” stand for variety comparison combination mark.
2.3 不同实验室对小麦品种遗传相似度检测准确度的GGE双标图分析
各实验室对55对小麦品种组合的SNP位点相似度检测结果与标准参照值相比较算得的“实验室-品种组合”正确度数据矩阵, 经GGE双标图分析绘制了实验室对品种组合间位点相似度检测的“正确度-精确度”功能图(图3-a)和“准确度排序”功能图(图3-b)。结果表明, (1)图3-a表达了各实验室的检测正确度和精确度, 各实验室检测的正确度由高到低依次排序为Lab8>Lab9>Lab1>Lab4>Lab7>Lab10>Lab5> Lab6>Lab2>Lab3, 其中Lab8、Lab9和Lab1的正确度最高, Lab4、Lab7、Lab10、Lab5和Lab6的正确度较高, Lab2和Lab3的正确度稍差。各实验室的检测精确度由高到低依次排序为Lab1>Lab8>Lab7> Lab10>Lab5> Lab9>Lab6>Lab4>Lab3>Lab2。其中, Lab1、Lab8、Lab7、Lab10、Lab5、Lab9、Lab6和Lab4的精确度好, 且相互间差异不大, 而Lab2和Lab3的精确度稍差。(2)图3-b中各实验室图标到标准参照图标(REF)的欧氏距离表示检测准确度, 各实验室准确度由高到低依次排序为Lab8>Lab1> Lab9>Lab4>Lab7>Lab10>Lab5>Lab6>Lab2>Lab3。其中, Lab8、Lab1和Lab9的准确度最高, Lab4、Lab7、Lab10、Lab5和Lab6的准确度较高, Lab2和Lab3的准确度稍差。
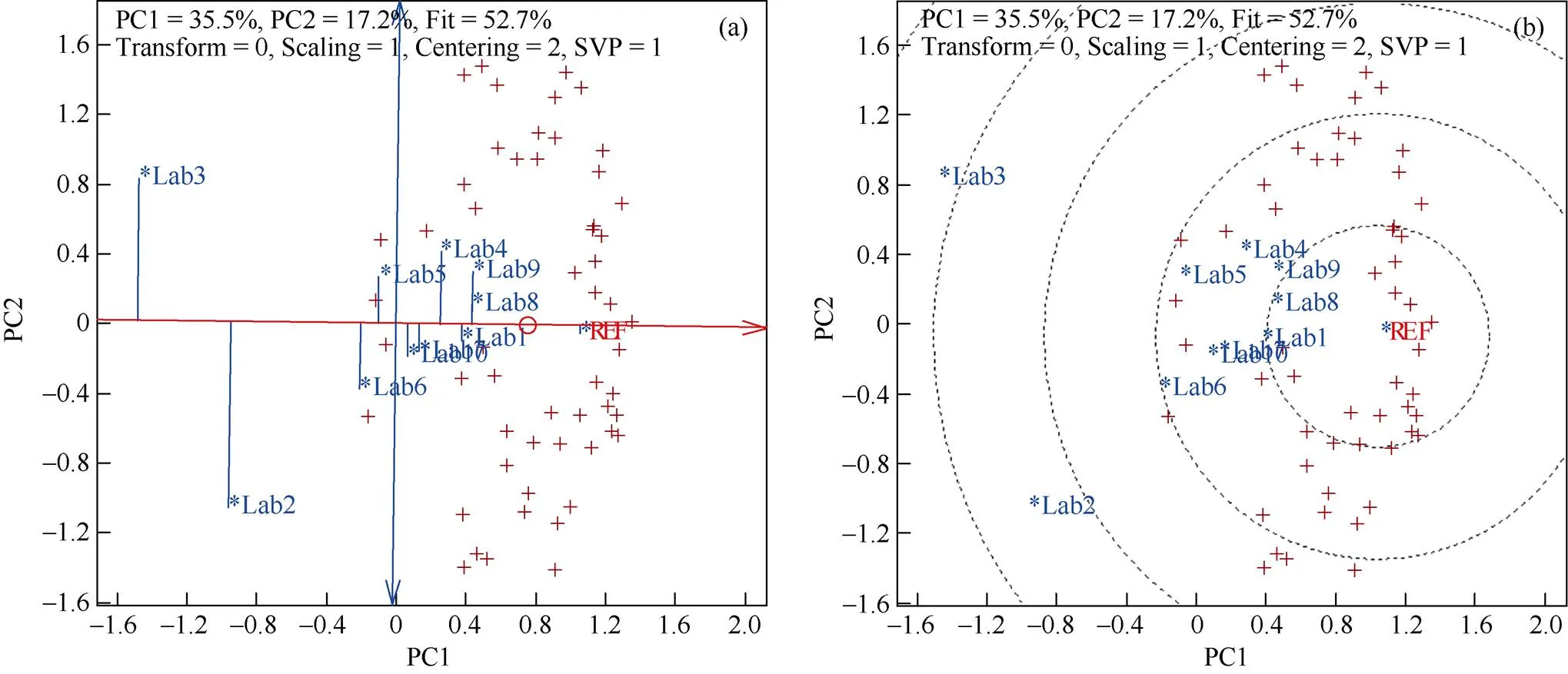
图3 实验室对小麦品种遗传相似度检测准确度GGE双标图分析的“正确度-精确度”功能图(a)和“准确度排序”功能图(b)
带“*”的实验室编号同表1, “+”表示品种组合图标。PC1相当于品种位点相似性检测的正确度, PC2的绝对值相当于精确度。图3-b中的同心圆圆心为标准参照坐标, 实验室图标到圆心的欧氏距离表示准确度, 距离越小则准确度越好。
Lab codes prefixed with star sign “*” are the same as those given in Table 1. The plus sign “+” stands for variety comparison combination mark. PC1 corresponds to the trueness in variety locus similarity detection and the absolute value of PC2 corresponds to the precision. The origin of concentric circles in Fig. 3-b is the standard reference mark, and the Euclidean distance from the variety combination mark to the origin represents the detection accuracy. The smaller the distance, the better the accuracy.
2.4 各实验室对小麦品种遗传相似度检测准确度及其容许误差估计
各实验室对小麦品种SNP位点检测的准确度参数及其相应的容许误差分析结果表明(表3), (1) 不同实验室的检测结果的正确度分布于96.8%~99.1%之间, 平均正确度为98.3%。各实验室检测正确度由高到低的排序为Lab8>Lab9>Lab1>Lab7>Lab4> Lab10>Lab5>Lab6>Lab2>Lab3。其中, Lab2和Lab3的检测正确度显著差于其余实验室。(2) 各实验室的检测精确度分布于1.1%~1.9%之间, 平均精确度为1.4%。各实验室检测精确度由好到差的排序为Lab8> Lab9>Lab1>Lab4>Lab7>Lab10>Lab5>Lab6>Lab2>Lab3。其中, Lab2和Lab3的检测精确度显著差于其余检测单位。(3) 各实验室的检测准确度分布于96.2%~98.5%之间, 平均准确度为97.7%。各实验室检测准确度由高到低的排序与正确度的排序相同。其中, Lab8、Lab9、Lab1和Lab7的准确度在98%以上, Lab4、Lab10、Lab5和Lab6的准确度在97%以上, Lab2和Lab3 的准确度最低, 显著低于其余实验室。(4) 各实验室检测正确度的容许误差分布于1.3%~ 1.9%之间, 平均为1.5%; 准确度的容许误差分布于1.5%~2.0%之间, 平均为1.7%。其中, Lab2和Lab3的检测正确度和准确度的容许误差均显著高于其余实验室。

表3 不同实验室SNP标记法检测准确度及其容许误差估计
同一列中标有相同小写字母的数据在0.05概率水平差异显著。
Different lowercase letters in the same row indicate significantly different at the 0.05 probability level.
3 讨论
3.1 基于SNP标记法鉴定农作物品种遗传相似度的检测误差问题
SNP标记技术近年来在农作物种子的真实性检测[21]、纯度检测[22]和指纹数据库构建[23]等方面被广泛研究和应用。2021年陆续颁布实施了小麦、玉米和水稻等主要农作物品种真实性鉴定SNP标记法行业标准[3-5], 其他农作物真实性鉴定的SNP标记法技术标准也在逐步研究和实施之中, SNP标记法在农作物种子真实性鉴定等领域中发挥着越来越重要的作用[1-2]。SNP标记法的检测可靠性虽然在理论上主要决定于分子标记的数量、质量及其最小等位变异频率(minor allele frequency, MAF), 不受外界环境条件的影响, 不同实验室或平台对相同检测样品都能获得同样的检测结果[1]。然而, 一些可能影响检测结果的因素, 如试样数量、DNA提取的质量、检测平台或仪器设备性能差异、操作人员对标准掌握的熟练程度差异等并不能完全被控制, 不同批次或单位检测结果间并不能做到完全一致, 检测误差仍不可能完全排除, 而这样的误差和不确定性必然影响SNP检测结果的准确性和应用。2023年ISTA报道了多实验室对豌豆品种SSR分子标记检测的验证试验, 实验室检测结果间的一致性只有约90%[7]。SNP标记法的检测准确度体现了检测结果的可靠性和可重复性, 而准确度又包括正确度与精确度(或精密度) 2个方面, 其中正确度指测试结果与真值或参照值之间的符合度, 精确度指测试结果之间的一致程度[8]。对于跨实验室的协同检测实验, 各实验室的检测结果与标准参照值相比, 即可计算检测正确度; 如没有设置标准参照值, 各实验室的平均检测结果则可以作为参照进行比较; 而同一单位检测时, 重复测试结果的平均值可以作为为参照。本研究由“小麦SNP标准”的制标单位提供标准参照值, 各实验室采用SNP标记法对统一提供的小麦种子样品进行检测, 结果表明实验室间检测的正确度、精确度和准确度均存在显著差异, 其中2个实验室的正确度、精确度和准确度均显著低于其他实验室。可见, SNP标记法的检测误差是客观存在的, 在制定判断品种间差异性的临界指标和阈值时应当考虑检测误差和置信区间。鉴于SNP标记法鉴定品种间遗传相似性主要依据品种间或品种与参照比较的相同或差异位点数(率)进行判断, 位点基因型比对结果以“1、0”统计, 多位点比对结果形成具有二项分布特征的数据集[17],而且SNP检测的不确定性通常为小概率事件, 故宜采用Wilson算法[18]计算准确度的容许误差。本研究表明, 各实验室检测正确度的容许误差分布于1.3%~ 1.9%之间, 平均为1.5%; 准确度的容许误差分布于1.5%~2.0%之间, 平均为1.7%, 说明通过对实验室的能力验证和操作人员的技术培训以提高检测准确度、降低容许误差区间是当前亟待解决的问题。
3.2 利用GGE双标图对农作物品种指纹数据检测准确度的可视化分析
基于SNP标记法等技术检测的DNA指纹数据通常都可能存在检测误差的问题, 而指纹数据的检测正确度、精确度和准确度等统计参数又比较抽象和不易理解。GGE双标图是农作物多环境品种试验中进行品种评价、试验环境评价和品种生态区划分的最高效、直观的统计和图形展示工具, 已经广泛应用于多环境品种试验数据处理和可视化分析[13-14,24]。GGE双标图的适用范围并不局限于多环境品种试验数据分析, 可以对所有二维数据进行可视化分析[25]。利用GGE双标图对农作物品种指纹数据的检测准确度进行可视化分析, 可以直观分析和展示各品种遗传相关性模式, 展示各品种组合或检测单位的检测正确度、精确度和准确度的关系, 以便更加直观地理解指纹检测数据的误差问题。本研究依据各实验室检测结果的相互比对分析形成的品种间位点相似度矩阵, 构建相当于GGE双标图“性状间关系”功能图[9,13,19]的“品种遗传关系”功能图和“品种遗传关系+误差”功能图(图1), 对品种间遗传相关模式进行可视化分析, 直观展示了品种间及品种组合间的遗传相关模式。同时, 基于“品种组合”和“实验室-品种组合”正确度矩阵构建的“正确度-精确度”功能图(图2-a和图3-a), 具有GGE双标图“均值-稳定性”功能图的特征与功能[20,26]。其中, AEA轴指向检测正确度大的方向; 通过原点垂直于AEA轴的AEC轴指向检测精确度差的方向, 越接近于AEA轴, 则精确度越好。在“正确度-精确度”功能图基础上构建的“准确度排序”功能图, 相当于GGE双标图的“理想环境”或“理想品种”功能图[20], 各品种组合或实验室图标到同心圆圆心的距离代表了检测准确度, 距离越小, 则检测越准确。各品种组合的“准确度排序”功能图(图2-b)展示了中科麦138/中科麦36和洛旱7号/洛旱11的检测准确度最高, 而济麦22/婴泊700的检测准确度相对较差。实验室“准确度排序”功能图(图3-b)直观地展示了各实验室准确度由高到低依次排序。本研究表明, GGE双标图技术应用于SNP标记检测数据, 可以更加直观、高效、科学地展示品种间遗传相关性模式和SNP标记检测的正确度、精确度和准确度的关系, 从而为GGE双标图在其他作物品种SNP标记检测数据分析中的应用提供了范例。
4 结论
基于小麦品种真实性鉴定的96个SNP位点在10个实验室的检测结果, 55对品种组合位点相似度检测的总体准确度约为98%。7组小麦品种组合间的遗传相似度在95%以上。洛旱7号/洛旱11相似度检测的准确度高, 晋麦47/临抗11准确度一般, 而济麦22/婴泊700的准确度较差。各实验室的检测准确度存在明显差异, Lab2和Lab3检测的正确度、精确度和准确度表现均显著差于其余实验室。各实验室检测正确度的容许误差分布于1.3%~1.9%之间, 平均为1.5%; 准确度的容许误差分布于1.5%~2.0%之间, 平均为1.7%。其中, Lab2和Lab3的容许误差均显著高于其余实验室。
[1] 徐云碧, 王冰冰, 张健, 张嘉楠, 李建生. 应用分子标记技术改进作物品种保护和监管. 作物学报, 2022, 48: 1853–1870. Xu Y B, Wang B B, Zhang J, Zhang J N, Li J S. Enhancement of plant variety protection and regulation using molecular marker technology., 2022, 48: 1853–1870 (in Chinese with English abstract).
[2] 李巧英, 郑戈文. SNP分子标记技术在农作物种子检测中的研究与应用. 中国种业, 2019, (11): 16–18. Li Q Y, Zheng G W. Research and application of SNP molecular marker technology in crop seed detection., 2019, (11): 16–18 (in Chinese).
[3] 庞斌双, 任雪贞, 刘丽华, 赵昌平, 张明明, 金石桥, 李宏博, 刘阳娜, 周泽宇, 张风廷, 张立平, 张胜全, 马锦绣, 权威, 王穆穆, 张旭, 侯建, 关海涛, 傅友兰, 王卫红. 小麦品种真实性鉴定 SNP标记法. 中华人民共和国农业行业标准, 2021, NY/T 4021-2021. Pang B S, Ren X Z, Liu L H, Zhao C P, Zhang M M, Jin S Q, Li H B, Liu Y N, Zhou Z Y, Zhang F T, Zhang L P, Zhang S Q, Ma J X, Quan W, Wang M M, Zhang X, Hou J, Guan H T, Fu Y L, Wang W H. Wheat (L.) variety genuineness identification: SNP based method. Agricultural Industry Standards of the People’s Republic of China, 2021, NY/T 4021–2021 (in Chinese).
[4] 王凤格, 晋芳, 田红丽, 易红梅, 赵久然, 金石桥, 杨扬, 王蕊,葛建镕, 支巨振, 赵建宗. 玉米品种真实性鉴定 SNP标记法. 中华人民共和国农业行业标准, 2021, NY/T 4022–2021. Wang F G, Jin F, Tian H L, Yi H M, Zhao J R, Jin S Q, Yang Y, Wang R, Ge J R, Zhi J Z, Zhao J Z. Maize (L.) variety genuineness identification: SNP based method. Agricultural Industry Standards of the People’s Republic of China, 2021, NY/T 4022–2021 (in Chinese).
[5] 魏兴华, 刘丰泽, 韩斌, 徐群, 冯旗, 赵妍, 支巨振, 周泽宇, 杨窑龙, 冯跃, 任雪贞, 王珊, 章孟臣. 水稻品种真实性鉴定 SNP标记法. 中华人民共和国农业行业标准, 2021, NY/T 2745–2021. Wei X H, Liu F Z, Han B, Xu Q, Feng Q, Zhao Y, Zhi J Z, Zhou Z Y, Yang Y L, Feng Y, Ren X Z, Wang S, Zhang M C. Rice (L.) variety genuineness identification: SNP based method. Agricultural Industry Standards of the People’s Republic of China, 2021, NY/T 2745–2021 (in Chinese).
[6] 田红丽, 张如养, 范亚明, 杨扬, 张云龙, 易红梅, 邢锦丰, 王凤格, 赵久然. Maize 6H-60K芯片在玉米实质性派生品种鉴定中的应用分析. 作物学报, 49: 2876–2885. Tian H L, Zhang R Y, Fan Y M, Yang Y, Zhang Y L, Yi H M, Xing J F, Wang F G, Zhao J R. Application of maize 6H-60K chip in identification of maize essentially derived varieties., 49: 2876–2885 (in Chinese with English abstract).
[7] International Seed Testing Association. Method Validation Reports on Rules Proposals for the International Rules for Seed Testing 2023 Edition, Wallisellen, Switzerland, 2023.
[8] 李成明, 冯士雍, 张震坤, 姜健, 周崎, 丁文兴, 宋武元, 于振凡, 李政军, 肖惠, 刘建斌, 陈玉忠. 测量方法与结果的准确度(正确度与精密度) 第6部分: 准确度值的实际应用. 中华人民共和国国家标准, 2009, GB/T 6379.6-2009. Li C M, Feng S Y, Zhang Z K, Jiang J, Zhou Q, Ding W X, Song W Y, Yu Z F, Li Z J, Xiao H, Liu J B, Chen Y Z. Accuracy (trueness and precision) of measurement methods and results-Part 6: Use in practice of accuracy values. National Standards of the People’s Republic of China, 2009, GB/T 6379.6-2009 (in Chinese).
[9] 严威凯. 品种选育与评价的原理和方法评述. 作物学报, 2022, 48: 2137–2154. Yan W K. A critical review on the principles and procedures for cultivar development and evaluation., 2022, 48: 2137–2154 (in Chinese with English abstract).
[10] Xu N, Qiao Y, Zhao S, Yang X, Li J, Fok M. Optimizing the test locations and replicates in multi-environmental cotton registration trials in southern Xinjiang, China., 2022, 62: 1866–1879.
[11] 许乃银, 李健. 利用GGE双标图划分长江流域棉花纤维品质生态区. 作物学报, 2014, 40: 891–898. Xu N Y, Li J. Ecological regionalization of cotton fiber quality based on GGE biplot in Yangtze River valley., 2014, 40: 891–898 (in Chinese with English abstract).
[12] Yan W. GGEbiplot: a Windows application for graphical analysis of multienvironment trial data and other types of two-way data., 2001, 93: 1111–1118.
[13] 严威凯. 双标图分析在农作物品种多点试验中的应用. 作物学报, 2010, 36: 1805–1819. Yan W K. Optimal use of biplots in analysis of multi-location variety test data., 2010, 36: 1805–1819 (in Chinese with English abstract).
[14] 许乃银, 王扬, 王丹涛, 宁贺佳, 杨晓妮, 乔银桃. 棉花纤维质量指数的构建与WGT双标图分析. 作物学报, 2023, 49: 1262–1271. Xu N Y, Wang Y, Wang D T, Ning H J, Yang X N, Qiao Y T. Construction of cotton fiber quality index and WGT biplot analysis., 2023, 49: 1262–1271 (in Chinese with English abstract).
[15] Jighly A, Hayden M, Daetwyler H. Integrating genomic selection with a genotype plus genotype × environment (GGE) model improves prediction accuracy and computational efficiency., 2021, 44: 3459–3470.
[16] 于振凡, 冯士雍, 刘文, 姜健, 丁文兴, 王斗文, 肖惠, 李成明.测量方法与结果的准确度(正确度与精密度) 第1部分: 总则与定义. 中华人民共和国国家标准, 2004, GB/T 6379.1-2004. Yu Z F, Feng S Y, Liu W, Jiang J, Ding W X, Wang D W, Xiao H, Li C M. Accuracy (trueness and precision) of measurement methods and results. Part 1: General principles and definitions. National Standards of the People’s Republic of China, 2004, GB/T 6379.1-2004 (in Chinese).
[17] 盖钧镒. 试验统计方法. 北京: 中国农业出版社, 2006. Gai J Y. Methods of Experimental Statistics. Beijing: China Agriculture Press, 2006 (in Chinese).
[18] Wilson E B. Probable inference, the law of succession, and statistical inference., 1927, 22: 209–212.
[19] Yan W, Kang M S, Ma B, Woods S, Cornelius P L. GGE biplot vs. AMMI analysis of genotype-by-environment data., 2007, 47: 643–655.
[20] Yan W. A systematic narration of some key concepts and procedures in plant breeding., 2021, 12: 724517.
[21] 刘丽华, 庞斌双, 刘阳娜, 李宏博, 王娜, 王拯, 赵昌平. 基于SNP标记的小麦高通量身份鉴定模式. 麦类作物学报, 2018, 38: 529–534. Liu L H, Pang B S, Liu Y N, Li H B, Wang N, Wang Z, Zhao C P. High-throughput Identification mode for wheat varieties based on SNP markers., 2018, 38: 529–534 (in Chinese with English abstract).
[22] 王立新, 季伟, 李宏博, 葛玲玲, 信爱华, 王丽霞, 常利芳, 赵昌平. 以DNA位点纯合率评价小麦品种的一致性和稳定性. 作物学报, 2009, 35: 2197–2204. Wang L X, Ji W, Li H B, Ge L L, Xin A H, Wang L X, Chang L F, Zhao C P. Evaluating uniformity and stability of wheat cultivars based on ratio of homozygous DNA locus., 2009, 35: 2197–2204 (in Chinese with English abstract).
[23] 田红丽, 赵紫薇, 杨扬, 范亚明, 班秀丽, 易红梅, 杨洪明, 刘少荣, 高玉倩, 刘亚维, 王凤格. 290个吉林省审定玉米品种SSR-DNA指纹构建及遗传多样性分析. 作物学报, 2022, 48: 2994–3003. Tian H L, Zhao Z W, Yang Y, Fan Y M, Ban X L, Yi H M, Yang H M, Liu S R, Gao Y Q, Liu Y W, Wang F G. Construction of SSR-DNA fingerprints and genetic diversity analysis of 290 maize varieties approved in Jilin province, China., 2022, 48: 2994–3003 (in Chinese with English abstract).
[24] Xu N, Fok M, Zhang G, Li J, Zhou Z. The application of GGE biplot analysis for evaluating test locations and mega-environment investigation of cotton regional trials., 2014, 13: 1921–1933.
[25] Yan W. Crop Variety Trials: Data Management and Analysis. New York: John Wiley & Sons, 2014.
[26] 许乃银, 李健. 棉花区试中品种多性状选择的理想试验环境鉴别. 作物学报, 2014, 40: 1936–1945. Xu N Y, Li J. Identification of ideal test environments for multiple traits selection in cotton regional trials., 2014, 40: 1936–1945 (in Chinese with English abstract).
Genetic similarity and its detection accuracy analysis of wheat varieties based on SNP markers
XU Nai-Yin1, JIN Shi-Qiao2,*, JIN Fang2, LIU Li-Hua3, XU Jian-Wen1, LIU Feng-Ze2, REN Xue-Zhen2, SUN Quan2, XU Xu1, and PANG Bin-Shuang3,*
1Institute of Industrial Crops, Jiangsu Academy of Agricultural Sciences, Nanjing 210014, Jiangsu, China;2National Agricultural Technical Extension and Service Center, Beijing 100125, China;3Institute of Hybrid Wheat, Beijing Academy of Agriculture and Forestry Sciences, Beijing 100097, China
The accuracy estimation of genetic similarity detection of crop varieties is an indispensable supplement and improvement to the application of SNP marker method in crop variety detection technology system. In this study, based on the cross-laboratory collaborative validation test data using SNP molecular marker method in 2021, the genetic similarity among wheat varieties and the accuracy of SNP molecular marker method in variety similarity detection were analyzed. The results showed as follows: (1) The overall accuracy of marker locus similarity detection among 55 wheat variety combinations by 10 laboratories was approximately 98%. (2) The genetic relationship between varieties view of GGE biplot delineated the genetic relationship between varieties. The genetic similarity between seven combinations of wheat varieties was over 95%, and the genetic similarity of other combinations was relatively lower. (3) The “trueness-precision” view and “accuracy ranking” view of GGE biplot identified that the similarity detection accuracy of the variety combination Jinmai 47/Linkang 11 was on average, Jimai 22/Yingbo 700 was relatively lower, while Luohan 7/Luohan 11 and other variety combinations were relatively high. (4) Significant differences were existed in detection accuracy among the 10 laboratories, and the performances in detection trueness, precision and accuracy of two laboratories were significantly worse than those of other laboratories. (5) The tolerance error of the trueness of each laboratory ranged from 1.3% to 1.9%, with an average of 1.5%. The tolerance error of accuracy was distributed between 1.5% and 2.0%, with an average of 1.7%. Among them, the tolerance errors of the detection trueness and accuracy of Lab2 and Lab3 were significantly worse than those of the other laboratories. In this study, the detection accuracy statistical model of SNP marker method in detecting crop variety similarity was constructed to analyze the detection accuracy and the corresponding tolerance error of variety combination in different laboratories, and the GGE biplot techniques were adopted to visualize the detection trueness, precision, and accuracy, so as to verify the accuracy and reliability of the detection method for variety locus similarity in each laboratory. Therefore, the findings in this study could provide the theoretical support and application examples for the accuracy evaluation of SNP marker detection technique system for genetic similarity among crop varieties.
wheat (L.); GGE biplot; SNP marker; genetic similarity; locus similarity; accuracy
10.3724/SP.J.1006.2024.31044
本研究由国家科技创新重大项目(2022ZD04019)资助。
This study was supported by the National Scientific and Technological Innovation Major Project (2022ZD04019).
金石桥, E-mail: jinshiqiao@agri.gov.cn; 庞斌双, E-mail: 1492196201@qq.com
E-mail: naiyin@126.com
2023-07-20;
2023-10-23;
2023-10-27.
URL: https://link.cnki.net/urlid/11.1809.S.20231027.1619.002
This is an open access article under the CC BY-NC-ND license (http://creativecommons.org/licenses/by-nc-nd/4.0/).

