花生含油量全基因组选择及近红外光谱筛选的育种技术探究
鲁 清 刘 浩 李海芬 王润风 黄 璐 梁炫强 陈小平 洪彦彬 刘海燕 李少雄
花生含油量全基因组选择及近红外光谱筛选的育种技术探究
鲁 清 刘 浩 李海芬 王润风 黄 璐 梁炫强 陈小平 洪彦彬 刘海燕 李少雄*
广东省农业科学院作物研究所 / 广东省农作物遗传改良重点实验室 / 国家油料作物改良中心南方花生分中心, 广东广州 510640
花生含油量对单位面积产油量至关重要。该性状受多个微效基因控制, 但可用的紧密连锁标记十分有限, 传统的分子标记辅助选择育种准确性不高。全基因组选择作为一种新的育种方法, 可实现对数量性状的早期预测; 近红外光谱分析可对作物品质性状(如含油量等)进行无损检测。通过两者优势互补, 建立花生含油量全基因组选择和近红外光谱筛选联合的育种技术, 探讨影响花生含油量全基因组选择预测准确性的因素, 为花生分子育种奠定理论基础。本研究以216个重组自交系为材料构建训练群体; 分别以139、464和505株F2、F3和F4为材料构建育种群体; 利用自主开发的“PeanutGBTS40K”液相芯片进行基因分型, 开展含油量全基因组选择育种模型分析; 通过联合全基因组选择和近红外光谱筛选技术, 开展花生含油量性状的育种应用, 并评价其育种效果。结果显示, 对训练群体进行基因分型后, 总共获得30,355个高质量SNPs, 并用于11个全基因组预测的模型选择分析。含油量预测准确性最高的模型为rrBLUP, 其次是randomforest和svmrbf。以重组自交系为预测群体, F2、F3和F4各世代含油量的预测准确性分别为0.116、0.128和0.119; 以重组自交系叠加上一轮的育种群体为预测群体, 各世代含油量的预测准确性分别为0.116、0.131和0.160。全基因组选择联合近红外筛选要比单独的全基因组选择对各世代的含油量选择效果提高1.8%、2.7%和3.4%; 与单独的近红外筛选相比, 差异不显著(0.10%、0.06%和0.07%); 而近红外筛选与全基因组选择相比, 含油量可显著提高1.7%、2.6%和3.3%。通过联合全基因组选择和近红外光谱筛选育种, F3和F4分别比F2的含油量提高1.2%和1.0%。F4总共获得16个入选改良株系, 有10个株系含油量≥55.0%, 其中2个株系(SF4_201和SF4_379)的理论产量分别比对照增产7.0%和11.1%。本研究通过建立花生含油量性状的全基因组选择-近红外光谱筛选联合育种技术, 可有效实现花生含油量性状的遗传改良。
花生; 含油量; 全基因组选择; 近红外光谱分析; 基因组育种值
我国是食用植物油生产和消费大国。由于快速增长的消费需求, 我国食用植物油综合自给率仅有30%左右, 进口依赖程度大。加之国际经贸摩擦、贸易壁垒等因素, 使得油料进口风险剧增, 油脂市场供给形势十分严峻, 迫切需要进一步提高国内油料生产能力。花生是我国重要的油料作物和经济作物之一。近年来, 我国花生年均种植面积在460万公顷左右, 居世界第二; 总产量保持在1700万吨以上, 位列世界第一[1]。在我国主要的油料作物中, 花生的单产和单位面积产油量最高, 是我国优势油料作物之一, 在保障我国食用植物油供给安全、稳定国内市场供给具有较大的潜力和优势[1]。含油量对单位面积产油量有最直接贡献。研究表明, 花生含油量每提高1%相当于产量提高2%左右, 效益可提高7%以上[2]。但是, 我国花生育成品种含油量普遍偏低(平均50%左右), 高油品种(含油量55%以上)缺乏[3], 尤其在我国南方沿海花生产区, 品种含油量长期徘徊在50%左右[4-6]。而我国花生种质资源鉴定中不乏有含油量超过55%的高油优异种质[7-8], 其中“潢川立杆”河南地方种含油量超过59%[9]。可见, 我国花生品种含油量的遗传改良还有很大的提升空间。
花生具有“地上开花地下结果”的特征, 这使得表型选择育种繁琐复杂、周期长; 而传统的分子标记辅助选择育种(Marker Assisted Selection, MAS)受限于有限的连锁标记[10], 并且MAS对于复杂数量性状选择准确性低[11-12]。显然, 利用常规的表型选择育种和传统的MAS技术开展花生数量性状(含油量等)的遗传改良在短期内难以取得突破。全基因组选择(Genomic Selection, GS)技术和近红外光谱(Near Infrared Ray, NIR)筛选技术的出现为花生含油量等品质性状的遗传育种提供了新方向。GS育种策略是利用覆盖整个基因组的遗传标记, 通过适当的统计学建模, 在训练群体中估计出每一个标记的效应值, 并在育种群体中对整个基因组遗传变异和遗传效应进行准确的预测, 估算个体基因组育种值(Genomic Estimated Breeding Value, GEBV), 根据GEBV对个体进行选留[13-14]。有研究表明, GS技术的遗传进展高于表型选择4%~25%, 单位遗传进展成本比传统育种低26%~65%[15]; 其选择标准是个体的基因组育种值, 对低遗传力性状具有很好的选择效果[16]。目前, GS技术已在家畜[17-19]、农作物[20-21]、果树[22]、林木[23-24]等育种上得到广泛应用。但是, GS在育种选择过程中不依赖个体表型, 其GEBV的预测准确性受标记密度、统计方法等因素影响, 可能造成遗传估计与真实表型产生偏差, 导致育种选留存在一定程度的“假阳性”[25]。NIR技术可以对作物种子品质性状(如含油量等)进行快速无损检测, 可实现单籽粒、低世代表型筛选[26]。通过GS和NIR技术互补可有效提升育种个体选留的准确性。栽培花生是异源四倍体作物(AABB, 2= 40), 基因组较大(约2.6 Gb)、结构复杂。以往对花生含油量的检测多采用常规的化学法。该方法需要一定的种子量进行研磨、萃取, 不仅操作繁琐而且难以在早代开展育种材料的选留。在分子育种方面, 花生含油量可用的连锁标记不多, 传统的MAS育种技术也是受限[3]。近年来, 随着花生野生种和栽培种基因组从头测序的完成[27-33], 研究者开发了多张高密度SNP或SSR物理图谱[34-36]; 加之多种高通量、自动化、大规模基因型检测平台开发完成, 并得到广泛应用[37-38]。这些都为花生开展GS育种提供了充分可行的条件。
本研究针对花生含油量性状, 以高油“93057”和低油“Y410”亲本获得的重组自交系(Recombinant Inbred Lines, RILs)为材料构建GS训练群体; 以待改良品种“航花2号”和高油亲本“93057”杂交后代为材料构建育种群体; 联合NIR筛选验证, 开发花生含油量性状的GS-NIR联合育种技术, 并探讨其育种应用效果, 为花生育种新技术的建立奠定基础。
1 材料与方法
1.1 试验材料
本研究中训练群体是2011年以高油材料“93057”为母本、低油材料“Y410”为父本杂交后, 每年春、秋两季种植, 通过单粒传直到F9代获得的216个RILs家系。本研究中育种群体是2020年以待改良品种“航花2号”为母本, 高油材料“93057”为父本杂交后获得的F2(2021年春季)、F3(2021年秋季)和F4(2022年春季)各世代分离群体, 分别包含139、464和505个单株。所有材料均种植在广东省农业科学院白云试验基地(广东省广州市白云区, 23°39′N, 113°44′E)。2016—2019年春季, 训练群体216份RILs家系每份材料种植4行, 每行长1.2 m, 行株间距0.2 m, 单粒播种, 随机区组3次重复, 常规田间管理。成熟后, 每个家系随机取小区中间的6株调查含油量表型。育种群体的各世代按照同样的密度单粒播种, 常规田间管理。成熟后, 调查相应世代每个单株的含油量表型。根据GS预测和NIR检测的结果, 含油量从高到低排序, 保留两者前10的单株编号, 合并后构成下一轮育种群体, 直到F4。利用瑞典波通DA7250近红外分析仪测定花生种仁的含油量。
1.2 基因分型
花生出苗60 d后取新鲜叶片, 利用植物DNA提取试剂盒(DP305-03, TianGen, 北京)按照操作说明步骤提取样本的基因组DNA。利用本团队开发的“PeanutGBTS40K”液相芯片(尚未发表), 委托博瑞迪生物技术有限公司(河北石家庄)进行试验材料的基因分型[39]。获得基因型后进行数据质控, 基本流程包括: 完整度过滤, 筛选基因型至少覆盖80%以上的个体标记; 杂合度过滤, 去除杂合率大于50%以上的标记; 最小等位基因频率过滤, 去除等位基因频率小于0.05的标记; 过滤多等位变异, 保留二等位变异基因型。
1.3 遗传力估计和群体结构分析
利用RILs家系的含油量表型和筛选后的基因型数据, 使用R软件包(rrBLUP)的kin.blup函数估算遗传方差(G2)和环境方差(E2)[40]。根据公式:2=G2/(G2+ (E2/)), 估算含油量性状的广义遗传力, 其中为样本重复数。使用gcta软件[41]基于过滤后的基因型进行群体结构主成分分析(Principal Component Analysis, PCA)。
1.4 GS模型选择
本研究利用训练群体的基因型和4个年份平均含油量表型对11种主要的GS模型, 包括rrBLUP、svmrbf、svmpoly、randomforest、pls、gblupD、gblupA、BayesA、BayesB、BayesC、BayesLasso进行训练, 并通过5倍交叉验证, 对各模型的预测准确性进行评估。其中, 5倍交叉验证方法为: 将训练群体样本随机分成5份, 4份作为训练集, 1份作为测试集。以均等的概率对子集进行建模训练, 每一次都统计测试集的预测值与真实值之间的皮尔森(Pearson)相关系数, 评估模型预测准确性。所有模型均使用R软件包实现, 其中 rrBLUP使用rrBLUP[40]的函数mix.solve; svmrbf和svmpoly使用kemlab[42]; randomforest使用randomForest[43]; pls使用pls (https://CRAN.R-project.org/package=pls); gblupD和gblupA使用rrblup[40]的函数kin.blup; Bayes A、Bayes B、Bayes C和Bayes Lasso均使用BGLR[44]。
为了探究不同训练群体大小对GS预测准确性的影响, 训练群体大小设置10%为梯度从小到大递增, 剩下的为育种群体, 每个训练群体大小重复100次。为了探究SNP标记数量对GS预测准确性的影响, 分别随机选择100、500、1000、5000、10,000、15,000、20,000、25,000和30,000的SNP数目, 采用5倍交叉验证进行训练, 每个标记密度重复100次。
2 结果与分析
2.1 训练群体及其亲本表型分析
亲本“93057”和亲本“Y410”是在前期花生种质资源鉴定评价过程中发现的2个含油量差异较大的材料。其中, “93057”平均含油量达55.4%, 而“Y410”仅为42.9% (图1-A)。以这2个含油量差异显著的材料为亲本, 通过杂交自交构建了包含216个RILs家系作为本研究的训练群体。2016—2019年的含油量表型分析显示该群体家系的平均含油量分别为52.1%、49.8%、48.9%和52.2%, 变异系数分别为5.9%、4.6%、4.0%和4.1%。Shapiro-Wilk正态分布检验显示, 其含油量在2017、2018和2019年均符合正态分布(图1-C), 表明该性状为典型的数量性状,且计算的广义遗传力为0.856。
2.2 训练群体基因分型及主成分分析
利用“PeanutGBTS40K”液相芯片对216个RILs家系进行基因分型。该芯片总共包含40,990个SNP标记, 通过数据质控后最终获得30,355个高质量SNP进行后续训练群体的全基因组选择模型分析。这些SNP在花生不同染色体分布如图2-A所示, 其中A07和B09分布的标记数量较多, SNP位点分别为3055个和3042个; 而A08和B07分布的标记数量较少, SNP位点分别为1355个和1293个; 平均密度为16.1个SNP Mb-1, 基因组覆盖率为99.7%, 位点平均最小等位基因频率为0.38, 杂合率约为0.92%。训练群体的PCA分析结果显示该群体没有明显的亚群结构, 前2个主成分PC1和PC2的方差解释率分别为11.26%和10.01% (图2-B)。
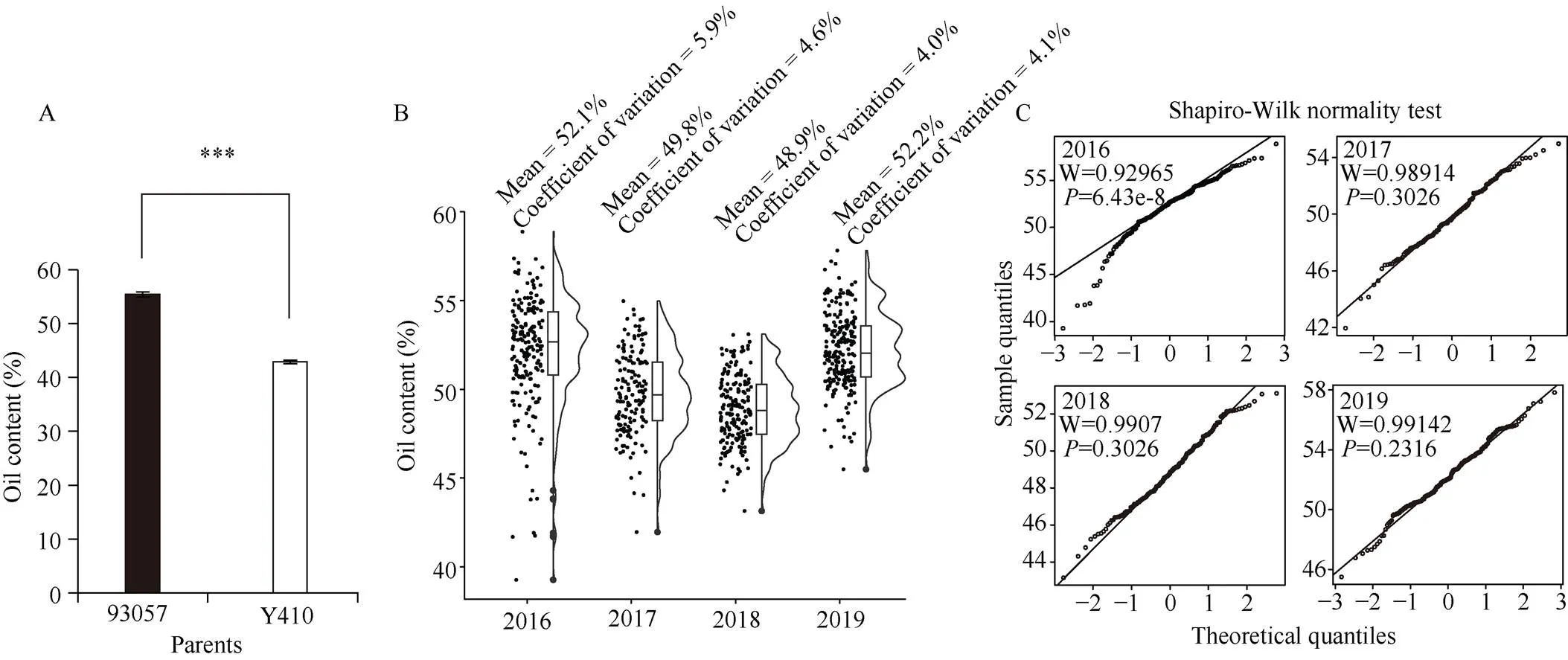
图1 训练群体亲本及216份家系表型分布
A: 高油亲本93057和低油亲本Y410的含油量统计; B: 216份重组自交系在4个不同年份的含油量分布; C: 216份重组自交系在4个不同年份的含油量正态性检验。*** 表示< 0.001。
A: oil content of high oil material 93057 and low oil material Y410; B: oil content distribution of 216 recombinant inbred lines in four years; C: the Shapiro-Wilk normality test of oil content distribution in four years. *** represent< 0.001.
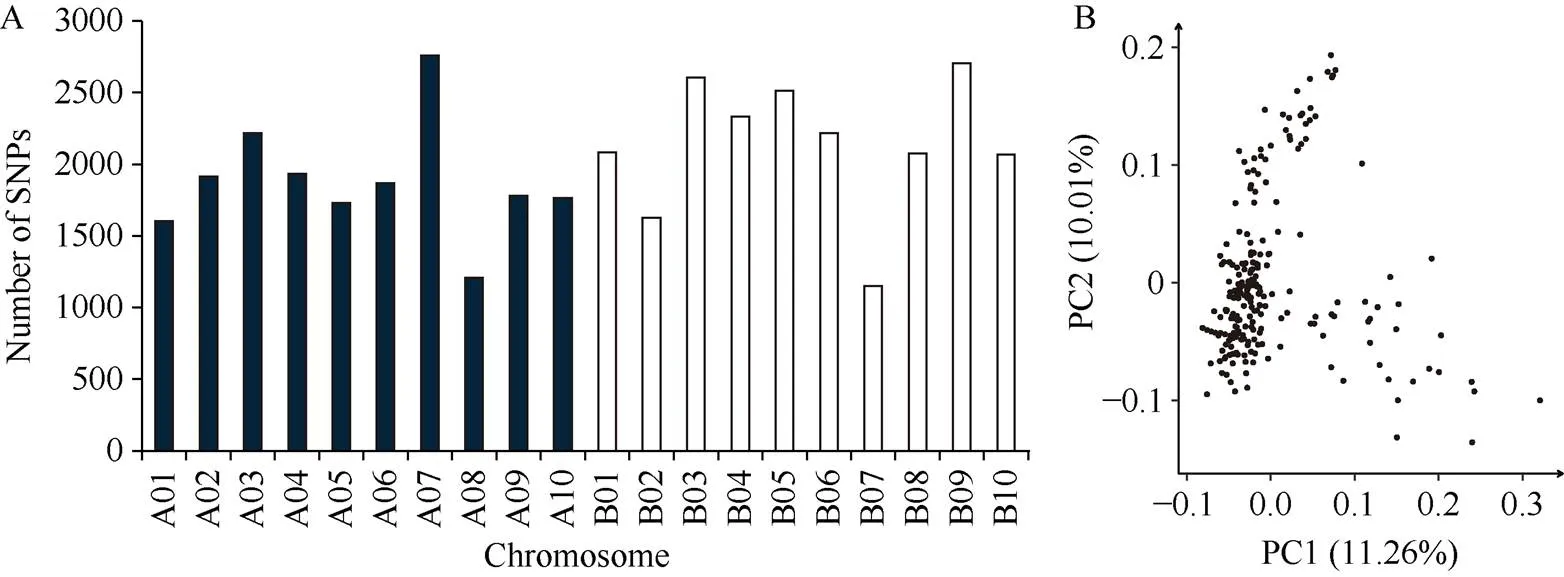
图2 训练群体SNP数目分布及主成分分析
A: SNP在花生20条染色体的分布; B: 训练群体主成分分析。
A: the distribution of SNP on 20 chromosomes; B: the principal component analysis of training population.
2.3 训练群体的不同GS模型的预测准确性评估
为了更好的比较不同模型的预测准确性, 本研究引入百果重和蛋白含量2个性状, 作为模型评估对照。利用30,355个SNP, 结合5倍交叉验证对各模型的预测准确性进行评估。结果显示, 在11个GS模型中rrBLUP模型对所有性状均具有最好的预测准确性, 其次是randomforest (图3-A)。其中, 含油量的预测准确性为0.331, 高于其他模型; 百果重和蛋白含量预测准确性分别为0.268和0.308。预测准确性最差的为gblupD模型, 其含油量性状的预测准确性为0.102。因此, rrBLUP模型可作为后续GS预测的最优模型。
通过随机抽样构建不同样本大小的训练群体, 探究训练群体样本量对含油量rrBLUP模型预测准确性的影响。如图3-B所示, 随着训练群体逐渐增大, GS预测准确性呈上升趋势; 当训练群体超过70%后, GS预测准确性增加幅度较小。同理, 通过对标记进行随机抽样, 研究不同标记数量对模型预测准确性的影响。由图3-C可知, 随着SNP标记数量的增多, GS预测准确性呈上升趋势; 当SNP标记数目达到5000个时, 其预测准确性达0.262; 继续增加SNP标记数量, 预测准确性增加比较缓慢。
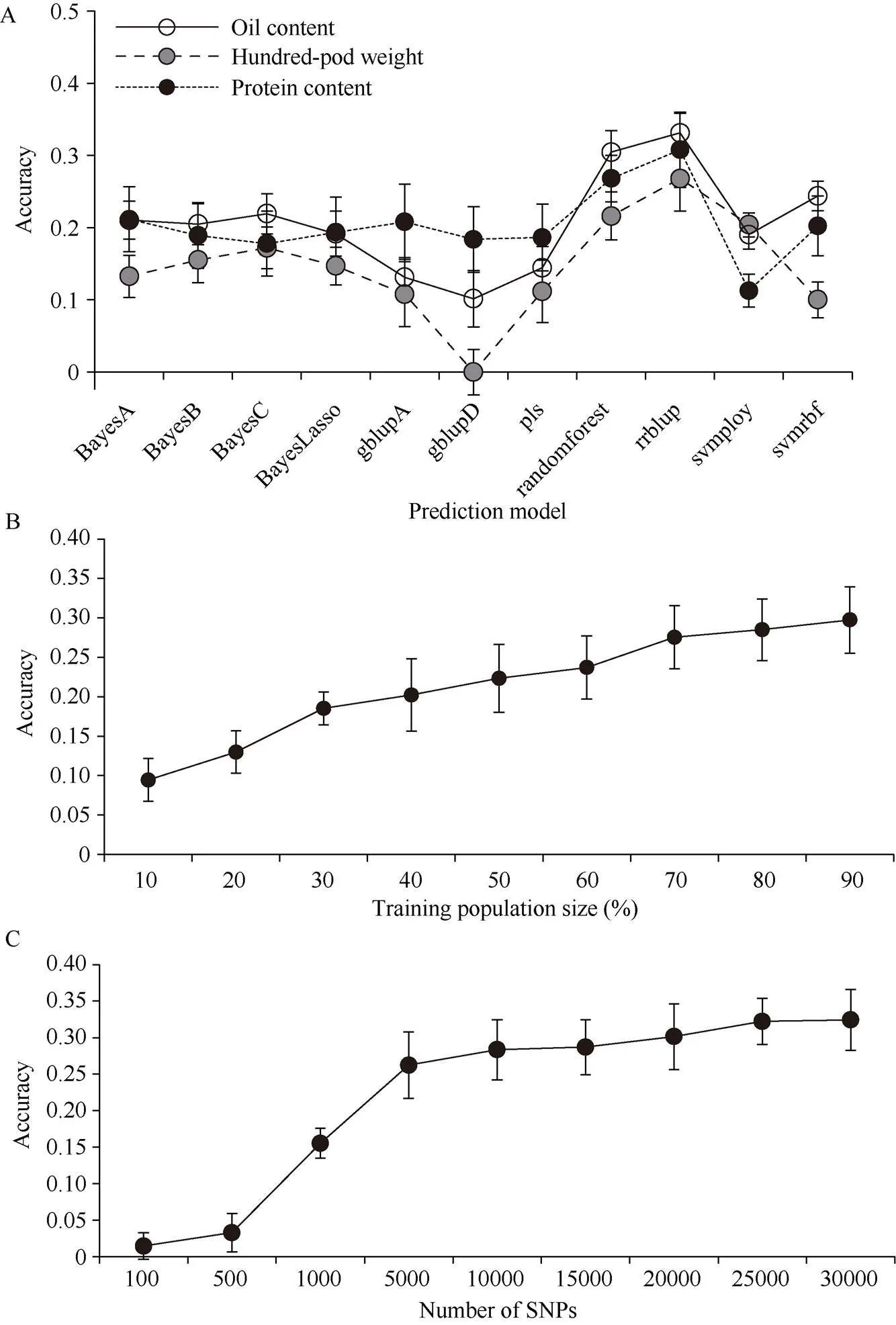
图3 含油量全基因组选择模型筛选及其预测准确性影响因素分析
A: 11个全基因组选择模型预测准确性比较; B: 训练群体不同样本大小的预测准确性比较; C: 不同标记数目的预测准确性比较。
A: comparison of accuracy among 11 genomic selection models; B: comparison of accuracy for different sample sizes of training populations; C: comparison of accuracy for different number of SNP markers.
2.4 最优模型对育种群体的育种值预测及育种决策
以高油“93057”和低油“Y410”亲本(图1-A)获得的RILs家系构建GS训练群体; 以待改良亲本“航花2号” (含油量49.4%)和高油亲本“93057”后代构建育种群体; 以筛选到的最优模型rrBLUP, 开展花生含油量GS预测准确性研究(图4-A)。利用RILs家系为训练群体, 分别以F2、F3和F4世代为育种群体, 探究不同世代的GS预测准确性。在本研究中, F2、F3和F4的预测准确性分别为0.116、0.128和0.119, 其预测准确性随世代的增加稍有提高(F3)而后降低(F4),但差异并不明显。以RILs家系为基础, 将上一代的育种群体添加进训练群体, 通过扩大训练群体样本量, 探究其预测准确性(图4-B~D)。随着训练群体样本量增大, 其预测准确性从0.116提升到0.160, 增幅达38.4% (图4-E~G)。因此, 通过扩大训练群体大小可有效提高预测准确性。
进一步, 本研究比较了各世代单独GS筛选、NIR筛选和GS-NIR联合筛选的育种效果。F2、F3和F4各世代, GS-NIR联合的育种方法分别比单独GS方法显著提高1.8%、2.7%和3.4%的含油量; 比单独NIR方法对含油量筛选差异不显著(0.10%、0.06%和0.07%); 而单独NIR比单独GS方法也可显著提高含油量(1.7%、2.6%和3.3%) (图4-H)。这表明, 对于每一世代的筛选, 利用GS-NIR联合的育种方法和单独NIR方法比仅依靠GS预测值筛选, 都可显著提高含油量选择效果, 其中GS-NIR方法选择效果最优。而在相同的育种方法筛选条件下, F4代对含油量的筛选与F3代无显著差异, 而F4和F3代对含油量的筛选与F2代差异显著或极显著(图4-H)。表明随着世代数的提高, 含油量的筛选效果有所降低; 在实际育种应用过程中, 可在较早世代开展含油量的GS-NIR决策。
2.5 基于GS-NIR育种技术的改良品系评价
每一轮GS后, 根据GEBV预测值和NIR检测的真实值, 分别将含油量从高到低排序, 保留两者前10的单株编号, 合并后构成下一轮育种群体, 直到F4, 种植成株系。据此, F2、F3和F4筛选后分别保留了15、17和16个单株。群体含油量结果显示F3和F4的平均含油量分别为51.9%和51.7%, 显著高于F2(50.7%); 而F3和F4的群体含油量无显著性差异(图5-A)。这表明在实际育种中, 可以在F3或F4对于花生含油量性状进行筛选决策。
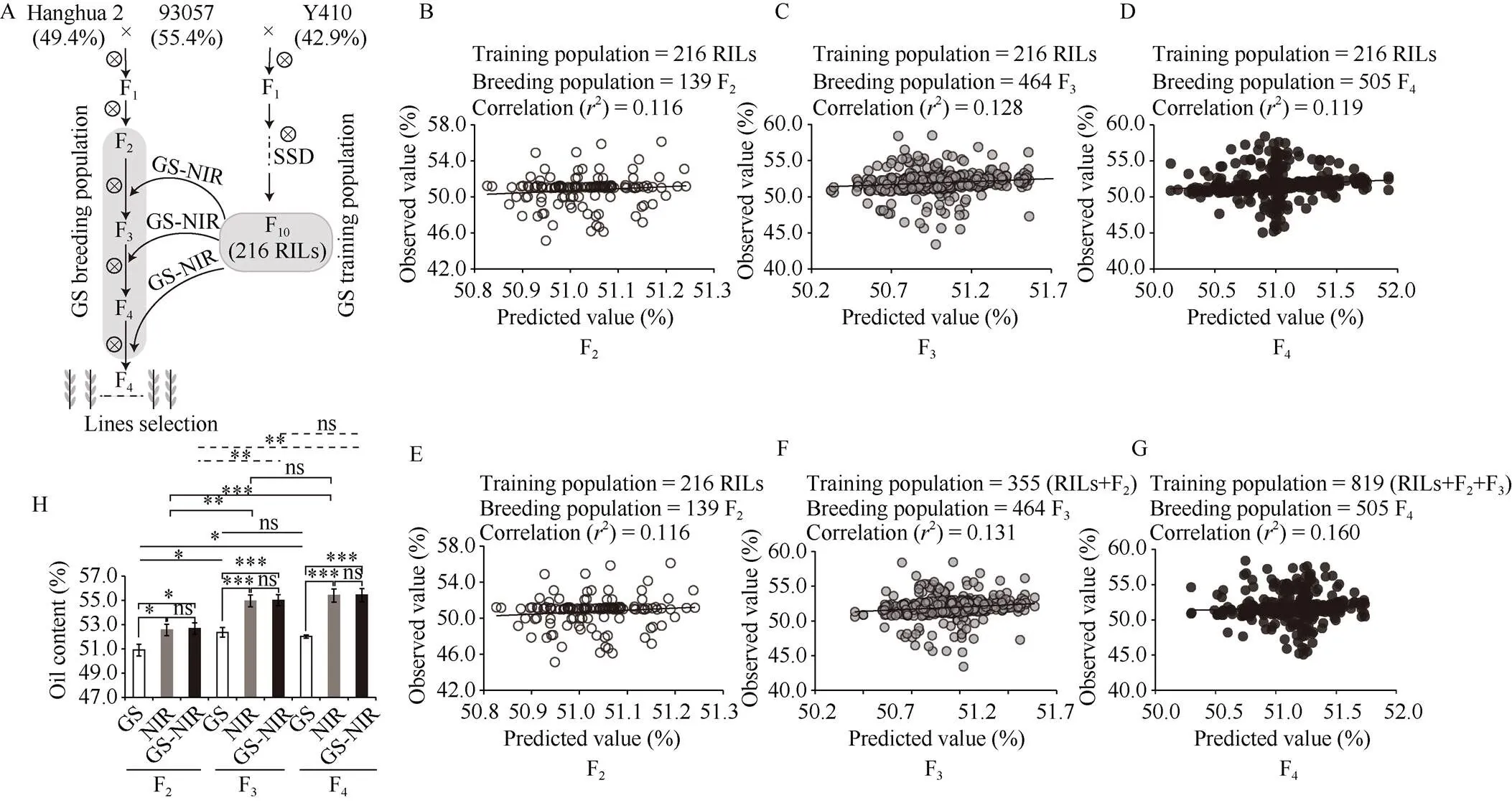
图4 花生含油量育种值预测及育种决策方法比较
A: GS-NIR联合的育种技术路线; B~D: 以RILs家系为训练群体, F2、F3和F4的预测值与观测值的相关性分析; E~G: 分别以RILs、RIL+F2和RIL+F2+F3为训练群体, F2、F3和F4的预测值与观测值的相关性分析; H: 单一的GS和NIR育种方法与GS-NIR联合的育种方法对不同世代入选单株含油量筛选比较。ns表示0.05; *、**和***分别表示在0.05、0.01和0.001概率水平差异显著。
A: GS-NIR joint breeding strategy; B-D: correlation analysis between predicted and observed values of F2, F3, and F4using RILs as the training population; E-G: correlation analysis between predicted and observed values of F2, F3, and F4using RILs, RIL+F2and RIL+F2+F3as the training population, respectively; H: comparison of GS, NIR and GS-NIR joint breeding methods for oil content in different generations. ns represents0.05; *, **, and *** represent significant difference at the0.05, 0.01, and 0.001 probability levels, respectively.
F4入选个体种植成株系, 对总共获得的16个改良株系进行综合评价。16个株系含油量范围在51.5%~56.8%之间, 平均含油量为54.6%。其中, 有10个高油株系(含油量≥55.0%), 分别是SF4_064 (56.6%)、SF4_074 (55.8%)、SF4_201 (56.8%)、SF4_ 203 (55.1%)、SF4_206 (55.0%)、SF4_213 (56.3%)、SF4_222 (55.5%)、SF4_359 (56.1%)、SF4_307 (56.0%)和SF4_379 (55.0%)。按照30万株 hm-2、鲜荚果50%含水率、80%收获率, 计算各改良株系及其亲本的理论产量。结果显示, 改良株系理论产量范围在2724.7~4957.9 kg hm–2, 平均产量为3828.0 kg hm–2。在10个高油株系中, 仅有2个株系的理论产量比对照“航花2号”增产, 分别是SF4_201 (4777.0 kg hm–2)和SF4_379 (4957.9 kg hm–2), 各增产7.0%和11.1%, 其余高油株系均减产11.1%~38.9%不等(图5-B)。表明花生多性状的协同改良具有相当大的难度, 需要开展多环境、跨组合试验筛选。因此, 探寻高通量、高效率、高准确性、低成本的现代育种技术是未来作物遗传改良的重要内容。
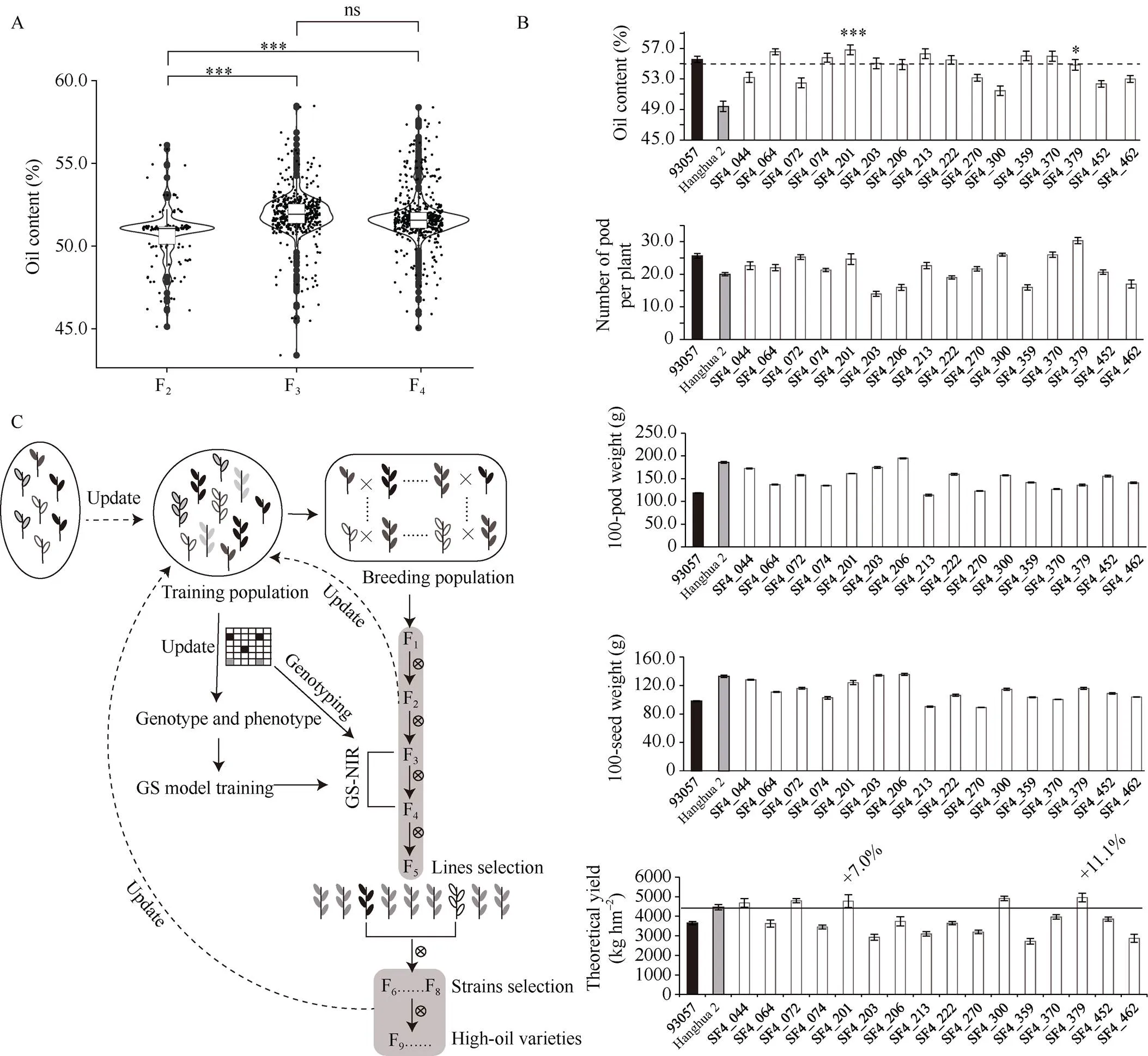
图5 花生含油量GS-NIR联合的育种策略及改良品系评价
A: 各世代的含油量比较; B: 16个改良株系综合评价; C: 花生含油量GS-NIR联合的育种策略。ns表示0.05; *和***分别表示在0.05和0.001概率水平差异显著。
A: comparison of oil content among different generations; B: evaluation of 16 improved lines; C: GS-NIR joint breeding strategy for peanut oil content improvement. ns represents0.05; * and *** represent significant difference at the0.05 and 0.001 probability levels, respectively.
2.6 花生含油量GS-NIR育种技术的参考策略
基于上述结果, 本研究提出了花生含油量GS-NIR联合育种的基本策略(图5-C)。第一, 构建含油量表型丰富的训练群体, 并不断扩充、更新训练群体, 增加遗传多样性; 第二, 根据育种需要, 选取合适的基因分型平台, 不断更新、添加有效的分子标记(如功能标记、主效QTL紧密连锁标记等); 第三, 在早世代开展一次GS选择决策(如在F3或F4), 可有效降低基因分型成本, 联合NIR对GS入选个体的含油量进行验证, 确保筛选决策的可靠性; 第四, 通过整合多轮基因型和表型数据, 不断更新和扩充训练群体, 有利于提高预测准确性。
3 讨论
3.1 作物不同育种方法的比较
作物遗传改良是提高作物产量、改善品质和增强抗性的主要途径。作物育种方法和育种技术的探索对提升作物遗传改良效率至关重要[45]。作物早期育种方法主要是依据个体的优异表型进行选留[45], 而作物表型往往是由基因型和环境互作产生的结果,仅有基因型效应是可以遗传的。因此, 传统的表型选择育种方法对农艺性状鉴定的准确性提出了极高的要求。对于一些易于观察的性状(如颜色性状等), 表型选择操作简单易行。但是, 作物育种中的绝大部分重要性状(如产量、品质、抗性等)往往都是由多基因控制的数量性状, 易受环境因素影响, 表型选择准确性不高[46]。另一方面, 传统的育种往往需要开展大规模的田间表型筛选, 而且依赖育种家多年的经验积累, 常常只有少部分育种材料得到田间测试和选择[47]。随着分子标记技术的发展, 利用MAS替代表型选择是提高作物育种效率的有效途径[48-49]。相对于表型选择, MAS技术方便快捷, 可在早世代对难以鉴定的性状进行选择, 缩短育种周期[50]。但是, MAS技术对主效基因控制的性状进行选择比较有效, 而对于多基因控制的数量性状仍然存在困难[51]。GS这一策略能够将几乎所有遗传变异都考虑进统计模型, 而不仅仅依赖显著性标记进行决策, 可实现对数量性状的有效选择[11]。目前, GS育种已在水稻、豌豆、橡胶树等作物或林木中得到育种应用[20-23]。本研究探究了花生GS育种及其准确性影响因素, 并结合NIR技术研究了GS联合NIR提高花生含油量的育种方法, 为花生GS育种提供了理论参考。
3.2 全基因组选择预测准确性的影响因素
花生含油量对单位面积产油量具有最直接的贡献, 属于多基因控制的数量性状[52]。本研究对花生含油量进行了GS选择, 其准确性在0.1~0.3左右, 低于大豆[53-54]、玉米[55-56]、水稻[57]等作物。这可能与GS统计模型、训练群体大小、标记密度、性状遗传力等因素有关。有研究表明, 对于植物而言在预测大量微效基因控制的复杂数量性状时, rrBLUP模型比较有效[58]。这与本研究结果基本一致。研究中同样发现, 通过加大训练群体大小、增多起始训练群体的世代可有效提高GS预测准确性(图4-E~ G)。Meuwissen等[13]的研究显示, 当训练群体大小从500增加到2200时, rrBLUP模型的准确性从0.579增加到0.732; 同时BayesB模型的准确性也从0.708上升到0.848。本研究将训练群体大小从216增加到819, rrBLUP的预测准确性从0.116增加到0.160, 增幅达38.4%。这表明在后续的GS育种中, 可通过积累、整合和共享不同实验室、不同项目、不同训练样本的基因型和表型等信息, 开展数据共享与开源育种, 达到有效提高预测准确性。标记密度也是影响GS预测准确性的重要影响因素。一般而言, 标记密度越密, 其预测准确性也越高[59], 但预测准确性并非线性增加[55]。这取决于标记与标记之间、或标记与QTL之间的连锁不平衡程度、群体类型、目标性状的复杂程度等[60]。在实际育种中, 过高的标记密度不一定能达到预期的选择效果, 反而会增加基因分型成本。因此, 选择合适的标记密度是平衡GS育种的经济性和准确性的重要内容。在玉米中的研究结果表明, 用于GS的SNP标记个数仅需500个即可获得较高的预测准确性[55]。基于本研究结果, 在花生上用于GS的SNP标记需要达到5000个, 其预测准确性变化趋势呈现平台期。这可能与栽培花生具有较大的基因组有关。另外, 由于世代间隔会积累更多的突变和重组, 也会影响GS选择的准确性, 必须定期更新训练群体。
3.3 全基因组选择、近红外筛选及两者联合的育种方法比较
本研究表明, 在同一世代条件下, GS-NIR联合方法和单独NIR方法对含油量的筛选效果均显著或极显著优于单独GS方法; 而GS-NIR联合方法比单独NIR方法对含油量的筛选差异不显著(图4-H)。一方面, 单独NIR方法虽然是开展含油量筛选的优异方法, 可以实现花生种仁品质性状的无损检测, 但其准确性依赖于高质量的预测模型[26], 并且单独的NIR育种依然属于传统的表型选择育种, 未考虑基因型对含油量表型的效应; 在实际育种应用中, 由于单独的NIR方法需要将所有的育种材料进行田间种植, 再进行品质检测, 这对于对小规模的育种群体是可行的, 而对于大规模育种群体的筛选, 极具挑战。另一方面, GS育种策略虽然是一种优异的育种方法, 可对大规模育种群体进行预测筛选, 大大降低田间种植规模, 已在动植物育种中得到广泛应用[17-24], 但其选择的标准是个体预测的GEBV值, 准确性受标记密度、统计方法等因素影响。由此可见, NIR和GS育种方法各有优缺点。本研究通过结合两者的技术优势, 探究GS-NIR联合的育种方法, 可实现比单独GS方法显著提升育种个体选留的准确性。目前, 基于单独的GS或NIR开展作物品质育种的报道较多[61-65], 而利用GS-NIR联合育种开展作物品质性状筛选, 未见报道。后续, 对于大规模育种群体, GS-NIR联合育种方法有望实现个体的提早选留, 减少育种的时间和空间成本。在相同的育种方法筛选条件下, F4代对含油量的筛选与F3代无显著差异(图4-H)。因此, 可在较早世代(F3或F4)开展一次含油量的GS选择, 既可保证GS的筛选效果又可避免多次GS基因分型, 造成育种成本的增加。本研究通过将前一轮的育种群体增加到训练群体, 用以更新训练群体, 结果可显著增加预测准确性(图4-E~G)。但是, 该策略是选择后代表型较好的个体发展的群体添加到训练群体中, 这可能导致训练群体遗传多样性变得狭窄[66]。GS在后代个体筛选决策时依据统计模型预测的基因组育种值, 其预测准确性受上述诸多因素影响。本研究认为, 对于一些复杂数量性状, 单单依赖GS进行决策, 可能会造成一定的选择假阳性或假阴性。为解决这一问题, 本研究通过联合GS技术和NIR筛选, 对GS入选个体再次进行NIR验证, 确保入选个体的目标表型准确性, 可以充分利用分子育种和表型选择的优势互补。基于本研究的结果, 利用GS-NIR策略可有效提高1.8%~3.4%的选择准确性(图4-H)。
3.4 全基因组选择的展望
综上所述, 作物GS的应用已经取得长足进步, 但依然面临诸多挑战。例如基因分型成本、基因型与环境互作、群体组成与结构、表型鉴定的准确性等等。随着现代分型技术的进步、统计方法的优化、高通量表型组学的发展等, 作物GS技术逐渐成熟和广泛应用为育种研究提供了新的机遇。在未来, 通过整合不同的农业大数据, 如基因组、泛基因组、表型组等数据, 借助人工智能算法等技术, 有望实现作物育种的规模化、自动化、智能化预测筛选, 精准导入目标性状基因, 实现分子设计育种。
4 结论
本研究开展了花生含油量性状的GS研究, 联合NIR筛选技术, 建立了花生含油量的GS-NIR联合育种技术流程, 可实现花生含油量性状的有效改良。通过扩大训练群体大小、适度控制标记密度、较早世代开展选择决策, 可实现GS选择效率和育种经济性的最优化。本研究为花生含油量性状的GS育种提供了重要的理论依据。
[1] 廖伯寿. 我国花生生产发展现状与潜力分析. 中国油料作物学报, 2020, 42: 161–166. Liao B S. A review on progress and prospects of peanut industry in China.,2020, 42: 161–166 (in Chinese with English abstract).
[2] 廖伯寿. 中国花生油脂产业竞争力浅析. 花生学报, 2003, 32(增刊1): 11–15. Liao B S. Analysis on the competitiveness of peanut oil industry in China., 2003, 32(S1): 11–15 (in Chinese with English abstract).
[3] 宋江春, 李拴柱, 王建玉, 张秀阁, 朱雪峰, 乔建礼, 向臻. 我国高油花生育种研究进展. 作物杂志, 2018, (3): 25–31. Song J C, Li S Z, Wang J Y, Zhang X G, Zhu X F, Qiao J L, Xiang Z. Advances in breeding of high oil peanut in China., 2018, (3): 25–31 (in Chinese with English abstract).
[4] 鲁清, 李少雄, 陈小平, 周桂元, 洪彦彬, 李海芬, 梁炫强. 我国南方产区花生育种现状、存在问题及育种建议. 中国油料作物学报, 2017, 39: 556–566.Lu Q, Li S X, Chen X P, Zhou G Y, Hong Y B, Li H F, Liang X Q. Current situation, problems and suggestions of peanut breeding in southern China.2017, 39: 556–566 (in Chinese with English abstract).
[5] 李少雄, 洪彦彬, 陈小平, 梁炫强. 广东花生生产、育种和种业现状与发展对策. 广东农业科学, 2020, 47(11): 78–83. Li S X, Hong Y B, Chen X P, Liang X Q. Present situation and development strategies of peanut production, breeding and seed industry in Guangdong., 2020, 47(11): 78–83 (in Chinese with English abstract).
[6] 杜普旋, 刘军, 陈荣华, 吴柔贤, 范呈根, 郭丹丹, 鲁清. 广东省花生种质资源收集与鉴定评价. 植物遗传资源学报, 2023, 24: 671–679. Du P X, Liu J, Chen R H, Wu R X, Fan C G, Guo D D, Lu Q. Systematic collection, identification and evaluation of peanut germplasm resources in Guangdong province., 2023, 24: 671–679 (in Chinese with English abstract).
[7] 姜慧芳, 段乃雄, 任小平, 孙大容. 花生种质资源的性状鉴定及综合评价进展. 花生科技, 1999, 38(增刊1): 144–147. Jiang H F, Duan N X, Ren X P, Sun D R.Progress in character identification and comprehensive evaluation of peanut germplasm resources., 1999, 38(S1): 144–147 (in Chinese with English abstract).
[8] 姜慧芳, 任小平, 王圣玉, 黄家权, 雷永, 廖伯寿. 野生花生高油基因资源的发掘与鉴定. 中国油料作物学报, 2010, 32: 30–34. Jiang H F, Ren X P, Wang S Y, Huang J Q, Lei Y, Liao B S.Identification and evaluation of high oil content in wildspecies., 2010, 32: 30–34 (in Chinese with English abstract).
[9] 苗利娟, 张新友, 黄冰艳, 董文召, 汤丰收, 刘娟, 张俊, 刘华,齐飞艳. 河南省花生农家品种资源农艺和品质性状分析. 植物遗传资源学报, 2016, 17: 854–860. Miao L J, Zhang X Y, Huang B Y, Dong W Z, Tang F S, Liu J, Zhang J, Liu H, Qi F Y. Evaluation of agronomic and quality traits in peanut (L.) landraces of Henan province., 2016, 17: 854–860 (in Chinese with English abstract).
[10] Moose S P, Mumm R H. Molecular plant breeding as the foundation for 21st century crop improvement., 2008, 147: 969–977.
[11] Heffner E L, Sorrells M E, Jannink J L. Genomic selection for crop improvement., 2009, 49: 1–12.
[12] Moreau L, Charcosset A, Hospital F, Gallais A. Marker-assisted selection efficiency in populations of finite size., 1998, 148: 1353–1365.
[13] Meuwissen T H, Hayes B J, Goddard M E. Prediction of total genetic value using genome-wide dense marker maps., 2001, 157: 1819–1829.
[14] Bhat J A, Ali S, Salgotra R K, Mir Z A, Dutta S, Jadon V, Tyagi A, Mushtaq M, Jain N, Singh P K, Singh G P, Prabhu K V. Genomic selection in theof next generation sequencing for complex traits in plant breeding., 2016, 7: 221.
[15] Wong C K, Bernardo R. Genome wide selection in oil palm: increasing selection gain per unit time and cost with small populations., 2008, 116: 815–824.
[16] Shikha M, Kanika A, Rao A R, Mallikarjuna M G, Gupta H S, Nepolean T. Genomic selection for drought tolerance using genome-wide SNPs in maize., 2017, 8: 550.
[17] Guo P, Zhu B, Xu L, Niu H, Wang Z, Guan L, Liang Y, Ni H, Guo Y, Chen Y, Zhang L, Gao X, Gao H, Li J. Genomic prediction with parallel computing for slaughter traits in Chinese Simmental beef cattle using high-density genotypes., 2017, 12: e0179885.
[18] Yang R, Xu Z, Wang Q, Zhu D, Bian C, Ren J, Huang Z, Zhu X, Tian Z, Wang Y, Jiang Z, Zhao Y, Zhang D, Li N, Hu X. Genome‑wide association study and genomic prediction for growth traits in yellow-plumage chicken using genotyping-by-sequencing., 2021, 53: 82.
[19] Ros-Freixedes R, Johnsson M, Whalen A, Chen C Y, Valente B D, Herring W O, Gorjanc G, Hickey J M. Genomic prediction with whole-genome sequence data in intensely selected pig lines., 2022, 54: 65.
[20] Toda Y, Wakatsuki H, Aoike T, Kajiya-Kanegae H, Yamasaki M, Yoshioka T, Ebana K, Hayashi T, Nakagawa H, Hasegawa T, Iwata H. Predicting biomass of rice with intermediate traits: Modeling method combining crop growth models and genomic prediction models., 2020, 15: e0233951.
[21] Bartholomé J, Prakash P T, Cobb J N. Genomic prediction: progress and perspectives for rice improvement., 2022, 2467: 569–617.
[22] Atanda S A, Steffes J, Lan Y, Al Bari M A, Kim J H, Morales M, Johnson J P, Saludares R, Worral H, Piche L, Ross A, Grusak M, Coyne C, McGee R, Rao J, Bandillo N. Multi-trait genomic prediction improves selection accuracy for enhancing seed mineral concentrations in pea., 2022, 15: e20260.
[23] Aono A H, Francisco F R, Souza L M, Gonçalves P S, Scaloppi Junior E J, Le Guen V, Fritsche-Neto R, Gorjanc G, Quiles M G, de Souza A P. A divide-and-conquer approach for genomic prediction in rubber tree using machine learning., 2022, 12: 18023.
[24] Freeman J S, Slavov G T, Butler J B, Frickey T, Graham N J, Klápště J, Lee J, Telfer E J, Wilcox P, Dungey H S. High density linkage maps, genetic architecture, and genomic prediction of growth and wood properties in., 2022, 23: 731.
[25] Misztal I, Aguilar I, Lourenco D, Ma L, Steibel J P, Toro M. Emerging issues in genomic selection., 2021, 99: skab092.
[26] 纪红昌, 邱晓臣, 柳文浩, 胡畅丽, 孔铭, 胡晓辉, 黄建斌, 杨雪, 唐艳艳, 张晓军, 王晶珊, 乔利仙. 花生籽仁含油量近红外模型的构建及其应用. 中国油料作物学报, 2022, 44: 1089–1097.Ji H C, Qiu X C, Liu W H, Hu C L, Kong M, Hu X H, Huang J B, Yang X, Tang Y Y, Zhang X J, Wang J S, Qiao L X. Construction and application of near infrared ray model for oil content prediction in peanut kernel.2022, 44: 1089–1097 (in Chinese with English abstract).
[27] Chen X, Li H, Pandey M K, Yang Q, Wang X, Garg V, Li H, Chi X, Doddamani D, Hong Y, Upadhyaya H, Guo H, Khan A W, Zhu F, Zhang X, Pan L, Pierce G J, Zhou G, Krishnamohan K A, Chen M, Zhong N, Agarwal G, Li S, Chitikineni A, Zhang G Q, Sharma S, Chen N, Liu H, Janila P, Li S, Wang M, Wang T, Sun J, Li X, Li C, Wang M, Yu L, Wen S, Singh S, Yang Z, Zhao J, Zhang C, Yu Y, Bi J, Zhang X, Liu Z J, Paterson A H, Wang S, Liang X, Varshney R K, Yu S. Draft genome of the peanut A-genome progenitor () provides insights into geocarpy, oil biosynthesis, and allergens., 2016, 113: 6785–6790.
[28] Bertioli D J, Cannon S B, Froenicke L, Huang G, Farmer A D, Cannon E K, Liu X, Gao D, Clevenger J, Dash S, Ren L, Moretzsohn M C, Shirasawa K, Huang W, Vidigal B, Abernathy B, Chu Y, Niederhuth C E, Umale P, Araújo A C, Kozik A, Kim K D, Burow M D, Varshney R K, Wang X, Zhang X, Barkley N, Guimarães P M, Isobe S, Guo B, Liao B, Stalker H T, Schmitz R J, Scheffler B E, Leal-Bertioli S C, Xun X, Jackson S A, Michelmore R, Ozias-Akins P. The genome sequences ofand, the diploid ancestors of cultivated peanut., 2016, 48: 438–446.
[29] Lu Q, Li H, Hong Y, Zhang G, Wen S, Li X, Zhou G, Li S, Liu H, Liu H, Liu Z, Varshney R K, Chen X, Liang X. Genome sequencing and analysis of the peanut B-genome progenitor ()., 2018, 9: 604.
[30] Yin D, Ji C, Ma X, Li H, Zhang W, Li S, Liu F, Zhao K, Li F, Li K, Ning L, He J, Wang Y, Zhao F, Xie Y, Zheng H, Zhang X, Zhang Y, Zhang J. Genome of an allotetraploid wild peanut: aassembly., 2018, 7: giy066.
[31] Chen X, Lu Q, Liu H, Zhang J, Hong Y, Lan H, Li H, Wang J, Liu H, Li S, Pandey M K, Zhang Z, Zhou G, Yu J, Zhang G, Yuan J, Li X, Wen S, Meng F, Yu S, Wang X, Siddique K H M, Liu Z J, Paterson A H, Varshney R K, Liang X. Sequencing of cultivated peanut,, yields insights into genome evolution and oil improvement., 2019, 12: 920–934.
[32] Bertioli D J, Jenkins J, Clevenger J, Dudchenko O, Gao D, Seijo G, Leal-Bertioli S C M, Ren L, Farmer A D, Pandey M K, Samoluk S S, Abernathy B, Agarwal G, Ballén-Taborda C, Cameron C, Campbell J, Chavarro C, Chitikineni A, Chu Y, Dash S, El Baidouri M, Guo B, Huang W, Kim K D, Korani W, Lanciano S, Lui C G, Mirouze M, Moretzsohn M C, Pham M, Shin J H, Shirasawa K, Sinharoy S, Sreedasyam A, Weeks N T, Zhang X, Zheng Z, Sun Z, Froenicke L, Aiden E L, Michelmore R, Varshney R K, Holbrook C C, Cannon E K S, Scheffler B E, Grimwood J, Ozias-Akins P, Cannon S B, Jackson S A, Schmutz J. The genome sequence of segmental allotetraploid peanut., 2019, 51: 877–884.
[33] Zhuang W, Chen H, Yang M, Wang J, Pandey M K, Zhang C, Chang W C, Zhang L, Zhang X, Tang R, Garg V, Wang X, Tang H, Chow C N, Wang J, Deng Y, Wang D, Khan A W, Yang Q, Cai T, Bajaj P, Wu K, Guo B, Zhang X, Li J, Liang F, Hu J, Liao B, Liu S, Chitikineni A, Yan H, Zheng Y, Shan S, Liu Q, Xie D, Wang Z, Khan S A, Ali N, Zhao C, Li X, Luo Z, Zhang S, Zhuang R, Peng Z, Wang S, Mamadou G, Zhuang Y, Zhao Z, Yu W, Xiong F, Quan W, Yuan M, Li Y, Zou H, Xia H, Zha L, Fan J, Yu J, Xie W, Yuan J, Chen K, Zhao S, Chu W, Chen Y, Sun P, Meng F, Zhuo T, Zhao Y, Li C, He G, Zhao Y, Wang C, Kavikishor P B, Pan R L, Paterson A H, Wang X, Ming R, Varshney R K. The genome of cultivated peanut provides insight into legume karyotypes, polyploid evolution and crop domestication., 2019, 51: 865–876.
[34] Pandey M K, Agarwal G, Kale S M, Clevenger J, Nayak S N, Sriswathi M, Chitikineni A, Chavarro C, Chen X, Upadhyaya H D, Vishwakarma M K, Leal-Bertioli S, Liang X, Bertioli D J, Guo B, Jackson S A, Ozias-Akins P, Varshney R K. Development and evaluation of a high density genotyping ‘Axiom_’ array with 58 K SNPs for accelerating genetics and breeding in groundnut., 2017, 7: 40577.
[35] Zhao C, Qiu J, Agarwal G, Wang J, Ren X, Xia H, Guo B, Ma C, Wan S, Bertioli D J, Varshney R K, Pandey M K, Wang X. Genome-wide discovery of microsatellite markers from diploid progenitor species,and, and their application in cultivated peanut ()., 2017, 8: 1209.
[36] Lu Q, Hong Y, Li S, Liu H, Li H, Zhang J, Lan H, Liu H, Li X, Wen S, Zhou G, Varshney R K, Jiang H, Chen X, Liang X. Genome-wide identification of microsatellite markers from cultivated peanut (L.)., 2019, 20: 799.
[37] Elshire R J, Glaubitz J C, Sun Q, Poland J A, Kawamoto K, Buckler E S, Mitchell S E. A robust, simple genotyping-by- sequencing (GBS) approach for high diversity species., 2011, 6: e19379.
[38] 徐云碧, 杨泉女, 郑洪建, 许彦芬, 桑志勤, 郭子锋, 彭海, 张丛, 蓝昊发, 王蕴波, 吴坤生, 陶家军, 张嘉楠. 靶向测序基因型检测(GBTS)技术及其应用. 中国农业科学, 2020, 53: 2983–3004. Xu Y B, Yang Q N, Zheng H J, Xu Y F, Sang Z Q, Guo Z F, Peng H, Zhang C, Lan H F, Wang Y B, Wu K S, Tao J J, Zhang J N. Genotyping by target sequencing (GBTS) and its applications., 2020, 53: 2983–3004 (in Chinese with English abstract).
[39] Guo Z F, Wang H W, Tao J J, Ren Y H, Xu C, Wu K S, Zou C, Zhang J A, Xu Y B. Development of multiple SNP marker panels affordable to breeders through genotyping by target sequencing (GBTS) in maize., 2019, 39: 37–49.
[40] Endelman J B. Ridge regression and other kernels for genomic selection with R package rrBLUP., 2011, 4: 250–255.
[41] Yang J, Lee S H, Goddard M E, Visscher P M. GCTA: a tool for genome-wide complex trait analysis., 2011, 88: 76–82.
[42] Karatzoglou A, Smola A, Hornik K, Zeileis A. Kernlab: an S4 package for kernel methods in R., 2004, 11: 721–729.
[43] Breiman L. Random forests., 2001, 45: 5–32.
[44] Perez P, de los Campos G. Genome-wide regression and prediction with the BGLR statistical package., 2014, 198: 483–495.
[45] 何红中, 周瑞洲. 中国作物育种技术发展的回望与思考. 科学, 2016, 68(4): 32–36.He H Z, Zhou R Z. Reflecting on the development of crop breeding technology in China., 2016, 68(4): 32–36 (in Chinese with English abstract).
[46] 刘忠松. 作物遗传育种研究进展: V. 表型选择与基因型选择. 作物研究, 2014, 28: 780–784.Liu Z S.Research progress in crop genetics and breeding: V. Phenotypic selection and genotype selection., 2014, 28: 780–784 (in Chinese with English abstract).
[47] Bernardo R. Testcross additive and dominance effects in best linear unbiased prediction of maize single-cross performance., 1996, 93: 1098–1102.
[48] Stuber C W, Goodman M M, Moll R H. Improvement of yield and ear number resulting from selection at allozyme loci in a maize population., 1982, 22: 737–740.
[49] Bernardo R, Yu J M. Prospects for genome wide selection for quantitative traits in maize., 2007, 47: 1082–1090.
[50] Hospital F, Moreau L, Lacoudre F, Charcosset A, Gallais A. More on the efficiency of marker-assisted selection., 1997, 95: 1181–1189.
[51] Xu Y, Crouch J H. Marker-assisted selection in plant breeding: from publications to practice., 2008, 48: 391–407.
[52] 江建华, 肖美华, 王晓帅, 于欢欢, 管叔琪, 倪皖莉. 花生含油量研究进展. 中国农学通报, 2012, 28(33): 1–6.Jiang J H, Xiao M H, Wang X S, Yu H H, Guan S Q, Ni W L. Recent progress in oil content ofL., 2012, 28(33): 1–6 (in Chinese with English abstract).
[53] 马岩松, 刘章雄, 文自翔, 魏淑红, 杨春明, 王会才, 杨春燕, 卢为国, 徐冉, 张万海, 吴纪安, 胡国华, 栾晓燕, 付亚书, 王曙明, 韩天富, 张孟臣, 张磊, 苑保军, 郭勇, Reif J C, 江勇, 李文滨, 王德春, 邱丽娟. 群体构成方式对大豆百粒重全基因组选择预测准确度的影响. 作物学报, 2018, 44: 43–52.Ma Y S, Liu Z X, Wen Z X, Wei S H, Yang C M, Wang H C, Yang C Y, Lu W G, Xu R, Zhang W H, Wu J A, Hu G H, Luan X Y, Fu Y S, Wang S M, Han T F, Zhang M C, Zhang L, Yuan B J, Guo Y, Reif J C, Jiang Y, Li W B, Wang D C, Qiu L J. Effect of population structure on prediction accuracy of soybean 100-seed weight by genomic selection., 2018, 44: 43–52 (in Chinese with English abstract).
[54] 唐友, 郑萍, 王嘉博, 张继成. 对比Bayesian B等多种方法的大豆全基因组选择应用研究. 大豆科学, 2018, 37: 353–358.Tang Y, Zheng P, Wang J B, Zhang J C. Application research for soybean genomics selection by comparing Bayesian B and other methods., 2018, 37: 353–358 (in Chinese with English abstract).
[55] 孙强, 任姣姣, 徐晓明, 李宗泽, 黄博文, 陈占辉, 吴鹏昊. 玉米株高和穗位高QTL定位和全基因组选择探究. 玉米科学, 2022, 30(4): 40–47.Sun Q, Ren J J, Xu X M, Li Z Z, Huang B W, Chen Z H, Wu P H. QTL mapping and genomic selection for plant height and ear height in maize., 2022, 30(4): 40–47 (in Chinese with English abstract).
[56] 许加波, 吴鹏昊, 黄博文, 陈占辉, 马月虹, 任姣姣. 利用F2:3家系来源单倍体定位玉米雄穗相关性状QTL及全基因组选择.作物学报, 2023, 49: 622–633.Xu J B, Wu P H, Huang B W, Chen Z H, Ma Y H, Ren J J. QTL locating and genomic selection for tassel-related traits using F2:3lineage haploids., 2023, 49: 622–633 (in Chinese with English abstract).
[57] 邱树青, 陆青, 喻辉辉, 倪雪梅, 张耕耘, 何航, 谢为博, 周发松. 水稻全基因组选择育种技术平台构建与应用. 生命科学, 2018, 30: 1120–1128.Qiu S Q, Lu Q, Yu H H, Nix M, Zhang G Y, He H, Xie W B, Zhou F S. The development and application of rice whole genome selection breeding platform., 2018, 30: 1120–1128 (in Chinese with English abstract).
[58] Wang X, Yang Z, Xu C. A comparison of genomic selection methods for breeding value prediction., 2015, 60: 925–935.
[59] Hayes B J, Bowman P J, Chamberlain A J, Goddard M E. Invited review: genomic selection in dairy cattle: progress and challenges., 2009, 92: 433–443 (in Chinese with English abstract).
[60] Habier D, Fernando R L, Dekkers J C. Genomic selection using low-density marker panels., 2009, 182: 343–353.
[61] Vinayan M T, Seetharam K, Babu R, Zaidi P H, Blummel M, Nair S K. Genome wide association study and genomic prediction for stover quality traits in tropical maize (L.)., 2021, 11: 686.
[62] Michel S, Kummer C, Gallee M, Hellinger J, Ametz C, Akgöl B, Epure D, Güngör H, Löschenberger F, Buerstmayr H. Improving the baking quality of bread wheat by genomic selection in early generations., 2018, 131: 477–493.
[63] Kumar S, Chagné D, Bink M C, Volz R K, Whitworth C, Carlisle C. Genomic selection for fruit quality traits in apple (Borkh.)., 2012, 7: e36674.
[64] Rao Y, Xiang B, Zhou X, Wang Z, Xie S, Xu J. Quantitative and qualitative determination of acid value of peanut oil using near-infrared spectrometry., 2009, 93: 249–252.
[65] 胡美玲, 郅晨阳, 薛晓梦, 吴洁, 王瑾, 晏立英, 王欣, 陈玉宁,康彦平, 王志慧, 淮东欣, 姜慧芳, 雷永, 廖伯寿. 单粒花生蔗糖含量近红外预测模型的建立. 作物学报, 2023, 49: 2498–2504. Hu M L, Zhi C Y, Xue X M, Wu J, Wang J, Yan L Y, Wang X, Chen Y N, Kang Y P, Wang Z H, Huai D X, Jiang H F, Lei Y, Liao B S. Establishment of near-infrared reflectance spectroscopy model for predicting sucrose content of single seed in peanut., 2023, 49: 2498–2504 (in Chinese with English abstract).
[66] Eynard S E, Croiseau P, Laloë D, Fritz S, Calus M P L, Restoux G. Which individuals to choose to update the reference population? Minimizing the loss of genetic diversity in animal genomic selection programs., 2018, 8: 113–121.
Research on oil content screen with genomic selection and near infrared ray in peanut (L.)
LU Qing, LIU Hao, LI Hai-Fen, WANG Run-Feng, HUANG Lu, LIANG Xuan-Qiang, CHEN Xiao-Ping, HONG Yan-Bin, LIU Hai-Yan, and LI Shao-Xiong*
Crops Research Institute, Guangdong Academy of Agricultural Sciences / Guangdong Provincial Key Laboratory of Crop Genetic Improvement / South China Peanut Sub-Center of National Center of Oilseed Crops Improvement, Guangzhou 510640, Guangdong, China
Oil content is a crucial trait for the yield of oil per unit area in peanut. This trait is controlled by multiple minor genes, and its avaliable tightly linked markers are very limited, resulting in low breeding accuracy in traditional molecular marker assisted selection. Genomic selection (GS), as a new breeding method, could achieve early prediction of quantitative traits. Near infrared ray (NIR) technology can non-destructively detect seed quality traits, such as oil content. By combining the advantages of the two breeding technologies, we have established a breeding technology that combined GS and NIR for breeding peanut oil content, and explored the factors that affected the accuracy of GS for peanut oil content. This study lays a theoretical foundation for peanut molecular breeding. Here, a total of 216 recombinant inbred lines were used as a training population. The F2(139), F3(464), and F4(505) were used to construct the breeding populations. Genotyping was carried out using the self-developed “PeanutGBTS40K” liquid chip. The breeding application of oil content was conducted using a GS and NIR jointed breeding technology, and evaluated its breeding effects. The results showed that after genotyping the training population, a total of 30,355 high-quality SNPs were obtained, and used for 11 GS models selection analyses. The rrBLUP model showed the highest accuracy, followed by randomforest and svmrbf. The GS prediction accuracy of F2, F3, and F4was 0.116, 0.128, and 0.119, respectively, using recombinant inbred lines as the training population. Accordingly, the prediction accuracy was 0.116, 0.131, and 0.160, respectively, using a superimposed training population. Compared with the GS, the GS-NIR can improve oil content by 1.8%, 2.7%, and 3.4% for each generation. Compared with the NIR, there was no significant difference (0.1%, 0.06%, and 0.07%). Compared with the GS, the NIR can significantly improve oil content by 1.7%, 2.6%, and 3.3% for each generation. Through the combined technologies, compared to F2, the oil content of F3and F4increased by 1.2% and 1.0%, respectively. Finally, a total of 16 improved lines were obtained in F4, of which 10 lines had oil content ≥ 55.0%. Among them, two lines (SF4_201 and SF4_379) had a theoretical yield increase of 7.0% and 11.1%, respectively, compared to the control variety. This study suggested that oil content could be effectively improved through GS combined with NIR in peanut.
peanut (L.); oil content; genomic selection; near infrared ray; genomic breeding value
10.3724/SP.J.1006.2024.34115
本研究由2022年省级乡村振兴战略专项资金种业振兴项目(2022-NPY-00-022), 广东省重点领域研发计划项目-现代种业(2020B020219003, 2022B0202060004), 财政部和农业农村部国家现代农业产业技术体系建设专项(花生, CARS-13), 广东省农业科学院农业优势产业学科团队项目(202104TD)和广东省农业科学院协同创新中心项目(XTXM202203)资助。
This study was supported by the Special Funds for the Revitalization of Agriculture through Seed Industry under the Provincial Rural Revitalization Strategy (2022-NPY-00-022), the Guangdong Provincial Key Research and Development Program-Modern Seed Industry (2020B020219003, 2022B0202060004), the China Agriculture Research System of MOF and MARA (Peanut, CARS-13), the Agricultural Competitive Industry Discipline Team Building Project of Guangdong Academy of Agricultural Sciences (202104TD), and the Project of Collaborative Innovation Center of GDAAS (XTXM202203).
李少雄, E-mail: lishaoxiong@gdaas.cn
E-mail: luqing2016@126.com
2023-07-06;
2023-10-23;
2023-11-09.
URL: https://link.cnki.net/urlid/11.1809.S.20231108.1431.002
This is an open access article under the CC BY-NC-ND license (http://creativecommons.org/licenses/by-nc-nd/4.0/).

