基于多粒度网络预测增强子-启动子相互作用
刘志豪, 王会青, 李浩琳, 韩家乐
(太原理工大学信息与计算机学院, 太原 030600)
增强子-启动子相互作用(EPⅠs)是指启动子与增强子跨越基因组发生协同作用,控制组织特异性基因表达的过程[1]。EPⅠs 失效将造成基因表达的破坏,甚至诱发严重疾病[2]。阿尔茨海默病(AD)的疾病变异风险与富集在小胶质细胞(大脑的主要免疫细胞)中的EPⅠs 变异率高度相关[3]。此外,通过对造血干细胞中的增强子或启动子位点进行基因编辑[4],增强EPⅠs 的表达水平,能够使红细胞中血红蛋白的表达持续增加,实现β地中海贫血疾病[5]的终生治疗。准确识别EPⅠs 对疾病来源追踪和发展基因疗法有重要意义。
近年来,深度学习方法通过增强子、启动子序列信息实现EPⅠs 的二分类预测,区分不同细胞系下的增强子-启动子相互作用。增强子、启动子序列不仅包含碱基级特征信息,还包含转录因子(TF)、共调节因子、染色质结构蛋白、调控元件[6]等元件级别的特征信息,从序列中提取这些不同层级的生物特征信息能有效预测EPⅠs。Jing 等[7]使用卷积神经网络(CNN)提取多个细胞系的碱基特征,并设计迁移学习的梯度反转层以减少细胞系特异性特征,用于跨细胞系的EPⅠs 预测。Min 等[8]提出的匹配启发式机制能够对提取的碱基特征和部分短基序特征进行特征强化,有效预测EPⅠs。Mao 等[9]对增强子、启动子序列进行注意力判别,识别过度表达的TF 结合位点、TF 对相互作用(TFs-pair Ⅰnteractions)等元件级特征,用于预测EPⅠs。以上研究通过增强模型对碱基层级特征或元件层级特征的提取能力,提升EPⅠs 预测效果,但未考虑碱基层级特征和元件层级特征对EPⅠs 预测的互补效果,缺乏对两种级别生物特征的联合分析。将原始序列粒化为细粒度和粗糙粒度,有助于提取碱基层级特征和元件层级特征,从不同层级表示、分析原始序列。因此,本文引入增强子、启动子序列的细粒度和粗糙粒度分别提取碱基层级、元件层级特征,通过融合不同层级间特征提升模型学习能力。
通过粒度选择,确定合适的粒度大小,有助于减少不同粒度间的冗余信息,提升模型的学习质量和学习效率[10]。Dong 等[11]为处理冗余特征和无关特征,提出基于粒度信息的特征选择算法模型,并根据分类精度的反馈自适应地找到最优粒度参数,获得更高的分类精度。Lin 等[12]通过邻域粗糙集获得包括不同粒度的所有特征排名,根据交叉熵算法的反馈选择最优特征子集。上述方法通过模型反馈,在候选粒度集中选择最优粒度或最优特征子集,避免学习过多冗余特征,有效提高任务的决策能力。为避免后续联合分析中学习过多冗余信息,本文需要选择合适的粗糙粒度。然而,由于增强子、启动子具有细胞特异性[13],不同细胞系的生物表现不同,序列组成也存在一定差异[14],在不同细胞系下选取相同的粗糙粒度,不利于学习不同细胞系的细胞特异性特征。因此,本文通过分类精度反馈从粗糙粒度候选集选取最优粗糙粒度,避免后续联合分析中提取过多冗余特征。此外,考虑到EPⅠs 的细胞特异性,对6 个细胞系分别进行粒度选择,选定不同细胞系的最优粗糙粒度,便于提取细胞特异性特征。
EPⅠs 的驱动过程涉及增强子的内部关联信息、启动子的内部关联信息、增强子-启动子间的互关联信息。通过提取序列的全局特征,捕获这3 种关联信息,能有效辅助EPⅠs 预测。Singh 等[15]提出SPEⅠD模型,使用CNN 和长短期记忆网络(LSTM)的混合结构提取全局特征,预测EPⅠs。Zhuang 等[16]提出的EPⅠsCNN,仅使用与SPEⅠD 相同的 CNN 结构,就得到与SPEⅠD 相似的性能,表明SPEⅠD 的预测能力主要源于对局部特征的提取,直接提取原始序列的全局特征的效果不理想。Zeng 等[17]提出EP2vec,将原始长序列拆分为固定短序列,结合梯度提升决策树预测EPⅠs,能更好地捕获全局信息,但对序列的拆分、填充造成部分特征丢失。此外,为提取局部特征,以上研究使用两个分支结构分别处理增强子、启动子序列,难以提取增强子-启动子间的关联信息。针对不同的特征提取任务,分别设计特定的子网络,能够避免不同信息流的学习干扰。因此,本文采用CNN 子网络和双层双向门循环单元(BiGRU)注意子网络,分别提取局部特征、全局特征,避免特征干扰。在双层BiGRU 注意子网络,使用掩源元件子序列和元件-全局递进策略,同时提取增强子内部关联、启动子内部关联和增强子-启动子间互关联等多种关联信息,获取更全面的全局特征。
综上,本文提出EPⅠs 预测模型EPⅠ-PBGA,在6 个细胞系分别进行粒度选择,确定最优粗糙粒度,并使用双层BiGRU 注意子网络和CNN 子网络分别提取序列的不同粒度特征。CNN 子网络使用双分支结构分别提取增强子、启动子原始序列的细粒度特征。双层BiGRU 注意子网络引入元件-全局递进策略处理掩源元件子序列,捕获多种元件特征关联作为粗糙粒度的全局关联特征。实验结果表明:同一细胞系下,选择不同粗糙粒度的模型表现出明显性能差异,提升了模型识别不同细胞系的细胞特异性特征的能力;EPⅠ-PBGA 在6 个不同细胞系数据集表现出较好性能,能够有效预测EPⅠs。
1 EPⅠ预测模型EPⅠ-PBGA
捕获不同粒度序列信息的EPⅠs 预测模型EPⅠ-PBGA 如图1 所示,使用双层BiGRU 注意子网络、CNN 子网络两个子网络。CNN子网络使用双分支结构提取细粒度特征信息。双层BiGRU 注意子网络处理元件子序列,提取粗糙粒度特征,并融合多种元件级特征关联信息来获取全局特征。

图1 EPⅠ-PBGA 模型架构Fig.1 Model framework of EPⅠ-PBGA
1.1 粗糙粒度选择
掩源元件子序列划分策略可寻找不同细胞系下粗糙粒度的最优选。原始序列经分割处理形成的多个子序列被称为元件子序列,模型接收不同尺度的元件子序列,根据分类精度确定最终的最优粗糙粒度。本文对元件子序列进行掩源处理,由增强子、启动子原始序列分别均匀划分得到的多个元件子序列,并不区分为启动子子序列或增强子子序列,而是掩盖来源进行混合处理,视作一个统一的元件子序列集SIn。掩源处理使双层BiGRU 子网络有能力同时学习增强子-增强子元件关联、启动子-启动子元件关联、增强子-启动子元件关联等3 种关联特征。由于本文将局部特征的提取交由CNN 子网络,双层BiGRU 子网络仅关注全局特征的获取,因此不必担心切分序列带来的特征丢失问题。
已知增强子、启动子的核心元件以及涉及的基因表达位点多处于几十bp 到几百bp 之间[6]。因此,将元件子序列的长度区间设定为50~500。通过多次不同设置的元件子序列切分操作,寻找在双层BiGRU 注意子网络中表现最好的元件子序列,视作该细胞系下的最优元件子序列。SIn由原始长序列划分得到,如式(1)所示,每个EPⅠs 序列划分为i个元件子序列,每个元件子序列将包含LSi个碱基。其中,元件子序列的个数i由增强子、启动子合并序列的总长度(L=5 000)和元件子序列长度LSi共同决定。通过实验,从特定集合中选取表现最优的LSi,作为该细胞系下的最优元件子序列长度,具体元件子序列长度集如式(2)所示。此外,受EPⅠs 的细胞特异性影响,不同细胞系下的最优元件子序列长度也可能不相同,需要分别在不同细胞系下确定最优元件子序列。
启动子、增强子序列是由4 个碱基组成的基因组序列:'A'(腺嘌呤)、'G'(鸟嘌呤)、'C'(胞嘧啶)和'T'(胸腺嘧啶)。由于每个向量之间的信息相互独立,将过长序列编码直接为独热向量(one-hot)使模型无法捕获序列中隐藏的关联信息。因此,本文使用模块dna2vec[18]处理切分后的元件子序列,dan2vec 在word2vec 词嵌入模型的基础上改进。相较于word2vec,dna2vec 使用人类基因组序列作为语料学习库,专用于DNA 序列编码。使用dna2vec 可以将 k-mers[19]处理后的DNA 序列嵌入到 100 维的连续向量空间,获得低维和高质量的向量。最终,每个元件子序列Si被编码为LSi*100 维的矩阵向量。
1.2 双层BiGRU 注意子网络
预处理得到的元件子序列集SIn被输入双层BiGRU 注意子网络,并通过元件特征提取层提取元件级特征,然后捕获多个元件级特征的潜在关联,获取基于粗糙粒度视角的全局关联特征。
首先,多个元件子序列被输入双层BiGRU 注意子网络的第一层作为元件特征提取层,通过独立的BiGRU 注意子模块分别处理不同的元件子序列,以提取元件子序列的碱基级特征。BiGRU 是一种双向循环神经网络,通过使用前向和后向两个方向的隐藏态,更好地理解序列上下文信息。增强子、启动子存在的双向转录[20]现象,因此必须从正向和反向两个角度看待元件子序列。BiGRU 的更新过程如式(3)~(7),由当前时刻输入和之前时刻输入ht−1共同决定ht。zt表示更新门向量,决定信息保留程度,rt表示重置门向量。其中,U是保留前一个隐藏状态向量ht−1的权重矩阵,ht−1是t时间的输入,· 是逐元素乘法,下标h、z、r分别表示为当前时刻、更新门和重置门。BiGRU 从正向和反向方向分别接受处理后的元件子序列,从而捕获双向特征的长期依赖关系,通过处理双向序列得到前向元件特征向量{h1,h2,···,ht,···,hL}和后向元件特征向量 {} ,合并为最终输出ht,如公式(7)所示,其中W和U均为权重向量;b为偏置向量;xt、ht分别为t时刻的输入向量和隐藏层状态;zt为更新门,rt为重置门。
为每个元件子序列分别赋予一个BiGRU 注意力模块,使模型对不同元件子序列独立处理,获取元件子序列的内部权重分数。本文获取的子序列特征向量与注意力分数加权,在首层得到最终的元件级特征向量Im,具体如公式(8)~(10)所示。其中,经BiGRU 处理生成输出hft,uft是hft经过单层MLP 得到的潜在表示。然后判断uft与上下文向量uω的相似度。并通过softmax 函数得到一个归一化的的重要性权重 αft。最终,计算 αft和hft的加权和得到最终的元件向量It,其中t∈(1,M) 。
因此,元件子序列集SIn经过元件特征提取层提取内部关联信息,被编码为元件特征向量集(I1,I2,···,It,···,Im)。
为捕获元件特征向量之间的潜在关联信息,在双层BiGRU 注意子网络的尾层,即元件-全局关联层接收元件特征向量集 (I1,I2,···,It,···,IM) 。模型将全部元件特征向量集 (I1,I2,···,It,···,IM) 交由一个共用的BiGRU 注意模块处理,将来自增强子和启动子的子序列打乱顺序后,均视作元件子序列集,使模型能够捕获增强子-增强子、启动子-启动子关联信息外,还有能力提取增强子-启动子间的关联信息增强模型提取全局关联信息的能力。
在全局关联层使用与元件特征提取层大体相同的BiGRU 注意模块,仅进行参数上的调整。通过注意力机制获取多个元件级特征向量间的注意力权值,区分元件特征向量对全局特征的重要程度,将特征差异较小的元件特征向量边缘化。具体如公式(11)~(12)所示,多个元件子序列向量集(I1,I2,···,It,···,IM)作为输入,交由全局关联层的BiGRU 注意模块处理,扁平向量V1是全局关联层的输出,同样也是双层BiGRU 注意子网络的最终输出,集成了粗糙粒度捕获的增强子、启动子元件间的多种潜在关联特征。
其中Ipt为注意力机制。
1.3 CNN 子网络
在细粒度视角,主要关注增强子、启动子序列的局部特征,如碱基、部分特定的子序列及用于结合蛋白质的TFs 等基序[21],这些基序能够促进EPⅠs。CNN 网络接收原始的增强子、启动子长序列,增强子和启动子原始序列分别被dna2vec 被编码为3 000×100 维、2 000×100 维的二维矩阵,作为网络输入,使用CNN 模块和BiGRU 注意模块的混合结构[22],提取细粒度层次下的高维特征信息。
由于在细粒度视角无需关注增强子-启动子间的关联关系,为了更好提取细粒度特征,CNN 子网络分离增强子、启动子学习通道。其中,CNN 模块包含一个卷积层和一个最大池化层,用于提取序列的局部特征。为保留主要特征,减少参数和计算量,本文使用一个最大池化层进行下采样,降低特征的输入维度。BiGRU 注意模块用来捕获处理经过CNN 模块提取的局部特征向量存在的上下文依赖关系,注意力机制用来识别细粒度层级的重要特征。
在CNN 子网络的末端,分别代表增强子和启动子的特征向量Ve和Vp合并为扁平V2,代表从增强子和启动子序列包含提取的细粒度特征信息。V2与双层BiGRU 注意子网络得到的向量V1合并输入全连接层,通过函数sigmoid 进行最终的EPⅠs 预测。
2 实验结果与分析
2.1 EPIs 数据集
本文使用来自 EPⅠANN[9]的基准数据集,如表1所示。该数据集包含6 种不同的细胞系,即GM12878(淋巴母细胞)、HeLa-S3(宫颈癌患者的外胚层细胞)、ⅠMR90(胎儿肺成纤维细胞)、K562(白血病患者的中胚层系细胞)、HUVEC(脐静脉内皮细胞)和 NHEK(表皮角质形成细胞)。6 个细胞系的正样本(真正的 EPⅠ)和负样本(非EPⅠ)比例约为1∶20,在EPⅠANN 的基准数据集中,增强子和启动子经过基因组扩选预处理,均分别扩展为2 000、3 000的定长序列并进行数据平衡处理[8]。对于同一细胞系的数据集,本文将阳性样本和阴性样本分别按9∶1 的比例分为初始训练集和测试集,并将初始训练集中的10%样本数据作为验证集,剩下的作为训练集,用于模型的调整和评估。
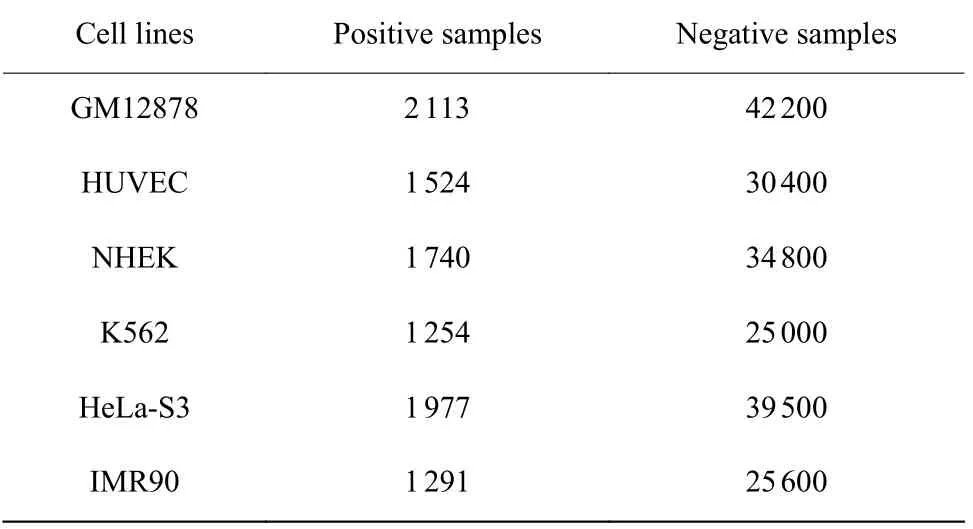
表1 6 个细胞系的EPⅠs 数据集Table 1 EPⅠs dataset in six cell lines
2.2 模型参数选择
由于本文使用的增强子、启动子序列过长,对多个数据集进行学习与评估需要较长的实验周期,本文综合实验评估标准和模型学习效率,对超参数进行调整,以减少参数量,提升学习效率。其中,在双层BiGRU 注意子网络的元件特征提取层,BiGRU 维度设置为50;在元件-全局关联层,BiGRU 维度设置为100。在CNN 子网络中,启动子和增强子的CNN卷积核设置均为40,滤波器为16;在最大池化层,将增强子、启动子池化步长分别确定为15、10;BiGRU维度设置为 50。训练批次epoch 设置为60,batchsize 设置为32,初始学习率设置为 5e−6,损失函数为交叉熵损失函数(binary_crossentropy),并使用0.5 的dropout 和批归一化,提高训练的稳定性。
2.3 粗糙粒度选择
为验证粗糙粒度选择对粗糙粒度编码模块提取特征信息的影响。通过选取不同的Length,改变粗糙粒度尺度并进行对比实验。在粒度选择分析中,EPⅠ-PBGA 的其他模块保持不变,根据性能表现确定最优粗糙粒度,结果如表2 所示,加粗部分为选取的最优粗糙粒度。

表2 不同细胞系下的粒度选择Table 2 Particle size selection under different cell lines
由表2 可知,不同的Length 对模型的性能表现有明显影响。在GM12878、ⅠMR90、HeLa-S3、 HUVEC数据集中,表现最好的Length 为100,且相较于50、200、500 等长度,其受试者工作特征曲线下面积(AUROC)、精准率-召回率曲线下面积(AUPR)、精准率和召回率的调和平均数(F1)分数均有明显提升,这说明粗糙粒度选择策略有效增强EPⅠ-PBGA 对粗糙粒度特征的学习能力。在K562 数据集中,当Length 为200 时EPⅠ-PBGA 有最佳的性能表现。这是由于不同细胞系下的增强子、启动子有较明显的EPⅠs 的细胞特异性,生物特征存在一定差异[14],导致特定细胞系下适合模型的Length 不同。此外,在NHEK 细胞系中选择不同的Length 时EPⅠ-PBGA 并没有表现出明显的性能差异,这说明NHEK 细胞系中,粗糙粒度选择并没有提升EPⅠ-PBGA 对粗糙粒度特征的学习能力。综上可知,对于大多数数据集,经过最优粗糙粒度选择的模型在性能表现上有所提升,提升了EPⅠ-PBGA 对粗糙粒度特征的学习能力,验证了粗糙粒度选择的必要性。
2.4 消融实验
为验证使用不同粒度的编码模块对模型的影响,选取HUVEC、ⅠMR90、NHEK 数据集对Fine(仅使用CNN 子网络)、Coarse(仅使用双层BiGRU 注意子网络)、Fine+Coarse(本文模型)进行消融实验,其中Fine+Coarse 和Coarse 均使用最优粗糙粒度。
本文以AUROC 作为模块贡献度的主要评价标准,由表3、表4 的性能表现来看,在HUVEC 细胞系中,Coarse 表现出更高的贡献度;在ⅠMR90 细胞系中,Fine 表现出更高的贡献度。在HUVEC、ⅠMR90数据集中,融合两种粒度信息的Fine+Coarse 性能比仅使用Fine 或Coarse 要好,验证了在HUVEC、ⅠMR90细胞系中Fine+Coarse 能有效融合不同粒度特征。此外,Fine+Coarse 并不适用于全部细胞系,通过比较AUROC 可知,在表5 的NHEK 细胞系中,选择最优粗糙粒度的Fine+Coarse的EPⅠ-PBGA 仅与Fine 持平,这表明在NHEK 细胞系仅在细粒度就存在丰富的特征信息,融合两种粒度特征并没有使EPⅠ-PBGA 学习到更丰富的特征信息。尽管在不同细胞系下细粒度和粗糙粒度对模型的贡献度有一定差异,但在绝大多数细胞系,使用Fine+Coarse 的模型相较于仅使用Fine 或Coarse 的模型表现出一定性能优势,提升了模型对增强子、启动子序列的学习能力。

表3 HUVEC 细胞系下的消融实验Table 3 Ablation experiment in HUVEC cell line
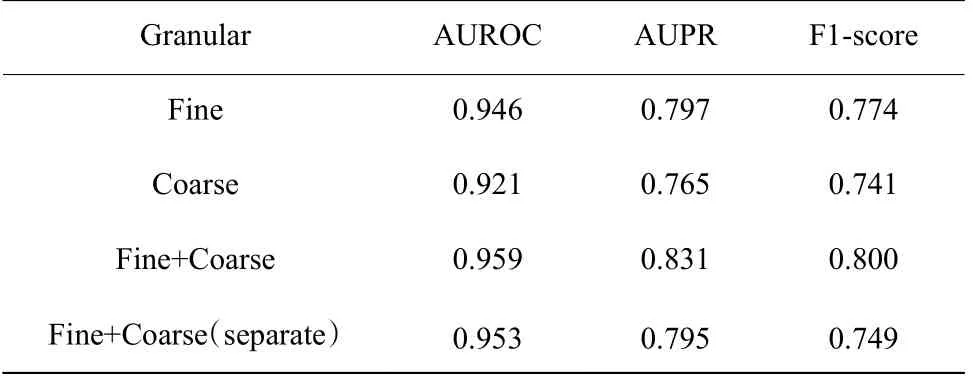
表4 ⅠMR90 细胞系下的消融实验Table 4 Ablation experiment in ⅠMR90 cell line

表5 NHEK 细胞系下的消融实验Table 5 Ablation experiment in NHEK cell line
在粗糙粒度编码模块中,本文通过元件子序列掩源处理的策略,捕获增强子和启动子之间的关联信息。为验证其有效性,在Fine+Coarse(separate)去除掩源元件子序列的处理,并在粗糙粒度分离增强子、启动子的学习过程。通过表3、表4、表5 可知,对比于Fine+Coarse(separate),Fine+Coarse 在HUVEC和ⅠMR90 细胞系的性能表现均有所提升;在NHEK细胞系中Fine+Coarse(separate)与Fine+Coarse的性能表现相近,这是由于NHEK 细胞系对粒度融合策略并不敏感。在HUVEC 和ⅠMR90 细胞系的性能表现验证了在大多数数据集中,元件子序列掩源处理的有效性。
2.5 对比实验
为了评估EPⅠ-PBGA 的有效性,使用采用了最优粗糙粒度的EPⅠ-PBGA 与SPEⅠD[15]、EPⅠsCNN[16]、EPⅠANN[17]、EPⅠDLMH[8]等EPⅠs 预测模型进行比较。结果如表6、表7 和表8 所示。EPⅠ-PBGA 在大部分细胞系中的性能表现总体优于对比方法。

表6 不同方法在6 个细胞系数据集下的AUROCTable 6 Performance of different methods in terms of AUROC on six cell lines
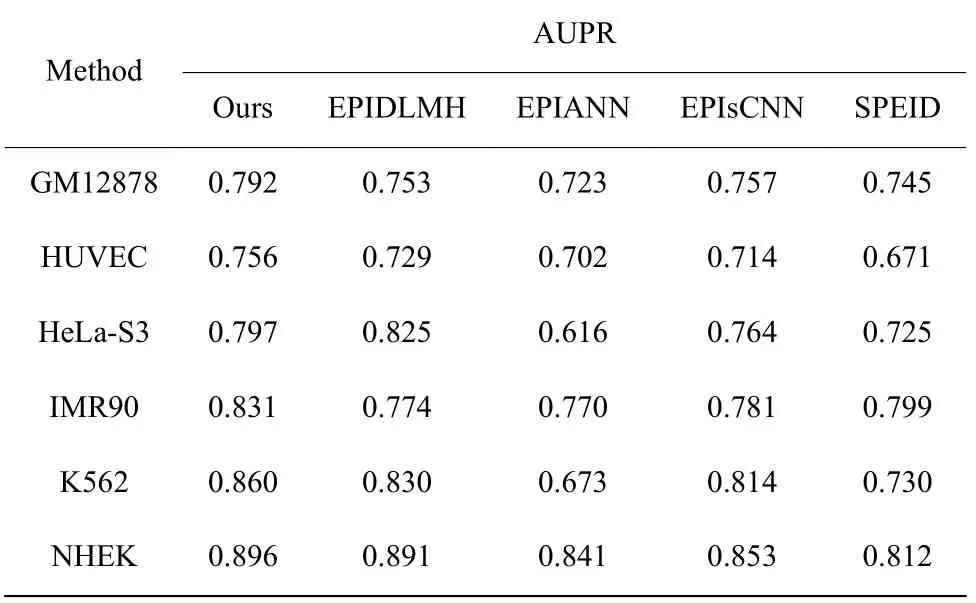
表7 不同方法在6 个细胞系数据集下的AUPRTable 7 Performance of different methods in terms of AUPR on six cell lines

表8 不同方法在六个细胞系数据集下的F1 分数Table 8 Performance of different methods in terms of F1-score on six cell lines
在以上对比方法中,SPEⅠD、EPⅠsCNN 均使用大量卷积,用于强化碱基、短基序等碱基级特征的提取能力;EPⅠDLMH 在SPEⅠD、EPⅠsCNN 等研究的基础上,在模型尾部通过启发式匹配机增强提取的高维特征,使EPⅠDLMH 在绝大部分数据集均优于SPEⅠD和EPⅠsCNN。而EPⅠANN 则通过建立增强子和启动子中的配对短区域,识别出更多的TFs 结合位点和TFs 相互作用等元件层级特征,用于预测EPⅠs。本文模型在大多数据集的性能表现优于亚军模型EPⅠDLMH,这是由于本文通过粒度选择提升模型对粗糙粒度特征的提取能力。由表2 可知,选择不同Length 的模型性能表现存在差异。相较于不同对比模型来说,在HUVEC 细胞系选择Length 为100 的本文模型的性能表现优于亚军模型EPⅠDLMH,选择Length 为50、200 的本文模型,其性能表现与EPⅠDLMH的性能表现相近;对比表2 和表6、表7、表8,例如在K562细胞系选择Length 为200 的本文模型的性能表现优于亚军模型EPⅠDLMH,当Length 为50、100、500 时本文模型的性能表现则低于EPⅠDLMH等方法。这说明选择合适的粗糙粒度能够有效提升模型学习质量,不合适的粗糙粒度反而影响模型学习。同时通过细粒度和粗糙粒度编码模块,有效融合不同层级特征,提升模型对序列的不同层级特征的学习能力。
3 结 论
本文提出双层BiGRU 注意网络EPⅠ-PBGA,在细粒度和粗糙粒度捕获多层次特征信息。通过使用掩源子序列划分策略,根据分类精度进行粒度选择,获取不同细胞系下的最优粗糙粒度。在粗糙粒度,双层BiGRU 注意子网络通过元件-全局策略处理掩源元件子序列,同时学习启动子-启动子、增强子-增强子、增强子-启动子元件间关联,而增强子-启动子元件间关联在过往研究中往往被忽略。此外,不同于SPEⅠD 和EPⅠsCNN 等方法使用大量卷积操作(1 024 个滤波器)提升了模型学习成本,本文通过两个子网络学习互补特征,使模型在保证学习能力的基础上,减少参数量和学习周期。但是,该模型存在一定的局限性,粒度选择依赖于参数的调整与实验设计,设计简单高效的粒度选择算法将是今后的研究方向。

