大豆球蛋白G3A1b抗原表位的预测及初步定位
段宇莹,席俊,王一超,付杨,吴枭,孙富宇,陈珍妮
河南工业大学 粮油食品学院,河南 郑州 450001
0 引言
食物过敏是当今世界食品安全的主要问题之一,据统计,全球约有2.5亿人遭受食物过敏的影响[1]。大豆是一种营养价值较高的高蛋白食品,日常食用可降低心血管疾病患病风险[2],但其引发的过敏问题不容小觑。目前有超过20种大豆蛋白被认为是过敏原,其中大豆球蛋白和β-伴大豆球蛋白约占大豆蛋白总过敏原的80%[3]。
大豆球蛋白由5个亚基(G1—G5)组成,相对分子质量约为 300~380 kDa,是由含酸性多肽链和碱性多肽链的亚基组合而成的六聚体[4]。研究[5]表明,酸性多肽链比碱性多肽链的抗原性高。蛋白质的抗原性是指抗原与其所诱导产生的抗体或致敏淋巴细胞特异性结合的能力,而抗原表位是认识蛋白质抗原性的基础[6],降低大豆球蛋白的抗原性需要定位其抗原表位。生物信息学软件作为目前常用的定位蛋白质抗原表位的生物工程软件,具有准确、可信度高、成本低等优势。付杨等[6]采用多个生物信息学软件,在G1亚基 A1a酸性多肽链中定位了24EQPQQN29等10个线性抗原表位。刘阳星月等[7]选取G2亚基的酸性多肽链A2作为研究对象,利用生物信息学方法,定位了37KPDNRIE43等8个B细胞的线性表位。陈慧彬等[8]在G5 亚基 A3 酸性多肽链中定位了90FEK-LQD109等6个线性表位。上述研究初步判定了大豆球蛋白潜在抗原表位的所在位点,对大豆球蛋白部分亚基的抗原性研究具有一定的推动作用。另外,王一超等[9]在所预测的抗原表位基础上,采用噬菌体展示等技术初步定位了G5 亚基A3 酸性多肽链被加工破坏的抗原表位,为精确定位大豆球蛋白致敏位点提供了依据。
综上所述,针对大豆球蛋白G1、G2、G5致敏亚基酸性多肽链的抗原性研究均有一定的进展,但目前国内并无针对G3亚基A1b酸性多肽链(G3A1b多肽链)抗原性的研究。基于此,本研究拟先根据A1b多肽链的氨基酸序列,重叠分段并设计合成引物,构建克隆载体和重组噬菌体;再利用T7噬菌体展示技术[11-14]表达目的蛋白,通过间接ELISA法检测抗原性,并制备热加工特异性吸收抗体鉴定被破坏的抗原表位,以期为后续G3A1b多肽链致敏表位的精确定位提供参考,为降低大豆球蛋白的抗原性提供理论指导,推动食品产业采用加工技术降低或去除大豆致敏性,制造让大豆过敏人群放心食用的食品。
1 材料与方法
1.1 主要材料与试剂
DNA 回收试剂盒、质粒提取试剂盒,天根生化科技有限公司;G3A1b多肽链、3个重叠分段(片段 1、 2、 3 )的上下游引物、IPTG、X-Gal,生工生物工程(上海)股份有限公司;感受态细胞E.coliJM109 Competent Cells、pMDTM18-T vector Cloning Kit、DNA Marker、T4 DNA Ligase、QuickCutTMEcoR Ⅰ、 QuickCutTMHind Ⅲ、 Ex-TaqTMVersion 2.0 ,宝日医生物技术(北京)有限公司;噬菌体试剂盒,美国Novagen公司;氨苄青霉素(Amp+)、羧苄青霉素(Carb+)、TMB单组分显色液,索莱宝科技(北京)有限公司;大豆球蛋白兔源多抗血清,参考姚利利等[15]的方法制备;羊抗兔IgG抗体,兰杰柯科技(北京)有限公司。
1.2 主要仪器与设备
Mastercycler nexus GSX1型PCR仪,德国Eppendorf 公司;Tanon-2500型凝胶成像系统,上海天能科技有限公司;LDZX-75KBS型立式压力蒸汽灭菌锅,上海申安医疗器械厂;SW-CJ-ID型超净工作台,苏州净化设备有限公司;SPX-150型生化培养箱,北京市永光明医疗器械有限公司;THZ-92A型台式恒温振荡器,上海跃进医疗器械有限公司;HH-1型恒温水浴锅,国华电器有限公司;D3024R型离心机,美国SCILOGEX;Multiskan FC型酶标仪,赛默飞世尔上海仪器有限公司。
1.3 实验方法
1.3.1G3A1b多肽链抗原表位预测使用NCBI (https:∥www.ncbi.nlm.nih.gov/nuccore/)查询大豆球蛋白G3A1b多肽链的氨基酸残基序列(共275个氨基酸):FREQPQQNECQIQRLNALKPDNRIESEG-GFIETWNPNNKPFQCAGVALSRCTLNRNALRRPSYT-NAPQEIYIQQGSGIFGMIFPGCPSTFEEPQQKGQSSRP-QDRHQKIYHFREGDLIAVPTGFAYWMYNNEDTPVV-AVSLIDTNSFQNQLDQMPRRFYLAGNQEQEFLQYQP-QKQQGGTQSQKGKRQQEEENEGGSILSGFAPEFLEH-AFVVDRQIVRKLQGENEEEEKGAIVTVKGGLSVISPP-TEEQQQRPEEEEKPDCDEKDKHCQSQSRN。
在PDB蛋白数据库中未找到X-射线晶体衍射构建的大豆球蛋白G3亚基立体结构,因此,本研究使用SWISS-MODEL在线预测软件构建G3A1b多肽链的三维结构,并根据该软件所得拉氏图(Ramachandran Plot)对所构建的蛋白质三级结构模型进行稳定性评估。使用SOPMA软件对G3A1b多肽链进行二级结构预测,利用DNAStar、BepiPred和ABCpred服务器对G3A1b多肽链进行线性表位预测,通过DiscoTope 2.0 Server服务器完成G3A1b多肽链构象表位的预测。
1.3.2G3A1b多肽链重叠分段及引物设计根据1.3.1综合预测结果,对大豆球蛋白G3A1b多肽链进行重叠分段,按照所公布的G3A1b多肽链序列将其分为3段,设计引物后使用Primer Premier 6.0等软件评估其可靠性。
1.3.3G3A1b多肽链基因扩增基因扩增PCR反应体系(50 μL)为:无菌水22 μL,上游引物1 μL,下游引物1 μL,模板1 μL,Ex-Taq premix 25 μL。反应程序设定为:94 ℃预变性5 min;94 ℃变性1 min,55 ℃退火1 min,72 ℃延伸1.5 min,设定30次循环;72 ℃再延伸10 min;4 ℃保存。对所得PCR产物进行2%琼脂糖凝胶电泳检测[16]。参照DNA回收试剂盒说明书回收G3A1b多肽链目的基因片段,回收后置于-20 ℃冰箱中保存[17]。
1.3.4G3A1b多肽链克隆载体构建、筛选及鉴定取4 μL按1.3.3方法所得DNA溶液,与1 μL pMDTM18-T vector载体、5 μL solution Ⅰ构建连接体系,于16 ℃恒温箱内过夜;将连接产物与E.coliJM109感受态细胞混合,冰浴30 min,于42 ℃条件下热激1.5 min后,再冰浴5 min,并加入SOC培养基混匀,于37 ℃摇床内振荡培养1 h;于4000 r/min 条件下离心4 min后,吸取400 μL上清液弃去;将剩余液体倒入固体培养基(含50 μg/mL Amp+,50 mg/mL IPTG,20 mg/mL X-Gal)内涂匀,待凝固后于37 ℃恒温培养箱内培养12~16 h;挑取白色单菌落,提取重组质粒并进行PCR鉴定(反应程序设定同1.3.3)和双酶切鉴定。
PCR鉴定体系(25 μL)为:无菌水10 μL,重组质粒1 μL,上游引物1 μL,下游引物1 μL,Ex-Taq premix 12 μL。双酶切反应体系(20 μL)为:EcoR Ⅰ 1 μL,Hind Ⅲ 1 μL,10 × Green Buffer 2 μL,重组质粒5 μL,无菌水11 μL。37 ℃水浴50 min。均使用2%琼脂糖凝胶电泳鉴定所得PCR产物。回收双酶切反应产物并测定其浓度、纯度,以确定所克隆DNA序列的准确性。于-20 ℃保存剩余回收双酶切反应产物。
1.3.5 目的基因噬菌体包装及鉴定1)噬菌体包装。取1.3.4所述方法回收的双酶切反应产物,与噬菌体载体臂构建包装体系[9],混匀后于16 ℃恒温箱内温育12~16 h以获得包装产物。在该产物中添加5 μL T7包装提取物并混匀,于22 ℃恒温培养箱内温育2 h后,加入54 μL LB液体培养基,终止反应。
2)重组噬菌体滴度测定。参照王一超等[9]的方法测定重组噬菌体的滴度。
3)重组噬菌体PCR鉴定。挑取1个噬菌斑溶解于20 μL TE溶液中,于65 ℃条件下加热10 min后,于12 000 r/min条件下离心1 min,取上清液进行PCR鉴定。重组噬菌体PCR鉴定体系(25 μL)为:无菌水10 μL,裂解质粒2 μL,T7 Select up primer 0.5 μL,T7 Select down primer 0.5 μL,Ex-Taq premix 12 μL。重组噬菌体PCR鉴定程序为:80 ℃预变性5 min;94 ℃ 变性50 s,55 ℃退火1 min,72 ℃延伸1 min,设定35次循环;72 ℃延伸6 min;4 ℃保存。对所得PCR产物进行2%琼脂糖凝胶电泳检测。
4)重组噬菌体扩增、保存及表达蛋白纯化。参照王一超等[9]的方法对重组噬菌体进行扩增、保存及表达蛋白纯化。
1.3.6 热加工破坏抗原表位特异性吸收抗体制备参考J.Xi等[18]的方法热加工处理大豆球蛋白抗原。大豆球蛋白质量浓度10 mg/mL,温度110 ℃,时间50 min,此时大豆球蛋白的抗原性最低。将热加工处理后的大豆球蛋白抗原与大豆球蛋白兔源多抗血清按质量比60∶1混匀后,于4 ℃条件下过夜,于7000 r/min条件下离心3 min,取上清液,即为特异性吸收抗体。
1.3.7 热加工破坏G3A1b抗原表位的ELISA鉴定取1.3.5所述方法所保存的重组噬菌体表达蛋白溶液,使用CBS溶液(碳酸盐缓冲溶液)将其稀释至50 μg/mL,并按每孔100 μL的量加入酶标板内,于4 ℃条件下过夜。取PBST缓冲液(磷酸盐吐温缓冲溶液)作为清洗液,每孔加入200 μL,清洗5次,每次加入清洗液均静置5 min后弃去。选用质量分数为5%的脱脂奶粉溶液作为封闭液,每孔200 μL,于37 ℃条件下温育2 h。按上述操作及剂量,用清洗液清洗5次孔板。分别选用大豆球蛋白兔源多抗血清和1.3.6中制备的特异性吸收抗体作为一抗,以未免疫的兔血清和PBS缓冲液(磷酸缓冲盐溶液)为阴性对照和空白对照,每孔100 μL,于37 ℃条件下温育1 h。按上述操作及剂量,用清洗液清洗5次孔板。每孔加入100 μL稀释8000倍的羊抗兔IgG抗体,于37 ℃条件下温育1 h。按上述操作及剂量,用清洗液清洗5次孔板。每孔加入100 μL TMB显色液避光显色3 min,再每孔加入50 μL H2SO4溶液(2 mol/L)终止反应。使用酶标仪检测待测物的OD450[19]。
2 结果与分析
2.1 大豆球蛋白G3A1b多肽链的同源建模及模型评估
通过SWISS-MODEL 在线预测软件找到与G3A1b同源的蛋白3ksc.2.A,其同源性达到72.8%,GMEQ(全局模型质量估计值)达到0.69。选择该同源蛋白为模板,参照刘阳星月等[7]的三级结构预测方法,在NCBI数据库中查询G3亚基氨基酸序列,获得的G3亚基三级结构模型如图1所示,G3A1b多肽链三级结构模型如图2所示。
G3A1b多肽链三级结构的拉氏图如图3所示。该图以肽平面绕Cα—N键旋转的角Φ为横坐标,绕Cα—C键旋转的角ψ为纵坐标[7,20-21],主要分为绿色区域(完全允许区域)、浅绿色区域(允许区域)和白色区域(不允许区域)。其中,落在完全允许区域和允许区域的氨基酸残基数量占总氨基酸残基数量的91.8%。通常情况下,当该比例超过90%时,表明该模型的构象符合立体化学规则,因为在这2个区域中氨基酸残基的非键合原子间的距离大于最小允许距离,构象稳定[6,22],这进一步表明通过SWISS-MODEL在线预测软件构建的G3A1b多肽链预测模型可用于后续构象表位的预测。
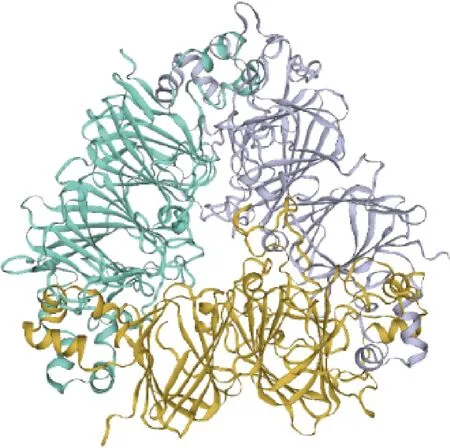
图1 G3亚基三级结构模型Fig.1 Tertiary structure of G3 subunit
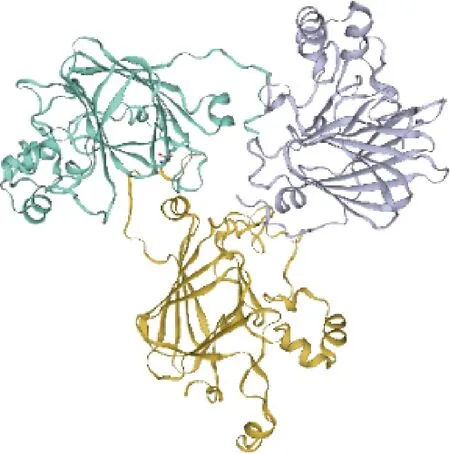
图2 G3A1b多肽链三级结构模型Fig.2 Tertiary structure of G3A1b polypeptide chain
2.2 G3A1b多肽链的B细胞抗原表位预测结果分析
2.2.1SOPMA预测结果二级结构的预测可为后续抗原表位定位提供一定的理论依据。SOPMA预测可明确蛋白质二级结构的多项相关信息,如G3A1b多肽链二级结构位点(见图4a),其中h为α-螺旋,e为β-折叠,t为β-转角,c为无规则卷曲);G3A1b多肽链二级结构在整个多肽链中的占比情况(α-螺旋占比20.36%,β-折叠占比17.09%,β-转角占比9.45%,无规则卷曲占比53.09%);G3A1b多肽链二级结构分布竖条纹图(见图4b));G3A1b多肽链二级结构位点峰图(见图4c))。基于抗原表位二级结构预测理论知识可知,β-转角和无规则卷曲是二级结构抗原表位预测的优势区域[23-24]。
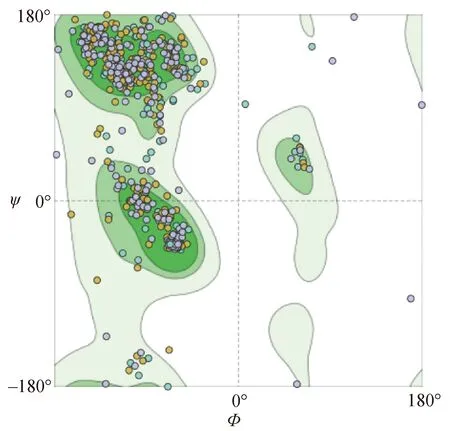
图3 G3A1b多肽链三级结构的拉氏图Fig.3 Ramachandran plot of the tertiary structure of G3A1b polypeptide chain
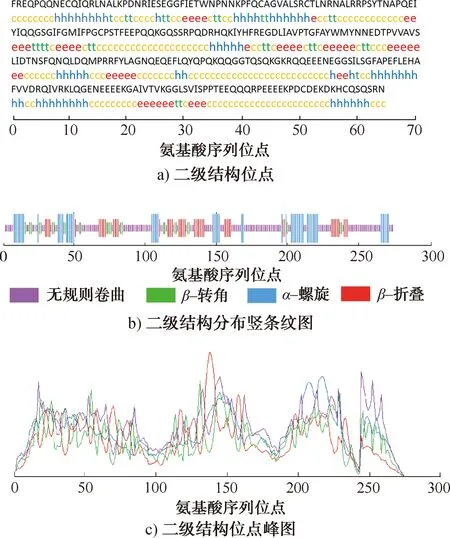
图4 G3A1b多肽链的SOPMA预测结果Fig.4 SOPMA prediction results of G3A1b polypeptide chain
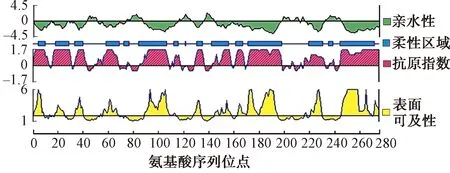
图5 基于G3A1b多肽链理化性质的DNAStar预测分析Fig.5 DNAStar prediction analysis based on physicochemical properties of G3A1b polypeptide chain
2.2.2DNAStar预测结果基于G3A1b多肽链理化性质的DNAStar预测分析如图5所示。由图5可知,G3A1b多肽链具有较高的疏水性。当抗原表位与抗体靠近时,相互间的正、负极性消失并失去亲水层,这促使二者进一步相互吸引甚至结合。因此,强疏水作用力对维系抗原表位和抗体结合的作用最大。结合表位预测理论知识[7],在综合抗原指数(Antigenic Index)、表面可及性(Surface Probability)、柔性区域(Flexible Regions)等因素后确定了13个最有可能成为抗原表位最佳位点的氨基酸序列,见表1。
2.2.3BepiPred预测结果BepiPred是基于对蛋白质的极性、疏水性、表面可及性、二级结构等物理性质,采用随机森林回归算法对给定抗原进行表位预测的服务器。将G3A1b多肽链作为给定抗原,将其氨基酸序列提交至BepiPred服务器后,可得到8个潜在抗原表位预测结果,见表2。
2.2.4ABCpred预测结果ABCpred是基于人工神经网络模型对蛋白质进行预测的服务器。将G3A1b多肽链序列信息输入ABCpred服务器后,预测出14个潜在抗原表位,见表3。
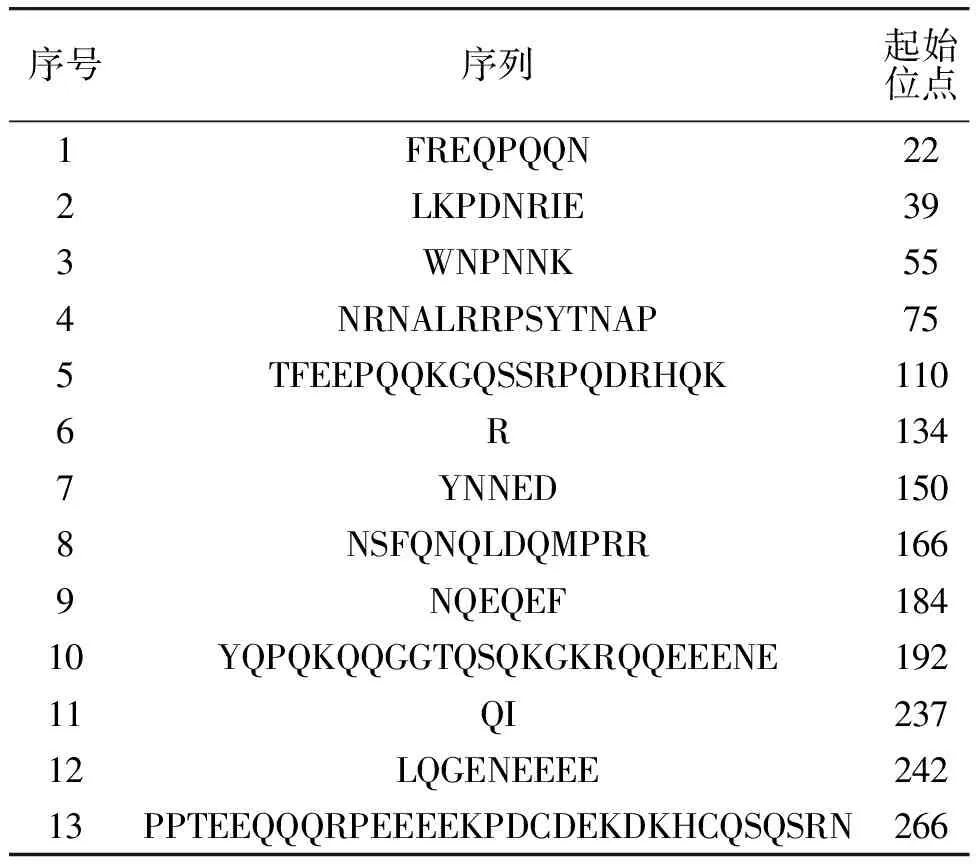
表1 G3A1b多肽链潜在抗原表位的DNAStar预测结果Table 1 DNAStar prediction results of potential antigenic epitopes of G3A1b polypeptide chain

表2 G3A1b多肽链潜在抗原表位的BepiPred预测结果Table 2 BepiPred prediction results of potential antigenic epitopes of G3A1b polypeptide chain
2.2.5G3A1b多肽链潜在线性抗原表位的综合预测结果DNAStar、BepiPred、ABCpred均是针对蛋白质的不同性质进行预测的,综合这3个服务器可使预测结果更完善,综合预测结果如图6所示。由图6可知,可确定27QQN29、39LKPDNRIE46、58NNK60、114PQQKGQSSRP123、134R、174QMPR177和184NQEQEF189这7个最有可能的线性抗原表位。利用PyMol软件进行三维空间展示,可更清晰地观测到这7个优势抗原表位在G3A1b多肽链中的具体分布位点,如图7所示。
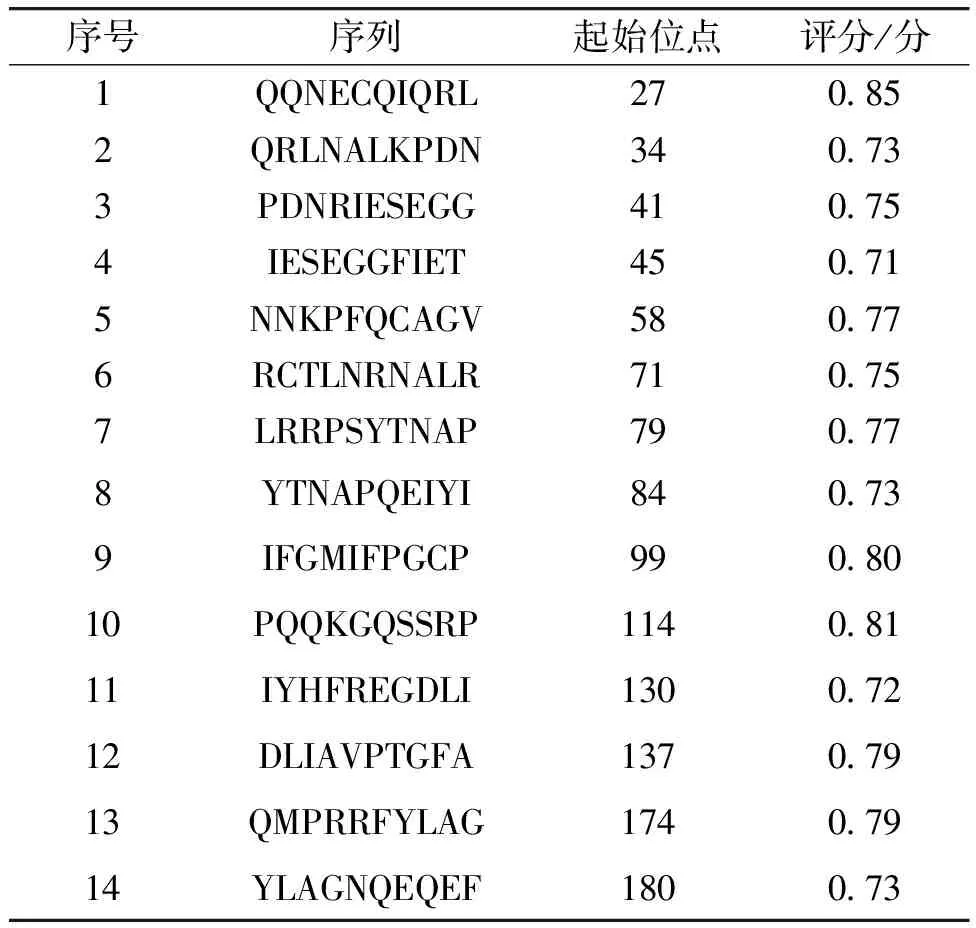
表3 G3A1b多肽链潜在抗原表位的ABCpred预测结果及评分Table 3 ABCpred prediction results and scores of potential antigenic epitopes of G3A1b polypeptide chain

图6 DNAStar、BepiPred、ABCpred综合预测结果Fig.6 DNAStar, BepiPred, ABCpred combined prediction results
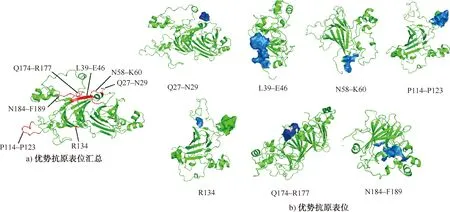
图7 G3A1b多肽链的预测抗原表位展示图Fig.7 Display of potential antigenic epitopes of G3A1b polypeptide chain
2.2.6DiscoTope2.0Server预测结果单独依靠G3A1b多肽链的三维模型及蛋白质序列信息进行线性表位预测存在片面性,有研究[25]表明,在所有B细胞表位中,线性表位只占很少一部分,而构象表位居多。G3A1b多肽链的DiscoTope 2.0 Server预测构象表位结果如图8所示,其中,红色字体均为潜在构象表位所在位点。结合上文二级结构预测结果可知,G3A1b多肽链的构象表位较多位于无规则卷曲处。
2.3 G3A1b多肽链的重叠分段与引物设计
依据生物信息学软件所预测的G3A1b多肽链线性表位及构象表位的分布进行重叠分段,确保每个重叠片段均存在一定量的线性表位和构象表位。使用Primer Premier 6.0软件设计G3A1b引物多肽链(见表4)。
片段一:1~120 aa
FREQPQQNECQIQRLNALKPDNRIESEGGFIET-WNPNNKPFQCAGVALSRCTLNRNALRRPSYTNAPQ-EIYIQQGSGIFGMIFPGCPSTFEEPQQKGQSSRPQDR-HQKIYHFREGDLIAV
片段二:71~200 aa
YIQQGSGIFGMIFPGCPSTFEEPQQKGQSSRPQD-RHQKIYHFREGDLIAVPTGFAYWMYNNEDTPVVAV-SLIDTNSFQNQLDQMPRRFYLAGNQEQEFLQYQPQK-QQGGTQSQKGKRQQEEENEGGSILS

图8 G3A1b多肽链的DiscoTope 2.0 Server预测构象表位结果Fig.8 DiscoTope 2.0 Server predicts conformational epitope results of G3A1b polypeptide chain

表4 G3A1b多肽链的引物设计Table 4 Primer design of G3A1b polypeptide chain
片段三:151~275 aa
LDQMPRRFYLAGNQEQEFLQYQPQKQQGGTQS-QKGKRQQEEENEGGSILSGFAPEFLEHAFVVDRQIV-RKLQGENEEEEKGAIVTVKGGLSVISPPTEEQQQRP-EEEEKPDCDEKDKHCQSQSRN
2.4 G3A1b多肽链的基因扩增及克隆载体的鉴定结果
G3A1b多肽链的基因扩增及克隆载体的鉴定结果如图9所示,其中,泳道M、1、2、3、4分别为DNA Maker、G3A1b-1、G3A1b-2、G3A1b-3、G3A1b,下同。由图9a)可观察到与预期目的片段大小一致的条带,1—4条带的长度分别为360 bp、390 bp、375 bp和825 bp。克隆载体的PCR鉴定结果(见图9b))和双酶切鉴定结果(图9c))所显示的片段大小均与预期一致。综上可知,G3A1b多肽链及其分段基因的克隆载体构建成功。
2.5 G3A1b多肽链重组噬菌体的鉴定结果
G3A1b多肽链重组噬菌体的鉴定结果如图10所示。由图10a)可知,重组噬菌体侵染E.ColiBLT5615后形成了噬菌斑,挑取单个斑块溶解于TE溶液中,于65 ℃条件下裂解后作为基因模板,并于PCR扩增后进行重组噬菌体的鉴定。由图10b)可观察到与预期相符的条带,表明G3A1b多肽链重组噬菌体构建成功。
2.6 热加工破坏G3A1b多肽链抗原表位的ELISA鉴定结果
G3A1b多肽链抗原性的ELISA鉴定结果如图11所示,其中,不同小写字母表示组间差异显著(P<0.05)。由图11可知,G3A1b-3的抗原性最强。综合G3A1b多肽链的理化性质和二级结构可知,G3A1b多肽链的疏水性较高,且G3A1b-3片段存在较多α-螺旋和β-折叠。这表明在分段前,可能存在较多抗原表位隐藏于内部,当G3A1b多肽链被分段表达后,原本积聚在内部的抗原表位容易暴露到外部。此外,G3A1b多肽链的构象表位超过半数位于G3A1b-3片段内,因而造成了G3A1b-3片段抗原性高于G3A1b抗原性的情况。
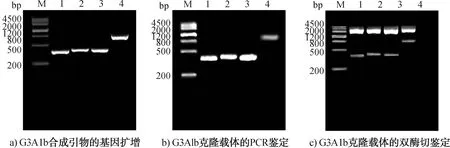
图9 G3A1b多肽链的基因扩增及克隆载体的鉴定结果Fig.9 Gene amplification of G3A1b polypeptide chain and identification results of cloning vector
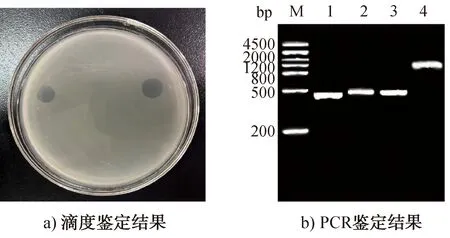
图10 G3A1b多肽链重组噬菌体的鉴定结果Fig.10 Identification results of recombinant phage G3A1b polypeptide chain
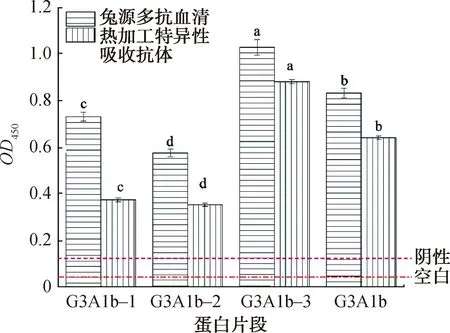
图11 G3A1b多肽链抗原性的ELISA鉴定结果Fig.11 ELISA results of antigenicity of G3A1b polypeptide chain
综上所述,G3A1b-3多肽链在热加工过程中被破坏程度相对明显。后续可根据G3A1b-3多肽链进一步进行重叠分段,这对精确定位加工破坏G3A1b多肽链抗原表位的探究提供了理论基础。
3 结论
本研究使用生物信息学软件对大豆球蛋白G3A1b进行潜在抗原表位预测,得出27QQN29、39LKPDNRIE46、58NNK60、114PQQKGQSSRP123、134R、174QMPR177和184NQEQEF189这7个可能的线性表位。结合DiscoTope 2.0 Server所预测的潜在构象表位对G3A1b进行重叠分段,分别为G3A1b-1(120 aa)、G3A1b-2(130 aa)和G3A1b-3(125 aa),采用PCR技术鉴定后将其构建克隆载体并进行筛选鉴定。而后构建重组噬菌体表达目的蛋白,进行抗原性检测及热加工破坏抗原表位的鉴定,鉴定结果显示G3A1b-3抗原性最强,且在热加工过程中被破坏得相对明显。这一发现为继续深入筛选影响大豆球蛋白G3A1b抗原性的表位区域提供了事实依据,缩小了探究G3A1b多肽链被加工破坏的抗原表位的定位范围,为精确定位大豆球蛋白G3A1b中致敏关键氨基酸提供了理论支持,为食品行业通过特定加工方法替换、消除或破坏该致敏氨基酸从而研发非致敏性大豆制品提供了参考。

