一株产红色素沙雷氏菌基因表达谱差异分析
李靖宇, 张肖冲, 李艳楠, 袁存霞, 刘建利, 金 多, 马志山
(1. 北方民族大学生物科学与工程学院, 银川750021;2. 宁夏特殊生境微生物资源开发与利用重点实验室, 银川750021)
肠杆菌科沙雷氏菌属(Serratia)细菌菌体为球状或短杆状,革兰氏染色阴性,无荚膜,有鞭毛,具有运动性,兼性厌氧菌,能产生多种胞外产物,包括脂肪酶、几丁质酶、核酸酶、蛋白酶和各种抗菌次级代谢产物,这些细菌广泛分布于土壤、沉积物、水、植物根系、动物表面及其胃肠道等环境中[1-3]。沙雷氏菌属包含30个种和4个亚种,其中许多成员可产生次级代谢产物,如普城沙雷氏菌(S.plymuthica)、红色沙雷氏菌(S.rubidaea)、黏质沙雷氏菌(S.marcescens)和嗜线虫沙雷氏菌(S.nemadiphila)可产生一种脂溶性红色素,被鉴定为灵菌红素,是一种具有独特三吡咯化学结构的生物碱次级代谢产物,在环境生物修复或制药工业中有潜在的应用价值[3-4]。与传统的化学方法相比,利用黏质沙雷氏菌生产灵菌红素具有经济、环保等优点,近年来受到广泛关注。然而,通过优化发酵参数,如培养基组成、pH值、温度以及培养周期以提高其天然菌株灵菌红素的产量用于商业目的仍是一个挑战[5]。研究表明黏质沙雷氏菌在28 ℃时能有效产生灵菌红素,在37 ℃或更高温度时急剧减少,充分说明灵菌红素的产生和温度有关[6]。沙雷氏菌属灵菌红素的生物合成依赖于pigA-N或pigA-O组成的pig基因簇,同时受到群体感应(Quorum sensing)的调控[3,7]。
群体感应是一种基因调控系统,它是随着种群密度的增加而受到激活[7]。在许多菌株中,色素的产生被群体感应所激活,而这种调节越来越被认为会对整体的细胞代谢产生重要影响[8]。由沙雷氏菌菌株产生的许多活性代谢物受到群体感应的调控,包括丁二醇发酵、胞外酶的产生、核酸酶,以及次级代谢产物,如生物表面活性剂、碳青霉烯、oocydin A和灵菌红素。此外,一系列重要的过程,如生物发光、运动性、产孢、毒力和生物膜的形成,也受到群体感应系统的调控[3]。以往的研究主要集中在群体感应、信号转导和双组分调节系统,以确定在灵菌红素产生中起重要作用的调节机制[6]。本文以一株产红色素沙雷氏菌为研究对象,在28 ℃能有效产生灵菌红素,对该温度条件下不同生长阶段该菌的转录组进行高通量测序分析,以期在转录组水平下探究产红色素沙雷氏菌的产色素相关基因表达谱差异。
1 材料与方法
1.1 菌株和培养基
产红色素沙雷氏菌(Serratiasp. L1) 菌株由本实验室从北方民族大学校园土壤中分离所得。
LB固体培养基:酵母浸粉5.0 g/L,胰蛋白胨10.0 g/L,NaCl 10.0 g/L,琼脂15.0 g/L加蒸馏水至1 000 mL,121 ℃灭菌20 min 后备用(液体培养基不含琼脂)。
1.2 生长曲线的测定
Serratiasp. L1菌株液体培养浓度达到OD600约1.070~1.122,按照1%接种量加入装有250 mL液体LB培养基的500 mL锥形瓶中(5个重复),另外设置1个不加菌LB液体培养基作为对照,在28 ℃和150 r/min条件下进行培养。每隔2 h在无菌条件下从锥形瓶中取5 mL培养液测定OD600值,取样完毕后立即将锥形瓶放回气浴恒温振荡器中继续培养,每次测定时用对照进行调零。用GraphPad Prism 5绘制生长曲线图。
1.3 菌株系统发育树构建
将液体培养菌株涂布于LB固体培养基上,在28 ℃条件下过夜培养,刮取适量菌种于无菌离心管中,送上海美吉生物医药科技有限公司进行菌种鉴定。16S rDNA PCR扩增反应体系:1.6 μL 2.5 mmoI/L dNTPs,1 μL 5 μmol/L 27F引物(5′-AGAGTTTGATCCT-GGCTCAG-3′),1 μL 5 μmol/L 1492R引物(5′- GGT-TACCTTGTTACGACTT -3′),2.0 μL 10 × ExTaq缓冲液,0.5 μL模板以及 0.2 μL 5 U ExTaq,最后加ddH2O到20 μL。反应条件:95 ℃预变性5 min;95 ℃变性30 s,56 ℃退火30 s,72 ℃延伸90 s,25个循环;72 ℃再延伸10 min。电泳条件:1%琼脂糖凝胶,120 V 电压电泳30 min。16S rDNA扩增产物使用ABI 3730XL基因测序仪进行双末端测序,根据abi测序峰图文件,手工去除两端低质量碱基序列,使用Seqman拼接两条序列,将拼接的序列在NCBI数据库中Blast比对,选择相似性高的序列与本研究序列保存为fasta格式文件,用MEGA-X做Maximum parsimony系统发育树(参数设置:Bootstrp method为999)。序列NCBI登录号:MT434357。
1.4 Serratia sp. L1不同生长阶段总RNA提取
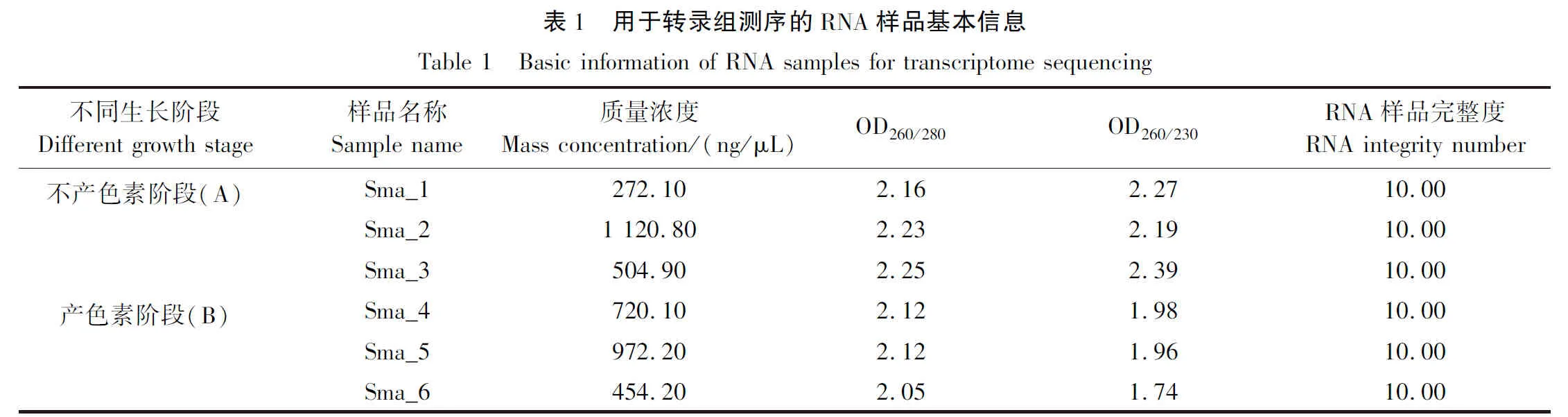
取固体培养基上生长5 h的Serratiasp. L1菌体作为不产色素阶段的代表样品,3个重复分别记为Sam_1、Sam_2和Sam_3,取培养24 h的菌体作为产色素阶段的代表样品,3个重复分别记为Sam_4、Sam_5和Sam_6。收集菌体时,在无菌操作条件下用无菌水分别将2个培养时间点的固体平板培养基表面的菌体收集于1.5 mL离心管中,6 000 r/min离心10 min。收集的菌体立即放入液氮罐低温处理30 min后,置于-80 ℃冰箱保存,用于后续总RNA的提取。
按照TRIzol®试剂使用说明书(Invitrogen),从菌体样品中提取总RNA,使用DNase I (TaKara)去除基因组DNA。用Agilent2100生物分析仪测定RNA质量,使用ND-2000(NanoDrop Technologies)进行定量。高质量的RNA样品(OD260/280=1.8~2.0、OD260/230≥2.0、RIN≥6.5、23S∶16S≥1.0、总量≥100 ng/μL、浓度≥2 μg)用于后续的建库(表1)。
1.5 构建cDNA文库与Illumina Hiseq测序
使用Illumina(San Diego,CA)的TruSeqTM RNA sample preparation Kit进行RNA文库构建。使用Ribo-Zero Magnetic kit (epicenter)去除rRNA,将mRNA随机断裂成200 bp左右的小片段,以mRNA为模板,利用随机引物和SuperScript double-stranded cDNA synthesis kit(Invitrogen,CA)反转录合成双链cDNA。在合成cDNA第二链时,用dUTP代替dTTP进行合成,合成的双链cDNA加入End Repair Mix将其补成平末端,5′端磷酸化,3′末端加上一个A碱基,连接Y字形测序接头。然后用UNG酶消除含有dUTP的cDNA第二链,从而使文库中只包含cDNA的第一链。然后用Phusion DNA polymerase(NEB)进行PCR扩增,扩增15个循环。TBS380(Picogreen)定量后,使用Illumina HiSeq X Ten(2×150 bp)进行RNA-seq双端测序。
1.6 Illumina Hiseq测序数据处理与分析
Illumina测序数据使用上海美吉生物医药科技有限公司(Shanghai Majorbio Biopharm Technology Co.,Ltd)云平台(www.majorbio.com)进行分析。首先对Illumina Hiseq测序原始数据进行质量控制和剪切,具体步骤如下:(1)去除 reads 中的adapter 序列;(2) 剪切去除5′端含有非A、G、C、T的碱基;(3) 剪掉reads末端测序质量低于Q20的碱基;(4)去除含N的比例达到10%的reads;(5) 舍弃去除adapter 及质量修剪后长度小于25 bp的序列。使用Bowtie(http:∥bowtie-bio.sourceforge.net/index.shtml)软件基于Burrows-Wheeler方法对得到的高质量reads与Serratiamarcescens(https:∥www.ncbi.nlm. nih.gov/ genome/?term=Serratia+marcescens)参考基因组进行比对,通过对字符串转换后得到的字符矩阵进行排序和变换来建立索引,将序列比对到参考基因组上,使用默认参数运行比对,获得用于后续分析的mapped reads,同时对比对结果进行质量评估。将参考基因组与KEGG数据库进行比对,进行基础功能注释,得到相应的功能注释信息。利用软件RSEM (http:∥deweylab.github.io/RSEM/)对基因的表达水平进行定量分析,定量指标为每百万读段中来自于某转录本的读段数,即Transcripts Per Million reads(TPM)。TPM的均一化过程使得不同样本中的总表达量一致,这样可以更直观地进行基因间表达量的比较。然后使用基于负二项分布的DESeq2 (http:∥bioconductor. org/packages/stats/bioc/DESeq2/)软件对原始计数进行统计分析,获得组间表达差异基因,默认参数:P-adjust<0.05和|log2FC|≥2。基于此,构建产色阶段显著上调基因集和显著下调基因集,并通过KEGG数据库对基因集中的基因进行功能分类注释,同时对基因集中的基因进行KEGG Pathway富集分析,当校正的P值(P-adjust)<0.05时,认为此KEGG Pathway功能存在显著富集情况。
2 结果与分析
2.1 产红色素沙雷氏菌菌株鉴定、进化树构建和生长曲线绘制
结果表明该菌在28 ℃培养5 h的条件下没有产生色素[图1(a)],培养24 h可在固体培养基形成产色素菌落[图1(b)],而温度提高到37 ℃后在固体培养基上不形成产色素菌落。在实验室液体培养条件下,28 ℃时会产生色素,但产色素不明显。其生长曲线见图1(c),基于生长曲线对数期和稳定期为后续分析该菌不同生长阶段,即不产色素和产色素,提供了取样时间参考。

(a)培养5 h;(b)培养24 h;(c)生长曲线。图1 Serratia sp. L1在28 ℃条件下菌落形态特征和生长曲线Figure 1 Morphological characteristics of colonies and growth curve of Serratia sp. L1 at 28 ℃
根据16S rDNA序列分析结果表明该菌与Serratiamarcescens相似性高达99%。通过对相似序列构建Maximum parsimony系统发育树,结果显示菌株L1与Serratiamarcescens其他菌株聚类在一起(图2)。
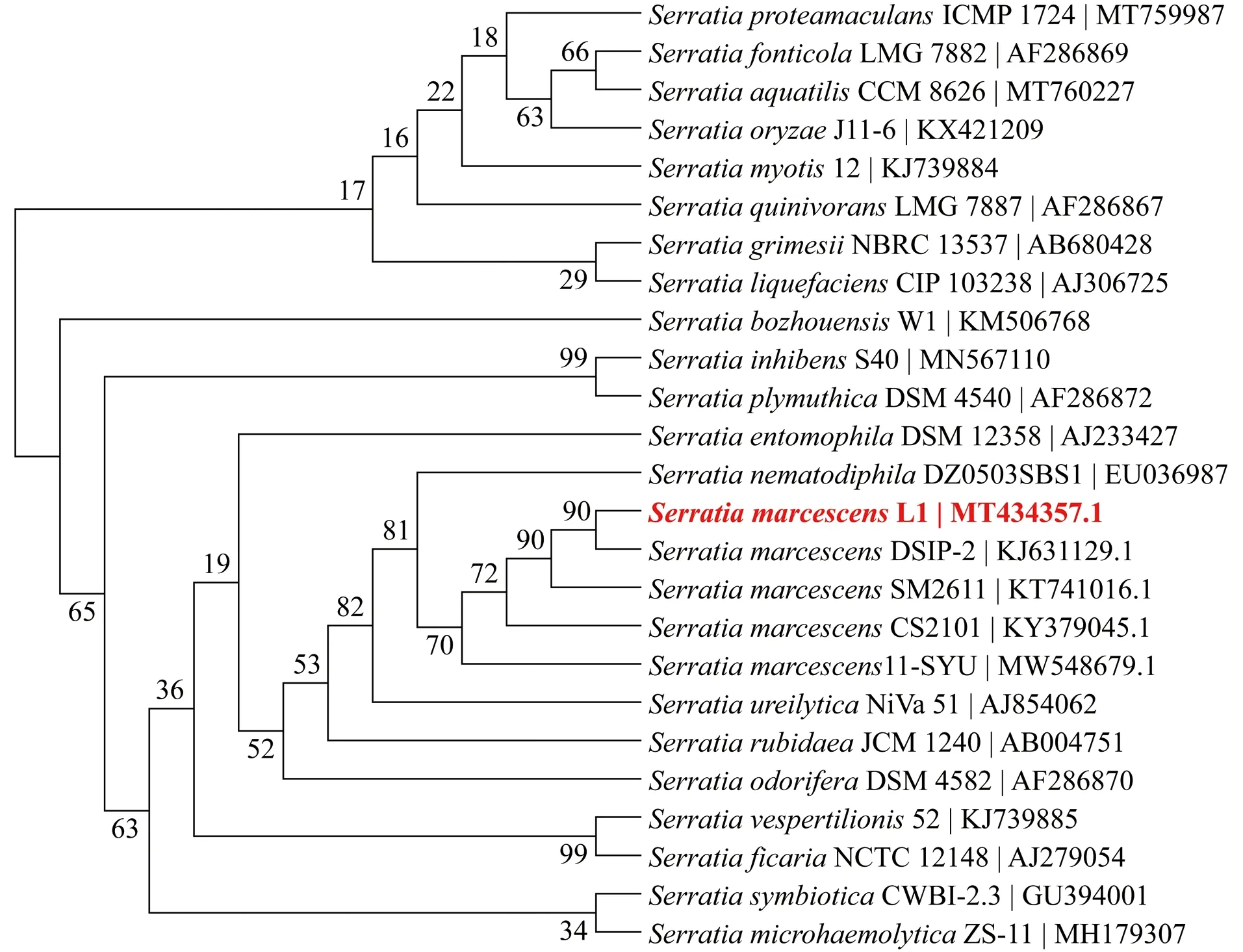
图2 基于16S rDNA产红色素Serratia sp. L1的Maximam Parsimony进化树Figure 2 The Maximam Parsimony phylogenetic tree of Serratia sp. L1 producing red pigment based on 16S rDNA
2.2 Serratia sp. L1菌株不同生长阶段转录组测序数据分析
为探究Serratiasp. L1菌株不同生长阶段基因表达谱差异特征,选取28 ℃条件下固体培养5 h菌体作为不产色素阶段的代表样品,24 h菌体为产色素阶段的代表样品进行转录组测序分析。不产色素和产色素阶段转录组分别平均获得40 386 475和38 944 075条原始reads。质控后分别平均获得40 000 741和38 081 085条高质量reads。与参考基因组比对后,不产色素和产色素阶段匹配上的reads数在clean reads中的平均百分比分别为90.66%和96.32%,CDS注释平均百分比分别为85.36%和90.94%,见表2。

根据转录组测序数据的PCA分析可知,第一轴(PC1)可解释91.99 %的组间差异,说明不产色素和产色素两个阶段的重复取样具有代表性,见图3(a)。在此前提下,分析了Serratiasp. L1菌株不同生长阶段基因表达谱差异特征,共获得显著差异基因2 512个,其中,上调基因1 317个,下调基因1 195个,见图3(b)。

图3 基于Serratia sp. L1不同生长阶段转录组的PCA分析(a)和差异基因表达谱(b)Figure 3 PCA analysis (a) and differential gene expression profile (b) based on transcriptome of Serratia sp. L1 at different growth stages
2.3 Serratia sp. L1菌株不同生长阶段差异基因表达谱KEGG注释分析
基于Serratiasp. L1菌株不同生长阶段差异基因集,对其进行KEGG数据库注释。结果(图4)表明:差异基因集中有141个基因属于氨基酸代谢途径;有13个基因属于其他次级代谢产物生物合成途径;有178个基因属于碳水化合物代谢途径;有95个基因属于能量代谢途径;有36个基因属于聚糖生物合成和代谢途径;有100个基因属于维生素和辅因子代谢途径;有42个基因属于其他氨基酸代谢途径;有23个基因属于萜类和聚酮化合物代谢途径;有54个基因属于核苷酸代谢途径;有17个基因属于异生物素生物降解和代谢途径。在差异基因集中,功能分类属于遗传信息处理、环境信息处理以及细胞过程的基因变化情况(图4)。

图4 基于Serratia sp. L1产色素阶段和不产色素阶段差异基因集的KEGG代谢通路注释Figure 4 KEGG metabolic pathway annotation based on differential gene set of Serratia sp. L1 between pigment producing stage and non-pigment producing stage
2.4 Serratia sp. L1菌株不同生长阶段差异基因表达谱KEGG功能富集分析
基于Serratiasp. L1菌株产色素阶段显著下调基因集进行KEGG Pathway富集分析,结果表明参与核糖体、鞭毛组装、TCA循环、肽聚糖生物合成、氨酰tRNA生物合成、原核生物碳固定、阳离子抗菌肽耐药性、细菌趋化性、细菌分泌系统、氧化磷酸化以及丙氨酸、天冬氨酸和谷氨酸代谢等基因存在显著功能富集趋势(图5、表3)。基于Serratiasp. L1菌株产色素阶段显著上调基因集进行KEGG Pathway富集分析,结果表明参与群体感应的基因存在功能显著富集趋势(图6、表3)。其中,一些与群体感应相关的基因注释信息见表4。双组分调节系统组氨酸激酶NarX和NarQ来感知硝酸盐/亚硝酸盐的含量,并将响应调节子NarL磷酸化,从而影响硝酸盐还原酶的表达(图7),这些基因的高表达表明沙雷氏菌L1生长后期局部环境中硝酸盐的含量可能会显著增加。然而,菌株之间对营养物质的需求受到局部环境可利用氮的限制,该菌生长后期感知外界环境低氮可利用性的GlnL组氨酸激酶以及响应调节子NarG表达量显著下降,导致参与硝酸盐同化的谷氨酰胺合成酶基因产物GlnA表达量显著下降(图7),说明该菌生长后期局部环境中氮源供应不足。
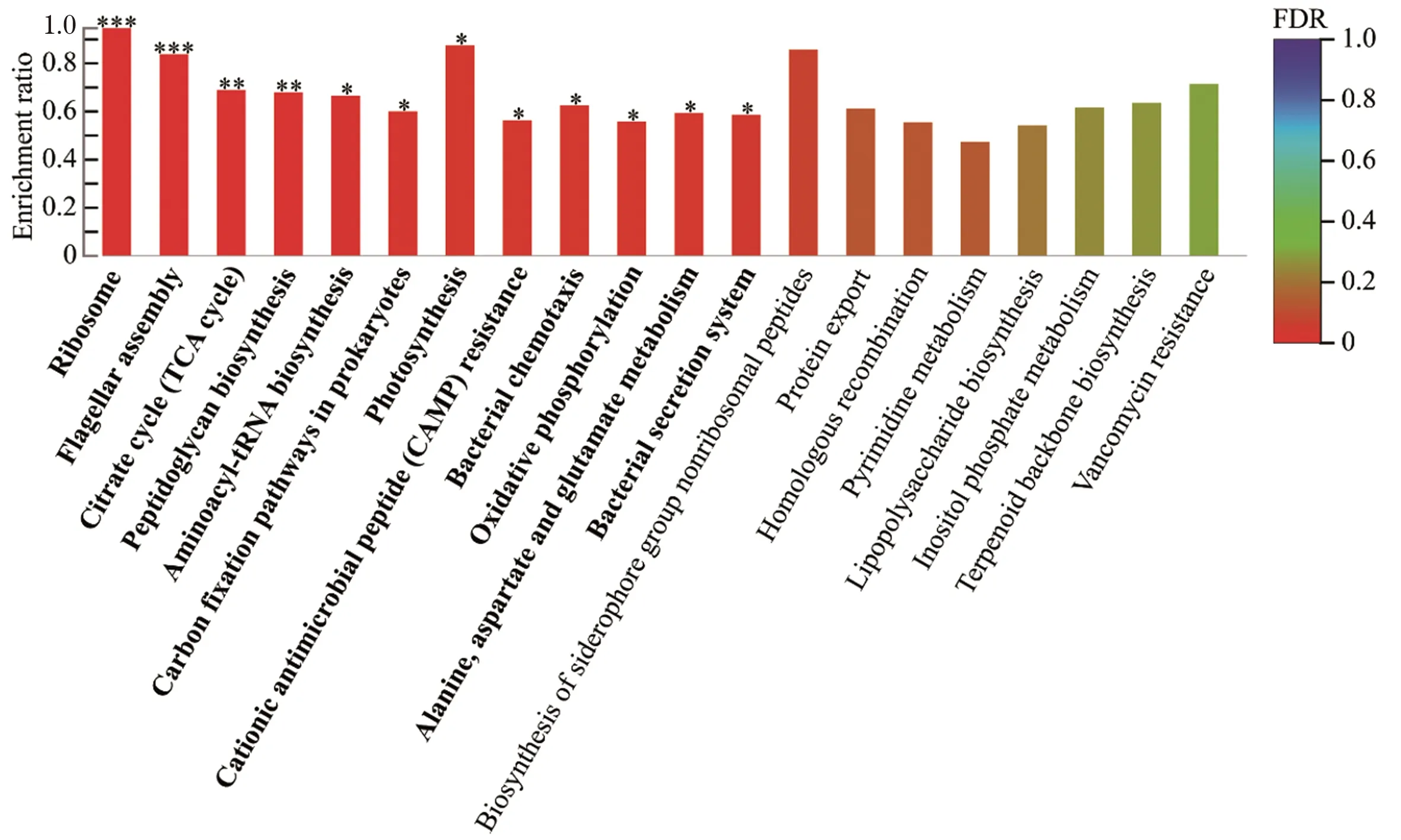
*** 表示P<0.001,** 表示P<0.01,* 表示P<0.05。图5 基于Serratia sp. L1产色素阶段显著下调基因集对基因进行KEGG pathway富集分析Figure 5 KEGG pathway enrichment analysis based on the significantly down regulated gene set of Serratia sp. L1 during the pigment production stage

*** 表示P<0.001,** 表示P<0.01,* 表示P<0.05。图6 基于Serratia sp. L1产色素阶段显著上调基因集对基因进行KEGG pathway富集分析Figure 6 KEGG pathway enrichment analysis based on the significantly up regulated gene set of Serratia sp. L1 during the pigment production stage

图7 Serratia sp. L1产色素阶段双组分系统显著变化相关基因Figure 7 Genes related to significant changes in two component system of Serratia sp.L1 during pigment production

3 讨论与结论
Serratiasp. L1菌株能够在28 ℃固体培养基上生长产生红色色素,此过程受到温度的调控,同时也受到群体感应的调控。灵菌红素的生产受到一系列环境信号的调节,如温度、磷酸盐限制和培养基成分[9]。群体感应是一种常见的调节细菌生理过程的机制,通过感知种群密度来控制各种生物功能,包括毒力因子的表达、次级代谢产物的产生、共生、生物膜的形成、生物发光、胞外多糖水平、抗生素产生、群集运动、细胞代谢、细胞表面电动特性、结合质粒的转移、个体的生存策略以及对变化环境的适应[10-11]。在其生长后期,固体培养基上的菌落局部环境变得不利于菌株生长,外界环境的改变刺激菌株高表达双组分调节系统以及群体感应系统的相关基因,如qseB、qseC、glrK、sdiA、lsrK、expR等(表4)。在沙雷氏菌中,已经描述了许多不同的LuxIR型系统,包括SwrIR(SerratialiquefaciensMG1)、SmaIR(Serratiasp. ATCC 39006)、SprIR (Serratiaproteamaculans)和SpnIR(SerratiamarcescensSS-1)[10]。在这些系统中,LuxI同源物是一种自诱导物合成酶,它催化S-腺苷甲硫氨酸(SAM)与酰基载体蛋白(ACP)之间的反应,从而产生自由扩散的酰基高丝氨酸内酯(AHL)作为自诱导物(AI)[12]。N-酰基高丝氨酸内酯(AHL)是由革兰氏阴性菌产生的一种群体感应信号分子。LuxI蛋白家族合成AHL、LuxR蛋白家族结合AHL并调节许多基因的表达,这些基因负责生物发光、色素或抗生素的产生等[9]。研究表明AHL自诱导物在高含量条件下会与同源细胞质LuxR转录因子结合激活luxI表达。当不被AI结合时,LuxR蛋白质会迅速降解[12]。本研究中沙雷氏菌L1在产色素阶段高表达自诱导物AI-2激酶lsrK,同时也高表达LuxR家族转录调节因子sdiA说明该菌在产生色素阶段启动了LuxIR型群体感应系统。这些基因的改变会影响沙雷氏菌整体细胞过程中结构基因的表达水平,特别是涉及能量代谢三羧酸循环的一些关键基因表达下调,如柠檬酸合成酶基因(gltA)、NADP依赖性异柠檬酸脱氢酶基因(icd)、2-酮戊二酸脱氢酶E1组分基因(sucA)等,参与氧化磷酸化过程的电子链蛋白编码基因显著下调,如细胞色素C泛醌氧化酶(cyoB、cyoA、cyoC)、F0F1 ATP合酶(atpB、atpA、atpF、atpD、atpH、atpC、atpG)、NADH醌氧化还原酶(nuoL、nuoM、nuoE、nuoF、nuoG、nuoH、nuoN)、琥珀酸脱氢酶(sdhC、sdhA、sdhB、sdhD)等。

综上所述,该菌能够在固体培养条件下受温度调控产生色素,转录组数据表明产色素过程同时受到群体感应系统的调节。然而,具体如何调节有待开展进一步的实验进行验证。

