细菌脲酶蛋白复合物UreABC的表达与纯化
李晓姣, 王加启, 赵圣国*, 程建波
(1.安徽农业大学 动物科技学院,安徽 合肥 230036;2.中国农业科学院北京畜牧兽医研究所 动物营养学国家重点实验室,北京 100193;3.太原市畜牧发展中心,山西 太原 030000)
脲酶(尿素酶,EC 3.5.1.5)可以催化尿素水解形成氨和二氧化碳[1]。在反刍动物中,瘤胃微生物分解尿素所产生的氨可以与酮酸结合形成可供机体利用的微生物蛋白,所以饲喂反刍动物尿素不仅可以节约饲料成本,还可以为其提供优质氮源[2]。但当脲酶活性太高,其分解尿素所产生的氨会超出瘤胃微生物的利用能力,导致动物氨中毒,因此也需要适当的限制脲酶的活性[3-4]。由于瘤胃尿素分解菌的重要作用,对尿素分解菌的研究一直受到广泛关注。虽然分子生态学和宏基因组学的应用,增加了人们对微生物多样性的了解,但对瘤胃中尿素分解菌的分布和多样性还知之甚少,百分之九十以上仍属于未被分类细菌[5]。Li 等[6]、Zhao等[7]通过厌氧滚管和调整培养基等技术,从牛瘤胃中分离得到5种尿素分解菌:溶糊精琥珀酸弧菌、布氏密螺旋体、白色瘤胃球菌、地衣芽胞杆菌和奇异变形菌。测序技术的快速发展,也为研究未培养微生物种类与功能提供了重要的研究方法。鉴于脲酶基因ureC是分析环境中的尿素分解菌的目标基因,Jin等[8]还通过Miseq方法进行ureC基因的扩增和测序,研究饲喂尿素对瘤胃菌群的影响,多样性分析结果表明,甲基球菌科(Methylococcaceae)、类芽胞杆菌科(Paenibacillaceae)、嗜甲基菌科(Methylophilaceae)、螺杆菌科(Helicobacteraceae)和梭菌科(Clostridiaceae)的ureC基因丰度较高,从而揭示了瘤胃中的优势尿素分解菌群。在此研究背景下,需要知道如何调控脲酶活性,才能为脲酶抑制剂的开发提供理论指导。细菌脲酶的活化需要结构蛋白和辅助蛋白的共同作用,结构蛋白包括UreA、UreB和UreC,辅助蛋白包括UreD/UreH、UreE、UreF和UreG。大多数细菌的脲酶由3个结构蛋白UreA、UreB和UreC形成 复合物(UreABC)3[9-10],如克雷伯氏菌(Klebsiellaaerogenes)和巴氏芽胞杆菌(Sporosarcinapasteurii)[11-12]。而幽门螺杆菌的脲酶则是由两个结构蛋白UreA和UreB形成大分子复合物( (UreAB)3)4[13]。脲酶蛋白的双核镍金属活性中心位于结构蛋白UreC中,因此结构蛋白处于脲酶活化的核心地位,且镍离子对其活化及蛋白构象的变化也具有重要作用。鉴于脲酶结构蛋白的独特生物功能,研究其蛋白结构、活性中心以及活化过程具有重要意义。中国农业科学院北京畜牧兽医研究所前期利用宏基因组测序技术筛选获得了瘤胃细菌脲酶基因簇,为了解脲酶的生物功能及蛋白结构,首先需要获得高纯度的蛋白。本研究克隆了脲酶的结构基因,并对其蛋白进行了表达纯化,以期为进一步研究蛋白结构、蛋白质之间的互作关系等提供参考。
1 材料与方法
1.1 材料
1.1.1 菌种与载体 大肠埃希菌XL10、BL21感受态细胞购自上海唯地,含His标签的pETDuet-1、pET28a+以及蛋白质分子质量标准(范围11~245 kD)均购自美国NEB。
1.1.2 试剂 高保真热启动DNA聚合酶、dNTP、NdeI、EcoRV、XhoI、EcoRI、NotI均购自 NEB公司;琼脂糖凝胶回收试剂盒、质粒小提试剂盒购自天根;His标签纯化柱histrap hp、脱盐柱HiPrep 26/10 Desalting、分子筛HiLoad 26/60 Superdex 200 column、分子筛蛋白标准品(Marker-Gel Filtration Calibration Kit)均购自美国GE通用电气;氨苄青霉素、硫酸卡那霉素购自美国Amresco。
1.1.3 仪器与设备 细胞破碎机(聚能JN-02C,广州聚能JNBIO);高速离心机(CR21GIII,日立Hitachi);蛋白纯化仪(GE AKTA Pure,美国GE通用电气);PCR扩增仪(伯乐T100,Bio-Rab);电泳仪(PowerPoc Basic,Bio-Rad)。
1.2 方法
1.2.1 脲酶基因的筛选 本研究前期构建了奶牛瘤胃微生物宏基因组,通过与已知脲酶基因对比,筛选获得了1个瘤胃脲酶基因簇,其中结构基因为ureA、ureB、ureC。
1.2.2 原核表达载体的构建 分别设计脲酶基因ureA、ureB、ureC的特异性引物,下划线为酶切位点:NdeI/XhoI、EcoRI/NotI、EcoRI/NotI,引物序列见表1。引物由生工生物工程(上海)股份有限公司合成。提取瘤胃微生物DNA后,以基因组DNA模板进行PCR扩增。反应体系(25 μL):5×HF缓冲液5 μL,上下游引物各0.25 μL,dNTP 0.5 μL,DNA模板0.5 μL,DNA聚合酶0.25 μL,双蒸水18.25 μL。PCR扩增程序:94 ℃预变性3 min;94 ℃变性30 s,60 ℃退火1 min,72 ℃延伸1 min,共30个循环;72 ℃延伸10 min。PCR产物经琼脂糖凝胶电泳检测后纯化回收。在37 ℃条件下,采用限制性内切酶EcoRI和NotI将ureB基因与载体pET28a+酶切2 h后,采用连接酶将酶切后的ureB与pET28a+室温连接30 min构成重组质粒pET28-ureB。将pET28-ureB转化到E.coliXL10挑取克隆,小量提质粒后,酶切后用1%琼脂糖凝胶电泳进行鉴定,将鉴定后的质粒送生工生物工程(上海)股份有限公司进行基因测序,验证目的条带是否与载体正确连接。ureC和载体pETDuet-1用EcoRI和NotI内切酶处理后,采用连接酶连接构成重组质粒pET-ureC(鉴定方法同pET28-ureB)。再将pET-ureC和ureA用NdeI和XhoI酶切后重新构成重组质粒pET-ureA&C(鉴定方法同pET28-ureB)。

表1 基因ureA、ureB、ureC的特异性引物
1.2.3 脲酶UreABC的表达及鉴定 将pET-ureC&A和pET28-ureB共同转化到大肠埃希菌E.coliBL21。涂板过夜培养后挑取1个单菌落于具有50 μg/mL氨苄青霉素和50 μg/mL硫酸卡那霉素的LB液体培养基中,37 ℃,250 r/min培养12 h。扩大培养至1 L体系,37 ℃,250 r/min培养至OD600为0.6~0.8,0.5 mmol/L IPTG诱导蛋白表达,16 ℃培养过夜,8 000 r/min 离心15 min,弃上清,收取菌体蛋白。将诱导的200 μL表达产物离心后保留沉淀,加入2×SDS上样缓冲液(125 mmol/L Tris-HCl pH 6~8、20%甘油、4% SDS、2%溴酚蓝),95 ℃煮沸10 min。变性后的蛋白用SDS-PAGE(12%的丙烯酰胺分离胶、4%丙烯酰胺浓缩胶)鉴定,电压120 V,电泳1 h,结束后考马斯亮蓝染色1 h,脱色液(10%乙酸)脱色后观察结果并拍照保存。
1.2.4 蛋白纯化及鉴定 将收集的菌体蛋白用20 mmol/L Tris-HCl、pH 7.5,500 mmol/L NaCl、1 mmol/L三氯乙基磷酸酯(TCEP)、40 mmol/L咪唑(Imidazole)重悬,高压(1 200 MPa)破碎12 000 r/min离心后保留上清。蛋白纯化仪(GE)及镍柱His-Trap进行His纯化(纯化参数设置见表2),洗脱时,纵坐标达到30 mAU开始收集蛋白,信号降到80 mAU停止收集,将不同的馏分蛋白进行电泳鉴定。为保证蛋白的正确折叠,将目标蛋白用脱盐柱HiPrep 26/10 Desalting置换到低盐溶液(20 mmol/L HEPES,pH 7.5,200 mmol/L NaCl, 1 mmol/L TCEP)中,纯化参数设置见表3。取1 mg置换后的蛋白用分子筛进一步纯化分离,并与蛋白标准品进行比较,纯化参数设置见表4。然后收集不同峰值的蛋白,进行SDS-PAGE鉴定。

表2 镍柱纯化的参数设置

表3 脱盐柱纯化的参数设置

表4 凝胶层析柱——分子筛纯化的参数设置
1.2.5 脲酶结构蛋白活性测定 脲酶活性测定采用苯酚-次氯酸钠比色法(靛酚蓝比色法)[14]。为了鉴定镍离子对脲酶结构蛋白的活化影响,设置添加镍离子的实验组及不添加镍离子的对照组,每组3个平行。挑取脲酶阳性克隆,加入15 mL LB培养基中诱导表达脲酶蛋白。8 000 r/min 离心15 min,沉淀用50 mmol/L HEPES缓冲溶液清洗两次后,收集菌体。再用10 mL 50 mmol/L HEPES缓冲溶液将菌体重悬,冰上超声破碎(220 V、30%、2 min)。12 000 r/min,4 ℃离心2 min,留取上清。将0.5 mL上清加入缓冲液(50 mmol/L HEPES,pH 7.0,50 mmol/L 尿素,150 mmol/L NaCl和100 mmol/L NaHCO3)中,实验组还需在缓冲液中添加100 μmol/L Ni2+,使终体积1 mL,37 ℃温育20 min。接着加入1.5 mL酚-硝普纳溶液和1.5 mL碱性次氯酸钠溶液均匀混合。37 ℃温育30 min,分光光度计在625 nm处测定吸光度。
2 结果与分析
2.1 脲酶基因的扩增
使用特异性引物对牛瘤胃液基因组DNA进行PCR扩增, PCR产物经琼脂糖凝胶电泳检测。由图1可知,在退火温度60 ℃时,均有明亮的条带,与标准 Marker对照发现,与理论上脲酶基因ureA、ureB、ureC分别的片段大小303、315、1 701 bp相符。
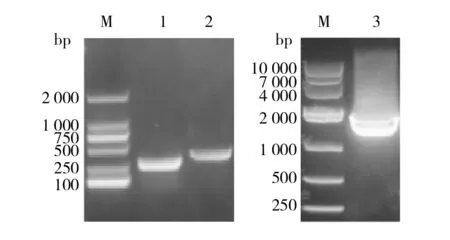
图1 基因ureA、ureB、ureC PCR产物琼脂糖凝胶电泳图Fig.1 PCR product agarose gel electrophoresis of ureA,ureB,ureC M:DNA Marker;1:ureA引物PCR扩增产物;2:ureB引物PCR扩增产物;3:ureC引物PCR扩增产物 M: DNA Marker; 1: PCR product of ureA primer; 2: PCR product of ureB primer; 3: PCR product of ureC primer
2.2 脲酶基因的克隆与鉴定
ureC、ureA、ureB的PCR扩增产物经过纯化、酶切、凝胶回收,形成重组质粒后转化至大肠埃希菌。将其摇菌提取pET-ureC、pET-ureC&A、pET28-ureB质粒后分别EcoRI/NotI、NdeI/EcoRV、EcoRI/NotI双酶切鉴定,结果均得到了与理论相符的线性载体和目的基因(分别为303、1 701、315 bp)两条带,初步判定为阳性克隆(图2),将筛选得到的阳性克隆质粒送至上海生工生物工程有限公司测序,结果表明,脲酶基因与质粒正确连接,确定为阳性克隆。
2.3 脲酶UreABC蛋白的表达及纯化
将正确克隆的pET-ureC&A和pET28-ureB质粒共同转化到大肠埃希菌BL21感受态细胞,用IPTG 诱导表达蛋白进行His纯化,洗脱时出现了两个主要的峰,为了判定目标蛋白所处的位置,将收集的不同馏分蛋白进行SDS-PAGE凝胶电泳检测,结果显示(图3),第1个峰主要包含His标签UreC与无标签UreA(约11 kD)2个条带,第2个峰包含3个条带,分别为含有His标签-UreC(约60 kD)、His标签-UreB(约11 kD)以及无标签的UreA。并且蛋白以可溶性形式表达,上清中含量很高,沉淀中含量较低。将第2个峰收集的蛋白通过脱盐柱,置换到低盐溶液。
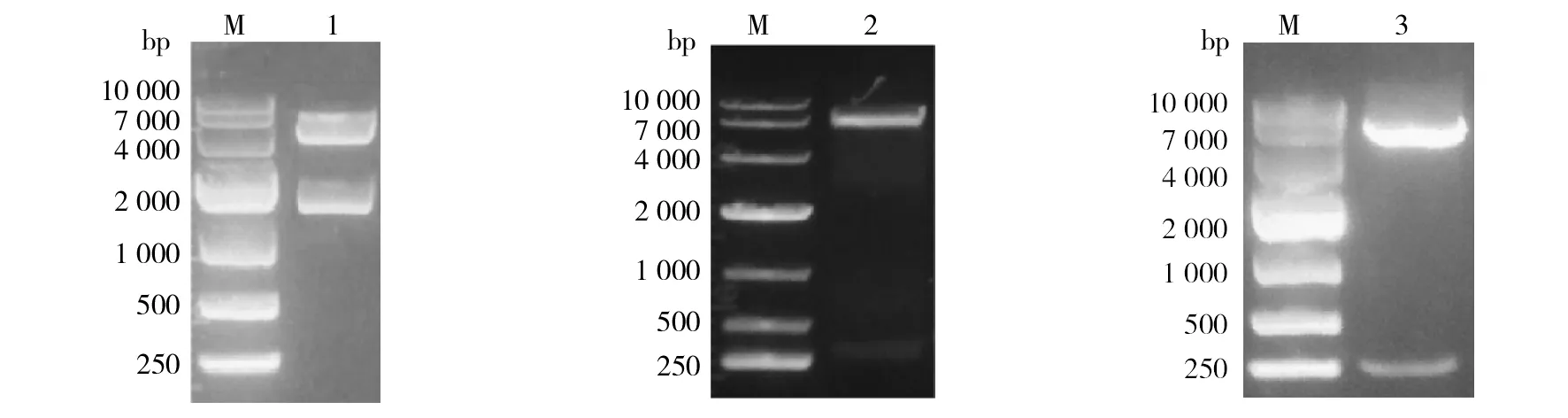
图2 pET-ureC、pET-ureC&A和pET28-ureB质粒双酶切琼脂糖凝胶电泳图Fig.2 pET-ureC,pET-ureC&A and pET28-ureB plasmid double digestion agarose gel electrophoresis M:DL10 000 Marker;1:pET-ureC质粒双酶切;2:pET-ureC&A质粒双酶切;3:pET28-ureB质粒双酶切 M: DL10 000 DNA Marker; 1: digestion plasmid pET-ureC; 2: digestion plasmid pET-ureC&A; 3: digestion plasmid pET28-ureB

图3 UreABC蛋白表达及镍亲和层析纯化Fig.3 Protein expression and purification(His-trep)of UreABC M:蛋白质分子质量标准; 1:破碎后的菌液; 2:破碎离心后的上清; 3:破碎离心后的沉淀; 4:镍亲和层析的流穿液;5:镍亲和层析的洗涤液; 6~8:镍亲和层析洗脱的第1个峰的3个馏分;9~14:镍亲和层析洗脱的第2个峰的6个馏分 M:Protein Marker ;1:broken bacterial liquid;2:supernatant after crushing;3:precipitated precipitation;4:flow through;5:wash;6-8:His-trep elution(three fractions of the first peak);9-14:His-trep elution(six fractions of the second peak)
根据NCBI数据库氨基酸比对分析,该蛋白可能以同源三聚体的形式存在,目标蛋白约240 kD,可以通过分子筛进一步纯化目标蛋白。结果见图4,出现6个主要的峰,通过与分子筛蛋白标准品的对比发现,目标蛋白应位于第5个峰处。通过SDS-PAGE鉴定(图5),峰5包含有目标蛋白复合物(UreABC)3。
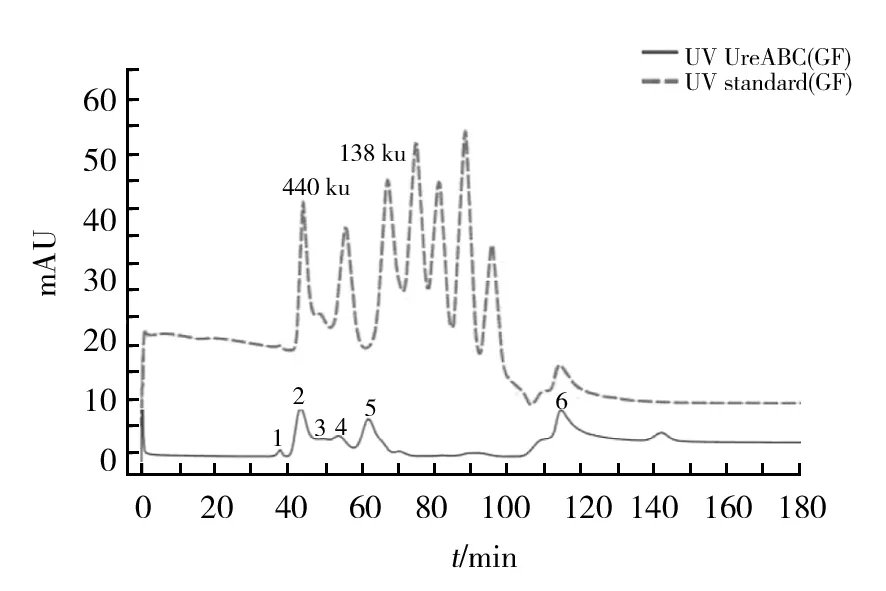
图4 蛋白标准品与蛋白UreABC 通过分子筛后的对比Fig.4 Comparison of protein standards and protein UreABC after size exclusion chromatography
2.4 脲酶活性测定
通过苯酚-次氯酸钠法测定镍离子对脲酶结构蛋白UreABC活性可知(图6),添加镍离子的脲酶蛋白是未添加镍离子脲酶蛋白活性的3.1倍,说明镍离子对脲酶蛋白发挥活性具有重要作用。

图5 UreABC蛋白用分子筛纯化后的电泳图Fig.5 Electropherogram of UreABC protein was purified by size exclusion chromatography M:蛋白质分子质量标准; 1:分子筛纯化后的第1个峰;2:分子筛纯化后的第2个峰;3:分子筛纯化后的第3个峰;4:分子筛纯化后的第4个峰;5:分子筛纯化后的第5个峰;6:分子筛纯化后的第6个峰 M: Protein Marker; 1: peak 1(purified by size exclusion chromatography); 2: peak 2(purified by size exclusion chromatography); 3: peak 3(purified by size exclusion chromatography); 4: peak 4(purified by size exclusion chromatography); 5: peak 5(purified by size exclusion chromatography); 6: peak 6(purified by size exclusion chromatography)
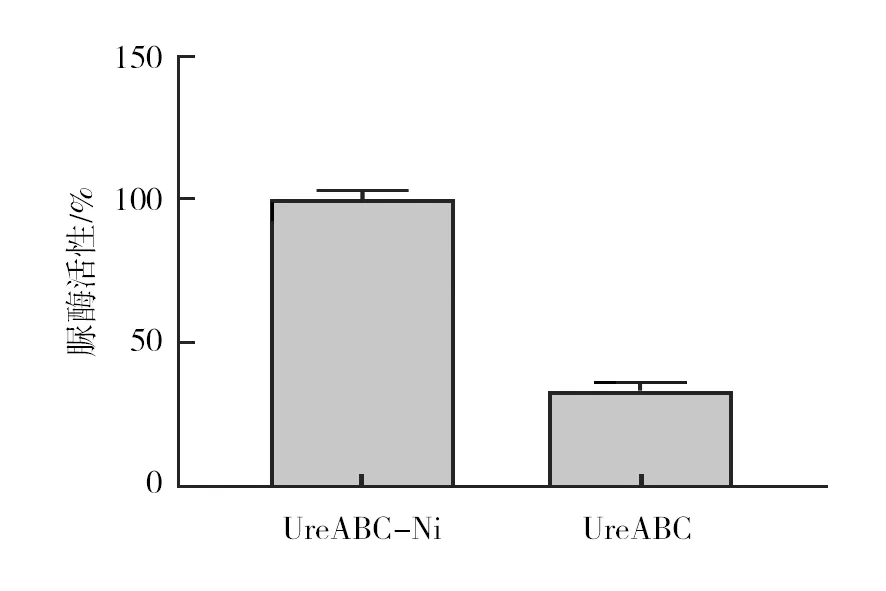
图6 Ni2+对脲酶结构蛋白UreABC活性的影响Fig.6 Effect of Ni2+ on the activity of urease structural protein UreABC
3 讨 论
本研究采用体外原核表达系统表达脲酶基因。成功构建了原核表达载体pET-ureC&A和pET28-ureB,将两个载体共同转化到大肠埃希菌BL21中诱导结构蛋白的表达。由于pET载体系列上所包含有的His标签,分子量较小,一般对目的蛋白的理化性质、蛋白结构影响较小且纯化过程简便。所以在构建表达载体时,ureC和ureA插入了双启动子表达载体的pETDuet-1,ureB插入了单启动子表达载体pet28a+,并通过镍亲和层析、凝胶过滤层析(脱盐柱、分子筛)逐步纯化蛋白复合物,最终得到了约240 kD的UreABC蛋白复合物。而Carter[15]在表达克雷伯氏菌的结构蛋白复合物时,先将野生型菌全长的基因簇插入pKK223-3载体中,之后以该重组质粒为DNA模板,采用针对结构基因簇ureABC所设计的引物进行PCR,最后将PCR产物插入pACT3载体诱导表达了结构蛋白复合物。上述两种方法均采用蛋白质体内结合的方法获得了蛋白复合物,但是表达后的蛋白比均不是1∶1∶1。杨少媛等[16]采用蛋白体外结合的方法,将FtsZ和MinC分别转化、纯化再混合,得到了FtsZ和MinC蛋白复合比例约为1∶1的复合物。但是采用针对结构基因簇ureABC所设计的引物对瘤胃液DNA进行PCR之后,将基因簇ureABC插入载体 pETDuet-1并且诱导蛋白表达,实验结果显示没有同时表达出脲酶蛋白UreA、UreB、UreC。此外,结构蛋白UreB、UreC单独表达时的可溶性较低,以包涵体的形式存在,并且性质不稳定,易降解,也不宜采用蛋白体外结合的方式获得蛋白复合物。
将细菌脲酶3个结构基因诱导共同表达蛋白后,可能会形成多种蛋白复合物。如图3所示,镍柱纯化之后洗脱时出现两个峰,第1个峰主要是UreC与UreA蛋白形成的复合物,因为UreA不带有His标签,只有其与UreC结合才可以被共同洗脱。第2个峰包含UreC、UreB及UreA三个蛋白,之所以出现第2个峰,是由于UreB表达时包含有His标签,当它们形成复合物后,与镍的结合能力更强,所以洗脱时所需要的咪唑浓度高于第1个峰。该结果说明3个结构基因在体内共同表达为蛋白复合物UreABC。其次,复合物条带比例不是1∶1∶1,洗脱下来的蛋白中UreC含量远远高于UreA和UreB的含量,推测有两种可能:首先,UreC和UreB本身携带有His标签,除了以复合物形式结合在镍柱上,还有单体形式的蛋白存在,所以UreA的条带最弱;其次,可能当3种蛋白共同存在时,UreC对镍柱的亲和能力最强,导致三者量的不均衡[16]。用分子筛分离纯化目标蛋白时,出现多个峰,根据SDS-PAGE结果显示,第1个和第2个峰蛋白浓度较低,且第2个峰复合物条带比例不是1∶1∶1。第3个、第4个和第5个峰的复合物条带比例均接近1∶1∶1。通过与蛋白标准品对比分析,约240 kD的目标蛋白应位于440~138 kD之间。但是,纯化后的蛋白在200 kD左右仍有杂蛋白,该杂蛋白与目标蛋白大小接近。王琼秀等[17]纯化原核表达的肿瘤坏死因子相关凋亡诱导配体蛋白时,通过硫酸铵初级分离和阳离子离子交换层析法获得了高纯度的蛋白。因此,若要提高本试验的蛋白纯度,应使用离子交换层析柱对蛋白进一步纯化。
脲酶的活化也是镍离子的金属组装过程,脲酶活性测定试验显示,获得了具有活性的脲酶蛋白,并且镍离子对脲酶结构蛋白的激活有重要作用。该过程中各辅助蛋白需要协同作用:UreE可以募集镍离子并将其传递给UreG,之后UreG可能将镍离子传递给UreF,穿过UreD/H通道,最终镍离子插入脲酶结构蛋白的活性中心[18-22]。基于结构蛋白在活化过程中的核心作用,其相关研究受到了广泛关注。目前可知UreC拥有双核镍金属活性位点,而UreA和UreB的生物功能仍然未知[23]。此外,多种外界因素均会影响脲酶的活性,包括Ni2+、pH、碳酸氢盐和GTP等[24]。有研究发现,在不含辅助蛋白的条件下,添加过量的镍离子和二氧化碳,可以适当激活脲酶蛋白。脲酶脱辅基蛋白首先与镍离子孵育,然后置于碳酸氢盐溶液中,则无法激活脲酶蛋白,这说明碳酸氢盐必须先于镍与脲酶蛋白结合才能使其激活[25]。目前获得了脲酶蛋白复合物,然后基于脲酶活性展开更深入的研究,以期为脲酶抑制剂的开发提供参考。

