红曲霉繁殖相关wetM基因的克隆与生物信息学分析
王炫栋, 杨孙玉悦, 胡凌鸣, 何智鹏, 丁允章, 蒋冬花
(浙江师范大学 化学与生命科学学院,浙江 金华 321004)
红曲霉(Monascusspp.)是我国历史悠久的真菌资源,在许多方面都有非常广泛的应用[1-2]。探究红曲霉的繁殖机制能够更好地调控红曲霉的生长发育。红曲霉繁殖过程中的子囊孢子和分生孢子在工业、食品业和基础研究中都有重要意义[3]。在工业上调控孢子产生数量可降低生产成本,控制污染。在食品业中,孢子的数量与次级代谢产物[4-5]的产量息息相关。在医学上,孢子是致病真菌散播的主要方式。因此,利用生物信息学探明红曲霉繁殖机制中相关基因的功能在各个方面都具有重要意义。在模式丝状真菌构巢曲霉(Aspergillusnidulans)中已经探明与繁殖相关的基因有brlA、wetA、abaA等,其相对应的转录调控蛋白家族也已有较多的研究[6-9]。但红曲霉中的繁殖调控路线和众多基因还不明确。本研究在红曲霉中发现了与构巢曲霉孢子发育相关的wetA(wet-white “湿-白样”)有较高同源性的基因,推测这个基因可能与红曲霉繁殖相关,命名为wetM(wet-white Monascus)基因。从红曲霉中首次克隆出wetM基因,并对此基因进行测序、基因调控转录蛋白生物信息学分析和结构功能的预测。
1 材料与方法
1.1 材料
1.1.1 菌株 从红曲米中筛选纯化保藏的紫色红曲霉(Monascuspurpureus)Mp-21菌株。
1.1.2 培养基 马铃薯葡萄糖琼脂(PDA)培养基[10]。
1.2 方法
1.2.1 紫色红曲霉菌株Mp-21活化 将保藏管Mp-21菌株的适量菌丝接种至PDA平板,30 ℃恒温培养7 d后,将菌落中的新生菌丝接种至新的培养基中,同样条件下继续培养,重复3次[11]。
1.2.2 紫色红曲霉Mp-21基因组DNA提取 使用OMEAGA真菌提取试剂盒,按照说明书操作提取紫色红曲霉Mp-21菌株基因组DNA。
1.2.3 紫色红曲霉Mp-21wetM引物设计 根据紫色红曲霉Mp-21转录组测序注释结果[12],筛选出与繁殖相关下游转录调控因子WetA蛋白的反转录序列(cDNA)具有较高同源性的Unigenes序列TRINITY_DN904_c0_g1。通过Primer 5.0软件,根据所得的Unigenes序列,设计后续实验中所需的引物[13-15]。引物wetM-F序列为TGCCGAGGTTTGGACAGTTA;引物wetM-R序列为ACGGATTGCCAGGCTCTATT。
1.2.4 紫色红曲霉Mp-21wetMPCR基因扩增和测序 得到引物后,使用高保真 LA Tap DNA polymerase,以紫色红曲霉Mp-21基因组DNA为模板进行PCR扩增反应[16-17]。反应体系:模版1 μL;LATaqDNA polymerase 0.25 μL;dNTP Mixture 4 μL;10×LA PCR Buffer 2.5 μL;brlM-F/wetM-F 1 μL;brlM-R/wetM-R 1 μL;ddH2O(Up to 25 μL)。反应条件:94 ℃ 预变性3 min;94 ℃变性 30 s,50 ℃退火30 s, 循环30次;72 ℃延伸3 min;72 ℃终延伸10 min。PCR扩增结束后,将产物进行琼脂糖凝胶电泳,电泳条件120 V、170 mA。电泳后在紫外光分析仪下分析并回收目的条带,随后使用Axygen DNA Gel kit试剂盒回收目的条带中的DNA。
1.2.5 紫色红曲霉Mp-21wetM基因生物信息学分析 将克隆回收的wetM基因片段送至上海生工进行测序。利用ExPASy网站中的Translate tool工具对测序结果进行氨基酸序列预测,分析其开放阅读框(ORF)。利用NCBI网站中的BLAST功能对测序结果进行比对,并提交到Conserved Domains中识别此蛋白序列中的保守结构域并分析。利用得到的ORF框序列,使用dnaMAN软件比对Mp-21wetM基因编码氨基酸序列与其他丝状真菌的繁殖相关因子WetA蛋白氨基酸序列的同源性和相关性。利用ExPASy网站中的ProtParam tool工具预测基因编码蛋白质的理化性质和一级结构,对氨基酸个数、分子量、等电点、亲水性等进行分析。通过PSIPRED 4.0软件分析wetM基因编码的WetM蛋白二级结构,该软件通过多序列比对方法提取具有同源性的序列二级结构以及分类信息来对提交序列进行二级结构预测。通过ExPASy网站中的Swiss-Model工具对蛋白结果进行同源比对建立蛋白质三级结构模型,根据模型对蛋白质的结构进行分析,预测其功能。
2 结果与分析
2.1 紫色红曲霉Mp-21 wetM基因克隆结果
紫色红曲霉Mp-21wetM基因克隆凝胶电泳结果如图1所示。wetM基因PCR克隆产物在电泳后只出现一条明亮的条带,出现在4 500 bp对应的位置。根据对应Marker,可初步判断产物片段长度和转录组测序中Unigenes长度大致相同。
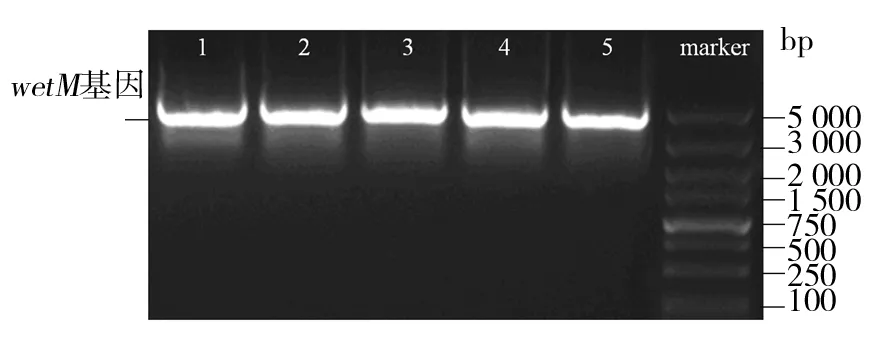
图1 紫色红曲霉Mp-21 wetM 基因克隆结果Fig.1 Cloning results of wetM genes of Monascus purpureus Mp-21
2.2 紫色红曲霉Mp-21 wetM基因分析
对所得测序结果中的转录序列进行氨基酸序列预测,利用ExPASy网站中的Translate tool工具得到ORF[18-19]。wetM基因氨基酸序列预测结果如图2所示,其中存在一个1 659 bp的完整ORF开放阅读框,理论负责翻译552个氨基酸。将ORF开放阅读框内的氨基酸序列提交到Conserved domains,分析结果显示WetM蛋白无明显保守结构域,再将该氨基酸序列提交到BLAST进行比对,比对结果显示WetM蛋白氨基酸序列与其他丝状真菌中的WetA蛋白有一定的同源性,其中同源性得分最高的是烟曲霉(A.fumigatus)的WetA蛋白。
将紫色红曲霉Mp-21wetM基因的ORF开放阅读框中的氨基酸序列与BLAST比对结果中同源性最高的费氏曲霉(A.fischeri)、烟曲霉(A.fumigatus)、乌达加瓦曲霉(A.udagawae)、棒曲霉(A.clavatus)、赭曲霉(A.ochraceoroseus)、兰贝利曲霉(A.rambellii)WetA蛋白氨基酸序列进行比对。结果显示,紫色红曲霉wetM基因转录调控的WetM蛋白与其他丝状真菌的WetA蛋白相似度在37.4%~39.5%范围内。其中相似度最高的是赭曲霉WetA蛋白序列,为39.5%;相似度最低的是费氏曲霉WetA与烟曲霉WetA序列,为37.4%。

图2 紫色红曲霉Mp-21 wetM 基因开放阅读框分析结果Fig.2 ORF analysis of wetM gene of Monascus purpureus Mp-21
WetM蛋白与其他丝状真菌在大部分区域相似度都较低,只有在氨基酸序列的最后90个氨基酸(460~552)中表现出极高的相似性,曲霉属真菌WetA蛋白氨基酸序列也同样具有这样的特点,即整体的蛋白氨基酸序列差异较大,但是末端的序列中相似度极高。在序列长度方面,WetM蛋白氨基酸序列与其他丝状真菌WetA蛋白氨基酸序列长度相近(图3)。
2.3 紫色红曲霉Mp-21 wetM基因编码蛋白质一级结构分析
利用ExPASy网站中的 ProParam tool工具对wetM基因编码蛋白质一级结构和理化性质进行进一步分析预测。预测该蛋白的原子总数为8 302个,分子式为C2605H4067N781O831S18,分子量为60 199.76,氨基酸数为552个,带正电荷的残基总数53个,带负电荷的残基总数51个,理论等电点(pI)为8.15,不稳定指数为79.97,脂肪族指数为57.66,亲水性平均值-0.712。预测蛋白中丝氨酸(Ser)的含量最高,占氨基酸总数的15.6%。在预估的半衰期中完整蛋白预计半衰期为哺乳动物网织红细胞内或体外环境下30 h,在酵母菌细胞内为>20 h,在大肠埃希菌细胞内>10 h。根据不稳定指数将WetM蛋白归类为不稳定蛋白。
2.4 紫色红曲霉Mp-21 wetM基因编码蛋白质二级结构分析
通过PSIPRED 4.0软件,利用多序列比对提取同源性序列对wetM基因编码蛋白质进行二级结构预测。平均预测准确率为78%,在所有蛋白质二级结构预测方法中居于领先地位[20-21]。按蛋白质二级结构分类标准,蛋白质二级结构主要包括α-螺旋(H)、β-折叠(E)、β-转角(T)和无规则卷曲(C),各种类型的二级结构并不是均匀分布在蛋白质中,在不同的蛋白质中二级结构的种类和数目也不尽相同。WetM蛋白的二级结构分析见图4,粉色代表α-螺旋,黄色代表β-折叠,灰色代表无规则卷曲,且图中蓝色越深则代表准确度越高。WetM蛋白质预测结果显示存在12个α-螺旋区域和2个β-折叠区域。
2.5 紫色红曲霉Mp-21 wetM基因编码蛋白质三级结构同源建模
通过ExPASy网站中的Swiss-Model功能将开放阅读框(ORF)导入,利用保守性高的蛋白质三级结构进行同源比对,建立蛋白质三级结构模型[22]。建模结构见图5。WetM蛋白的保守功能区域含有多个结构类型,属于非典型结构域。

图3 紫色红曲霉Mp-21 WetM蛋白序列与其他丝状真菌WetA蛋白序列比对Fig.3 Comparison of protein sequences of WetM of Monascus purpureus Mp-21 with those of WetA of other filamentous fungi 其他丝状真菌:费氏曲霉、烟曲霉、乌达加瓦曲霉、棒曲霉、赭曲霉、兰贝利曲霉 other filamenteus fungi:A. fischeri, A. fumigatus, A. udagawa, A. clavatus, A. ochraceus, A. lambelli

图4 紫色红曲霉Mp-21 WetM蛋白序列二级结构预测Fig.4 Prediction of secondary structure of Monascus purpureus Mp-21WetM protein

图5 紫色红曲霉Mp-21 wetM基因编码 蛋白质保守区三级结构预测图Fig.5 Prediction of the tertiary structure of protein conserved region encoded by wetM genes of Monascus purpureus Mp-21
3 讨 论
首次从紫色红曲霉Mp-21中克隆出红曲霉繁殖相关基因wetM,并通过基因测序和开放阅读框分析得到了该基因的ORF序列,对ORF序列进行氨基酸预测及蛋白结构功能预测。对其编码产生的WetM蛋白进行生物信息学分析,分析角度大致从保守结构域、蛋白同源性分析、蛋白各级结构预测等方面出发。通过各项分析预测可知,WetM蛋白无明显保守结构域,将WetM蛋白与其他丝状真菌的WetA蛋白比对后发现,具有一定的同源性,蛋白氨基酸序列差异较大,但在末端则表现出高度相似。通过分析WetM蛋白的一级结构和理化性质,总结其分子式、理论等电点、亲水性平均值并预测不稳定指数后将该蛋白归类至不稳定蛋白。WetM蛋白的二级结构分析可知该蛋白存在12个α-螺旋区域和2个β-折叠区域。根据同源建模得到的蛋白质三级结构模型发现,WetM蛋白的保守功能区域含有多个结构类型,属于非典型结构域。
本研究在一定程度上可以完善相应家族蛋白的研究,以及目前尚未明晰的红曲霉繁殖途径。针对繁殖相关的基因及调控产生的相关蛋白结构与功能进行研究,对红曲霉繁殖调控手段即控制子囊孢子和分生孢子的产生提供了理论参考,能够在与红曲霉相关的生产中更科学地调控生产条件,对红曲霉在各个产业中的应用具有积极作用。