纳米孔测序技术在疾病检测中的研究进展
张皓博,樊晓旭,刘蒙达,迟庆安,2,刘婷婷,亓 菲,3,孙淑芳,范伟兴
(1.中国动物卫生与流行病学中心,国家动物结核病参考实验室,山东青岛 266032;2.海南省动物疫病预防控制中心,海南海口 571100;3.青岛农业大学动物医学院,山东青岛 266109)
纳米孔测序技术(nanopore sequencing)为近几年兴起的第三代测序技术,由牛津纳米孔技术公司(Oxford Nanopore Technologies,ONT)开发。纳米孔测序技术与以往的测序方法不同,当长的单链DNA穿过稳定在电阻聚合物膜中的蛋白质纳米孔时,就可以直接检测核苷酸,无需进行主动的DNA合成。通过在电阻聚合物膜中设置电压,传感器可以实时检测到随着DNA分子穿过孔的离子电流变化,从而判定核苷酸的类别及修饰情况[1]。ONT还开发了MinION纳米孔测序仪。MinION是一款大小与手机相当的USB设备,可对病原进行现场快速测序[2],极大提高了分析质量和速度,有助于实现疾病的“早干预、早处置”。
利用纳米孔测序技术,人们可将DNA或RNA序列,通过纳米孔进行实时单分子测序。该技术高通量、实时、灵活、超长读长、低成本,可直接测序DNA或RNA,无需PCR扩增和样品化学标记,极大简化了测序试验流程[3]。该技术不受实验室环境限制,可应用于野外环境,如高山、丛林、北极和空间站等[4]。本文就纳米孔测序技术的原理、应用及在疾病检测中的优势及未来发展进行综述。
1 原理
1.1 测序基本原理
纳米孔测序技术,以插入在聚合物膜中的生物纳米孔为基础,将膜浸没在电解质溶液中以生成高电阻,再加以适当电压,使离子仅能通过纳米孔以形成电位差,从而产生电流。当单个核苷酸进入纳米孔时,会导致电流发生特征性扰动。这种扰动与核苷酸碱基大小和化学性质相关,因而通过检测这种扰动,可实现对DNA或RNA序列的测定。在MinION设备核心部分,DNA解旋酶将DNA解开,使一条链穿过蛋白质孔,而每个DNA碱基的独特构象都会引起电流的特征性扰动,从而读出序列(图1-A)。
纳米孔起源于库尔特计数和离子通道的发现,在外部施加电压的情况下,尺寸略小于孔径的颗粒可以穿过纳米孔。纳米孔可以嵌入生物聚合物膜中,将含有电解质的反应器分隔为顺式和反式隔室(图1-B)。在偏置电压下,溶液中的电解质例子通过电泳穿过纳米孔,从而产生例子电流信号,根据信号扰动,实现纳米孔测序。
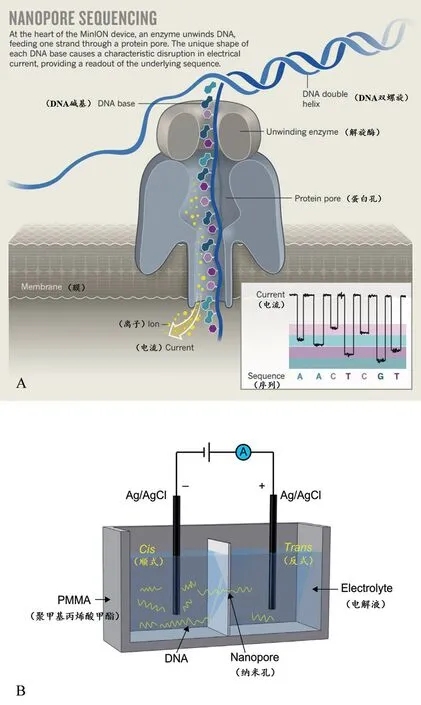
图1 纳米孔测序原理示意图[3,5]
1.2 DNA片段修饰
试验[6]表明,可以通过施加适当电势来引导单链DNA片段穿过纳米孔,但必须对DNA片段和纳米孔进行轻微修饰,以实现DNA通过生物聚合物纳米孔的过程可控,从而确保信号可测量。为了使DNA片段到达纳米孔并控制片段通过纳米孔的速度,首先需要将片段化的DNA转化为5'磷酸化和3'dA尾平末端修复的DNA,实现对DNA片段的修饰,然后通过DNA片段A和T突出端与测序所需的引导接头Y连接。引导接头载有一个运动蛋白,可以控制DNA穿过纳米孔以及与后续核苷酸序列相结合,这是DNA片段附着到电阻聚合物膜上和在纳米孔中移动所必需的,从而使DNA捕获灵敏度提高了20 000倍。
1.3 测序读取方式
目前,ONT提供了3种测序读取方式。不同方式的读取会影响测序的可信度。最简单的方法是单向读取[one-directional(1-D)read],无论样品是单链还是双链,都仅读取1条链(模板链)。
如果是双向读取[two-directional(2-D)read],则样品必须在文库制备过程中为双链或变为双链。与1-D仅读取模板链的碱基相比,2-D方式中,使用发卡接头将其连接到双链DNA互补链,测序时紧接着模板链通过纳米孔,因此互补链的碱基也被读取,从而能够在1次读取期间对两条链进行测序。通过发卡接头引起的电流特征性扰动,使用适当的生物信息学和数据处理技术,使读取结果可以在生物信息上实现正向和反向阅读。
1-D和2-D的组合读取方式称为“1-D2”。与2-D读取方式相同,在1-D2方式中,样品也必须是双链或变成双链的,需要1-D2接头和引导接头,但不需要发卡接头。测序时,先读取模板链,然后将运动蛋白直接连接到互补链的反向1-D2接头上,直接进行反向读取。与1-D相比,由于两条链的碱基都被读取且利用生物信息学进行了比较,因此2-D和1-D2读取方式增加了测序的可靠性[7]。
2 测序仪
2007年,ONT公司获得了纳米孔测序专利许可,并于2010年开始进行DNA测序工作。在2012年基因组生物技术进展会议上,ONT公布了第一款商用的纳米孔测序设备MinION(图2)。MinION是一款仅为90 g的便携式设备,芯片多达2 048个纳米孔,通过专用集成电路(ASIC)将其分成512组,测序可在数分钟内完成,仪器价格低至1 000美元。作为纳米孔测序的主要测序仪,人们对其寄予了极大的希望和关注。2014年以来,ONT凭借MAP(MinION access programme)项目,将MinION应用于多个领域[8]。目前,ONT又发布了新的MinION Mk1设备和试剂。该设备结合了MinION的实时、快速、便携等优点,并具有更高通量、更强大的集成计算以及高分辨率的触摸屏,提供了一个可在任何地方进行DNA和RNA测序的分析解决方案。

图2 MinION和MinION Mk1C测序仪
3 应用
3.1 细菌学检测
许多研究已表明,纳米孔测序具有良好的通用性和适用性。MinION不仅可以直接在实验室使用,也可以在实验室外的多种环境中广泛使用。研究人员已将MinION带到北极,研究多年冻土冰楔中微生物群落的基因组特征[9],以及在位于加拿大阿尔伯塔省的北约反恐技术中心的军事训练场检测生物制剂[10],甚至在国际空间站利用病毒、细菌和小鼠DNA来研究评价MinION在太空中表现[4]。
MinION也已用于许多与细菌相关的传染病研究。研究人员利用MinION对16S rRNA和ITS基因进行测序,成功鉴定了临床分离的细菌和真菌菌株[11],使用9.5版的芯片能够获得99%的准确性,这意味着在临床实验室,可采用MinION进行靶向测序。在一项研究[12]中,利用MinION从脑膜炎患者的血液培养物中分离样品,进行16S rRNA的靶向测序,分类分析证实,传染源是一种很少引起人类脑膜炎的人兽共患病原体——胎儿弯曲杆菌。此外,纳米孔测序还能够检测细菌导致免疫逃避的突变位点,这已在百日咳杆菌全基因组测序研究[13]中得到证实。
纳米孔测序也应用于鉴定临床病例中难以确定传染源的病原菌。在一项研究[14]中,从被诊断为感染性心内膜炎患者的心脏瓣膜组织中,提取了7种DNA分离物进行纳米孔全基因组测序,结果在所有样品中均能检测到链球菌、Q热柯克斯体和巴尔通体,而通过传统微生物学检测均为阴性。
3.2 病毒学检测
纳米孔测序通常以覆盖整个基因组的检测方式来鉴定临床病毒感染。在2015年的一项研究[15]中,早期版本的MinION(错误率约为30%)成功鉴定出了3种痘病毒株。另一项研究[16]报道了利用纳米孔测序,从血液样本中检测到基孔肯雅病毒、埃博拉病毒和丙型肝炎病毒,从样本收集到得出报告整个过程仅耗时6 h。在埃博拉疫情期间,西非使用了MinION仪器现场对病毒进行基因组测序,实现了实时监测疫情[17]。虽然埃博拉病毒变异较快,但纳米孔测序技术可以在医疗条件较差的地区帮助医疗人员鉴定病原体,监测患者对疫苗和治疗的反应。同样,在南美利用MinION测序分析了寨卡病毒的起源和传播[18],从而为研究寨卡病毒感染提供了帮助。同样,如果对流感病毒的整个基因组进行纳米孔测序,追踪新型流感毒株,也可为制备更有效的疫苗提供基础。
3.3 抗生素耐药性检测
在临床诊断中,不仅要鉴定病原体,还要快速准确判断病原微生物的耐药性,以提高抗菌治疗效果。超长读长的纳米孔测序可以识别抗性基因的存在,从中分析测定细菌的耐药型。一项研究[19]利用纳米孔测序实时分析方法,鉴定了40例肺炎克雷伯菌血液分离株的抗生素耐药型,发现纳米孔实时分析方法与传统鉴定方法的一致性为77%,重要的是,如果将这种实时方法用于临床患者治疗,则使用抗菌药物的治疗时间中位数相比传统方法缩短了约20 h。其他临床研究[20]已阐明:抗生素耐药基因在埃希氏菌、沙门氏菌、克雷伯菌和肠球菌中具有广泛的特征性;传统的测序平台(二代测序)读长较短,需要通过后期拼接组装完成完整的质粒序列测定,而纳米孔测序平台可直接对这些质粒进行测定解析。纳米孔测序产生的长序列还可以检测大肠杆菌多重耐药菌株中可移动的耐药元件。在2015年的一项研究[21]中,研究人员使用早期版本的MinION来检测肠杆菌、鲍曼不动杆菌和金黄色葡萄球菌的耐药元件,尽管存在一定的错误率,但纳米孔测序可以在整个基因组中准确发现存在的耐药元件。此外,由于纳米孔测序可以实现较长的读长,因此鉴定抗生素耐药基因相比二代测序所需的读数阈值更低。
3.4 动物疫病检测
与人类医学研究相比,纳米孔测序在动物疫病检测中的应用处于起步阶段,目前主要用于快速诊断和疫病溯源。例如,在非洲猪瘟(ASF)检测中,O'Donnell等[22]在MinION仪器上,开发了ASF病毒测序快速分析工具ASF-FAST,使用ASF-FAST可捕获足够的病毒序列,在测序10 min内完成解析192 kb基因组[23]。ASF-FAST工具展示了MinION实时测序功能的优势,并进一步扩展应用于仔猪病毒性肠道疾病的准确鉴定和诊断[24]。MinION不仅可以用于快速诊断禽霍乱病原,还可以实时鉴别LPS结构以进行准确的靶向疫苗接种,确保免疫效果[25]。牛呼吸道疾病(BRD)通常是由自身免疫力下降及暴露于多种病原共同诱导的,目前是北美和澳大利亚养殖场中牛死亡的最常见原因。仅在北美,BRD导致的产业损失超过10亿美元。McCabe等[26]利用MinION,在10 min内完成文库制备,在测序的第一个小时内产生了足够的读数,能够鉴定引起BRD的3种病毒;此外,McCabe等还发现,MinION具有在环境中快速诊断病毒的潜力。
3.5 应用中的瓶颈
虽然纳米孔测序已成功应用于病原及抗生素耐药性检测,但是必须承认,在实际应用中还存在一些瓶颈问题。问题之一是,DNA移位速度过快(约1~3 μs/碱基),这限制了纳米孔测序对单个核苷酸碱基的识别,从而影响测序精度[6]。过去几年,已研究开发出各种方法来减慢移位速度。其中:修饰方法包括用链霉亲和素修饰以固定多核苷酸[27]以及分子接头β-CD与修饰的α-HL通道通过二硫键偶联,能够成功捕获和鉴定4个单核苷酸碱基[28];物理方法包括降低电压[29]、降低溶液温度[30]、提高黏度[30]和提高盐浓度[31]。目前,将纳米孔测序和其他短读测序结合起来也是一种潜在的解决方案,可以帮助获得更高质量的基因组信息[32]。然而,通过纳米孔从DNA长链中读取单核苷酸信息仍然是一个挑战。
纳米孔测序应用面临的另一个重要问题是,尽管人们开发了许多微生物核酸提取方法,但样品制备过程仍需要优化。由于临床样品成分非常复杂,宿主中核酸污染的比例很高,导致微小的微生物种群可能被覆盖和干扰[33]。因此,需要进一步改进和优化样品收集、核酸提取、文库制备以及测序和数据处理方案,同时需要设置外部的质量控制措施。
4 疾病检测方面的优势
4.1 检测碱基修饰
天然DNA分子可能带有不同类型的碱基修饰(如甲基化)。第二代测序技术(next-generation sequencing,NGS)因在文库制备过程中使用PCR扩增,所以无法检测到修饰信息,不能直接检测天然DNA中的碱基修饰,而纳米孔测序技术可对天然DNA和RNA进行单分子测序,从而可以直接检测碱基修饰。Wescoe等[34]证明了单通道纳米孔可以区分胞嘧啶的所有5种修饰情况,包括胞嘧啶(C)、5-甲基胞嘧啶(5-mC)、5-羟基胞嘧啶、5-甲酰基胞嘧啶(5-fC)和5-羧基胞嘧啶(5-caC),准确率高达92%~98%。这种方法已被用于在较小的基因组中绘制表观遗传图谱,如结核菌株的甲基化模式,以检测疾病发展进程。纳米孔测序技术用于检测人类基因组DNA碱基修饰仍在试验中。Euskirchen等[35]研究表明,表观遗传学信息可能具有直接的临床疾病检测价值。在研究中,他们已使用碱基修饰和序列数据来区分不同类型的脑肿瘤。此外,纳米孔测序还提供了检测RNA表观修饰的平台。表观遗传修饰不仅影响DNA,还影响RNA,从而对转录和翻译产生功能性作用。
4.2 超长读长、高分辨率和实时测序
纳米孔测序的读长远远超过了NGS平台。在大肠杆菌中,1-D的读长可以超过300 kb,而2-D的读长也高达60 kb。由于超长的读长,使其比NGS具有更高的分辨率。Jain等[36]利用MinION解决了人类Xq24参考序列中50 kb的缺口。之前这个序列中的缺口是无法确定的,因为它包含一段4.8 kb的串联重复序列。最近一项研究[37],对人关节感染的超声处理样品进行NGS和纳米孔全基因组测序,结果纳米孔测序比NGS可发现更多的病原。当研究人类基因组鉴定结构变异时,尤其是检测癌症[38],存在很多拷贝数变异以及基因重复、缺失、插入、倒位和易位等情况,短读长的NGS在装配时会出现很多错误,需要百万次读取来校正,而平均读长为8 kb的纳米孔测序,只需要几百次就能可靠地检测结构变异。在RNA表达分析方面,NGS的读长较短,需要将cDNA组装成长的转录本,这对于鉴定同工型的转录本存在困难,而纳米孔测序通过测到全长的cDNA,就避免了此问题。例如,Bolisetty等[39]使用MinION来检测果蝇Dscam1基因中的RNA剪接变体。该基因是自然界中最复杂的可变剪接基因,具有18 612种可能的同工型,长度从1 806 bp到1 860 bp。该研究使用MinION检测到了7 000多个Dscam1异构体,比对一致性大于90%,而平均读长为450 bp的NGS,不能鉴定出这些同工型,表明纳米孔测序具有更高的分辨率。
目前,除了纳米孔测序,没有其他平台可以进行实时分析。对于临床或现场检测而言,相比NGS和Sanger测序,纳米孔测序具有明显的优势,可在数小时或更短的时间内获取和分析DNA或RNA序列。此外,纳米孔测序的文库制备更加简单,不受环境限制,便于携带,这进一步加快了检测时间,有助于迅速诊断疾病并及时进行针对性治疗。目前,这种优势主要用于研究,虽然还未完全应用于常规临床诊断,但未来可对每个患者样本进行测序,并以标准品为对照。这些信息将有助于更好地了解人类疾病的潜在机制,并最终帮助提供个性化的治疗方案。
4.3 强大的生物信息学支持
ONT提供了许多生物信息工具进行数据分析,以优化MinION测序结果的准确性。对于具有生物信息学专业知识的用户来说,可以下载Guppy工具包,并通过计算机上的命令行界面处理FAST5文件,将其转换为FASTQ。Guppy可以为用户提供几种用于碱基调用的算法,可以将原始读取精度提高3%以上。Guppy工具包还包括下游分析组件,如多线操作、接头的修整和对齐等。除了简单的分类之外,在Github(面向开源及私有软件项目的托管平台)上,还有很多更复杂性的工具供用户使用,包括medaka、Tombo、Pomoxis和nanopolish,它们分别用于序列校正,从原始测序数据中识别修饰的核苷酸,基因组装配和基因组装配中的错误校正[40]。
ONT的目标是能够在任何环境下,可由任何人对任何生物进行分析。为了支持该目标,ONT创建了Metrichor公司。该公司开发了图形用户界面工具,作为其EPI2ME平台的一部分,供生物信息学知识有限的用户使用。EPI2ME中的应用程序允许用户进行许多不同的分析,包括微生物样本的系统分类、抗性基因ID的注释以及与参考基因组的比对等。总体而言,ONT的这些应用程序无需通过输入命令行运行软件,而其他平台尚不提供这种功能。另一种简便的平台是One Codex。它是一个在线平台,可为任何基于微生物的样品提供分类学分析、抗性基因预测和基因组比对[41]。One Codex数据库包括61 000个细菌、48 000个病毒、1 800个真菌、1 900个古细菌和200个原生动物基因组。它提供了针对16S、5S、23S、gyrB、rpoB、18S、28S和ITS基因分析的数据库。抗生素耐药部分包括200个基因,可分析每个基因的同源性、覆盖率和读取深度。对于不想为微生物分类投入太多时间或资源的实验室而言,One Codex是一种进行快速、安全分析的最佳选择。
5 展望
目前,纳米孔测序仍面临许多挑战。与NGS相比,纳米孔测序最大的局限性是读取精度相对较低,因此在单核苷酸变异(SNV)检测中,纳米孔测序并不是最佳方案。有研究将纳米孔测序用于SNV基因分型,但仍需要较高的覆盖度。纳米孔测序尽管测序精度不如NGS,但为全基因组测序诊断传染病提供了许多解决方案,已成功用于人类和动物病原体检测、抗生素耐药基因检测和混合微生物群落鉴定。随着准确性的提高和性能的不断改进,加之在成本、测序时间和生物信息学分析方面的明显优势,纳米孔测序有望在临床实验室成为疾病检测的重要选择。

