锌指蛋白521 在肾母细胞瘤的表达及转录调控预测研究
胡师华,韦康来
肾母细胞瘤(Nephroblastoma,WT)是儿科最常见的恶性肿瘤之一,并且是最常见的肾癌类型(占所有小儿肾脏肿瘤的95%以上)。近年来,经过肿瘤研究和多模式治疗的显著进步,医学上对WT的诊治水平得到明显提升。然而,仍有相当一部分特别是患有复发性疾病的患者存活率相对较低,外科手术等治疗方式亦对小儿造成较大痛苦,许多WT 患者仍然面临与治疗相关的问题[1,2]。研究WT 相关基因和调控因子及其在肿瘤中的作用和产生影响的途径机制,依据能够识别或影响肿瘤生长的有效指标,进行推测和调控肿瘤生物学发生发展及复发情况,可以对WT 患儿进行前瞻性鉴定和更积极的治疗。而关于转录因子及其在各种病理生理过程中的作用的研究为癌症诊断和治疗增加了新途径[3]。
转录因子(TF)是一种能够在某些基因上游特异性结合的蛋白质,可以调节相应靶基因的表达,驱动一系列的细胞活动。有基础研究发现TF 在发育过程中起核心作用,决定细胞命运的转录因子的失活是致癌的重要步骤[4]。为此,我们拟从的TF 的角度研究调控机理并预测信号通路涉及的重要调控基因,探讨其在WT 中致病的机制,以支持WT 的诊断与治疗。染色质免疫共沉淀技术(ChIP)可以用于研究TF 结合位点或组蛋白特异性修饰位点,ChIP-Seq 技术是将ChIP 与第二代测序技术相结合,高效地在全基因组范围内检测与组蛋白修饰、转录因子等互作的DNA 区段,通过将测序结果精确定位到基因组上可以获取全基因组范围内与组蛋白、转录因子等互作的DNA 区段信息。
锌指蛋白521(Zinc finger protein 521,ZNF521)定位于18 号染色体(18q11.2)上,亚细胞定位分析表明ZNF521 蛋白主要在细胞核中表达。我们前期基于本院三个WT 样本测序数据做了多指标的计算生物学分析,结果显示ZNF521 在WT 中的表达明显高于非肿瘤肾组织。锌指结构由多个半胱
氨酸和/或组氨酸组成,通过锌离子来维持结构的稳定性。锌指通过与目的基因的DNA 或RNA 结合,或锌指之间的相互作用来调控基因表达,在影响细胞增殖分化凋亡、调节胚胎发育过程中发挥重要的作用[5,6]。然而,尚未有ZNF521 在WT 的文献报道。ZNF521 在WT 的作用和机制仍需探索。在本研究中,我们基于多个数据库来源的RNA-seq 数据分析ZNF521 在WT 组织中的表达水平,探讨ZNF521 可能发挥的调控作用,同时通过ChIP-Seq 数据及通路分析探讨其在WT 发展过程中可能调控的靶基因及其参与的信号通路,为进一步探索WT 诊断与治疗相关标记物提供理论参考。
1 材料与方法
1.1 肾母细胞瘤和非肿瘤肾组织之间ZNF521 的差异表达及临床意义
1.1.1 TARGET和GTEx数据库中肾母细胞瘤和非肿瘤肾组织之间ZNF521 的差异表达 基于TARGET(https://ocg.cancer.gov/programs/target)获取ZNF521在肾母细胞瘤组织(WT)和非肿瘤肾组织(NK)的TPM 表达值。同时,从GTEx(https://gtexportal.org/)数据库收集ZNF521 在正常肾组织中的TPM 表达值以增加补充对照组织。将TPM 表达值进行log2(x+0.001)标准化处理。通过计算ZNF521 在RNAseq 数据中的表达情况,绘出表达量散点图和受试者工作特征(ROC)曲线,评估WT与NK之间ZNF521的差异表达和区分肿瘤组织的能力。曲线下面积(AUC)数值越接近于1 表示诊断的准确性越高。
1.1.2 其他数据库中肾母细胞瘤和非肿瘤肾组织之间ZNF521的差异表达 通过检索GEO(http://www.ncbi.nlm.nih.gov/geo/)、Arrayexpress(https://www.ebi.ac.uk/arrayexpress/)和文献数据库收集样本数据,提取纳入研究中有关ZNF521 在WT 和正常肾组织中的基本信息,以及表达和诊断数据(N、M±SD、TP、FP、FN 和TN)。ZNF521 在每个研究中的表达情况和区分WT 和非肿瘤肾组织的能力将以小提琴图和ROC 曲线的方式展示。
为了进一步说明ZNF521 在WT 中的表达水平和针对肿瘤组织的区分能力,使用Meta 分析对TARGET 和GTEx 的RNA-seq 数据以及芯片数据进行评估。提取出每项研究中的M±SD 值,计算其标准平均差(SMD)及其95%置信区间。I2大于50%且P 值小于0.05 表明有显著的异质性,应该使用随机效应模型计算SMD,否则用固定效用模型。进行Begg"s Test 和Egger"s test 用于评价Meta分析中纳入研究的发表偏倚。如果P>0.05,则认为不存在显著的发表偏倚。最后进行诊断准确性试验Meta 来综合判断ZNF521 区分WT 和非肿瘤肾组织的能力,通过敏感度和特异度确定最佳cut-off 值,计算出每项研究中的TP、FP、FN 和TN 值,从而汇总诊断性Meta 分析,计算出汇总敏感性、特异性、阴性似然比、阳性似然比、诊断比值比,最后绘制综合受试者工作曲线(SROC),获得SROC的AUC。
1.2 肾母细胞瘤中ZNF521 的相关基因筛选及其功能注释
使用符合纳入条件的GEO 数据集以及TARGET-GTEx 的WT 数据,用Pearson 相关分析分别计算出各个数据集中和ZNF521 表达相关的基因(r>0.3,P<0.05)并取交集。为了解这些相关基因的生物学功能,利用Clusterprofiler 包对这些显著相关的基因进行基因本体富集分析(gene ontology,GO)、京都基因与基因组百科全书通路分析(Kyoto encyclopedia of genes and genomes,KEGG)和疾病富集分析(disease ontology,DO)[7,8]。利用R 软件对这些显著相关的基因进行可视化作图,包括分子功能(molecular function,MF)、细胞组成(cellular component,CC)、生物过程(biological process,BP)的GO 分析,KEGG 分析和DO 分析,P值 小于0.05被认为具有显著意义。
1.3 ZNF521的潜在靶基因预测及其功能富集分析
通过Cistrome DB 平台下载ZNF521的ChIP-Seq数据,根据使用BETA 和FastQC software2(http://www. bioinformatics. babraham.ac.uk/projects/fastqc/)预测出putative targets 和score 数值筛选出的结果,将每个实验数据的结果合并以预测ZNF521 的潜在靶基因[9,10]。使用R 软件的limma 包将纳 入 的GEO芯片数据集进行差异分析,用voom函数计算的TARGET 和GTEx 的测序样本中的差异基因(cutoff 标准:P<0.05,log2|FC| >1)[11]。结合芯片和测序数据,选择在不同数据集中出现过3 次及以上的差异基因作为WT 的差异基因。
然后将基于ChIP-Seq 数据预测的潜在靶基因、ZNF521 相关基因、芯片和测序数据筛选出的WT 差异基因这三个部分取交集,交集部分作为ZNF521的最佳潜在靶基因。最后,为了系统了解疾病分子机制,使用STRING v11 工具[12]对ZNF521 最佳潜在靶基因进行功能注释,构建蛋白互作网络(PPI)。根据GO 条目分析选择可能涉及WT 相关生物过程的基因,基于GTEx-TARGET 数据使用离差平方和法对各基因表达值合并,进行相关性分析,并通过UCSC(http://genome.ucsc.edu)观察最佳潜在靶基因的转录起始位点附近是否有ZNF521的结合信号峰。
1.4 统计分析
文章中的统计分析由SPSS 21.0(SPSS Inc.,Chicago,IL,USA),R 版 本4.0.3(https://www.rproject.org/)、GraphPad Prism 7.0,Stata 14.0(http://www.stata.com)和MetaDiSc 1.4 完成对实验数据进行统计分析和绘图。分析ZNF521 在WT 和NK 组之间的表达水平,两个连续变量之间的比较先进行正态性检验Kolmogorov-Smirnov,如两组数据正态分布(α>0.10),进行方差齐性检验,方差齐(P>0.10)进行两样本t检验,方差不齐则用近似t检验;如任何一组数据呈偏态,使用Mann-WhitneyU检验。结果以平均值±标准差表示。统计显著性定义为P值<0.05。
2 结 果
2.1 肾母细胞瘤和非肿瘤肾组织之间ZNF521 的差异表达和临床意义
2.1.1 TARGET 和GTEx 数据库中ZNF521 在肾母细胞瘤和非肿瘤肾组织之间的差异表达分析 从TARGET 数据库下载ZNF521 在126 例WT 细胞瘤组织(WT)和6 例非肿瘤肾组织(NK)的TPM 表达值,从GTEx 数据下载ZNF521 在45 例正常肾组织中的TPM 表达值。由于数据呈偏态,选择Mann Whitney test。结果显示在TARGET 和GTEx 数据中,ZNF521 在WT 呈显著高表达(NKvsWT:1.167±0.106vs6.526±0.095,P<0.001,图1.A),诊断WT 的受试者工作特征曲线下面积(AUC)为1.000(95%CI:1.000~1.000,P<0.001,图1.D)。
2.1.2 综合多个数据库来源的肾母细胞瘤和非肿瘤肾组织之间ZNF521 的表达分析及临床意义 收集到4 个WT 芯片数据集:GSE73209、GSE11024、GSE4530、GSE2712,其中有2 个包含ZNF521 数据的WT 相关基因芯片被纳入:GSE73209(6NKvs32WT)和GSE11024(12NKvs27WT)。芯片数据中ZNF521 的表达分析结果显示ZNF521 在WT 中呈显著高表达。在GSE73209 数据集中,NKvsWT 为7.699±0.208vs8.913±0.137(P<0.001,图1.B),AUC为0.932(95%CI:0.850-1.000,P<0.001,图1.E);在GSE11024 数 据 集 中,NKvsWT 为6.147±0.055vs9.865±0.281(P<0.001,图1. C),AUC 为0.929(95%CI:0.834~1.000,P<0.001,图1.F)。2 个芯片与GTEx-TARGET 数据合计共3 项研究,包含了185例WT 组织和69 例非肿瘤肾组织。基于这3 项研究,我们利用Meta 分析验证ZNF521 在WT 组织中的表达。异质性分析的结果显示I2=95.7%,P<0.001,提示存在较大异质性,因此选用随机效应模型进行。分析结果显示ZNF521 在WT 组织中呈显著高表达(SMD=3.385,95%CI:1.024~5.747,P=0.005,图2.A)。漏斗图分析显示纳入研究不存在显著的发表偏倚,Begg 检验(P=0.602)和Egger 检验(P=0.477)结果均无显著统计学差异。通过敏感度和特异度计算,GTEX-TARGET 数据的最佳截断值为3.250,GSE73209 数 据 的 最 佳 截 断 值 为8.479,GSE11024 数据的最佳截断值为7.357,诊断准确性实验meta 分析结果显示:汇总灵敏度和特异度分别为0.940 和1.000;汇总阳性似然比和阴性似然比分别为28.690 和0.060;汇总诊断比值比为508.86(图2.B-F),使用四格表数据拟合计算得到SROC 的AUC 为0.990(95%CI:0.970~0.990,表1)。

表1 诊断准确性实验meta 分析使用的四格表资料
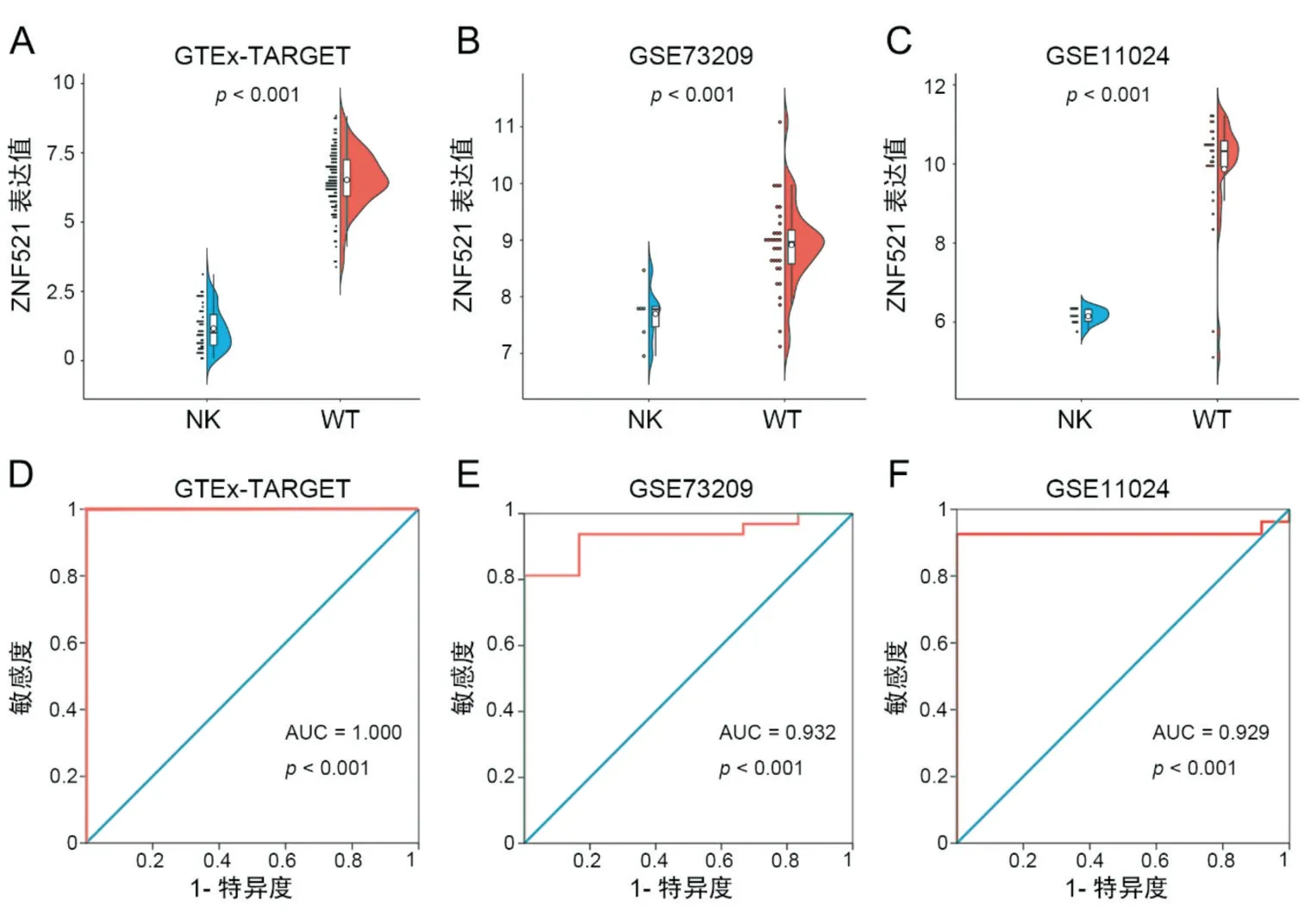
图1 ZNF521 的表达情况及诊断效能评价 A-C:小提琴图展示ZNF521 在肾母细胞瘤(WT)的表达显著高于正常组织(NK)。D-F:受试者工作特征曲线(ROC)显示ZNF521 诊断肾母细胞瘤的ROC 曲线下面积(AUC)有统计学意义
2.2 肾母细胞瘤中ZNF521 的相关基因筛选及其功能注释
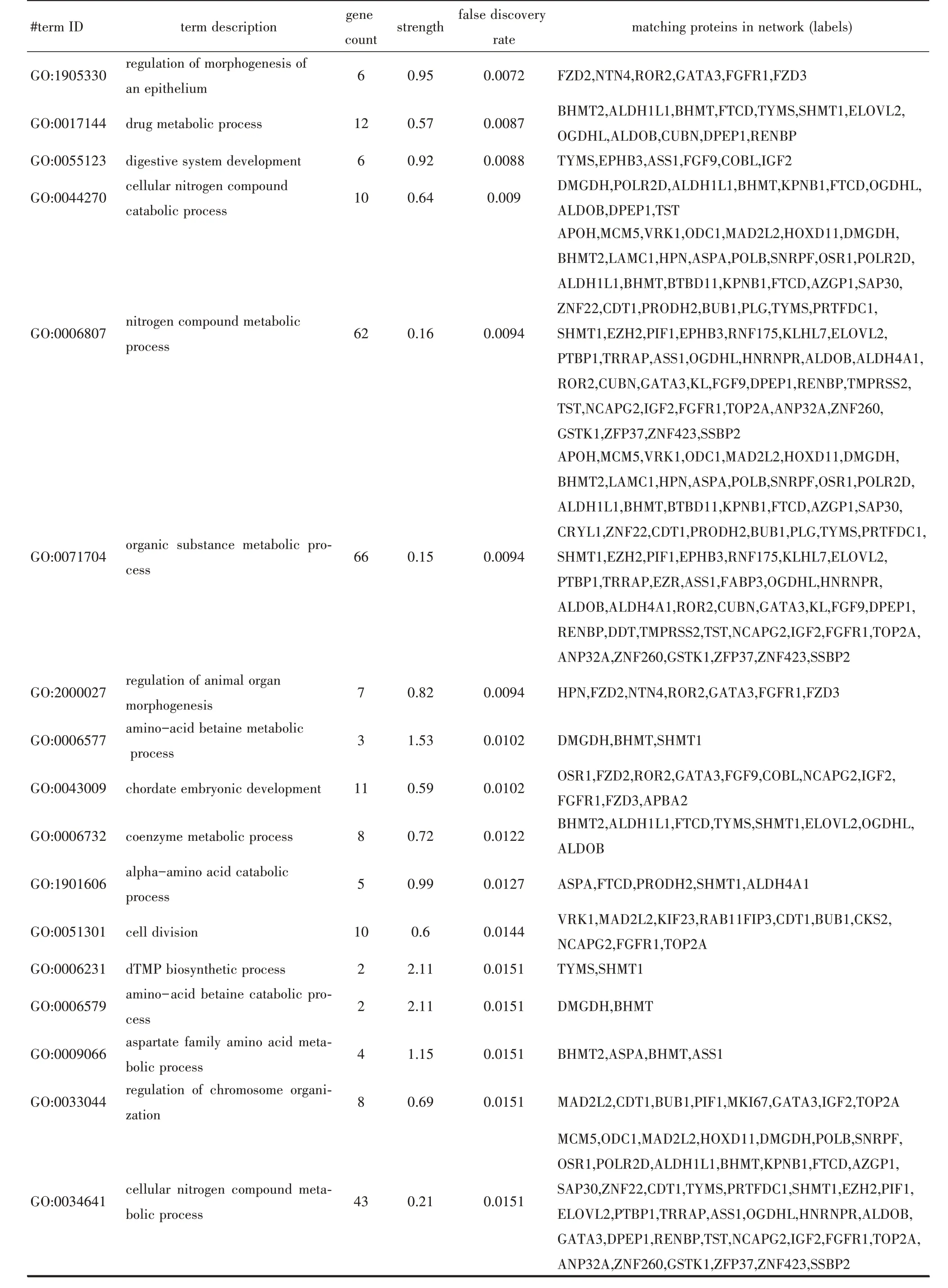
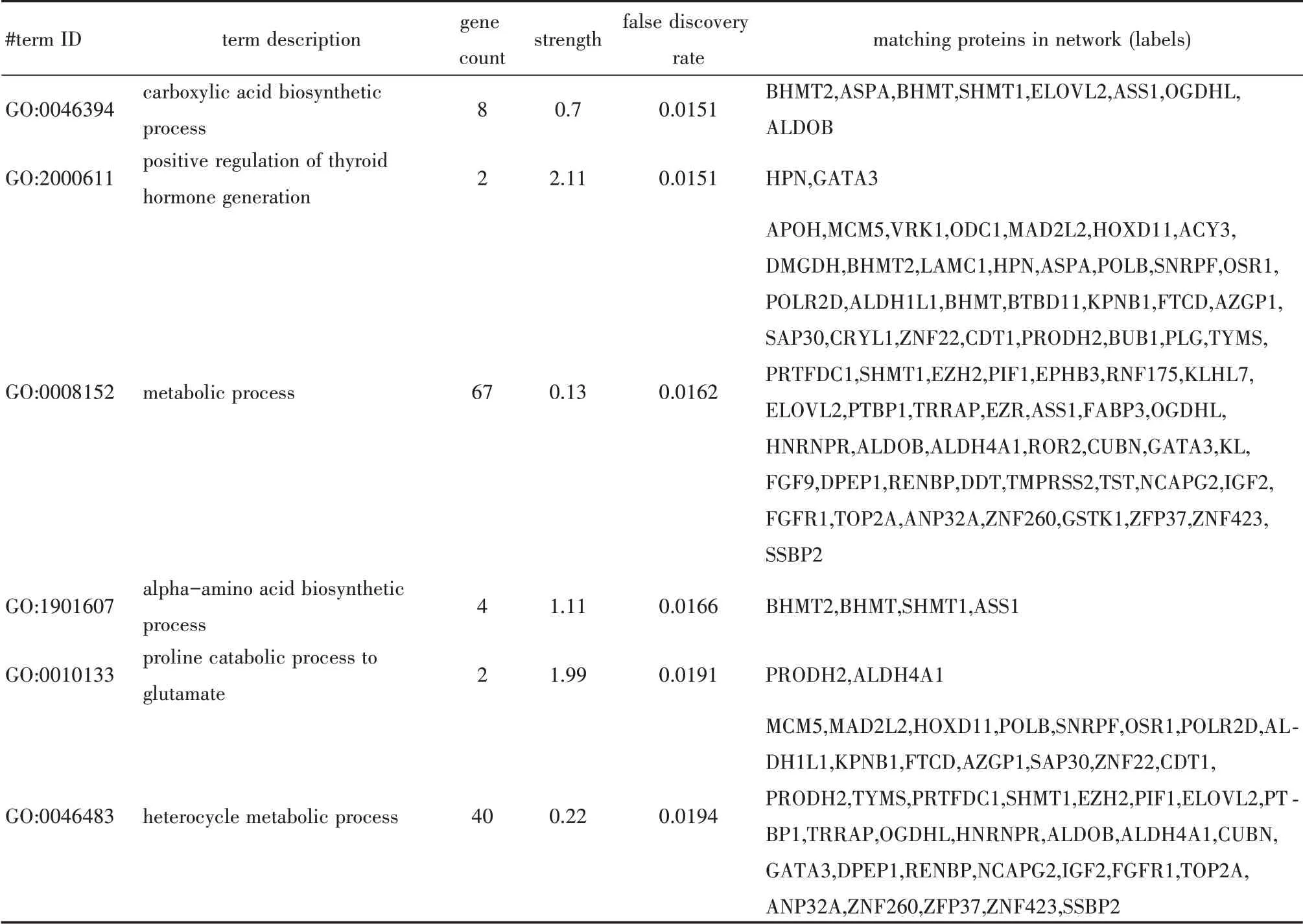
我们对GTEx-TARGET、GSE73209、GSE11024三个数据集进行相关性分析,分别得到ZNF521 相关基因38245 个、7201 个、11692 个。最终交集得到3359 个作为ZNF521 最有可能的相关基因,分别进行GO 注释,KEGG 通路分析和DO 分析。GO富集分析显示,显著富集的生物过程包括DNA 复制、肾脏发育和有丝核分裂。分子功能分析中,基因与“辅酶结合”、“DNA 依赖性ATP 酶活性”、“微管蛋白结合”最为相关。根据细胞定位分析,相关基因最显著富集于染色体、着丝粒区域和顶端细胞(图3.A-C)。KEGG 富集分析显示ZNF521 相关基因主要与细胞周期、DNA 复制和脂肪酸降解相关(图3.D)。DO 分析显示对泌尿系统癌症、胃肠系统癌症、常染色体遗传病显著相关(图3.E)。
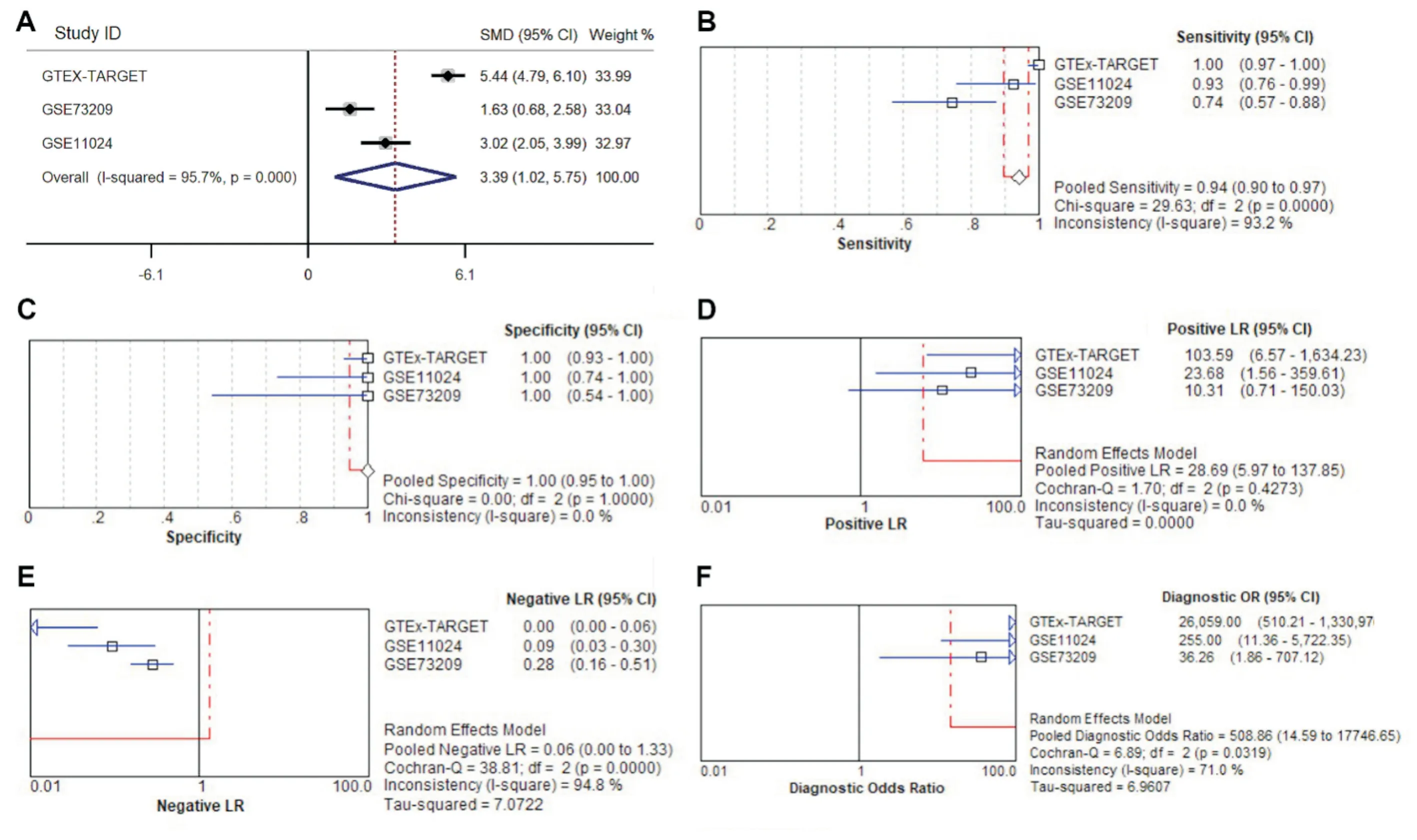
图2 综合数据库来源肾母细胞瘤和正常组织ZNF521 表达水平的森林图 A. Meta 分析评估TARGET-GTEx 以及GEO 芯片数据的森林图。诊断准确性试验分析森林图:B. 敏感性分析;C. 特异性分析;D. 阴性似然比;E. 阳性似然比;F. 诊断比值比
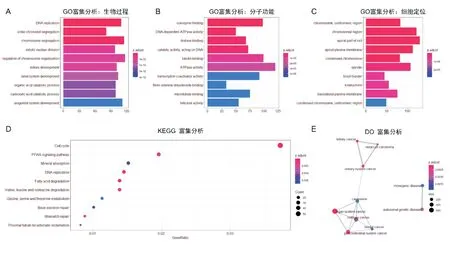
图3 ZNF521 表达显著相关基因的富集分析结果 A. 基因本体富集分析的生物过程;B. 基因本体富集分析的分子功能;C. 基因本体富集分析的细胞定位;D. 京都基因与基因组百科全书通路富集分析通路分析;E. 疾病富集分析结果
2.3 ZNF521 的最佳潜在靶基因预测及其功能富集分析
从Cistrome DB 网站检索获取到2 份ZNF521 相关的ChIP-seq 数据,依据score>0.01,筛选得到2411个ZNF521 潜在靶基因。同时,运算GTEx-TARGET、GSE73209、GSE11024、GSE4530、GSE2712 数据集来筛选差异基因。将各个数据集进行交集处理,筛选出在不同数据集出现3 次以上的差异基因,共得到1346 个作为WT 差异基因,其中393 个上调基因和953 个下调基因。将ZNF521 潜在靶基因、ZNF521 相关基因与WT 差异表达基因三部分取交集,最终得到101 个最佳潜在靶基因。通过构建PPI(P<0.001),发现网络中的FGFR1 涉及泌尿生殖系统发育、胚胎发育调控等多个生物过程,且与ZNF521 表达呈强相关,FGFR1 转录起始位点附近有ZNF521 的ChIP-Seq 数据的结合信号(图4.A-C,表2)。
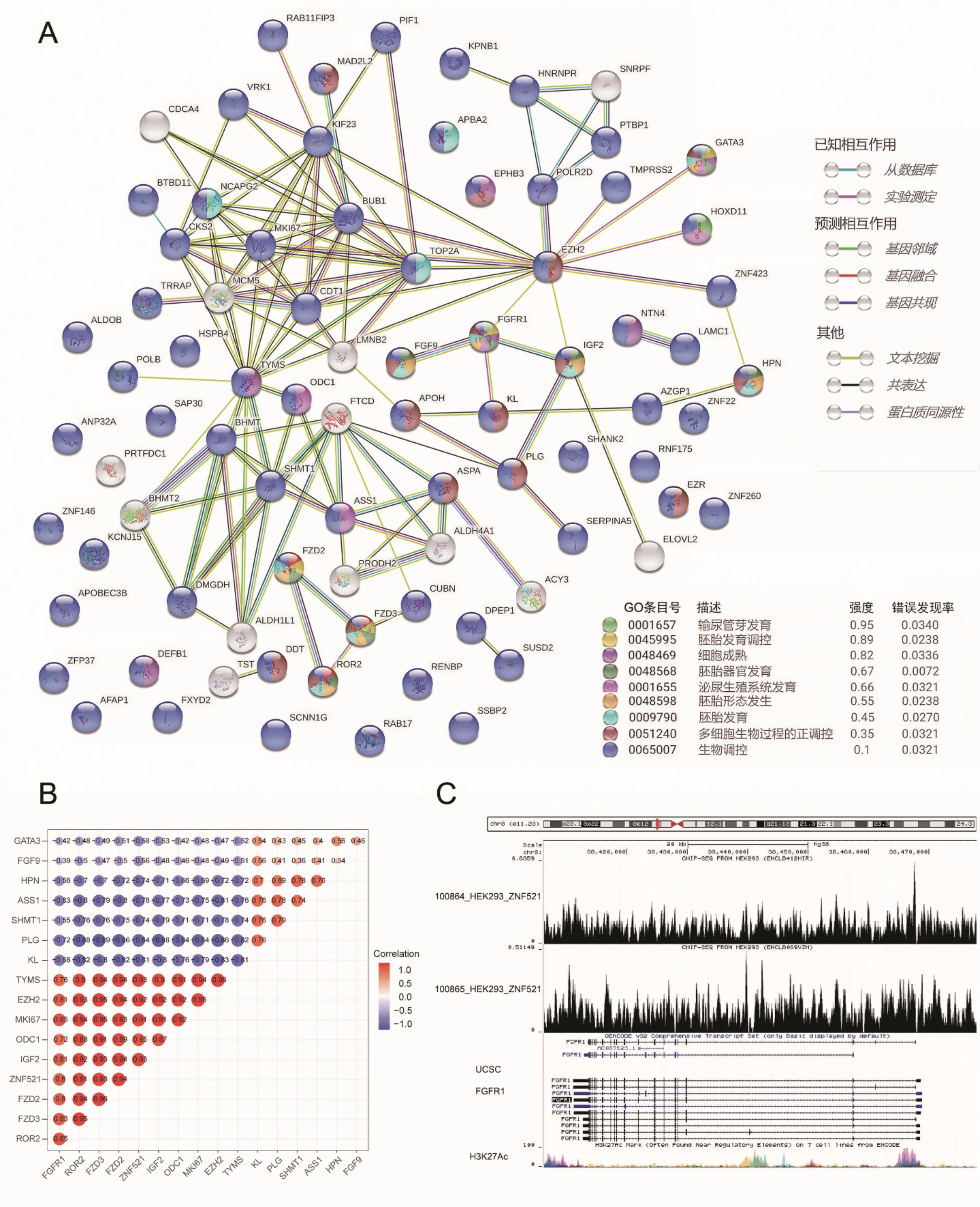
图4 A.构建ZNF521 最佳预测靶基因的蛋白质互作网络;B. ZNF521 与部分最佳预测靶基因的相关性热图;C. 通过UCSC浏览ChIP-Seq 数据中ZNF521 与最佳潜在靶基因FGFR1 的靶向结合位点
#term ID term description gene count strength false discovery rate matching proteins in network (labels)GO:1905330 6 0.95 0.0072 GO:0017144 GO:0055123 GO:0044270 regulation of morphogenesis of an epithelium drug metabolic process digestive system development cellular nitrogen compound catabolic process 12 6 10 0.57 0.92 0.64 0.0087 0.0088 0.009 GO:0006807 nitrogen compound metabolic process 62 0.16 0.0094 GO:0071704 organic substance metabolic process 66 0.15 0.0094 GO:2000027 0.82 0.0094 FZD2,NTN4,ROR2,GATA3,FGFR1,FZD3 BHMT2,ALDH1L1,BHMT,FTCD,TYMS,SHMT1,ELOVL2,OGDHL,ALDOB,CUBN,DPEP1,RENBP TYMS,EPHB3,ASS1,FGF9,COBL,IGF2 DMGDH,POLR2D,ALDH1L1,BHMT,KPNB1,FTCD,OGDHL,ALDOB,DPEP1,TST APOH,MCM5,VRK1,ODC1,MAD2L2,HOXD11,DMGDH,BHMT2,LAMC1,HPN,ASPA,POLB,SNRPF,OSR1,POLR2D,ALDH1L1,BHMT,BTBD11,KPNB1,FTCD,AZGP1,SAP30,ZNF22,CDT1,PRODH2,BUB1,PLG,TYMS,PRTFDC1,SHMT1,EZH2,PIF1,EPHB3,RNF175,KLHL7,ELOVL2,PTBP1,TRRAP,ASS1,OGDHL,HNRNPR,ALDOB,ALDH4A1,ROR2,CUBN,GATA3,KL,FGF9,DPEP1,RENBP,TMPRSS2,TST,NCAPG2,IGF2,FGFR1,TOP2A,ANP32A,ZNF260,GSTK1,ZFP37,ZNF423,SSBP2 APOH,MCM5,VRK1,ODC1,MAD2L2,HOXD11,DMGDH,BHMT2,LAMC1,HPN,ASPA,POLB,SNRPF,OSR1,POLR2D,ALDH1L1,BHMT,BTBD11,KPNB1,FTCD,AZGP1,SAP30,CRYL1,ZNF22,CDT1,PRODH2,BUB1,PLG,TYMS,PRTFDC1,SHMT1,EZH2,PIF1,EPHB3,RNF175,KLHL7,ELOVL2,PTBP1,TRRAP,EZR,ASS1,FABP3,OGDHL,HNRNPR,ALDOB,ALDH4A1,ROR2,CUBN,GATA3,KL,FGF9,DPEP1,RENBP,DDT,TMPRSS2,TST,NCAPG2,IGF2,FGFR1,TOP2A,ANP32A,ZNF260,GSTK1,ZFP37,ZNF423,SSBP2 HPN,FZD2,NTN4,ROR2,GATA3,FGFR1,FZD3 GO:0006577 1.53 0.0102 GO:0043009 regulation of animal organ morphogenesis amino-acid betaine metabolic process chordate embryonic development 7 3 1 0.59 0.0102 GO:0006732 0.72 0.0122 GO:1901606 0.99 0.0127 GO:0051301 GO:0006231 GO:0006579 1 8 5 1 0.6 2.11 2.11 0.0144 0.0151 0.0151 DMGDH,BHMT,SHMT1 OSR1,FZD2,ROR2,GATA3,FGF9,COBL,NCAPG2,IGF2,FGFR1,FZD3,APBA2 BHMT2,ALDH1L1,FTCD,TYMS,SHMT1,ELOVL2,OGDHL,ALDOB ASPA,FTCD,PRODH2,SHMT1,ALDH4A1 VRK1,MAD2L2,KIF23,RAB11FIP3,CDT1,BUB1,CKS2,NCAPG2,FGFR1,TOP2A TYMS,SHMT1 DMGDH,BHMT GO:0009066 1.15 0.0151 BHMT2,ASPA,BHMT,ASS1 GO:0033044 coenzyme metabolic process alpha-amino acid catabolic process cell division dTMP biosynthetic process amino-acid betaine catabolic process aspartate family amino acid metabolic process regulation of chromosome organization 0.69 0.0151 GO:0034641 cellular nitrogen compound metabolic process 0 2 2 4 8 43 0.21 0.0151 MAD2L2,CDT1,BUB1,PIF1,MKI67,GATA3,IGF2,TOP2A MCM5,ODC1,MAD2L2,HOXD11,DMGDH,POLB,SNRPF,OSR1,POLR2D,ALDH1L1,BHMT,KPNB1,FTCD,AZGP1,SAP30,ZNF22,CDT1,TYMS,PRTFDC1,SHMT1,EZH2,PIF1,ELOVL2,PTBP1,TRRAP,ASS1,OGDHL,HNRNPR,ALDOB,GATA3,DPEP1,RENBP,TST,NCAPG2,IGF2,FGFR1,TOP2A,ANP32A,ZNF260,GSTK1,ZFP37,ZNF423,SSBP2
GO:0046394 GO:2000611 GO:0008152 GO:1901607 GO:0010133 GO:0046483 carboxylic acid biosynthetic process positive regulation of thyroid hormone generation metabolic process alpha-amino acid biosynthetic process proline catabolic process to glutamate heterocycle metabolic process 8 2 67 4 2 40 0.7 2.11 0.13 1.11 1.99 0.22 0.0151 0.0151 0.0162 0.0166 0.0191 0.0194 BHMT2,ASPA,BHMT,SHMT1,ELOVL2,ASS1,OGDHL,ALDOB HPN,GATA3 APOH,MCM5,VRK1,ODC1,MAD2L2,HOXD11,ACY3,DMGDH,BHMT2,LAMC1,HPN,ASPA,POLB,SNRPF,OSR1,POLR2D,ALDH1L1,BHMT,BTBD11,KPNB1,FTCD,AZGP1,SAP30,CRYL1,ZNF22,CDT1,PRODH2,BUB1,PLG,TYMS,PRTFDC1,SHMT1,EZH2,PIF1,EPHB3,RNF175,KLHL7,ELOVL2,PTBP1,TRRAP,EZR,ASS1,FABP3,OGDHL,HNRNPR,ALDOB,ALDH4A1,ROR2,CUBN,GATA3,KL,FGF9,DPEP1,RENBP,DDT,TMPRSS2,TST,NCAPG2,IGF2,FGFR1,TOP2A,ANP32A,ZNF260,GSTK1,ZFP37,ZNF423,SSBP2 BHMT2,BHMT,SHMT1,ASS1 PRODH2,ALDH4A1 MCM5,MAD2L2,HOXD11,POLB,SNRPF,OSR1,POLR2D,ALDH1L1,KPNB1,FTCD,AZGP1,SAP30,ZNF22,CDT1,PRODH2,TYMS,PRTFDC1,SHMT1,EZH2,PIF1,ELOVL2,PTBP1,TRRAP,OGDHL,HNRNPR,ALDOB,ALDH4A1,CUBN,GATA3,DPEP1,RENBP,NCAPG2,IGF2,FGFR1,TOP2A,ANP32A,ZNF260,ZFP37,ZNF423,SSBP2#term ID term description gene count strength false discovery rate matching proteins in network (labels)
3 讨 论
目前,WT 的发病机制尚不明确,现有研究认为基因组不稳定是引发WT 的重要因素,其中伴有许多癌症相关基因的异常表达。这些基因在癌症中异常表达的原因包括TF 失调。转录因子结合的区域可以是基因的启动子,增强子或者其他的调控区域。如果结合后RNA 聚合酶的结合增强导致该基因的表达增多,反之则阻碍该基因的表达。转录因子调控大量下游靶基因,影响细胞增殖、分化、凋亡和细胞周期等生理活动。
目前已发表的ZNF521 与癌症相关的文献中,有研究证明ZNF521 在肝细胞癌组织中下调,并通过miR-802调控的Runx2转录抑制和AKT失活抑制肝细胞癌的生长,提示其对肝细胞癌的潜在治疗价值。ZNF521 能通过调节miR-204-5p,抑制胃癌细胞凋亡,促进胃癌细胞增殖、迁移和侵袭[13]。此外,ZNF521 有助于髓母细胞瘤细胞的克隆生长、迁移和致瘤性[14],亦可与GLI1 和GLI2 相互作用,增强髓母细胞瘤中Sonic Hedgehog 通路的活性[15]。
我们利用TARGET、GTEx 和GEO 的数据集整合分析了ZNF521 在WT 的表达水平。通过筛选相关基因和预测靶基因,进行功能富集分析,提供对ZNF521 基因表达模式的了解,以探索优化的诊断治疗WT的靶标。首先,通过针对WT芯片及测序数据,我们发现在WT 患者的ZNF521 在RNA 水平上均显著上调。然后我们使用这些芯片和测序数据计算出ZNF521 相关基因,其相关基因的高分通路是细胞周期和DNA 复制通路,并且与染色体调控、肾脏系统发育等生物过程显著相关。综合ZNF521潜在靶基因和WT 差异基因,最后得到ZNF521 的最佳潜在靶基因,如FGFR1、IGF2、ODC1、MKI67、EZH2 等均与ZNF521 呈现相同的上调趋势。其中,我们发现FGFR1 涉及细胞代谢、细胞分裂、胚胎发育,并且在PPI 中与存在相似功能的IGF2、EZH2 存在潜在互作关系。
由于现有研究中ZNF521 的ChIP-seq 数据只有两份能进行可视化,并且WT 的研究相对较少,还需要加大样本量来降低数据的异质性。后续我们将联合ZNF521 及其预测靶基因的表达,收集临床信息如实验室和影像学检查的诊断资料、无病生存率等预后数据进行综合分析,可以为WT 的筛查、早期诊断及预后评价提供更有价值的信息。
综上所述,我们通过整合分析发现转录因子ZNF521 及相关基因与WT 密切相关,并探讨了它影响WT 发生的可能机制。ZNF521 的相关基因和靶基因的功能研究为探索WT 的相关分子机制和基因治疗提供理论依据。

