乳腺癌变过程中Pim1和Notch1差异性表达的临床价值
熊世双 ,陈晓文,穆亚男,张德瑞,蒋金芳,李军,曹玉文
0 引言
Pim1分子是丝/苏氨酸激酶家族重要成员,与多种肿瘤进展关系密切[1-4],Pim1在乳腺癌中异常表达,其高表达与乳腺癌TNM分期、淋巴结转移、ER表达密切相关[5]。Pim1调控基因转录激活,Notch1分子被确定为Pim1下游目标[6]。乳腺癌和前列腺癌细胞Pim1激酶促进Notch1磷酸化,增强Notch1核定位和转录活性[7]。目前,Pim1和Notch1在乳腺导管上皮细胞癌变过程中的表达状况以及与乳腺癌预后的关系研究甚少,因此,我们应用qRTPCR、免疫组织化学及数据库检测Pim1和Notch1在乳腺导管上皮细胞癌变过程中的表达,分析与乳腺癌临床病理参数和预后的关系,以期为乳腺癌早期诊断及临床治疗提供一定的依据。
1 资料与方法
1.1 资料
收集2000年1月—2010年9月石河子大学医学院第一附属医院病理科石蜡包埋组织乳腺癌旁正常组织(ANBT,距乳腺癌组织至少5 cm以上)37例、乳腺普通型导管增生(UDH)40例、乳腺导管原位癌(DCIS)32例和乳腺浸润性导管癌(IDC)200例。所有患者术后均经病理确诊为浸润性乳腺癌,排除术前放化疗或内分泌治疗和合并有其他严重疾病患者。同时收集患者临床病理资料,包括组织分级、TNM分期、淋巴结转移及ER、PR、HER2表达。所有患者签署知情同意书,石河子大学第一附属医院伦理委员会审批。
1.2 方法
1.2.1 引物设计与合成 使用Oligo7.0设计引物,引物序列:Pim1:上游引物:5′-GAGAAGGACCGGATTTCCGAC-3′,下游引物:5′-CAGTCCAGGAGCCTAATGACG-3′;Notch1:上游引物:5′-TGGACCAGATTGGGGAGTTC-3′,下游引物:5′-GCACACTCGTCTGTGTTAC-3′;β-actin:上游引物:5′-CTCCATCCTGGCCTCGCTGT-3′,下游引物:5′-GCTGTCACCTTCACCGTTCC-3′。由上海生工生物工程公司合成引物。
1.2.2 qRT-PCR检测Pim1和Notch1 mRNA的表达 乳腺组织中总RNA用RNeasy FFPE试剂盒(目录号73504,QIAGEN,德国)提取,根据Reverse Transcription Kit,加入反转录酶Superscript Ⅲ,反转录出cDNA,在带有Fast SYBR Green Master Mix的7500 Fast Real-Time PCR系统(Applied Biosystems,Carlsbad,CA,美国)上进行qRT-PCR。qRT-PCR反应条件即50℃温育2 min,95℃预变性5 min,95℃变性10 s,62℃退火延伸30 s,40个循环。以人β-actin mRNA的表达进行归一化,即ΔCt=CtPim1/Notch1-Ctβ-actin。
1.2.3 免疫组织化学Envision二步法检测Pim1和Notch1蛋白表达 蜡块连续3 μm厚切片,脱蜡、水化、柠檬酸盐高压抗原修复,加一抗(Pim1抗体,Abcam,1:100;或Notchl抗体,SIGMA,1:300),4℃冰箱孵育过夜,加二抗、DAB显色、苏木精对比染色、中性树脂封片。显微镜下观察,两位资深病理医师双盲法判读结果,阳性结果根据阳性细胞百分比和染色强度分值相乘,得分范围0~12分,中位值4为分界值,≥4分为高表达,<4分为低表达。
1.2.4 Oncomine在线数据库(www.oncomine.org)[8]分析Pim1和Notch1 mRNA表达 数据库中设置限定条件:(1)基因:Pim1/Notch1;(2)肿瘤类型:乳腺癌;(3)分析类型:癌和正常组织;(4)数据类型:mRNA;(5)临界值设定(P<0.05;倍数变化=所有;基因排名=前10)。采用箱状图展示结果。
1.2.5 GEPIA在线数据库(http://gepia.cancer-pku.cn/) 分析Pim1和Notch1蛋白在乳腺组织中的表达,结果以箱式图表示。
1.2.6 UALCAN在线工具(http://ualcan.path.uab.edu) 分析Pim1和Notch1蛋白与乳腺癌患者临床病理参数的关系[9]。
1.2.7 Kaplan-Meier Plotter数据库(http://kmPlot.com/analysis/) 对Pim1和Notch1表达与乳腺癌预后关系进行生存分析[10]。数据库中设置条件:(1)肿瘤:乳腺癌;(2)基因:Pim1/Notch1;(3)生存期:总生存期(OS)/无复发生存期(RFS)。
1.3 统计学方法
用SPSS22.0软件进行统计学分析。Pim1和Notch1 mRNA水平以及蛋白的表达用均数±标准差描述。组间差异比较用非参秩和(两组间用Mann-Whitney U检验、三组及以上用Kruskal-Wallis H)检验,两变量相关性分析用Pearson秩相关,构成比及其他资料差异的分析用卡方检验,P<0.05为差异有统计学意义。
2 结果
2.1 Pim1和Notch1在乳腺癌变过程中的表达
qRT-PCR结果显示Pim1 mRNA在乳腺癌旁正常组织、普通型导管增生、导管原位癌、浸润性导管癌组织中的表达水平无明显变化(P=1.105),见图1A,四组中两两比较差异无统计学意义(P>0.05),见图1B,Oncomine数据库发现Pim1 mRNA在乳腺癌中表达略高于正常组织,差异无统计学意义(P=0.063),见图1C。Notch1 mRNA在从正常到浸润癌组织中的表达逐渐升高,差异有统计学意义(P=0.000),见图1D,各组中两两比较,除ANBT和UDH组外,其余各组差异有统计学意义(P<0.05),见图1E,数据库显示Notch1 mRNA在乳腺癌中水平也高于正常乳腺组织,差异无统计学意义(P=0.213),见图1F。
Pim1蛋白免疫组织化学结果显示其定位于乳腺上皮细胞核,见图2A,从正常到浸润癌四组中的阳性表达逐渐降低,见图2B,组间两两比较,Pim1蛋白在ANBT中表达明显高于DCIS(P=0.008)和IDC(P=0.019)组,在UDH中表达亦明显高于DCIS(P=0.005)和IDC(P=0.004)组,见图2C,GEPIA数据库发现乳腺癌中Pim1蛋白水平显著低于正常组织,见图2D。Notch1蛋白定位于乳腺上皮细胞质,见图2A,从正常乳腺到浸润癌组中表达逐渐升高,见图2E,各组中进行两两比较,除DCIS和IDC组(P=0.001)外,其余各组间差异有统计学意义,见图2F,GEPIA数据库显示正常组织Notch1蛋白水平高于乳腺癌组织,差异无统计学意义(P>0.05),见图2G。Pim1和Notch1蛋白在乳腺癌中表达相关性分析,显示二者呈显著负相关(r=-0.385,P=0.001),见表1。
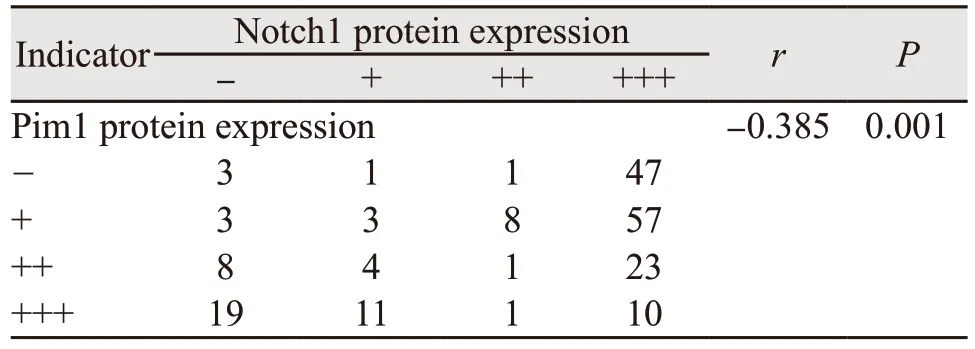
表1 Pim1和Notch1蛋白表达相关性Table 1 Association between Pim1 and Notch1 of protein expression
2.2 Pim1和Notch1表达与乳腺癌患者临床病理参数的相关性
Pim1 mRNA表达与乳腺癌患者淋巴结转移、TNM晚期显著相关。Notch1 mRNA与组织学3级和淋巴结转移明显相关,Pim1蛋白低表达与乳腺癌高分级、淋巴结转移、ER阴性、PR阳性和HER2阴性显著相关。Notch1蛋白高表达与淋巴结转移明显相关,见表2。UALCAN数据库发现Pim1 mRNA在淋巴结转移、TNM晚期、ER与PR和HER2阳性的表达显著降低,见图3A~C。Notch1 mRNA与淋巴结转移、TNM晚期、ER与PR和HER阴性显著相关,见图3D~F。

图1 乳腺组织Pim1和Notch1 mRNA表达Figure 1 Pim1 and Notch1 mRNA expression in breast tissues
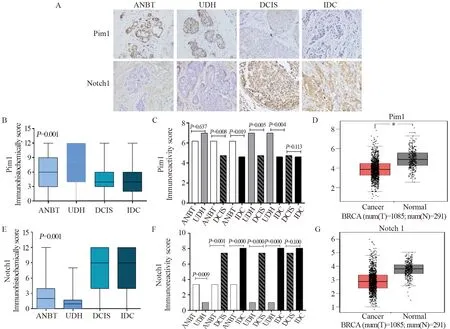
图2 乳腺组织Pim1和Notch1蛋白表达Figure 2 Pim1 and Notch1 protein expression in breast tissues
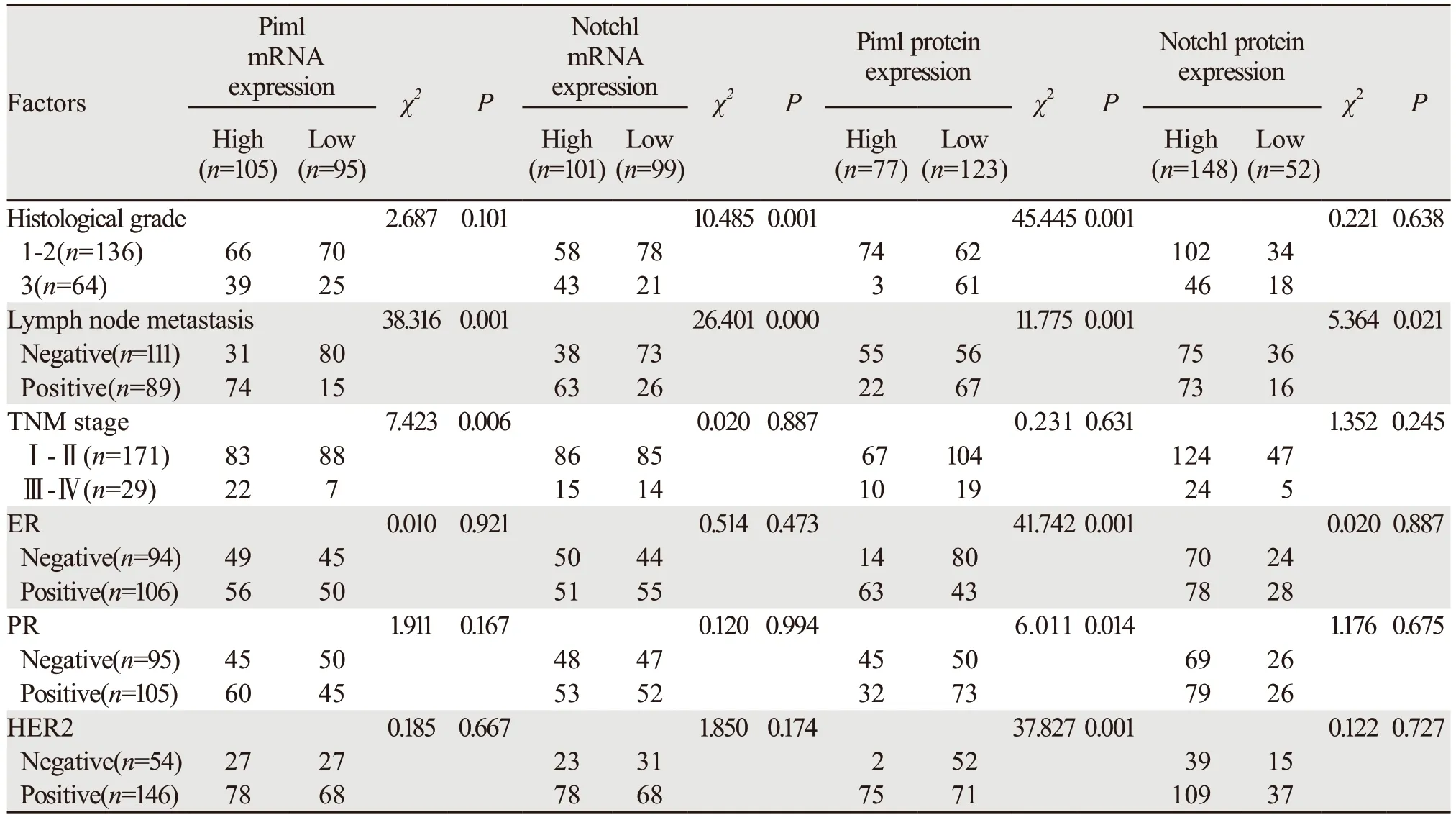
表2 Pim1和Notch1中蛋白和mRNA与乳腺癌患者临床病理参数的相关性Table 2 Correlation of Pim1/Notch1 protein and mRNA with clinicopathological features of breast cancer patients
2.3 Pim1和Notch1表达与乳腺癌患者预后的关系
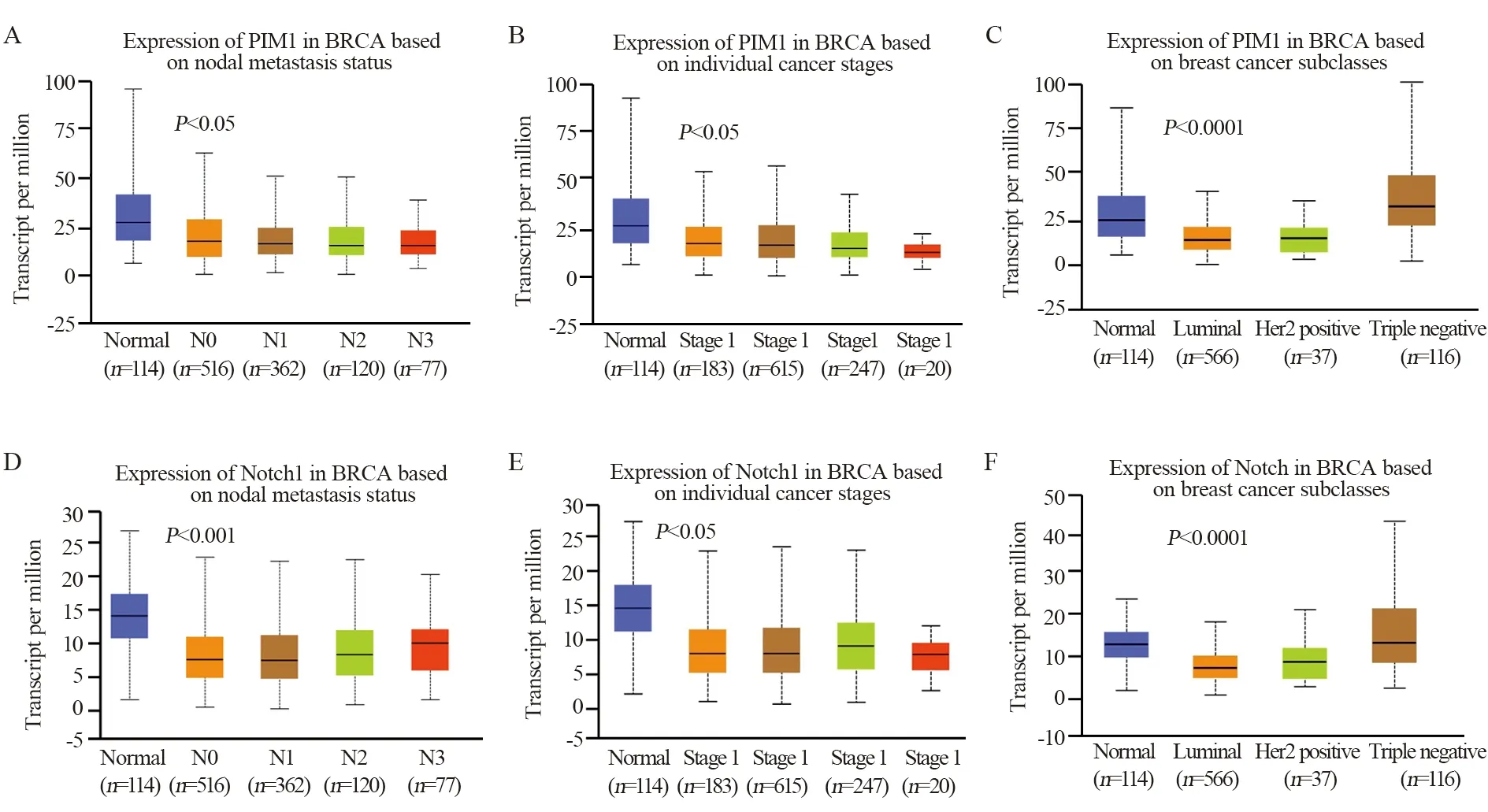
图3 UALCAN数据库中Pim1、Notch1 mRNA表达与乳腺癌临床病理参数关系Figure 3 Relation between Pim1,Notch1 mRNA expression and clinicopathological parameters of breast cancer patients from UALCAN database
Kaplan-Meier Plotter对Pim1 mRNA与预后数据进行挖掘,结果显示,209193_at基因芯片中,Pim1低表达的乳腺癌患者总生存期及无复发生存期明显缩短(P=0.000),见图4A~B。218902_s_at基因芯片中,Notch1高表达患者总生存时间显著减少(P=0.025),见图4C,无复发生存期缩短,见图4D。
3 讨论
有文献报道Pim1 mRNA在乳腺癌等多种恶性肿瘤中表达升高[7,11-13],也有研究发现Pim1在非小细胞肺癌中下调[14],说明Pim1 mRNA在不同肿瘤中表达水平不一致,可能受多种因素的影响。Pim1蛋白在乳腺癌变过程中的表达逐渐降低,尤其在浸润性乳腺癌中显著低表达,GEPIA数据库也验证了该结果,提示Pim1蛋白在乳腺导管上皮细胞癌变过程中可能充当抑癌基因角色,Pim1蛋白表达对乳腺组织来说是一个保护因素,也有文献报道Pim1蛋白促进乳腺细胞癌变和增殖[15-16]。Pim1表达与乳腺癌患者临床病理特征的相关性分析显示,Pim1 mRNA表达与淋巴结转移、TNM晚期显著相关,提示Pim1 mRNA表达可能促进乳腺癌恶性进展及转移,有学者报道Pim1参与了三阴性乳腺癌的细胞增殖、迁移和凋亡,沉默Pim1基因可以促进肿瘤细胞凋亡并增强对化疗的敏感度[17],说明Pim1作为癌基因参与乳腺肿瘤的进展。
UALCAN数据库显示,Pim1 mRNA在TNM晚期和淋巴结转移的患者中显著降低,与本研究结果相符,提示Pim1可能承担着减缓乳腺癌恶性程度的角色。以上研究结果出现不一致,可能与乳腺癌样本量、分子分型、检测方法及敏感度等有关,提示需要进一步扩大样本量、分层细化研究对象、标准化检测方法等。我们进一步使用Kaplan-Meier Plotter数据库显示Pim1蛋白低水平的乳腺癌患者总生存期和无复发生存期明显降低,提示Pim1分子是乳腺癌预后的一个保护性因素。本研究结果及数据库分析提示,Pim1分子与乳腺癌变、进展和预后密切相关。
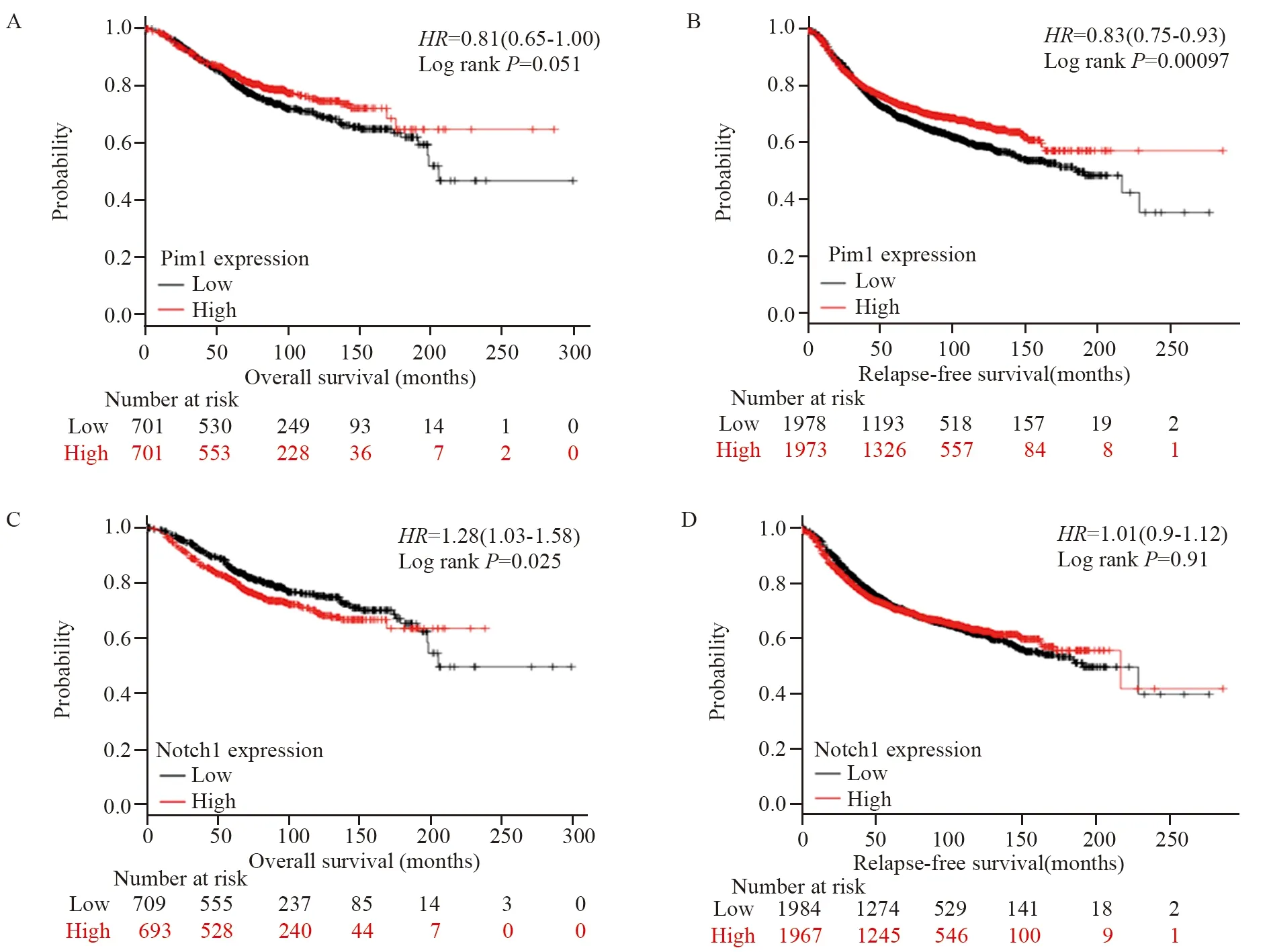
图4 Kaplan-Meier Plotter数据库中Pim1和Notch1 mRNA表达与乳腺癌预后关系Figure 4 Relation between Pim1/Notch1 mRNA expression and prognosis of breast cancer patients from Kaplan-Meier Plotter database
Notch1基因在乳腺导管上皮细胞癌变过程中的表达有明显变化,Notch1 mRNA水平呈显著增高趋势,与Oncomine数据库分析Notch1 mRNA在正常组和浸润癌组中的差异性表达趋势相同,提示可能作为癌基因角色促进乳腺癌变。Notch1蛋白在乳腺癌变组织中表达逐渐升高,与Notch1 mRNA表达一致,进一步提示Notch1发挥癌基因的作用,这与GEPIA数据库分析结果相反,这可能与不同检测人群、标本数量等有关,此外,本研究结果和文献报道[18-19]均发现Notch1分子在正常上皮的水平分别显著低于普通型增生、原位癌和浸润癌组,在原位癌和浸润癌组中的表达差异无统计学意义,推测Notch1分子可能在乳腺早期癌变阶段起促进作用,与乳腺癌晚期浸润无明显相关性。分析Notch1与乳腺癌患者临床病理参数的关系发现,Notch1 mRNA和蛋白表达均与淋巴结转移显著相关,与文献报道结果[7,13,20]相同,表明Notch1表达可能促进乳腺癌浸润和转移,此外,Notch1 mRNA与乳腺癌高分级密切相关,数据库发现Notch1 mRNA在TNM晚期、三阴性乳腺癌患者中表达显著增高,还有研究显示Notch1蛋白表达与组织学高分级和高分期显著正相关[21],与本研究结果相似,提示Notch1基因表达与乳腺癌发生转移等恶性进展的指标明显有关。Kaplan-Meier Plotter数据库分析显示Notch1高表达的乳腺癌患者总生存时间更短,与文献报道[15,22-23]的研究结果相一致,说明Notch1分子为促癌因子,高水平Notch1分子可能与乳腺癌预后不良相关。上述研究提示Notch1在乳腺癌发生发展过程中可能起着促进作用。
我们进一步分析Pim1和Notch1蛋白在乳腺癌组织中表达的相关性,结果显示二者呈显著负相关,提示Pim1和Notch1分子在乳腺癌变进展过程中可能起着负性协同作用,本研究结果仅观察到Pim1和Notch1分子的表达相关性,虽然目前有一篇文献报道Pim1磷酸化调控下游分子Notch1[7],但二者的具体关系和作用机制将是我们下一步的研究方向。
本研究初步分析了Pim1和Notch1在乳腺癌中的表达和临床意义,为探讨Pim1和Notch1信号通路在乳腺癌发生发展中的作用机制提供了前期研究基础,为乳腺癌预后判断提供了理论依据。

