基于灰度信息约束水平集的胰腺组织分割方法
宫照煊,杨东升,乔建忠,覃文军,4
1(中国科学院 沈阳计算技术研究所有限公司,沈阳 110168)
2(沈阳航空航天大学 计算机学院,沈阳 110136)
3(东北大学 计算机科学与工程学院,沈阳 110169)
4(医学影像智能计算教育部重点实验室,沈阳 110819)E-mail:gzxgzxppy@163.com
1 引 言
胰腺是人体最重要器官之一,具有内、外分泌功能.近年,胰腺疾病的发病率逐年上升,尤其是胰腺癌,为致死率最高的肿瘤之一,5年生存率小于1%.CT检查为诊断胰腺病变最主要的检查方式,利用CT数据早期准确的诊断胰腺病变可以有效提高病人生存率.在CT图像上进行胰腺分割作为这一切重要前提,其研究与发展意义重大[1].胰腺组织主要位于后腹膜,形态、位置变异较大,且周围有大量邻近组织,如胃、十二直肠、脾脏及大血管,这些组织在CT图像上与胰腺紧密相连且密度相似.近年来国内外学者提出了多种胰腺分割方法,Cai等人[2]提出了一种基于深度学习的胰腺组织自动分割方法,该方法在深度学习模型中引入了图融合模型,提高了网络模型的求解精度.Zhou等人[3]使用局部区域模型对胰腺区域进行限定,然后在限定区域利用深度学习模型训练得到胰腺组织.Farag等人[4]提出了一种超像素及图像堆标记的模型实现胰腺组织的提取.Li等人[5]提出了一种双向循环序列卷积神经网络方法实现胰腺分割,该模型可以直接分割三维胰腺组织,因考虑了二维层之间的相关性,可以有效提高网络的求解精度.Karasawa等人[6]提出了一种基于多图普的胰腺组织提取方法,该方法利用胰腺组织周围的血管结构选择与其相似的未标记影像图谱,再利用多图普配准实现胰腺组织的提取.王鑫[7]利用水平集模型实现胰腺组织的提取,该方法结合胰腺的形状先验知识构建水平集模型,通过水平集曲线演化实现胰腺组织的精确提取.吴娇龙[8]提出了一种基于形状约束和种子迁移生长的胰腺分割方法,该方法通过区域生长方法进行分割,并利用胃部具有的规则形状特征进行条件约束,对目标区域胰腺实现精确提取.牛帅[9]提出了一种基于低秩分解和多图谱的胰腺分割方法,结合低秩分解和Hessian增强方法获取不同的图谱,最后利用图谱融合实现胰腺组织的提取.
近年来,水平集方法[10-13]受到广大学者的关注,水平集方法不受目标拓扑结构的影像,在影像组织分割中具有较大的优势.因此,本文使用水平集方法实现胰腺组织的自动提取,首先利用直方图均衡化提高胰腺组织与其周围组织的对比度,然后使用深度学习模型实现部分胰腺组织的提取,统计提取的胰腺组织信息并设计灰度信息约束项,结合长度项和图像信息项共同建立水平集模型,通过对水平集模型的演化获取最终的胰腺组织分割结果.算法流程图见图1.
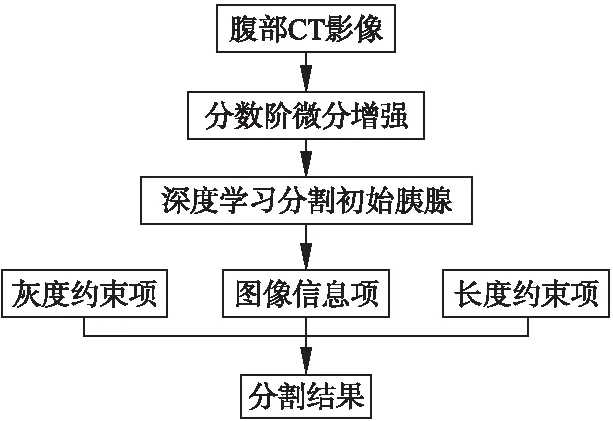
图1 基于灰度信息约束水平集的胰腺分割算法流程图
2 方 法
胰腺在CT影像中与其周围的组织(胃、十二直肠、肝脏)灰度差异较小,直接在原始影像上分割胰腺很难得到理想的分割效果.为了提高胰腺在影像中的对比度,本文利用分数阶微分增强方法对CT影像进行预处理,在增强后的影像中提取胰腺可以有效降低分割难度.
2.1 分数阶微分增强
分数阶微分可以使图像的边缘信息更加突出,纹理信息更加清晰,并能非线性保留图像平滑区域信息.对于信号图像w(m,n),增强后图像可表示为:
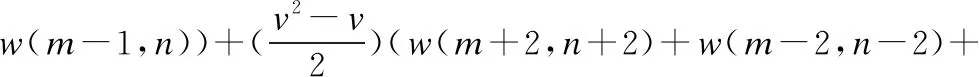
w(m-2,n))+(3v2-v3-2v)(w(m-3,n-3)+
w(m+3,n+3)+w(m-3,n+3)+w(m+3,n-3))
(1)
图2展示了分数阶微分增强后的结果.从图中可以看到增强后的图像中(图2(b))胰腺的轮廓更加清晰,与原图像(图2(a))相比,增强后的影像中胰腺具有更高的对比度.

图2 分数阶微分增强结果
2.2 深度学习模型
本文采用U-net深度学习模型[14]实现胰腺组织的提取.U-net网络由下采样层和上采样层组成.主要由卷积层和池化层以及上采样层组成.利用U-net模型只需要少量数据训练网络就能得到胰腺组织的初始分割结果,初始胰腺分割结果的边界可作为后续水平集模型的初始轮廓曲线.通过统计初始胰腺分割结果的灰度信息可以得到胰腺组织的灰度值范围:
Ilow=μ+θ1·σ,Ihigh=μ+θ2·σ
(2)
其中Ilow和Ihigh分别为利用深度学习模型得到的初始胰腺的灰度上限和灰度下限,μ和σ分别为初始胰腺区域的均值和方差,θ1和θ2为常数.
2.3 水平集模型
Li等人提出了一种基于距离规则化的水平集演化模型(Distance Regularized Level Set Evolution,DRLSE)[15],本文在DRLSE模型的基础上引入灰度信息约束项,可以使水平集曲线更准确地演化至胰腺组织的边缘.本文模型的能量函数定义如下:
ε(φ)=αEintensity(φ)+EDRLSE(φ)
(3)
其中Eintensity(φ)为灰度信息约束项,EDRLSE(φ)为DRLSE模型的能量项,α为非负常数.通过最小化该能量模型,使水平集曲线逐渐演化到胰腺区域边缘,从而实现胰腺组织的精确分割.
DRLSE模型
DRLSE模型由三部分组成:长度约束项,面积约束项以及规则项.能量表达式为:
EDRLSE(φ)=λ1R(φ)+λ2L(φ)+λ3A(φ)
(4)
(5)
(6)
(7)
其中λ1,λ2,λ3为非负常数.公式(5)-公式(7)分别为规则项,长度约束项以及面积约束项.规则项可以保持符号距离函数的连续性,此项可以有效避免水平集重新初始化问题[16].长度项用来约束演化曲线的长度,当曲线收敛至目标边缘时长度项的取值最小,同时该项也可以使演化曲线的表面更加光滑.面积项可以加速曲线的演化,当演化曲线离目标边缘较远时,该项可以有效提升曲线演化的速度.公式(5)和公式(6)的H(x)和δ(·)分别为赫维赛德(Heaviside)函数及其导数,定义为:
(8)
(9)
根据公式(5)-公式(7),公式(4)可以重写为:
(10)
利用梯度下降流最小化公式(10),可以得到:
(11)
灰度信息约束项
为了规则化零水平集,需要得到水平集曲线φ的长度:
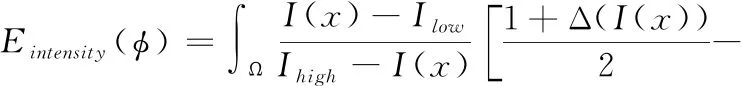
(12)
其中:
(13)
利用梯度下降流最小化总能量函数E(φ),可以得到:
(14)
公式(14)为本文提出的水平集演化模型,通过多次迭代可以使水平集曲线向胰腺组织的边缘收敛,从而实现胰腺分割.
3 实验结果
本文的数据集来自ISICDM2018胰腺分割挑战赛上的公开数据集,我们选取了20组病人的CT影像,随机选取每组病人数据中的含有胰腺组织的2D影像.实验参数设置如下:λ1=0.2,λ2=2,λ3=-1,α=0.1,θ1=-1,θ2=3.
3.1 评估指标
本文选用以下两种评估指标来度量本文提出的方法及其它方法的分割结果:
1)Dice系数
(15)
S和G分别为自动分割结果及医生手动勾画的金标准结果.Dice系数用来度量分割结果的准确性,是一种被广泛使用的评估指标,该度量同时考虑了分割结果得到的正确候选点及错误候选点.Dice系数越趋近于1代表分割结果越接近专家手动勾画结果.
2)正阳率(TPR)
(16)
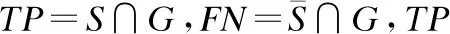
3.2 结果展示
图3展示了本文方法与其它几种水平集方法的分割结果.初始化曲线选择为胰腺内部的一个方块区域(图3(a)),CV模型(图3(b))可以得到全部的胰腺组织,但同时也存在过分割现象;GAC模型(图3(c))也得到了部分胰腺组织,但曲线在演化时却无法准确地收敛至目标的边缘.IVC模型(图3(d))可以得到完整的胰腺组织,但图中胰腺的左下方区域灰度差异与胰腺的灰度值差异很小,该部分为非胰腺区域,IVC模型无法准确地将这部分区域分离出来,且得到的胰腺边缘表面不平滑,呈锯齿状.DRLSE模型(图3(e))可以得到大部分胰腺组织,但受初始化曲线影响没有准确地收敛至胰腺尾部.本文方法可以准确地使曲线收敛至胰腺边缘,灰度信息约束项能够有效使将胰腺与其灰度相似的区域分离.
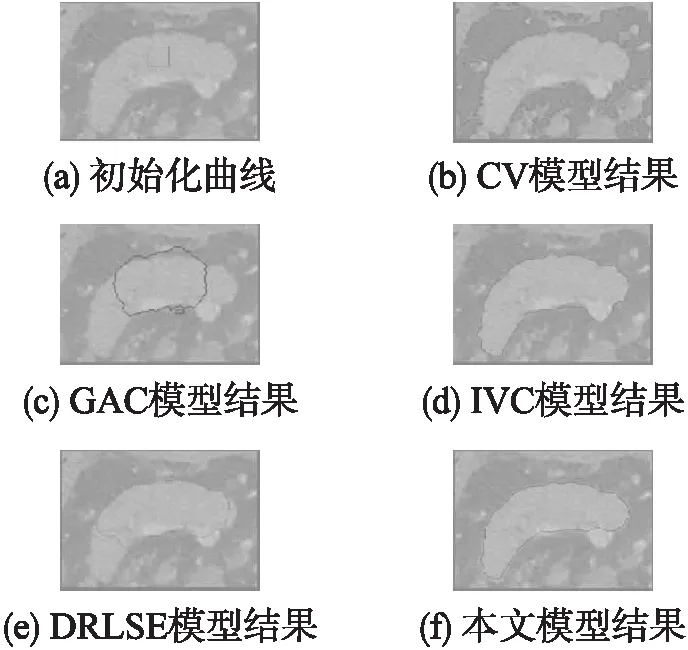
图3 本文方法与其它方法的胰腺分割结果对比
表1展示了本文方法得到的评估结果,多数数据得到的DC值都在0.85以上,TPR值在0.8以上,说明本文提出的胰腺分割方法可以较准确地分割出胰腺组织.数据9和16的DC值和TPR值都较低,这两组病例中的胰腺有病变的部分,且与周围的肝脏组织粘连在一起,故本文方法对这两组病例的分割效果欠佳.
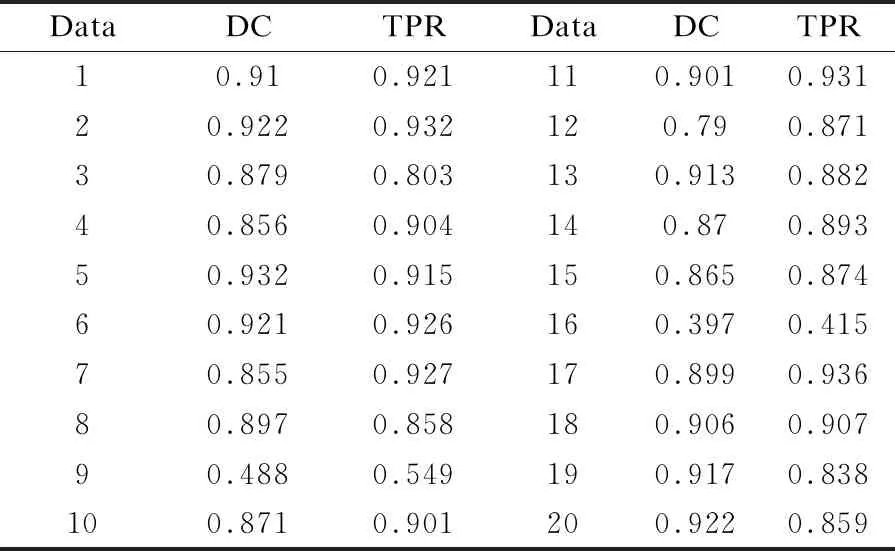
表1 DC和TPR值结果评估
图4展示了本文方法与深度学习方法的结果对比,本文方法是在深度学习结果的基础上结合水平集模型进行精确分割.从图中可以看到,深度学习方法得到的dice值中线为0.72,而本文方法采用水平集优化后得到的dice值中线为0.82.可见本文方法在深度学习模型的基础上引入基于灰度信息约束的水平集方法可以获得更加精确的分割结果.
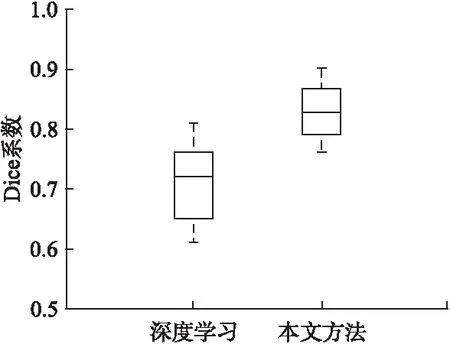
图4 深度学习与本文方法的dice值比较
图5比较了本文方法与其他五种方法的dice值和可预测正样本值(TPR).从图中可以看到,CV模型的dice中值为0.45,TPR中值为0.44;GAC模型的dice中值为0.42,TPR中值为0.44;LBF模型的dice中值为0.61,TPR中值为0.46;IVC模型的dice中值为0.68,TPR中值为0.62;DRLSE模型的dice中值为0.67,TPR中值为0.62;本文方法的dice中值为0.81,TPR中值为0.83.本文方法得到的结果可以得到更准确的胰腺分割结果.
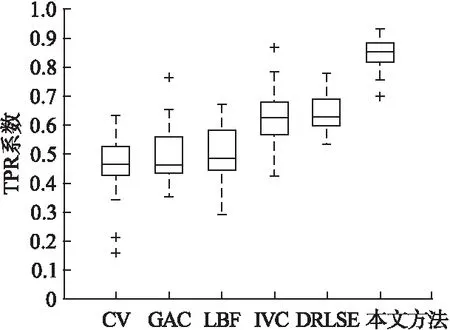
图5 不同胰腺分割方法的dice值及TPR值比较
4 结 论
本文提出了一种基于灰度信息约束的胰腺组织分割方法.利用深度学习提取部分胰腺组织,统计这部分信息并设计了基于胰腺灰度的信息约束项,提取出的胰腺轮廓作为水平集的初始演化曲线.文中提出的灰度约束项能够有效地使水平集曲线收敛至胰腺组织的边缘,长度约束项可以使演化曲线更光滑.实验部分将本文方法与其他五种水平集方法做了比较,并利用两种度量指标评价各个方法,结果表明本文方法可以更准确、有效地提取胰腺组织.未来的研究工作将集中在水平集的算法优化上,考虑结合GPU技术减少水平集方法的运行时间.

