六种蚊虫DNA条形码序列分析*
陈光辉 王孟文 李 焱 刘 东 廖 凌**
(1.新疆大学生命科学与技术学院,新疆乌鲁木齐 830046; 2.喀什海关技术中心, 新疆喀什 844000; 3.喀什大学生命与地理科学学院,新疆喀什 844000)
蚊类是重要的医学昆虫(窦洪举,1987;王创新,2013),有些种类是严重疾病的传播媒介(刘建利,2015;尹授钦,2016),严重危害人类健康(陆宝麟, 2000;张光学, 2008;边长玲, 2009;Wangetal., 2012)。口岸蚊类监测表明,我国口岸蚊类以按蚊属、库蚊属、伊蚊属为主(聂维忠,2011;Wangetal., 2012;方义亮,2013;郭玉燕等,2017)。目前,需要重点防治的蚊类有中华按蚊Anophelessinensis、雷氏按蚊Anopheleslesteri、大劣按蚊Anophelesdirus、微小按蚊Anophelesminimus、淡色库蚊Culexpipienspallens尖音库蚊淡色亚种、致倦库蚊Culexpipiensquinquefasciatus、三带喙库蚊Culextritaeniorhynchus、白纹伊蚊Aedesalbopictus、埃及伊蚊Aedesaegypti等(陆宝麟,2001;邵柏,2002;周健明,2013;赵宣等,2017)。
由于蚊类的医学媒介重要性,对其快速准确鉴定成为口岸流行性传染病防控监测与控制的基础,也是疾病预防与控制、国境卫生检疫的核心环节。传统形态学鉴定方法虽然具有研究方便、直观等特点(梁帆等,2017),但是受技术人员的经验积累、专业水平和主观认识、标本完整性与发育阶段等因素限制(吴宏华,2013),而且蚊科存在大量复合组(体)、种团和近缘种,如赫坎按蚊复合种团(Anopheleshyrcanuscomplex)、尖音库蚊复合种团(CulexpipiensComplex)、大劣按蚊种团(Anophelesdiruscomplex)等,种团内的近缘种形态差异甚小,对于形态较难区分的复合体成员种(或隐种),仅使用传统的形态分类方法鉴定较为困难(陆宝麟,1999;周华云,2004)。这都使得传统媒介蚊种鉴定方法鉴定周期长、鉴定效率低等问题长期存在,不利于口岸蚊虫的高效防控;因此,需要发展更加安全、快速、准确、灵敏的检疫鉴定技术。
分子鉴定方法能够实现媒介生物的快速鉴定,该方法是以核酸碱基排列顺序为基础而建立的(高琪,2005),即将分子生物学技术如RAPD、PCR-SSCP和AFLP等,结合多种分子标识如微卫星、线粒体基因(COI、COII、16SRNA等)和核糖体序列(ITS)等,实现蚊类的分子系统学鉴定(陆宝麟等,1999;彭淑琼等,2010;付文博等,2013);该方法以DNA序列为研究对象,具有准确度高、简便快速、且不受发育阶段的影响等优点(张媛等,2011;周青松等,2013;宋南等,2013;陈光辉等,2017),已经被广泛应用于各物种的分类学鉴定(肖金花等,2004;韦健红等,2010;陈光辉等,2018)。目前在蚊类线粒体基因中,常选用COI、COII、16SRNA鉴定物种(郭玉燕等,2017);在核糖体基因中,常选用ITS1、ITS2 和18S rDNA 鉴定物种。例如,多数物种的COI序列的种内遗传距离大多小于2%,而种间遗传差异相对较高(Folmeretal., 1994;Hebertetal., 2003b; Wardetal., 2005; Hajibabaeietal.,2006;张媛,2011),能够广泛用于动物种及种群研究。由于ITS1、ITS2等基因选择压力小,变异较大,种内遗传差异相对其他基因较高,适合物种及其种群的研究(黄华平等,2006);ITS1基因序列种内遗传距离有时会超过3%,也可以有效区别物种(Robertetal., 2013)。
蚊类分子系统学方面的研究主要集中于所在属、种及种内复合组的研究,并且多集中于mtDNA-COI、ITS2基因,对ITS1、Cytb等基因的研究较少。本研究通过传统分类学确定未知蚊类大致类群,检索或设计引物,扩增出蚊虫的ITS1、ITS2、Cytb、COI基因序列,获得的相关分子数据作为形态学特征的补充,实现目标蚊种的快速鉴定;同时,通过已知种类通用引物来获得未知基因序列,丰富了蚊类ITS1、ITS2、COI核酸数据库,为蚊虫快速鉴定、监测预警及后续防控提供理论基础。

表1 样品信息Tab.1 Sample information
1 材料与方法
1.1 标本采集
蚊类的采集:2017和2018年8月中旬前后,在黄昏至凌晨蚊类出没期,在喀什市阳光小区河边和小亚郎水库边蚊虫较多的水边架设蚊虫采集网采集样品;部分蚊类标本系喀什海关口岸送检样品,样品获得信息如表1所示。依据文献(陆宝麟,1997;宋明昌,2004)初步确定为双翅目蚊科,形态鉴定结果由通过蚊虫能力验证的专家进行核实;能力验证样本为未知虫足,将蚊虫样本整理后保存于含有99%酒精的离心管中,并放置于冰箱4 ℃冷藏保存,做为DNA提取材料。
1.2 基因组 DNA 提取和PCR 扩增
1.2.1基因组 DNA 的提取: DNA提取参照邹志文等(2011)Chelex改进方法;对单只蚊虫样品用双蒸水清洗干净(清洗2次,期间涡旋震荡取出杂质),将清洗干净的样品置于洁净滤纸上干燥;挑取单头样品置于1.5 mL的离心管中,用灭菌玻璃研磨棒充分研磨;加灭菌双蒸水0.5 mL,涡旋混匀,冰箱-20 ℃冷却10 min后,12 000 r/min离心5 min,弃上清,重复2~3次;沉淀中加入150 μL悬浮好的5%的Chelex-100溶液,56 ℃水浴2 h;涡旋5~10 s,置100 ℃加热10 min;取出后涡旋溶液5~10 s,10 000 r/min离心5 min,上清液即可作为PCR反应的DNA模板,-20 ℃保存备用(Walshetal.,2011)。此法提取的DNA无法电泳检测(周月琴等,2003)。
1.2.2基因片段PCR 扩增: 引物由北京奥科生物技术公司合成,具体引物序列及参考文献如表2所示。扩增体系总体积为25 μL,包括1.5 U Taq酶,0.25 mmol/L dNTP,1×反应缓冲液,2.0 mmol/L Mg2+,上下游引物各为通用引物0.3 μmol/L,DNA 模板50 ng。反应程序为:95 ℃预变性 5 min;95 ℃变性 45 s,55 ℃或58 ℃(ITS1)退火 1 min,72 ℃ 延伸 1 min,循环 40次,循环结束后 72 ℃延伸 7 min。PCR产物直接送到安徽通用生物技术有限公司进行测序(双脱氧法)。
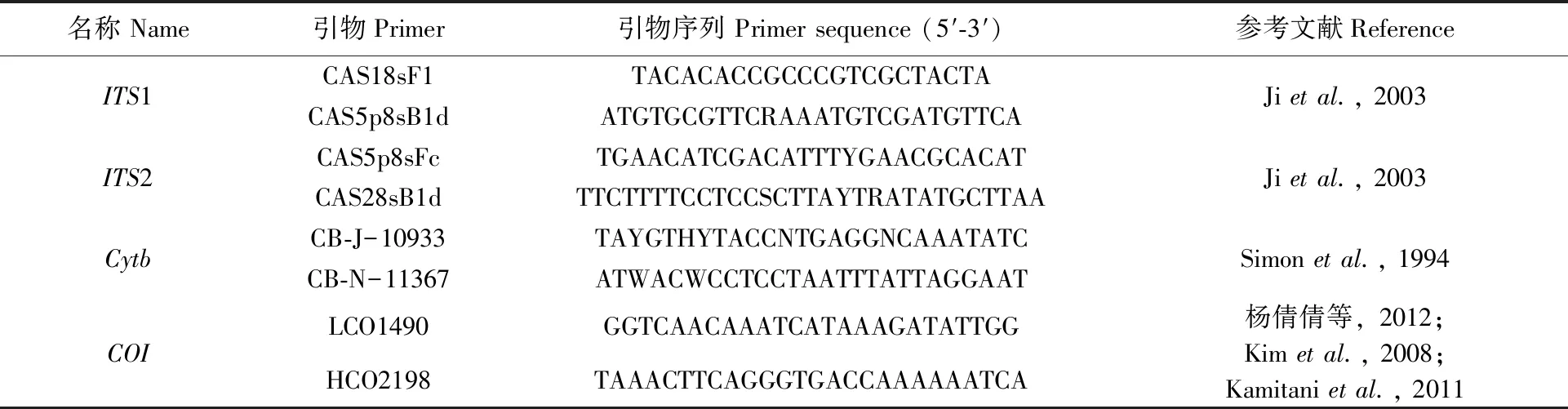
表2 PCR 扩增引物设计及参考文献Tab.2 PCR amplification primers and references
1.3 序列数据处理及分子系统树构建
测序获得的基因序列用 DNA star Package 中的Editseq软件进行正反链拼接匹配校正。在NCBI(https://www.ncbi.nlm.nih.gov/)网站上运行 BLAST程序进行序列同源性比较,确定所获得的序列是否是昆虫的ITS1、ITS2、Cytb、COI基因序列。根据相似度和种内遗传距离对样品进行分子鉴定。结合从GenBank 中比对的与所获得的ITS1、ITS2、Cytb、COI序列同源性较高的蚊类的相关序列,用ClustalX1.83软件比对,非保守区域去除。用分子进化遗传分析软件MEGA6.0(Kumaretal., 2004;金倩等,2013)分析各物种间DNA序列的差异,统计分析碱基含量和使用频率。基于 Kimura-2-Parameter模型,用邻近法(NJ, Neighbour-Jioning)构建分子系统树,1 000次循环估计系统树中节点的自举置信水平(Bootstrap confidence level,BCL)。
2 结果
2.1 分子鉴定
测序获得了14个蚊虫样品的部分ITS1、ITS2、Cytb、COI基因的部分序列,依据表3中在NCBI数据库中查阅的相关序列的最高得分、覆盖度、相似度、可能物种排序的等信息进行鉴定。覆盖度较低,且序列相似度低于种内遗传距离的补数时,确定为新的条形码序列,即数据库中未检索到相关高度相似序列,核酸序列与NCBI数据库中的相关序列相似度较低,确定为该蚊虫样品的新序列。本文获得的蚊虫核酸序列39条,其中ITS1 基因10条、ITS2基因9条、Cytb基因10条、COI基因10条;由于实验操作水平有限和样品有限有些样品PCR扩增未获得相关目的片段,未获得的序列在表4中用“/”标出。
对14个蚊虫样依据序列相似性和种类遗传距离进行分子鉴定,共鉴定出6种蚊虫,其中样品1、2、3、4、11、12鉴定为凶小库蚊,样品5、7、8鉴定为尖音库蚊(能力验证样品),样品6、10鉴定为里海伊蚊,样品9鉴定为赫坎按蚊,样品13鉴定为致倦库蚊(喀什机场截获),样品14 鉴定为白纹伊蚊。样品鉴定结果、对应样品编号、所获得的基因片段如表3所示。
2.2 ITS1、ITS2、Cytb、COI碱基含量统计
将所测序列进行拼接校对,在NCBI 网站上运行BLAST 程序可以确定测得序列为昆虫的ITS1、ITS2、Cytb、COI基因序列。通过与 GenBank中的相关基因序列进行比较,结合 GenBank 中下载的与该ITS1、ITS2、Cytb、COI序列同源性较高的蚊类的序列(本文中所获得的核酸序列均能反应是蚊虫中的相关基因的序列),用 ClustalX1.83 软件进行比对,将非保守区域去除,测序拼接后仅以表4中的序列长度进行分析。通过DNAman软件分析本文所获得的各个基因序列所有位点中AT碱基含量和GC碱基含量如表4所示。
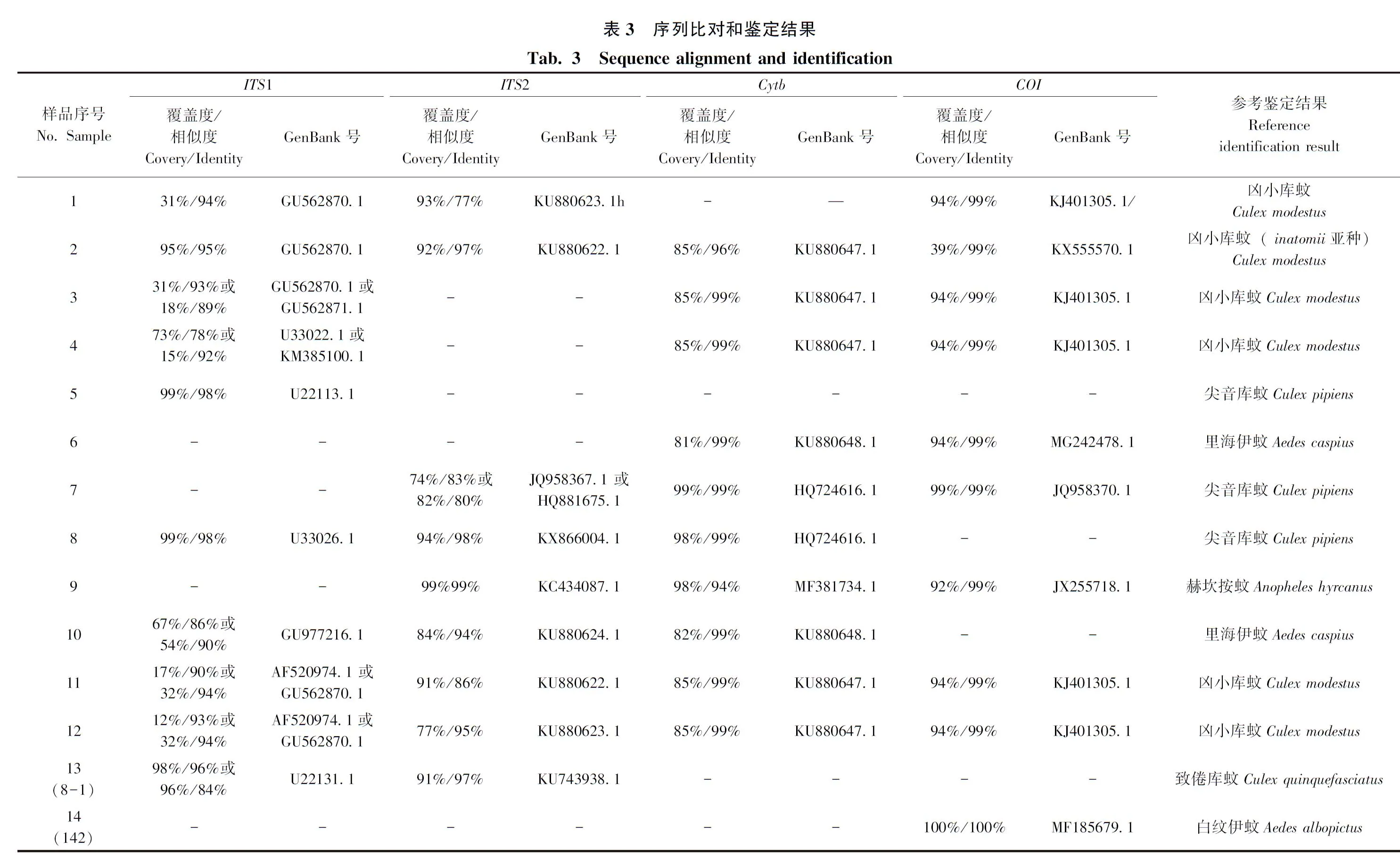
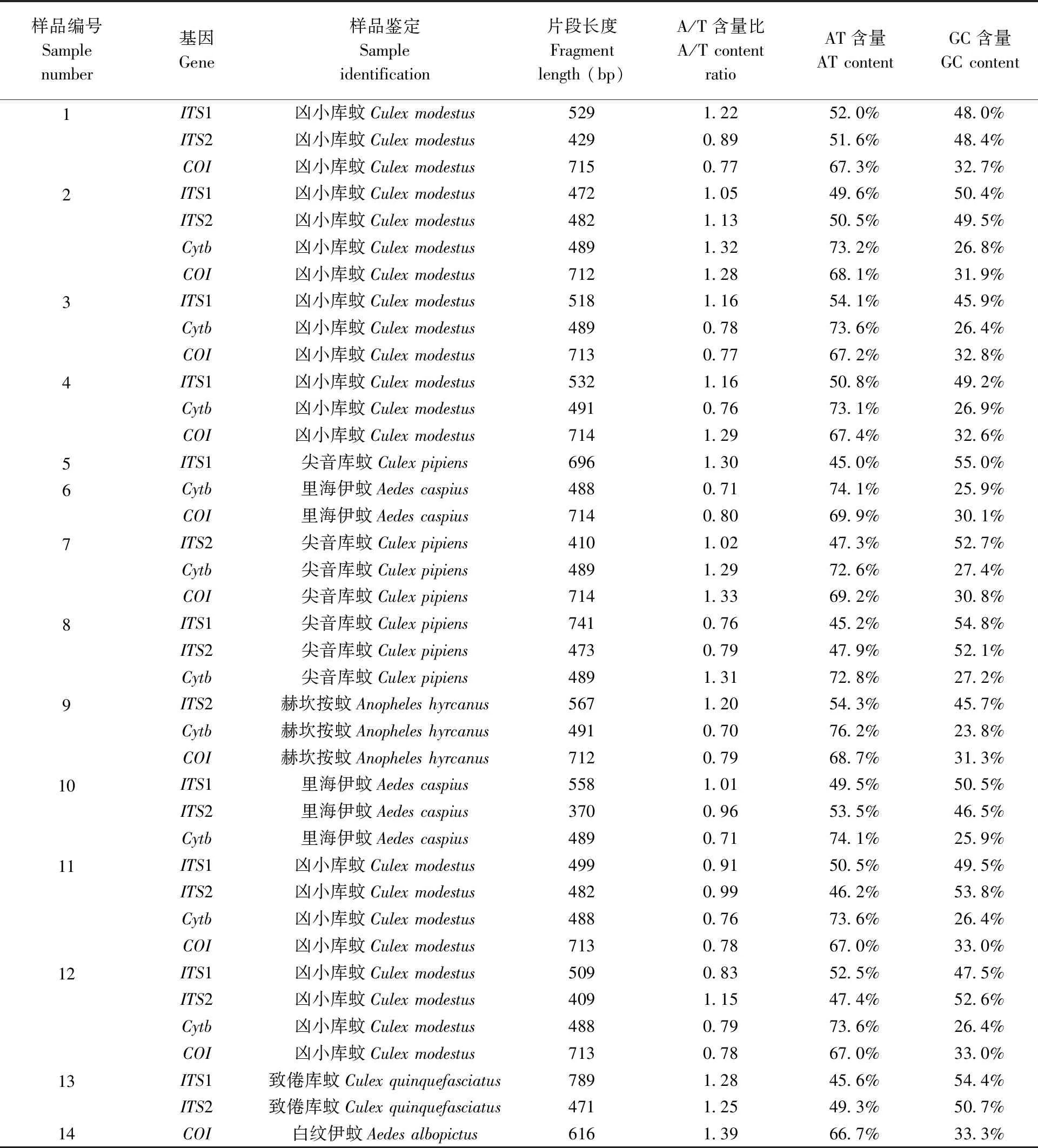
表4 核苷酸使用频率统计Tab.4 Statistics on nucleotides
2.3 系统进化树构建结果
测序获得的样品COI、Cytb、ITS2序列,结合与GeneBank 中相似性性较高的蚊类的相关序列,基于 Kimura-2-Parameter模型,用邻近法(NJ, Neighbour-Jioning)构建了COI、Cytb、ITS2基因的分子系统树,系统树如图1、2、3所示。结果表明数据库中某特定蚊虫所在科、属的分子数据越丰富,构建出来的系统进化树越能
体现进化程度,本文中获得的COI、Cytb序列相对较ITS2多,因此系统进化树反应出的进化关系较丰富;而ITS1数据较少系统进化树构建较为困难且不能反应出的进化关系。COI、Cytb、ITS2相似性比对均有较高的相似性,如样品多数COI基因序列与NCBI中相关序列覆盖度较高且相似性高达99%,利用构建系统进化树,也利于分子鉴定。
3 讨论
3.1 分子鉴定结果分析
蚊类种类鉴定依据种内遗传距离序列相似性进行。拼接校对所测序列,在 NCBI 网站上运行BLAST程序,依据序列相似性进行鉴定;鉴定结果如表3所示,最高得分、覆盖度、相似度、可能物种等都是鉴定的依据。对达到相似度标准的序列,可以鉴定到种;未到标准的为新序列。样品多数COI基因序列与NCBI中相关序列相似性高达99%,大于或等于鉴定水平98%,因此可以鉴定为该种类昆虫,即根据Hebert(2003a,b)中提出的具有足够核酸信息的COI基因种内遗传距离小于等于0.02时,可将其鉴定为同一种类,在此NCBI数据中的最高得分、覆盖度、相似度(遗传距离的补数);例如,样品14与NCBI中MF185679.1白纹伊蚊COI基因的相似度为100%,因此样品14确定为白纹伊蚊;再如样品5、8的ITS1序列与NCBI中的序列相似度高达98%,因此可以鉴定为凶小库蚊。ITS1,ITS2相似度大于97%、Cytb,COI相似度大于98%视为相似度达到物种鉴定标准或水平,即能够实现准确鉴定。如样品5、8的ITS1序列,如样品2、8、9、13的ITS2序列,与NCBI中的序列相似度大于97%,鉴定为相似度最高的蚊类物种;如样品2、3、4、6、7、8、10、11、12的Cytb序列,样品1、2、3、4、6、7、9、11、12、13的COI序列,与NCBI中的序列相似度大于98%,鉴定为相似度最高的蚊类物种;且与图1、2系统进化树中的各自已知种类聚为一簇,能够对鉴定结果进行较好的印证。单个序列或多个序列的鉴定结果是一致的,可以确定每个样品都进行了准确鉴定,鉴定结果如表4所示。样品13出现ITS1相似度为96%,ITS2相似度为97%,ITS1相似度未达到鉴定水平要求,不能作为准确鉴定的参考,在此选在ITS2作为鉴定参考,因此样品13鉴定为致倦库蚊;相似度较低时,需要在进行其他相关基因的扩增并获得序列,以验证结果的准确性,如样品13需要进一步做COI或其他基因来验证。ITS1、ITS2、Cytb序列相似度低于鉴定水平,说明数据库中关于实验样品相关蚊类的ITS1、ITS2、Cytb序列还不够丰富,本研究获得的ITS1、ITS2、Cytb新序列较多,新的COI序列较少,在表3中标出,获得的新的序列有助于后续利用ITS1、ITS2、Cytb序列进行快速鉴定。同时也说明蚊类COI序列研究的较多,该类蚊虫的ITS1、ITS2、Cytb基因序列有待丰富。
3.2 ITS1、ITS2、Cytb、COI碱基含量分析
核酸使用率统计表明,Cytb、COI基因序列的AT含量明显高于GC含量AT含量,表现明显的A+T碱基偏嗜,且A与T含量相当,符合昆虫线粒体基因碱基组成的基本特征。ITS1、ITS2、Cytb、COI基因均与NCBI数据库中的相关基因相对应,因此分析表明这4类基因序列均能在分子水平上对蚊类进行检测分析。
3.3 系统进化树构建与分析
文中该类样品的ITS1相似度较低,达到种内遗传距离鉴定标准的序列较少,说明文中该类样品的ITS1数据较少,利用该序列进行快速鉴定有鉴定失败的风险,说明文中该类样品的该基因有待进一步丰富,本文没有构建文中该类样品的ITS1基因的系统进化树;相反文中该类样品的COI基因达到种内遗传距离鉴定标准的序列较多,说明文中该类样品的COI数据较多,利用该基因进行快速鉴定大都能够实现快速鉴定。文中该类样品的ITS2、Cytb系统系统进化树数据介于ITS1和COI之间,且覆盖度较低,不利于建树分析遗传距离(如样品2的Cytb基因覆盖度低时表达出来的核酸信息不足,可能和其他种类通过少量信息耦合,建树时不能准确的反映进遗传距离),说明有待补充和丰富,以便于以后在COI基因扩增测序不利时,利用ITS1、ITS2和Cytb基因对该类样品进行快速鉴定。从COI、ITS2和Cytb各自基因NJ系统进化树可以分析得知:鉴定的目标蚊种均能和NCBI数据中相应蚊种COI、ITS2和Cytb基因聚成一簇,遗传进化分支清晰明确。但是COI基因尖音库蚊有两个分支,且不能聚成一簇;凶小库蚊和尖音库蚊的其中一支遗传距离较近,明显聚成一簇;Cytb基因;尖音库蚊和凶小库蚊之间的关系与COI基因类似,其中样品2COI(Culexmodestus)和Cytb(Culexinatomii(modestus))基因分别簇与不同的分支;而ITS2基因样品2则与Culexmodestus聚为一簇;以上分歧说明样品的分子鉴定有待进一步分析。
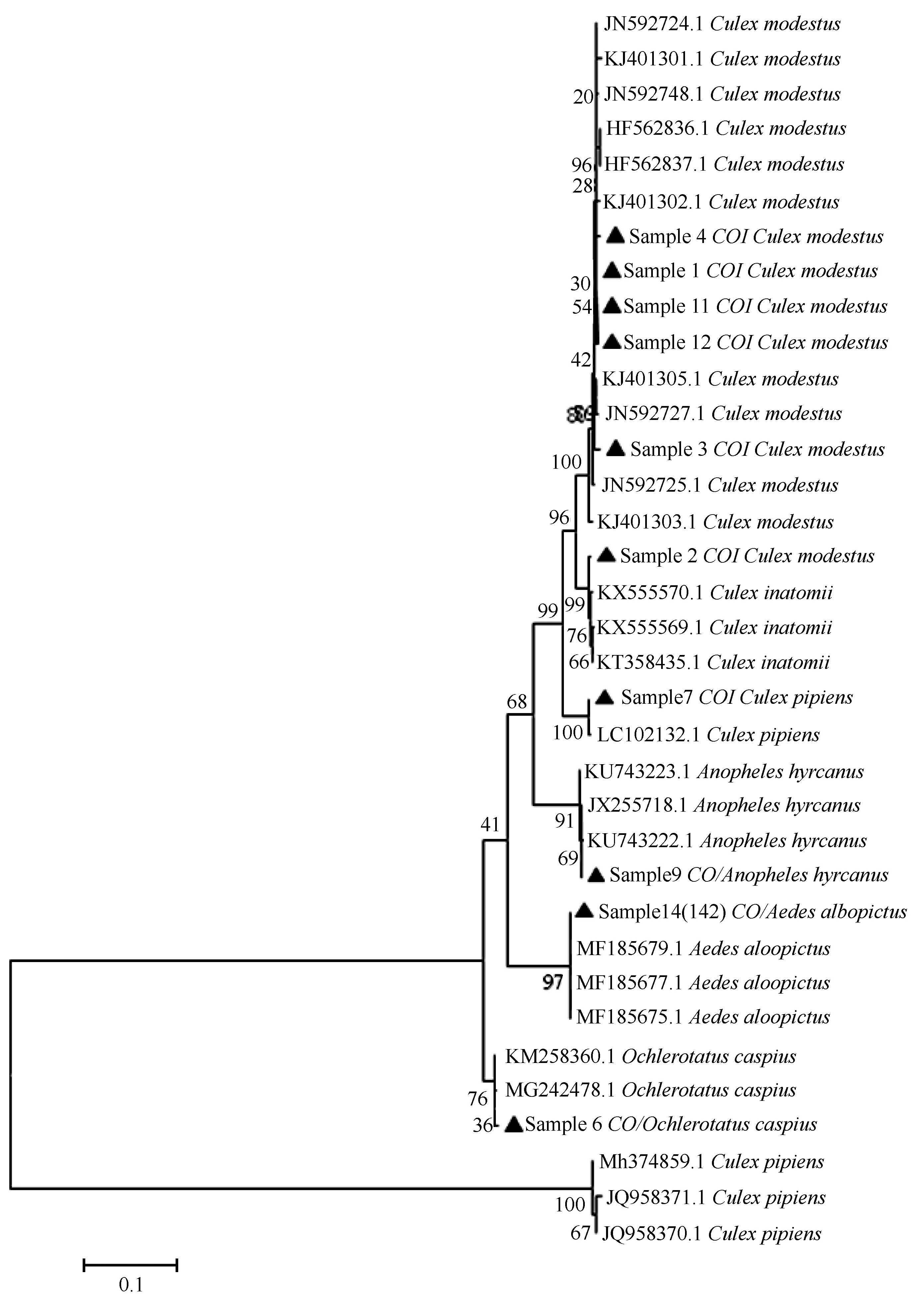
图1 部分蚊种的COI基因 NJ 分子系统树Fig.1 NJ phylogenetic tree of COI gene from partial mosquito species注:图中数字表示置信度;我们的样本COI用实心三角标出。 Note:Integer indicates bootstrap confidence values,and our samples are marked in solid triangles.

图2 部分蚊虫的CytB基因 NJ 分子系统树Fig.2 NJ phylogenetic tree of CytB gene of partial mosquito species注:图中数字表示置信度;我们的样本用实心三角标出。 Note:Integer indicates bootstrap confidence values,and our samples are marked in solid triangles.
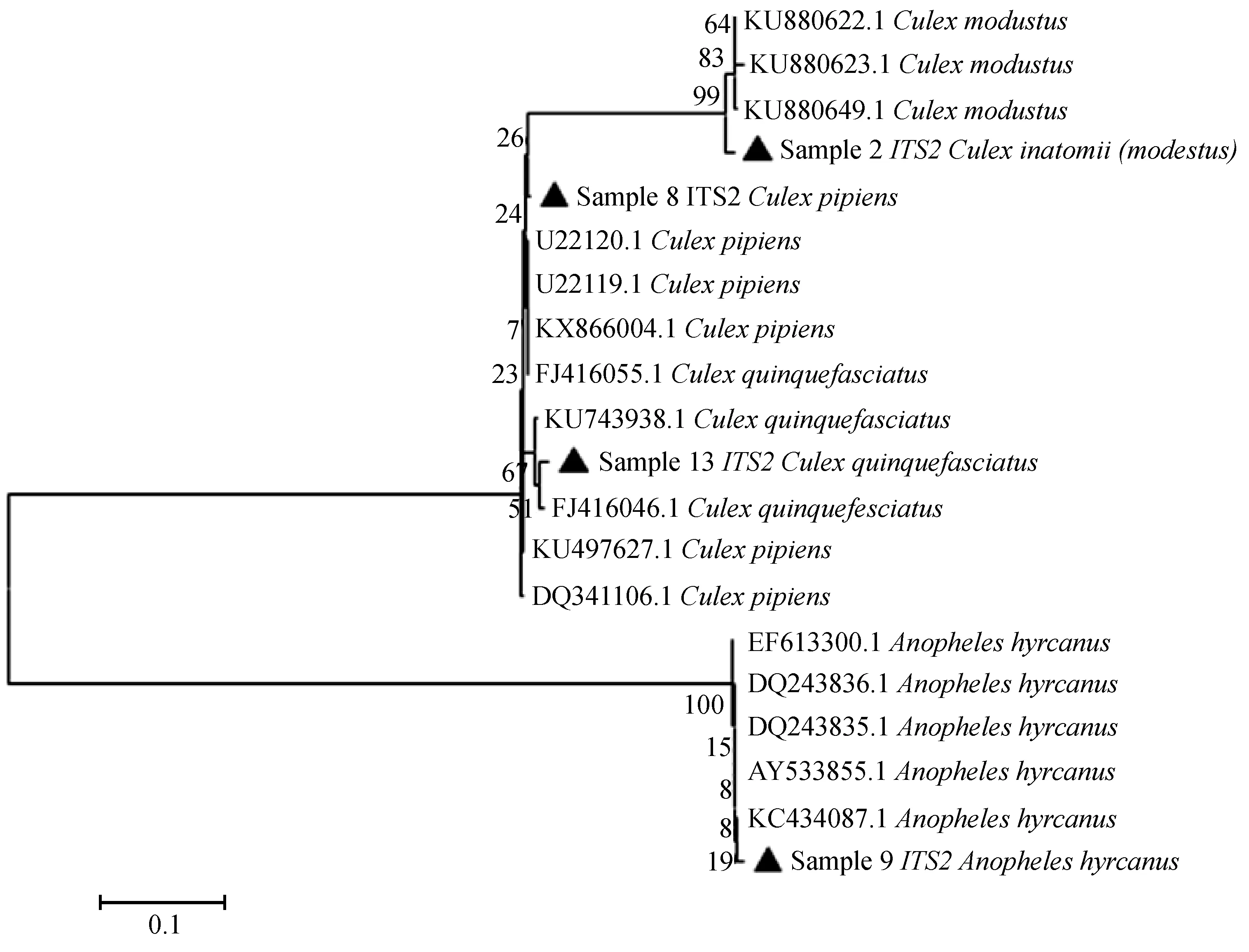
图3 部分虫类ITS2序列NJ分子系统树Fig.3 NJ phylogenetic tree of ITS2 gene of partial mosquito species注:图中数字表示置信度;我们的样本ITS2用实心三角标出。 Note:Integer indicates bootstrap confidence values,and our samples are marked in solid triangles.
本研究利用DNA条形码技术,结合形态学分类方法,通过提取蚊类DNA,检索或设计适用于蚊类相关基因扩增的引物进行PCR扩增并测序,获得了文中一些类蚊虫样本的ITS1、ITS2、Cytb、COI基因的序列,根据种内遗传距离和NJ树分析表明这些基因序列都可作为特定蚊虫DNA条形码数据;各种类相关基因各自成簇,遗传进化关系相对清晰;该研究获得了几种蚊虫的DNA 分子特征,丰富了蚊虫基因序列的数据库,为快速鉴定提供多条新的参考序列。

