应用基因芯片筛选HBV 对肝癌细胞脂代谢相关基因影响的初步研究
梅 勇,魏 柏,杨盛力,占 静
华中科技大学同济医学院附属梨园医院普外科,湖北 武汉 430077
近年来不断有学者揭示异常的脂代谢与肝脏病变密切相关,一方面致癌因素诱发代谢异常,促使细胞恶性转化;另一方面异常的代谢参与了癌细胞的增殖、侵袭和转移。肝癌是最常见的恶性肿瘤之一,目前已明确乙型肝炎病毒对肝癌的发生、发展密切相关[1],但HBV感染对于脂代谢的影响尚未见报道。因此本研究以整合了HBV DNA的HepG2.2.15细胞来模拟被HBV感染状态[2],利用基因芯片技术研究肝癌细胞HepG2.2.15和HepG2的脂肪代谢相关基因表达差异,通过寻找差异基因表达为后续研究HBV对肝癌脂肪代谢相关基因对肝癌生长、转移和耐药等的影响提供新的思路。
1 材料与方法
1.1 材料肝癌细胞株HepG2细胞和HepG2.2.15细胞由华中科技大学同济医学院附属协和医院卢银平教授馈赠。基因芯片为Affymetrix公司的GeneChip primeview human;基因芯片杂交、洗涤和染色试剂盒购自GeneChip Hybridization Wash and Stain Kit;RNA质检为Agilent RNA 6000 Nano试剂盒;胎牛血清、DMEM培养液、RNA提取试剂Trizol和PCR试剂Power SYBR®Green PCR Master Mix购自Life Technologies公司;PCR引物购自Invitrogen公司。
1.2 细胞系和细胞培养肝癌细胞株HepG2和HepG2.2.15采用含质量浓度为100 g/L胎牛血清的DMEM,置于37 ℃,体积分数为5%的CO2培养箱内培养。当生长浓度为60%~80%融合时用于后续实验。
1.3 RNA抽提和质检采用Trizol法进行样品的总RNA抽提,抽提所得总RNA送至上海吉凯生物公司使用NanoDrop 2000和Agilent Bioanalyzer 2100质检,合格样本进入基因芯片和RT-PCR实验。
1.4 基因芯片检测差异基因质检合格的RNA样品由上海吉凯生物公司使用Affymetrix公司提供的GeneChip primeview human 100 format 芯片进行基因芯片分析。基因表现有显著差异的筛选标准为必须符合 |FC|>2,根据KEGG中所有pathway的基因信息,将差异基因进行富集分析,根据P值排序后,选取前十位加以展示。
1.5 RT-PCR验证基因芯片结果采用Tirzol法提取细胞RNA,利用分光光度计测定RNA的浓度和纯度后取2 μg的mRNA逆转录为cDNA,根据sybrGreen Master Mix操作说明进行定量PCR反应,检测CYP1A1、CYP1B1和AHR的表达。设3个平行孔。检测基因引物如表1所示。
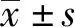

表1 验证基因引物序列及扩增片段长度Tab 1 The sequence and length of gene primer
2 结果
2.1 总RNA质控提取HepG2细胞及HepG2.2.15细胞的总RNA,其A260/A280为1.7~2.2,RIN≥7.0, 28S/18S>0.7,证实已提取到高纯度的RNA。
2.2 基因芯片分析差异表达基因与生物学信息分析以肝癌细胞株HepG2和HepG2.2.15为研究对象,通过基因芯片筛选到显著差异表达(|FC|>2)的基因1 263个[3],脂代谢基因有92个,其中表达上调的基因53个,表达下调的基因39个,涉及细胞生长、增殖、分化、运动和耐药等多种分子功能。将差异基因进行富集分析,根据P值排序后,我们选取前十位加以展示(见表2~表3)。
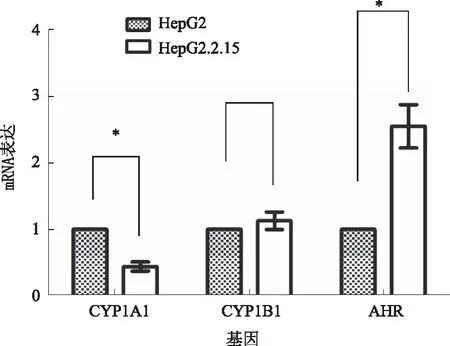
2.3 RT-PCR验证基因芯片中表达差异基因根据既往研究结果[4]和本次差异表达基因,选择了3个基因利用RT-PCR检测mRNA水平从而进行验证。结果显示,与HepG2细胞比较,CYP1A1在HepG2.2.15细胞中的表达降低,CYP1B1未见明显变化,AHR显著升高(见图1)。可见上述基因表达变化与基因芯片一致,说明芯片结果可靠。

表2 HBV介导的差异表达基因Tab 2 Analysis of HBV induced differentially expressed genes
续表2

基因名称基因编号FC基因生物功能DLK18788-11.036673NotchsignalingpathwayPVRL325945-12.10701lensmorphogenesisincamera-typeeyeCRIP11396-14.727744apoptoticprocessNDRG465009-15.420665responsetostressFST10468-17.917171negativeregulationoftranscriptionfromRNApolymeraseⅡpromoterHOXD13231-18.290617transcription,DNA-dependentABCG29429-22.710133ATPcatabolicprocess
表3 与脂代谢相关的差异表达基因Tab 3 Analysis of differentially expressed genes of lipid metabolism related genes
注:*P<0.05。
3 讨论
现已证实HBV感染与肝癌的发生、发展相关,有学者指出HBV参与了DNA重排、增殖凋亡、细胞周期等过程的调节[5]。与传统的研究方法相比,基因芯片可以获得大量差异表达基因谱及大量差异表达基因的信息,从而实现从全局预测这些基因的功能和基因间的相互关系[6]。
越来越多的报道显示,脂代谢异常和肿瘤发生存在相关性[7-8],肝脏作为脂代谢的重要脏器,过度的脂质堆积会导致肝脏脂肪变,最终发展为肝癌。本研究结果显示,涉及脂代谢的基因有92个,具有表达上调的基因53个,表达下调的基因39个,涉及细胞生长、增殖、分化、运动和耐药等多种分子功能。
在本研究中表达上调的基因中最为明显的是PON1、PLA2G、PRKD1等。PON1主要由肝脏合成的一个急性期负调控蛋白,不仅调节胆固醇的转运[9-10],还可改变氧化应激反应和脂质过氧化物反应的应对能力[11],目前已有相关报道显示PON1多态性与肿瘤的相关性,有望成为肿瘤诊疗新的靶标[12]。PLA2G是磷脂酶家族中的一员,在生物界分布广泛,目前已知PLA2G的功能大多与炎症反应、免疫反应、细胞增殖、缺血性损伤等相关[13]。已有文献报道PLA2G2A被认为是一个潜在的抑癌基因,可调节肿瘤的侵袭和转移,但其抗肿瘤的具体机制尚未明确。PRKD1(PKD1)广泛表达于各组织器官中,参与调控细胞内多条信号传导通路,调节细胞增殖、凋亡、侵袭、血管生成等生物学行为。有研究表明,PRKD1表达异常与前列腺癌、乳腺癌、消化道肿瘤相关,推测PRKD1是RAS-NF-κB、PI3K/Akt、JNK等信号通路的重要节点[14-15],使得通过调控PRKD1来治疗肿瘤成为可能。
在HepG2/HBV细胞中表达下调的脂代谢相关基因中有代表性的是AKR1C2、AGMO、CYP1A1等。AKR1C2为人类醛酮还原酶超家族成员之一,在不同肿瘤中表达水平不一,可能参与调控细胞周期、增殖、上皮细胞向间质细胞的转变等生物学过程[16-17],但具体调控通路值得进一步探索。AGMO(TMEM195)是一类跨膜蛋白,其家族成员参与了肿瘤的发生、发展[18],但AGMO生理学功能及在肿瘤病理演进中的作用仍不清楚。CYP1A1是细胞色素P450家族成员之一,现报道CYP1A1多态性和肿瘤易感性及药物代谢相关[19],其机制有待进一步探索。
有研究[4]表明,肿瘤组织周围或内部的脂代谢异常最终影响肿瘤的发展及恶化。目前在动物模型中已经得到证实HBV通过脂质能量通路可能影响肿瘤生长、增殖及代谢,以脂代谢异常相关基因为靶点改变肝癌生长、凋亡等特性也逐渐受到广泛关注。本实验采用RT-PCR检测了CYP1A1、CYP1B1和AHR基因在HepG2.2.15及HepG2中的异常表达,验证HBV参与了肝癌脂代谢的调节,同时也表明基因芯片结果可信,但其具体调节通路还有待进一步探索。
本研究应用基因芯片筛选了HBV对肝癌细胞脂代谢相关基因表达的影响,在基因功能分析中获取了HBV可能作用的与脂代谢相关的靶基因,此外实验采用了RT-PCR检测了HBV对目前常见脂代谢基因表达的影响,验证了芯片结果的可靠性,提示这些基因可能成为临床逆转肝癌生长的新靶点,为深入探讨HBV在肝癌脂代谢中的作用提供了新的线索。然而本研究中采用的芯片结果主要反映了基因在转录水平的变化,仍具有一定局限性,尚需进一步研究。

