植物RNA结合蛋白研究进展
张在宝,李婉杰,李九丽,张弛,胡梦辉,程琳,袁红雨
植物RNA结合蛋白研究进展
张在宝1,2,李婉杰1,李九丽1,张弛1,胡梦辉1,程琳1,袁红雨1,2
(1信阳师范学院生命科学学院,河南信阳 464000;2信阳师范学院河南省茶学重点实验室,河南信阳 464000)
在真核生物中,RNA结合蛋白(RBPs)是一类重要的转录后调控因子,通过与RNA结合形成核糖核蛋白复合物来调节真核生物细胞的RNA代谢过程,包括RNA的转移、修饰、翻译及降解。RNA结合蛋白广泛存在于动物、植物以及微生物中,约占真核生物基因编码蛋白的2%—8%。近年来,对RNA结合蛋白的研究已成为备受关注的热点。RNA结合蛋白与人类健康密切相关,许多RNA结合蛋白的突变都会导致人类疾病。RNA结合蛋白(尤其是三角状五肽重复区蛋白)不仅在植物中大量存在,而且作为重要的调控因子在RNA代谢、生长发育以及应激反应过程中发挥重要作用,这已引起人们的广泛关注。相对于动物RNA结合蛋白的大量研究,植物RNA结合蛋白的功能研究还相对较少。文中详细总结了近几年植物RNA结合蛋白的功能研究、作用机制以及同其他RNA结合蛋白之间的相互关系,并在此基础上重点阐述了5类RNA结合蛋白家族在植物中的功能研究进展,包括富含丝氨酸-精氨酸的RNA结合蛋白(SR蛋白)、富含甘氨酸的RNA结合蛋白(GR-RBPs)、三角状五肽重复区蛋白(PPR蛋白)、DEAD-box RNA解旋酶(DEAD-box RHs)以及RNA分子伴侣。主要在拟南芥、水稻和小麦等模式植物或经济作物中对上述5类植物RNA结合蛋白的功能基因进行介绍,总结每类RBPs在植物的RNA代谢、生长发育以及逆境胁迫响应过程中的重要作用,从而为基础研究和农业生产实践提供了重要的理论依据。在这5类RBPs中,SR蛋白主要作为重要的选择性剪接因子参与RNA代谢,从而在植物的生长发育和胁迫响应中发挥关键的调节作用;许多GR-RBPs家族成员具有功能多样性,一方面可能通过介导植物激素信号通路来调节植物的胁迫耐受性和各种生长发育过程;另一方面作为RNA分子伴侣参与RNA折叠反应并因此在低温和干旱等非生物胁迫响应过程中发挥关键作用。PPR蛋白主要参与线粒体和叶绿体的RNA代谢,调节植物的胁迫响应和生长发育过程;DEAD-box RHs作为细胞核和细胞器重要的RNA剪接因子,在植物生长发育以及非生物胁迫响应中发挥多种功能;作为非特异性RNA结合蛋白的RNA分子伴侣,通过参与RNA折叠反应而维持RNA分子的正常功能。此外,前4类RNA结合蛋白中有许多RBPs具有RNA分子伴侣活性,这使得同一蛋白可能具有功能多样性,从而赋予植物在逆境下具有较强的胁迫耐受性。
RNA结合蛋白;RNA代谢;植物生长发育;应激反应
转录及转录后水平的基因表达调控对真核生物生长发育和响应外部环境刺激至关重要。真核生物基因表达的转录后调控包括RNA加工、前体RNA剪接、RNA的细胞核输出、RNA的稳定、翻译和凋亡,这些过程被称为RNA代谢。在RNA代谢途径中,大量的蛋白质因子——RNA结合蛋白(RNA-binding proteins,RBPs)与RNA结合形成核糖核蛋白(ribonucleoprotein,RNP)复合物来调节RNA代谢过程。每一种RNA分子都有许多RNA结合蛋白与之结合来稳定、保护、组装或转移它[1-2]。RNA结合蛋白具有多个保守的基序和结构域,包括RNA识别基序(RNA recognition motif,RRM)、锌指基序、K同源性结构域(K-homology domain,KH)、冷休克结构域、富含甘氨酸的区域、富含精氨酸的区域、RD重复序列和SR重复序列,这些结构域相互结合构成了各种特异性RBPs[3-4]。
RNA结合蛋白广泛存在于动物、植物以及微生物中,约占真核生物基因编码蛋白的2%—8%[5]。根据蛋白结构,RNA结合蛋白可以分为7个蛋白家族:富含丝氨酸-精氨酸的RNA结合蛋白(SR蛋白)、富含甘氨酸的RNA结合蛋白(GR-RBPs)、三角状五肽重复区蛋白(PPR蛋白)、DEAD-box RNA解旋酶(DEAD-box RHs)、叶绿体RNA剪接与核糖体成熟结构域蛋白(chloroplast RNA splicing and ribosome maturation domain protein,CRM蛋白)、S1结构域蛋白(S1 domain containing-protein,SDPs)和细菌冷休克蛋白(bacterial cold shock protein,CSPs)。在哺乳动物中,RNA结合蛋白与人类疾病如肿瘤、神经系统疾病等密切相关。例如RNPC1(RRM家族的RBP)通过调节癌细胞中mRNA的稳定性而实现靶标的差异表达,其异位表达会诱导细胞周期停滞,抑制乳腺肿瘤细胞增殖,并通过调节诱导的逆转上皮-间质转化(epithelial-mesenchymal transition,EMT)进一步抑制肿瘤细胞的迁移和侵袭[6]。TDP-43是核糖核蛋白(Heterogeneous nuclear ribonulcleoprotein,hnRNP)家族成员,其存在于海马神经元的树突中并能够结合数千个RNA分子,参与miRNA代谢,对神经元的生长不可或缺[7]。植物的RNA结合蛋白在植物生长发育以及应激反应过程中发挥重要作用,目前对RNA结合蛋白在植物中的功能和作用机制的研究还比较少。前人对植物RBPs的研究主要集中在核编码的靶向叶绿体或线粒体的RBPs、富含甘氨酸的RBPs(GR-RBPs)以及三角状五肽重复区蛋白(PPR蛋白)。大量的研究表明植物RBPs作为重要的调控因子在RNA代谢、植物生长发育以及胁迫响应中发挥重要作用,从而引起人们对植物RBPs的广泛关注。本研究阐述了一些与植物(主要是拟南芥)生长发育以及胁迫响应相关的RBPs的功能、作用机制和相互联系,重点总结了5类RBP家族在植物生长发育和胁迫反应中的重要作用(表1和表2),包括SR蛋白、GR-RBPs、PPR蛋白、DEAD-box RHs以及RNA分子伴侣。
1 富含丝氨酸―精氨酸的RNA结合蛋白(SR蛋白)家族
SR蛋白家族在动植物中都是非常重要的一类RNA结合蛋白,并且在高等真核生物中高度保守。SR蛋白参与基因转录后调控的关键步骤,是前体mRNA组成型和选择性剪接的必需因子。SR蛋白的结构特征包括:N端有1个或2个RNA识别基序(RRM),C端是富含丝氨酸和精氨酸的二肽(dipeptide rich in serine and arginine,SR)结构域。研究较多的动物SR蛋白大多参与mRNA的剪接过程。与人类的12个SR蛋白相比,植物中SR蛋白的种类和数量较多一些,比如拟南芥和水稻中分别有19和22个SR蛋白,大豆具有25个SR蛋白,但是关于这些植物SR蛋白的研究还很少。目前,植物中的研究表明SR蛋白在植物的生长发育和胁迫响应中起着关键作用。
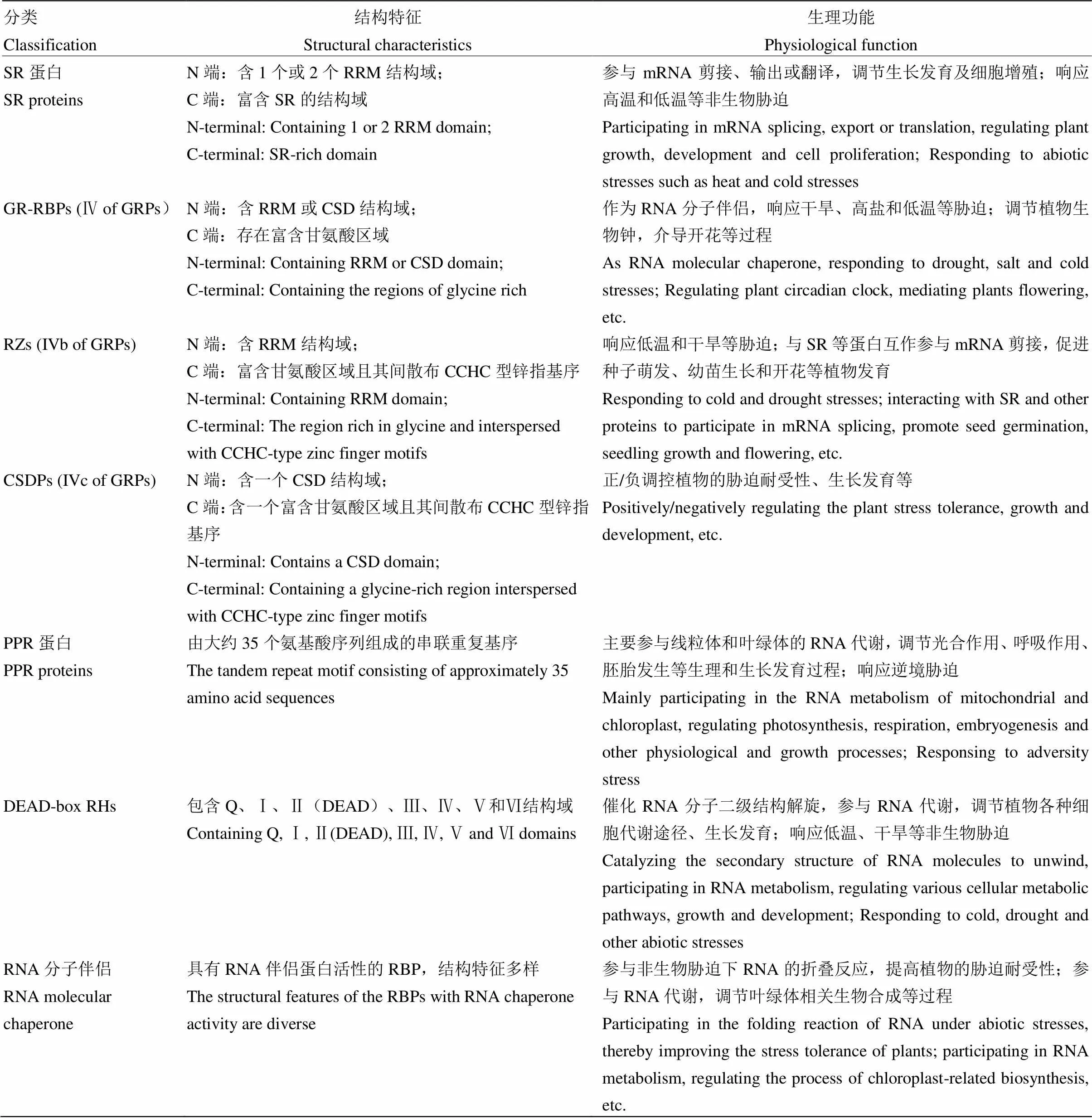
表1 植物RNA结合蛋白的分类、结构特征及主要功能
RRM:RNA识别基序;SR:富含丝氨酸和精氨酸的二肽;CSD:冷休克结构域
RRM: RNA recognition motif; SR: dipeptide rich in serine and arginine; CSD: cold shock domain
水稻SRp32、SRp33a、SRp33b、SCL26、RSZp23、RSZ36和RSZ37a通过参与mRNA的剪接、输出或翻译过程,调节水稻生长发育的各个方面[8];拟南芥RS40和RS41能够调节自身基因前体mRNA和其他SR基因的选择性剪接[9-10],AtRSZ33也具有类似的剪接模式。此外,AtRSZ33的异位表达会导致植物发育异常,比如加快细胞增殖、改变细胞伸长和分裂极性等[11],这表明植物SR蛋白可能通过mRNA代谢过程而实现对植物生长发育的调节。SR蛋白在RNA代谢中的可变性使其在非生物胁迫下具有较强的胁迫弹性,例如温度胁迫和激素处理均能改变SR蛋白的选择性剪接,如受激素影响的SR1、SR34b和SCL33等蛋白,这表明非生物胁迫和激素可能通过改变SR基因转录本的结构而影响它们的选择性剪接功能、剪接效率和靶向性[12]。
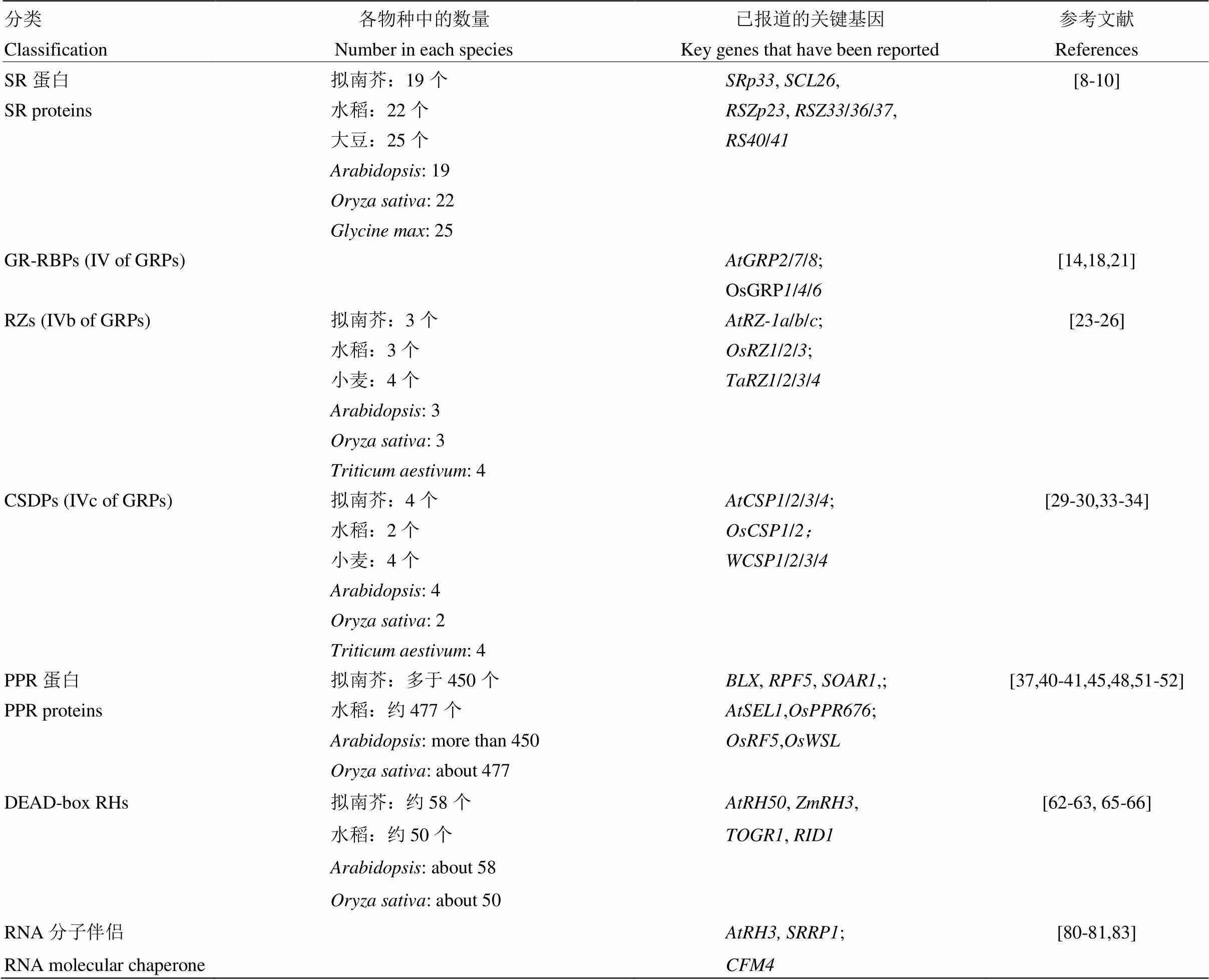
表2 植物RNA结合蛋白的分类、物种分布及研究现状
植物SR蛋白往往通过与同源或异源蛋白的相互作用来发挥重要的剪接功能。例如水稻RSp29和SCL26相互作用以增加mRNA的剪接效率;RSZ36和SRp33b相互作用以改变自身和其他SR蛋白的选择性剪接模式[8];RS40和RS41蛋白与含KH结构域的RNA结合蛋白HOS5等相互作用共同调节前体mRNA的加工[10]。目前,对植物SR蛋白的研究仍处于初级阶段,很多与之相关的功能和机制尚未得到解析,高等真核生物的SR蛋白具有较高的序列保守性,这或许是研究植物SR蛋白可有效利用的一种捷径。
2 富含甘氨酸的RNA结合蛋白(GR- RBPs)家族
植物中富含甘氨酸的蛋白质(GRPs)以高含量的甘氨酸为特征(20%—70%),共分为4类,其中只有第Ⅳ类GRPs(GR-RBPs)具有RNA结合功能,是RNA结合蛋白[13]。GR-RBPs在植物中广泛存在,参与调节植物生长发育和响应非生物胁迫过程。GR-RBPs的结构特征是在C-末端存在富含甘氨酸的区域,N-末端存在RNA识别基序(RRM)或冷休克结构域(CSD)。
拟南芥基因组编码十多种GR-RBPs,这些GR-RBPs在生长发育以及各种胁迫反应中发挥重要作用。具有RNA分子伴侣活性的和在水稻中的过表达有助于提高水稻的干旱耐受性和种子产量,并且其转基因植株对生长发育无任何副作用,因此,它们可以作为一种潜在的调节基因来改善干旱胁迫下的水稻生长发育[14]。AtGRP7能增强拟南芥对干旱和高盐的耐受性,它通过非ABA依赖型的方式调节气孔开度以控制水分蒸腾,从而介导植物的非生物胁迫反应[15]。此外,AtGRP7能够通过调节保卫细胞中的mRNA输出而提高植株的低温耐受性[15]。AtGRP7是丁香假单胞菌Ⅲ型效应物HopU1的直接靶标,其和AtGRP8不仅能提高拟南芥的免疫功能[16-17],而且还能作为生物钟调节器共同调节昼夜节律[18]。AtGRP7和AtGRP8在自主开花途径中能够调节植物开花,在突变体和反转义植株中开花阻遏蛋白(FLOWERING LOCUS C,FLC)被上调,并由此导致晚花表型。与此相反,过表达会降低转录,从而导致早花[19]。AtGRP2以非ABA依赖型的方式促进盐或冷胁迫下的种子萌发和幼苗生长,提高拟南芥盐和低温耐受性[20]。综上所述,拟南芥的GR-RBPs主要作为RNA分子伴侣在非生物胁迫响应中发挥重要作用,同时其可能通过介导植物激素信号通路而调节植物的生长发育。
水稻和拟南芥的GR-RBPs在功能上具有保守性,它们在植物响应低温过程中主要作为RNA分子伴侣发挥调节作用。3个水稻GR-RBP蛋白(OsGRP1、OsGRP4和OsGRP6)能够弥补冷休克期间低温敏感型大肠杆菌突变体的生长缺陷表型,以及恢复冷胁迫下拟南芥突变体的缺陷表型[21]。KIM等[21]研究发现OsGRP4和OsGRP6通过介导mRNA从细胞核向细胞质的运输过程来参与水稻的低温胁迫响应。
2.1 含有CCHC型锌指基序的GR-RBP—RZ家族
在GRPs家族的4类蛋白中,第Ⅳ类GRPs又分为a、b、c和d 4个不同的子类,含有CCHC型锌指基序的GRPs属于Ⅳb亚类(RZs家族)。RZs在N-末端含有RRM结构域,在C-末端有富含甘氨酸的区域且其间散布着CCHC型锌指基序。不同物种中的RZs在结构和功能上具有保守性,参与植物的生长发 育和胁迫响应过程。
拟南芥基因组编码3个RZ基因,分别被命名为、和。在低温胁迫下强烈上调,在干旱和脱落酸胁迫下则与之相反。在低温下AtRZ-1a促进种子萌发和幼苗生长,而其突变体中的上述过程明显受到抑制[22-23]。在盐或干旱胁迫条件下,AtRZ-1a则抑制拟南芥种子萌发和幼苗生长,说明AtRZ-1a的调节方式具有环境特异性[22-23]。AtRZ-1b和AtRZ-1c通过与其他蛋白如SR蛋白相互作用,调节前体mRNA剪接,在植物发育的许多方面发挥重要作用。和双突变植株表现出多种发育缺陷,如种子萌发和根生长延迟、晚花、少花、植株高度降低、叶尺寸减小以及锯齿状叶等[24]。转录组分析发现许多关键的发育调节基因和激素相关基因的表达水平在双突变体中发生改变,这是导致双突变体出现多种异常表型的原因。同时,AtRZ-1b和AtRZ-1c是内含子剪接所必须的,AtRZ-1b和AtRZ-1c促进第一个内含子的高效剪接并抑制转录,从而调节植物开花[24]。
水稻基因组也编码3个RZ基因(),其转录水平均受低温胁迫诱导,受干旱或盐胁迫的影响微弱。上述3个基因中只有能够提高拟南芥的低温耐受性,表明其功能存在一定差异[25]。小麦基因组编码4个RZ基因(),它们在非生物胁迫下植物的种子萌发、幼苗生长和低温耐受性方面发挥不同功能。盐胁迫下,过表达的3种转基因拟南芥的种子萌发均被抑制;干旱胁迫下,过表达或的转基因拟南芥种子萌发受到抑制;过表达的转基因拟南芥幼苗生长在低温或盐胁迫下被显著抑制,而过表达则提高了拟南芥的低温耐受性[26]。由此推测不同物种的RZs在参与植物的生长发育以及胁迫反应过程中,存在一定的功能保守性。
2.2 含有冷休克结构域的GR-RBPs—CSDPs家族
CSDPs家族属于GR-RBPs的Ⅳc亚类。冷休克结构域(cold shock domain,CSD)是与RNA、ssDNA和dsDNA结合有关的高度保守的核酸结合结构域,在细菌、动物和植物中广泛存在。细菌冷休克蛋白由单一的CSD组成,在低温下被诱导,并在低温胁迫反应过程中发挥重要作用[27]。植物CSD蛋白的结构特征是在N端含有一个CSD,C端含有一个富含甘氨酸的区域,并在富含甘氨酸的区域中散布着不同数量的CCHC型锌指基序[28]。CSDPs同样在植物发育和胁迫响应中发挥重要作用。
拟南芥中存在4种CSD蛋白,分别为AtCSP1、AtCSP2、AtCSP3和AtCSP4。其中,AtCSP2表达量最高,AtCSP3是目前研究最深入的,AtCSP2与AtCSP4的同源性最高,且在某些方面具有功能冗余。AtCSP1在干旱或盐胁迫下植物的种子萌发和生长中发挥作用[29]。AtCSP2通过CBF依赖性途径负调节植物的低温耐受性,其过表达导致植物的低温耐受性降低,此外在低温耐受性和开花时间的调控方面与AtCSP4存在功能冗余。另外,过表达导致植株的形态和生长发育发生改变,如植株矮小,开花延迟和果荚变短[30],AtCSP2还能够通过调节GA和ABA代谢基因的表达来负调控种子萌发[28]。AtCSP3参与植物盐和干旱胁迫耐受性的调控,过表达能够显著提高盐和干旱胁迫下的植株存活率[31]。AtCSP4通过调节与发育相关的基因表达而在长角果发育的晚期阶段起重要作用,过表达导致MADS-box和胚乳发育基因的表达在花和果实发育过程中发生异常改变,转基因植株表现出长角果缩短和种子成熟度低等缺陷表型[32]。
小麦也编码4种CSD蛋白(WCSP1—WCSP4),其中WCSP1是第一个被发现的CSD蛋白,WCSP1能赋予低温敏感型大肠杆菌较强的耐受性[33]。水稻编码2种CSD蛋白,分别为OsCSP1和OsCSP2,它们表现出核酸结合活性并改善了大肠杆菌突变体的低温敏感性[34]。此外,小麦WCSPs和水稻OsCSPs在植物发育中也发挥重要作用[33-34],但仍未见相关的研究报道。目前,关于植物CSD蛋白的研究还很少,虽然在植物中已经鉴定出许多CSD蛋白,但对它们功能的解析程度还不够深入。
3 三角状五肽重复区蛋白(PPR蛋白)家族
PPR蛋白是一种序列特异性RNA结合蛋白,它们以序列特异性方式结合RNA,是目前研究最深入和最重要的一类RNA结合蛋白之一。PPR蛋白家族成员是mRNA转录后靶向细胞器的关键调控者,主要参与线粒体和叶绿体的RNA代谢过程。PPR蛋白几乎涉及植物细胞器RNA代谢的所有方面,包括RNA编辑、RNA剪接、RNA稳定以及翻译过程。PPR蛋白由大约35个氨基酸序列经串联重复组成,并且随后折叠成一对反向平行的α螺旋。PPR蛋白主要被分为2个亚类,P类和PLS类。P类PPR蛋白具有规范的35个氨基酸基序,但有些除了PPR基序外,还会在末端存在额外的结构域,以行使特异性功能;而PLS类的PPR蛋白具有3种不同类型的PPR基序,其长度各不相同:P(35个氨基酸)、L(35—36个氨基酸)和S(约31个氨基酸)。PLS类PPR蛋白一般会包含额外的结构域,根据这些结构域的不同,又分为不同的亚家族,包括PLS、E1、E2、E+和DYW等家族,不同类型的PPR蛋白可能具有不同的功能[35-36]。PPR蛋白主要存在于植物中,在其他真核生物中较少,人类基因组中只有不到10个PPR蛋白。在植物中,拟南芥基因组含有超过450个PPR蛋白,水稻中大约有477个,除这两类外,其他物种如玉米中含量相对较少。PPR蛋白在细胞器RNA加工、CMS植物的育性恢复、光合作用、呼吸作用、胚胎发生、植物发育以及胁迫响应过程中发挥重要作用。目前已经有部分植物PPR蛋白被报道,但仍有大部分PPR蛋白的分子功能未得到解析。下面主要总结了近几年关于拟南芥、水稻和玉米PPR蛋白的功能研究进展。
在拟南芥中,靶向线粒体的PPR蛋白BLX[37]、AtGRS1[38]和SLO4[39]参与调节线粒体内含子的剪接,并对植物早期胚胎发育和植株生长发挥重要作用。还有一些靶向线粒体的 PPR蛋白,它们不仅在RNA代谢过程中发挥重要作用,而且在改善细胞质雄性不育和逆境胁迫等方面也具有重要功能。如RPF5蛋白除了参与特定mRNA的成熟过程外,也能够部分恢复细胞质雄性不育系的育性[40]。SOAR1是拟南芥响应非生物胁迫重要的正调控蛋白,过表达能显著提高植物的干旱、盐和低温的胁迫耐受性,并且不会影响植物的正常生长和发育。SOAR1还是ABA信号转导的关键负调控因子,通过整合ABA依赖型和非依赖型的信号传导途径来调节植物的应激反应[41-42]。AtSLG1在RNA编辑、植物生长发育、ABA处理和干旱胁迫反应中发挥重要作用[43]。AtPGN在植物响应逆境胁迫过程中发挥重要作用。突变体表现出对坏死性真菌病原体的易感性以及对脱落酸、葡萄糖和盐胁迫的超敏感性。基因功能研究揭示PGN在胁迫反应过程中通过调节线粒体的活性氧动态平衡来发挥作用[44]。靶向叶绿体的拟南芥PPR蛋白,它们除了在叶绿体内含子剪接过程中起至关重要的作用外,在生长发育以及光合作用中也发挥重要作用。如AtSEL1参与叶绿体发育所需的质体基因的表达调控,在突变体中,光合作用的相关蛋白减少且叶绿体发育受损[45];AtPPR2在胚胎发育的第一次有丝分裂和细胞增殖过程中发挥重要作用[46];此外还有叶绿体发育及光合作用所必须的SVR7蛋白,其基因突变体()显示叶绿体ATP合酶亚基的积累受到影响。综上所述,拟南芥PPR蛋白在细胞器mRNA代谢、植物生长发育以及胁迫反应等方面发挥关键作用[47]。
在水稻中,许多PPR蛋白除了具有基本的RNA编辑功能外,还参与植物发育的各个方面,如花粉发育、幼苗生长和叶绿体发育,并且在改善细胞质雄性不育和非生物胁迫等方面也发挥重要作用。如OsPPR676蛋白是水稻生长和花粉发育所必需的,OsPPR676对于质体atpB亚基的产生是必不可少的,其通过影响ATP合酶的活性在脂肪酸、碳水化合物以及其他有机物的生物合成中发挥关键作用[48]。OsPPR6参与叶绿体中与光合作用相关的基因转录物的剪接并调节早期叶绿体的生物合成,是水稻叶绿体生物合成所必需的调节因子[49]。OsPGL1通过参与线粒体和叶绿体中特定位点的RNA编辑而调节相关的生物过程[50]。OsRF5通过与富含甘氨酸的蛋白质GRP162相互作用而恢复红莲型细胞质雄性不育系的育性[51]。OsWSL[52]和OsWSL4[53]是叶绿体重要的剪接因子,影响水稻早期叶片发育,且OsWSL能提高植物对ABA、盐和糖胁迫的耐受性。OsV4在低温胁迫下早期的叶绿体发育中起重要作用[54]。
近期发现玉米PPR蛋白在基因功能上存在一定的相似性。玉米PPR蛋白的种类较少,且大多靶向线粒体。如靶向线粒体的EMP10[55]、EMP11[56]、DEK10[57]和DEK35[58]蛋白对线粒体内mRNA的不同位点进行RNA编辑和剪接,是维持线粒体功能和胚胎、胚乳发育不可或缺的一类PPR蛋白。另外,PPR78[59]和PPR2263[60]对线粒体mRNA进行编辑和剪接,对线粒体发挥功能和种子发育具有重要作用。近几年,尽管已经鉴定出许多植物PPR蛋白,但由于植物中PPR蛋白本身基数较大的缘故,还有很多发挥重要功能的PPR蛋白未被研究。此外,目前关于PPR蛋白作用的详细地分子机制还不清楚,需要进一步深入探究。
4 DEAD-box RNA解旋酶(DEAD-box RHs)家族
RNA解旋酶(RH)是能够改变RNA结构的酶。RHs分为6个超家族(SF1—SF6),其中超家族Ⅱ(SF2)是最大的解旋酶家族,主要由DEAD-box RHs组成,DEAD-box RHs具有RNA结合活性,是RNA结合蛋白。DEAD-box RHs可以催化RNA分子中二级结构的解旋,从而影响有机体中RNA代谢过程,它们在调节各种细胞代谢途径中发挥重要作用。DEAD-box RNA解旋酶由Q、Ⅰ、Ⅱ(DEAD)、Ⅲ、Ⅳ、Ⅴ和Ⅵ结构域组成,这些结构域具有不同的功能,有的具有解旋功能,有的具有RNA结合活性,有的具有其他特异性功能。在真核生物中,植物编码的DEAD-box RHs更多,拟南芥编码约58个,水稻约50个,人类基因组中约有36个。目前植物中的DEAD-box RHs研究主要集中在拟南芥和水稻上,DEAD-box RHs除了在RNA代谢方面发挥重要作用外,在植物生长发育和非生物胁迫中也行使重要功能。
4.1 DEAD-box RHs在RNA代谢中起着至关重要的作用
大多数DEAD-box RHs在RNA代谢过程中发挥作用,这些DEAD-box RHs不仅是细胞核RNA代谢中必不可少的调节因子,有些也参与线粒体和叶绿体的RNA代谢过程。如AtRH9参与线粒体mRNA中Ⅱ组内含子的剪接[61];AtRH50参与叶绿体23S和4.5S rRNA的加工[62];ZmRH3参与rRNA的生物合成以及叶绿体中mRNA的剪接[63];RCF1[64]和RID1[65]是拟南芥前体mRNA必要的剪接因子;水稻TOGR1[66]在高温下能维持rRNA的稳定性。综上所述,DEAD-box RHs在RNA代谢中起着重要作用。
4.2 DEAD-box RHs在植物生长发育和非生物胁迫反应中具有不同作用
许多DEAD-box RHs在植物生长发育过程中也具有重要作用。如RID1在配子体发育过程中发挥重要作用[65];AtRH57参与植物种子萌发和幼苗生长过程中葡萄糖和脱落酸信号交联的糖应答过程[67];拟南芥AtRH36[68]和水稻OsRH36[69]参与雌配子的有丝分裂过程,它们在功能上具有保守性;OsRH2和OsRH34参与调控水稻株高、花粉和种子发育[70],综上所述,DEAD-box RHs在植物生长发育的多个过程中发挥重要的调节作用。
DEAD-box RHs在非生物胁迫中的潜在作用也日益被发现。如RCF1参与植物低温感应基因的调控并增加植株的低温耐受性[64];AtRH7参与植物发育和拟南芥的低温耐受性[71];过表达促进了水稻在高温胁迫下的生长[66];靶向线粒体的拟南芥AtRH9和AtRH25的过表达抑制了高盐浓度条件下的种子发芽,其中的过表达也能增强植株的低温耐受性[72];RH8与PP2CA相互作用,通过ABA依赖型信号传导来调节干旱胁迫反应[73];叶绿体靶向的BrRH22在高盐和干旱胁迫下促进拟南芥种子发芽和植株生长[74]。以上结果表明DEAD-box RHs在非生物胁迫反应中扮演关键作用。尽管DEAD-box RHs在植物中已取得较大进展,但仍有很多DEAD-box RHs未被鉴定,此外关于DEAD-box RHs参与的信号转导调控网络还不清楚,因此,未来还要进一步确定它的RNA靶标及其与其他调节信号的交联机制。
5 RNA分子伴侣
同蛋白质一样,RNA分子需要正确地折叠成相应的结构才能发挥正常功能,然而RNA分子由于其固有的动力学和热力学折叠问题而容易错误折叠成非功能性结构。RNA分子伴侣是非特异性的RNA结合蛋白,通过对错误折叠的RNA进行结构重排来促进RNA的正确折叠,以保证RNA代谢过程的顺利进行[75]。RNA分子伴侣在生物体如细菌、病毒、动物和植物的生长和发育中起着关键作用,其中对RNA分子伴侣在植物中的研究远远落后于其他3类生物。许多富含甘氨酸的RNA结合蛋白(GR-RBPs)以及DEAD-box RNA解旋酶(DEAD-box RHs)家族成员具有RNA分子伴侣活性,它们在植物生长发育和胁迫反应中发挥重要作用,并且有些已被确定在逆境胁迫下作为RNA分子伴侣起作用。
具有RNA分子伴侣活性的RNA结合蛋白参与植物胁迫响应。当细胞暴露于低温时,RNA分子伴侣的作用更加突出,错误折叠的RNA分子在低温下变得过度稳定,在缺少RNA分子伴侣的帮助下不能呈现天然构象。研究证明GR-RBPs的某些家族成员在低温反应期间表现出RNA分子伴侣活性,如拟南芥的AtGRP2[20]和AtGRP7[21];水稻的OsGRP1、OsGRP4和OsGRP6[21]等蛋白。另外,在GR-RBPs家族中,含有CCHC型锌指基序的GR-RBPs(RZs)成员也被发现具有RNA分子伴侣活性,如拟南芥AtRZ-1a[76]、水稻OsRZ2[25]和小麦TaRZ2[77]等蛋白。GR-RBPs家族的CSDP1[78]在提高拟南芥低温耐受性方面也起RNA分子伴侣作用。以上结果表明具有RNA分子伴侣活性的GR-RBPs家族成员在植物胁迫反应中发挥重要作用。在细胞响应低温过程中,GR-RBPs的结构域序列和总体折叠方式对于GR-RBPs的RNA分子伴侣活性至关重要[79]。DEAD-box RHs家族的某些成员在植物胁迫反应中也具有RNA分子伴侣活性,如低温胁迫下的AtRH25蛋白[72];另外由具有RNA分子伴侣活性的拟南芥AtRH3 所调节的内含子剪接对于叶绿体功能、植物的生长和胁迫反应是至关重要的[80]。
RNA分子伴侣在植物的生长和发育中起着关键作用,尤其是一些靶向叶绿体的RNA分子伴侣。如靶向叶绿体的SRRP1影响拟南芥叶绿体转录物的剪接和加工[81];靶向叶绿体的SDP影响rRNA加工、叶绿体生物合成及光合作用[82],这些对拟南芥的正常生长是至关重要的;还有CFM4通过参与叶绿体rRNA的加工在拟南芥生长和应激反应中发挥作用[83]。综上所述,在植物生长和发育过程中,许多具有RNA分子伴侣活性的RNA结合蛋白在RNA代谢的调节过程中发挥重要作用。目前,已有许多证据表明RNA分子伴侣在植物生长发育以及胁迫反应中发挥重要作用,但关于RNA分子伴侣的研究还不够深入,接下来不仅要继续挖掘更多具有RNA分子伴侣活性的RBPs,还要研究RNA分子伴侣如何识别底物RNA以及它们如何与其他蛋白协作共同参与转录后的RNA代谢过程,从而调节植物生长发育并响应环境胁迫。
6 结论与展望
植物中的RBPs作为重要的转录后调控因子,在RNA代谢、植物生长发育以及胁迫反应过程中发挥重要作用。在本文所论述的五类植物RBP家族中,SR蛋白主要作为重要的选择性剪接因子在植物的生长发育和胁迫响应中起着关键作用;GR-RBPs家族的成员普遍具有功能多样性,其可能通过参与介导植物激素信号通路调节植物生长发育的多个方面,并主要作为RNA分子伴侣在多种胁迫反应中发挥重要作用;PPR蛋白主要参与线粒体和叶绿体的RNA代谢,调节植物生长发育以及胁迫反应过程;DEAD-box RHs可作为细胞核和细胞器重要的RNA剪接因子,并在植物生长发育以及非生物胁迫反应中发挥多种功能;RNA分子伴侣作为非特异性的RNA结合蛋白,通过参与RNA折叠反应保证RNA代谢过程的顺利进行,此外,许多RBPs具有RNA分子伴侣活性,它们作为RNA分子伴侣调节植物的生长发育以及胁迫反应。总的来说,上述五类RNA结合蛋白在植物中的作用集中体现在RNA代谢和RNA折叠反应方面,并通过这两个方面的作用,影响相关基因的表达,从而调节胚胎发育、幼苗生长等生长发育以及响应干旱、低温等逆境胁迫。
近年来,关于RBPs在植物生长发育和胁迫反应中的研究正在迅速增加,目前,关于SR蛋白以及RNA分子伴侣的研究还很少,对于相对研究较多的GR-RBPs和PPR蛋白来说,应进一步深入确定它们的RNA靶标及其参与的信号转导调控网络。很多RBPs在高等真核生物中具有较强的保守性,因此,可以利用植物中的某些蛋白与动物RNA结合蛋白的同源性去寻找植物中具有潜在功能的RBPs。此外,随着近年来转录组测序技术的发展使我们能够获得植物在某一生理状态下的转录组信息,从而通过对RNA结合蛋白突变体或逆境胁迫中的转录组进行组学分析,预测RNA结合蛋白的潜在功能和可能参与的信号调控网络。未来关于RBPs的研究应该更加系统和全面化,除了继续挖掘更多的具有重要功能的RBPs外,还应关注RBPs作用的RNA靶标,探究RBPs识别底物RNA的生化机制,以及RBPs与其他调控因子交联而共同调节植物生命活动的信号调控网络。
[1] MITCHELL S F, PARKER R. Principles and properties of eukaryotic mRNPs., 2014, 54(4): 547-558.
[2] SINGH G, PRATT G, YEO G W, MOORE M J. The Clothes Make the mRNA: Past and present trends in mRNP fashion., 2015, 84: 325-354.
[3] LORKOVIC Z J, BARTA A. Genome analysis: RNA recognition motif (RRM) and K homology (KH) domain RNA-binding proteins from the flowering plant., 2002, 30(3): 623-635.
[4] LEE K, KANG H. Emerging roles of RNA-binding proteins in plant growth, development, and stress responses., 2016, 39(3): 179-185.
[5] GLISOVIC T, BACHORIK J L, YONG J, DREYFUSS G. RNA-binding proteins and post-transcriptional gene regulation., 2008, 582(14): 1977-1986.
[6] XUE J Q, XIA T S, LIANG X Q, ZHOU W, CHENG L, SHI L, WANG Y, DING Q. RNA-binding protein RNPC1: acting as a tumor suppressor in breast cancer., 2014, 14: 322.
[7] SEPHTON C F, YU G. The function of RNA-binding proteins at the synapse: implications for neurodegeneration., 2015, 72(19): 3621-3635.
[8] ISSHIKI M, TSUMOTO A, SHIMAMOTO K. The serine/ arginine-rich protein family in rice plays important roles in constitutive and alternative splicing of pre-mRNA., 2006, 18(1): 146-158.
[9] KALYNA M, SIMPSON C G, SYED N H, LEWANDOWSKA D, MARQUEZ Y, KUSENDA B, MARSHALL J, FULLER J, CARDLE L, MCNICOL J, DINH H Q, BARTA A, BROWN J W. Alternative splicing and nonsense-mediated decay modulate expression of important regulatory genes in., 2012, 40(6): 2454-2469.
[10] CHEN T, CUI P, CHEN H, ALI S, ZHANG S, XIONG L. A KH-domain RNA-binding protein interacts with FIERY2/CTD phosphatase-like 1 and splicing factors and is important for pre-mRNA splicing in., 2013, 9(10): e1003875.
[11] KALYNA M, LOPATO S, BARTA A. Ectopic expression ofreveals its function in splicing and causes pleiotropic changes in development., 2003, 14(9): 3565-3577.
[12] PALUSA S G, ALI G S, REDDY A S. Alternative splicing of pre-mRNAs ofserine/arginine-rich proteins: regulation by hormones and stresses., 2007, 49(6): 1091-1107.
[13] LEWINSKI M, HALLMANN A, STAIGER D. Genome-wide identification and phylogenetic analysis of plant RNA binding proteins comprising both RNA recognition motifs and contiguous glycine residues., 2016, 291(2): 763-773.
[14] YANG D H, KWAK K J, KIM M K, PARK S J, YANG K Y, KANG H. Expression ofglycine-rich RNA-binding protein AtGRP2 or AtGRP7 improves grain yield of rice () under drought stress conditions., 2014, 214: 106-112.
[15] KIM J S, JUNG H J, LEE H J, KIM K A, GOH C H, WOO Y, OH S H, HAN Y S, KANG H. Glycine-rich RNA-binding protein 7 affects abiotic stress responses by regulating stomata opening and closing in., 2008, 55(3): 455-466.
[16] NICAISE V, JOE A, JEONG B R, KORNELI C, BOUTROT F, WESTEDT I, STAIGER D, ALFANO J R, ZIPFEL C. Pseudomonas HopU1 modulates plant immune receptor levels by blocking the interaction of their mRNAs with GRP7., 2013, 32(5): 701-712.
[17] FU Z Q, GUO M, JEONG B R, TIAN F, ELTHON T E, CERNY R L, STAIGER D, ALFANO J R. A type III effector ADP-ribosylates RNA-binding proteins and quells plant immunity., 2007, 447(7142): 284-288.
[18] SCHMAL C, REIMANN P, STAIGER D. A circadian clock-regulated toggle switch explains AtGRP7 and AtGRP8 oscillations in., 2013, 9(3): e1002986.
[19] STREITNER C, DANISMAN S, WEHRLE F, SCHONING J C, ALFANO J R, STAIGER D. The small glycine-rich RNA binding protein AtGRP7 promotes floral transition in., 2008, 56(2): 239-250.
[20] KIM J Y, PARK S J, JANG B, JUNG C H, AHN S J, GOH C H, CHO K, HAN O, KANG H. Functional characterization of a glycine-rich RNA-binding protein 2 inunder abiotic stress conditions., 2007, 50(3): 439-451.
[21] KIM J Y, KIM W Y, KWAK K J, OH S H, HAN Y S, KANG H. Glycine-rich RNA-binding proteins are functionally conserved inandduring cold adaptation process., 2010, 61(9): 2317-2325.
[22] KIM Y O, KIM J S, KANG H. Cold-inducible zinc finger-containing glycine-rich RNA-binding protein contributes to the enhancement of freezing tolerance in., 2005, 42(6): 890-900.
[23] KIM Y O, PAN S, JUNG C H, KANG H. A zinc finger-containing glycine-rich RNA-binding protein, atRZ-1a, has a negative impact on seed germination and seedling growth ofunder salt or drought stress conditions., 2007, 48(8): 1170-1181.
[24] WU Z, ZHU D, LIN X, MIAO J, GU L, DENG X, YANG Q, SUN K, ZHU D, CAO X, TSUGE T, DEAN C, AOYAMA T, GU H, QU L J. RNA binding proteins RZ-1B and RZ-1C play critical roles in regulating Pre-mRNA splicing and gene expression during development in., 2016, 28(1): 55-73.
[25] KIM J Y, KIM W Y, KWAK K J, OH S H, HAN Y S, KANG H. Zinc finger-containing glycine-rich RNA-binding protein inhas an RNA chaperone activity under cold stress conditions., 2010, 33(5): 759-768.
[26] XU T, GU L, CHOI M J, KIM R J, SUH M C, KANG H. Comparative functional analysis of wheat () zinc finger- containing glycine-rich RNA-binding proteins in response to abiotic stresses., 2014, 9(5): e96877.
[27] GRAUMANN P L, MARAHIEL M A. A superfamily of proteins that contain the cold-shock domain., 1998, 23(8): 286-290.
[28] SASAKI K, KIM M H, KANNO Y, SEO M, KAMIYA Y, IMAI R.cold shock domain protein 2 influences ABA accumulation in seed and negatively regulates germination., 2015, 456(1): 380-384.
[29] PARK S J, KWAK K J, OH T R, KIM Y O, KANG H. Cold shock domain proteins affect seed germination and growth ofunder abiotic stress conditions., 2009, 50(4): 869-878.
[30] SASAKI K, KIM M H, IMAI R. Arabidopsis COLD SHOCK DOMAIN PROTEIN 2 is a negative regulator of cold acclimation., 2013, 198(1): 95-102.
[31] KIM M H, SATO S, SASAKI K, SABURI W, MATSUI H, IMAI R. COLD SHOCK DOMAIN PROTEIN 3 is involved in salt and drought stress tolerance in., 2013, 3: 438-442.
[32] YANG Y, KARLSON D T. Overexpression ofaffects late stages of embryo development in., 2011, 62(6): 2079-2091.
[33] RADKOVA M, VITAMVAS P, SASAKI K, IMAI R. Development- and cold-regulated accumulation of cold shock domain proteins in wheat., 2014, 77: 44-48.
[34] CHAIKAM V, KARLSON D. Functional characterization of two cold shock domain proteins from., 2008, 31(7): 995-1006.
[35] MANNA S. An overview of pentatricopeptide repeat proteins and their applications., 2015, 113: 93-99.
[36] XING H, FU X, YANG C, TANG X, GUO L, LI C, XU C, LUO K. Genome-wide investigation of pentatricopeptide repeat gene family in poplar and their expression analysis in response to biotic and abiotic stresses., 2018, 8(1): 2817.
[37] SUN Y, HUANG J, ZHONG S, GU H, HE S, QU L J. Novel DYW-type pentatricopeptide repeat (PPR) protein BLX controls mitochondrial RNA editing and splicing essential for early seed development of., 2018, 45(3): 155-168.
[38] XIE T, CHEN D, WU J, HUANG X, WANG Y, TANG K, LI J, SUN M, PENG X. Growing slowly 1 locus encodes a PLS-type PPR protein required for RNA editing and plant development in., 2016, 67(19): 5687-5698.
[39] WEISSENBERGER S, SOLL J, CARRIE C. The PPR protein SLOW GROWTH 4 is involved in editing of nad4 and affects the splicing of nad2 intron 1., 2017, 93(4/5): 355-368.
[40] HAULER A, JONIETZ C, STOLL B, STOLL K, BRAUN H P, BINDER S. RNA processing factor 5 is required for efficient 5' cleavage at a processing site conserved in RNAs of three different mitochondrial genes in., 2013, 74(4): 593-604.
[41] JIANG S C, MEI C, LIANG S, YU Y T, LU K, WU Z, WANG X F, ZHANG D P. Crucial roles of the pentatricopeptide repeat protein SOAR1 inresponse to drought, salt and cold stresses., 2015, 88(4/5): 369-385.
[42] MEI C, JIANG S C, LU Y F, WU F Q, YU Y T, LIANG S, FENG X J, PORTOLES COMERAS S, LU K, WU Z, WANG X F, ZHANG D P.pentatricopeptide repeat protein SOAR1 plays a critical role in abscisic acid signalling., 2014, 65(18): 5317-5330.
[43] YUAN H, LIU D. Functional disruption of the pentatricopeptide protein SLG1 affects mitochondrial RNA editing, plant development, and responses to abiotic stresses in., 2012, 70(3): 432-444.
[44] LALUK K, ABUQAMAR S, MENGISTE T. Themitochondria-localized pentatricopeptide repeat protein PGN functions in defense against necrotrophic fungi and abiotic stress tolerance., 2011, 156(4): 2053-2068.
[45] PYO Y J, KWON K C, KIM A, CHO M H. Seedling Lethal1, a pentatricopeptide repeat protein lacking an E/E+ or DYW domain in, is involved in plastid gene expression and early chloroplast development., 2013, 163(4): 1844-1858.
[46] LU Y, LI C, WANG H, CHEN H, BERG H, XIA Y. AtPPR2, anpentatricopeptide repeat protein, binds to plastid 23S rRNA and plays an important role in the first mitotic division during gametogenesis and in cell proliferation during embryogenesis., 2011, 67(1): 13-25.
[47] ZOSCHKE R, QU Y, ZUBO Y O, BORNER T, SCHMITZ- LINNEWEBER C. Mutation of the pentatricopeptide repeat-SMR protein SVR7 impairs accumulation and translation of chloroplast ATP synthase subunits in., 2013, 126(3): 403-414.
[48] LIU Y J, LIU X, CHEN H, ZHENG P, WANG W, WANG L, ZHANG J, TU J. A plastid-localized pentatricopeptide repeat protein is required for both pollen development and plant growth in rice., 2017, 7(1): 11484.
[49] TANG J, ZHANG W, WEN K, CHEN G, SUN J, TIAN Y, TANG W, YU J, AN H, WU T, KONG F, TERZAGHI W, WANG C, WAN J. OsPPR6, a pentatricopeptide repeat protein involved in editing and splicing chloroplast RNA, is required for chloroplast biogenesis in rice., 2017, 95(4/5): 345-357.
[50] XIAO H, XU Y, NI C, ZHANG Q, ZHONG F, HUANG J, LIU W, PENG L, ZHU Y, HU J. A rice dual-localized pentatricopeptide repeat protein is involved in organellar RNA editing together with OsMORFs., 2018, 69 (12): 2923-2936.
[51] HU J, WANG K, HUANG W, LIU G, GAO Y, WANG J, HUANG Q, JI Y, QIN X, WAN L, ZHU R, LI S, YANG D, ZHU Y. The rice pentatricopeptide repeat protein RF5 restores fertility in Hong-Lian cytoplasmic male-sterile lines via a complex with the glycine-rich protein GRP162., 2012, 24(1): 109-122.
[52] TAN J, TAN Z, WU F, SHENG P, HENG Y, WANG X, REN Y, WANG J, GUO X, ZHANG X, CHENG Z, JIANG L, LIU X, WANG H, WAN J. A novel chloroplast-localized pentatricopeptide repeat protein involved in splicing affects chloroplast development and abiotic stress response in rice., 2014, 7(8): 1329-1349.
[53] WANG Y, REN Y, ZHOU K, LIU L, WANG J, XU Y, ZHANG H, ZHANG L, FENG Z, WANG L, MA W, WANG Y, GUO X, ZHANG X, LEI C, CHENG Z, WAN J. WHITE STRIPE LEAF4 encodes a novel p-type PPR protein required for chloroplast biogenesis during early leaf development., 2017, 26 (8): 1116.
[54] GONG X, SU Q, LIN D, JIANG Q, XU J, ZHANG J, TENG S, DONG Y. The riceencoding a novel pentatricopeptide repeat protein is required for chloroplast development during the early leaf stage under cold stress., 2014, 56(4): 400-410.
[55] CAI M, LI S, SUN F, SUN Q, ZHAO H, REN X, ZHAO Y, TAN B C, ZHANG Z, QIU F.encodes a mitochondrial PPR protein that affects the cis-splicing of nad2 intron 1 and seed development in maize., 2017, 91(1): 132-144.
[56] REN X, PAN Z, ZHAO H, ZHAO J, CAI M, LI J, ZHANG Z, QIU F. EMPTY PERICARP11 serves as a factor for splicing of mitochondrial nad1 intron and is required to ensure proper seed development in maize., 2017, 68(16): 4571-4581.
[57] QI W, TIAN Z, LU L, CHEN X, CHEN X, ZHANG W, SONG R. Editing of mitochondrial transcripts nad3 and cox2 by Dek10 is essential for mitochondrial function and maize plant development., 2017, 205(4): 1489-1501.
[58] CHEN X, FENG F, QI W, XU L, YAO D, WANG Q, SONG R.encodes a PPR protein that affects cis-splicing of mitochondrial nad4 Intron 1 and seed development in maize., 2017, 10(3): 427-441.
[59] ZHANG Y F, SUZUKI M, SUN F, TAN B C. The mitochondrion- targeted PENTATRICOPEPTIDE REPEAT78 protein is required for nad5 mature mRNA stability and seed development in maize., 2017, 10(10): 1321-1333.
[60] SOSSO D, MBELO S, VERNOUD V, GENDROT G, DEDIEU A, CHAMBRIER P, DAUZAT M, HEURTEVIN L, GUYON V, TAKENAKA M, ROGOWSKY P M. PPR2263, a DYW-Subgroup Pentatricopeptide repeat protein, is required for mitochondrial nad5 and cob transcript editing, mitochondrion biogenesis, and maize growth., 2012, 24(2): 676-691.
[61] KOHLER D, SCHMIDT-GATTUNG S, BINDER S. The DEAD-box protein PMH2 is required for efficient group II intron splicing in mitochondria of., 2010, 72(4/5): 459-467.
[62] PAIERI F, TADINI L, MANAVSKI N, KLEINE T, FERRARI R, MORANDINI P, PESARESI P, MEURER J, LEISTER D. The DEAD-box RNA helicase RH50 is a 23S-4.5S rRNA maturation factor that functionally overlaps with the plastid signaling factor GUN1., 2018, 176(1): 634-648.
[63] ASAKURA Y, GALARNEAU E, WATKINS K P, BARKAN A, VAN WIJK K J. Chloroplast RH3 DEAD box RNA helicases in maize andfunction in splicing of specific group II introns and affect chloroplast ribosome biogenesis., 2012, 159(3): 961-974.
[64] GUAN Q, WU J, ZHANG Y, JIANG C, LIU R, CHAI C, ZHU J. A DEAD box RNA helicase is critical for pre-mRNA splicing, cold-responsive gene regulation, and cold tolerance in., 2013, 25(1): 342-356.
[65] OHTANI M, DEMURA T, SUGIYAMA M.root initiation defective1, a DEAH-box RNA helicase involved in pre-mRNA splicing, is essential for plant development., 2013, 25(6): 2056-2069.
[66] WANG D, QIN B, LI X, TANG D, ZHANG Y, CHENG Z, XUE Y. Nucleolar DEAD-Box RNA helicase TOGR1 regulates thermotolerant growth as a Pre-rRNA chaperone in rice., 2016, 12(2): e1005844.
[67] HSU Y F, CHEN Y C, HSIAO Y C, WANG B J, LIN S Y, CHENG W H, JAUH G Y, HARADA J J, WANG C S. AtRH57, a DEAD-box RNA helicase, is involved in feedback inhibition of glucose-mediated abscisic acid accumulation during seedling development and additively affects pre-ribosomal RNA processing with high glucose., 2014, 77(1): 119-135.
[68] HUANG C K, HUANG L F, HUANG J J, WU S J, YEH C H, LU C A. A DEAD-box protein, AtRH36, is essential for female gametophyte development and is involved in rRNA biogenesis in., 2010, 51(5): 694-706.
[69] HUANG C K, YU S M, LU C A. A rice DEAD-box protein, OsRH36, can complement anmutant, but cannot functionally replace its yeast homolog Dbp8p., 2010, 74(1/2): 119-128.
[70] HUANG C K, SIE Y S, CHEN Y F, HUANG T S, LU C A. Two highly similar DEAD box proteins, OsRH2 and OsRH34, homologous to eukaryotic initiation factor 4AIII, play roles of the exon junction complex in regulating growth and development in rice., 2016, 16: 84.
[71] HUANG C K, SHEN Y L, HUANG L F, WU S J, YEH C H, LU C A. The DEAD-Box RNA helicase AtRH7/PRH75 participates in Pre-rRNA processing, plant development and cold tolerance in., 2016, 57(1): 174-191.
[72] KIM J S, KIM K A, OH T R, PARK C M, KANG H. Functional characterization of DEAD-box RNA helicases inunder abiotic stress conditions., 2008, 49(10): 1563-1571.
[73] BAEK W, LIM C W, LEE S C. A DEAD-box RNA helicase, RH8, is critical for regulation of ABA signaling and the drought stress response via inhibition of PP2CA activity., 2018, 41(7): 1593-1604.
[74] NAWAZ G, LEE K, PARK S J, KIM Y O, KANG H. A chloroplast-targeted cabbage DEAD-box RNA helicase BrRH22 confers abiotic stress tolerance to transgenicplants by affecting translation of chloroplast transcripts., 2018, 127: 336-342.
[75] WOODSON S A. Taming free energy landscapes with RNA chaperones., 2010, 7(6): 677-686.
[76] KIM Y O, KANG H. The role of a zinc finger-containing glycine-rich RNA-binding protein during the cold adaptation process in., 2006, 47(6): 793-798.
[77] XU T, HAN J H, KANG H. Structural features important for the RNA chaperone activity of zinc finger-containing glycine-rich RNA- binding proteins from wheat () and rice ()., 2013, 94: 28-35.
[78] KIM J S, PARK S J, KWAK K J, KIM Y O, KIM J Y, SONG J, JANG B, JUNG C H, KANG H. Cold shock domain proteins and glycine-rich RNA-binding proteins fromcan promote the cold adaptation process in., 2007, 35(2): 506-516.
[79] KWAK K J, PARK S J, HAN J H, KIM M K, OH S H, HAN Y S, KANG H. Structural determinants crucial to the RNA chaperone activity of glycine-rich RNA-binding proteins 4 and 7 induring the cold adaptation process., 2011, 62(11): 4003-4011.
[80] GU L, XU T, LEE K, LEE K H, KANG H. A chloroplast-localized DEAD-box RNA helicase AtRH3 is essential for intron splicing and plays an important role in the growth and stress response in., 2014, 82: 309-318.
[81] GU L, JUNG H J, KIM B M, XU T, LEE K, KIM Y O, KANG H. A chloroplast-localized S1 domain-containing protein SRRP1 plays a role inseedling growth in the presence of ABA., 2015, 189: 34-41.
[82] HAN J H, LEE K, LEE K H, JUNG S, JEON Y, PAI H S, KANG H. A nuclear-encoded chloroplast-targeted S1 RNA-binding domain protein affects chloroplast rRNA processing and is crucial for the normal growth of., 2015, 83(2): 277-289.
[83] LEE K, LEE H J, KIM D H, JEON Y, PAI H S, KANG H. A nuclear-encoded chloroplast protein harboring a single CRM domain plays an important role in thegrowth and stress response., 2014, 14: 98.
The Research Progress of Plant RNA Binding Proteins
ZHANG ZaiBao1,2, LI WanJie1, LI JiuLi1, ZHANG Chi1, HU MengHui1, CHENG Lin1, YUAN HongYu1,2
(1College of Life Sciences, Xinyang Normal University, Xinyang 464000, Henan;2Henan Key Laboratory of Tea Plant Biology, Xinyang Normal University, Xinyang 464000, Henan)
In eukaryotes, RNA-binding proteins (RBPs) are an important class of post-transcriptional regulators that direct and regulate the RNA metabolism. RBPs together with RNA to form ribonucleoprotein complexes have been reported to play critical roles in many RNA processes, including translocation, modification, translation and degradation. RBPs are widely present in animals, plants and microorganisms, accounting for about 2%-8% of the proteins encoded by eukaryotic genes. In recent years, the researches on RNA-binding proteins have become a hot topic. RBPs have been reported to involved in many human diseases by mutation and genetic analysis. The large number of RBPs in plants has also been reported, and they played similar important functions in plant RNA metabolism. However, our understanding of the roles and mechanisms of action of plant RBPs is less well studied than in animals. In this review, we will discuss recent progresses of multiple RBP family members that play essential roles in RNA metabolism during plant growth, development and stress responses. Five classes of plant RBP families were discussed, including serine-arginine-rich RNA-binding proteins (SR proteins), glycine-rich RNA-binding proteins (GR-RBPs), pentatricopeptide repeat proteins (PPR proteins), DEAD-box RNA helicase (DEAD-box RHs) and RNA chaperones. The critical roles of these plant RBPs in RNA metabolism during plant growth, development, and stress responses were summarized. Functions as an alternative splicing factor during RNA metabolism, SR proteins play important roles in plant growth and stress response. GR-RBPs family members displayed functional diversity: many of them regulate plant stress tolerance and various growth and development processes by mediating plant hormone signaling pathways and others mediate abiotic stress response acting as RNA chaperones. PPR proteins are the most widely studied and they mainly involved in RNA metabolism of mitochondria and chloroplasts. As important RNA splicing factors of cell nuclei and organelles, DEAD-box RHs play variety of functions in plant growth, development and abiotic stress response. RNA chaperones are non-specific RBP that maintain the normal function of RNA molecules by facilitate RNA folding via structural rearrangement of misfolded RNAs.
RNA binding proteins; RNA metabolism; plant growth and development; stress response
10.3864/j.issn.0578-1752.2018.21.001
2018-06-04;
2018-08-08
河南省教育厅项目(18A180031)、河南省自然科学基金(182300410063)、信阳师范学院南湖学者计划
张在宝,Tel:0376-6391380;E-mail:zaibaozhang79@163.com
(责任编辑 李莉)

