长链非编码RNA SBF2-AS1在肺癌组织中的表达及作用研究*
杜玮,周建龙,叶丹
(1.广州军区广州总医院 内六科,广东 广州 510010;2.广州市番禺区何贤纪念医院,广东 广州 511400)
长链非编码RNA(long noncoding RNA,lncRNA)是一类转录本长度<200 nt的RNA分子,其不编码任何的蛋白质,而是以RNA的形式存在。lncRNA早期普遍被认为是基因组转录的噪音,不具有任何生物学功能[1-3]。随着近年研究的深入,一些lncRNA的功能被逐渐阐明,发现lncRNA在表观遗传水平的调控、转录调控及转录后调控,对肿瘤的发生发展及胚胎干细胞上面都发挥重要作用[1-3]。已有数据表明SBF2-AS1作为1个新的lncRNA在肺癌组织中高表达[4],因此本研究选其为实验对象,验证其在肺癌组织中的表达情况,并研究其与肺癌病例特征的关系,并进一步研究探讨其对肺癌细胞生物学功能的影响。
1 资料与方法
1.1 一般资料
收集2011~2015年广州军区广州总医院肺癌组织样本,纳入标准:①经病理确诊的肺癌患者;②患者术前均未行放、化疗。手术切除癌组织及癌旁组织立即放入液氮,而后置入-80℃冰箱冷冻保存。所有标本均经病理检查确诊。患者签署知情同意书并获医院伦理委员会审核通过。
1.2 主要试剂
RNA逆转录试剂盒、实时荧光定量聚合酶链反应(qRT-PCR)试剂盒(大连宝生物工程有限公司),MTT细胞增殖及细胞毒性检测试剂盒(上海碧云天生物技术有限公司),PE-Annexin-V/7-AAD细胞凋亡试剂盒(南京凯基生物科技发展有限公司),Lipofectamine RNAimax reagent(美国 Invitrogen 公司)。
1.3 方法
1.3.1 总RNA的提取及qRT-PCR检测 采用Trizol法提取肺癌及癌旁组织总RNA,分光光度计法测定提取RNA的浓度及纯度。按照TaKaRa逆转录试剂盒说明书2步法完成逆转录反应,得到cDNA。qRT-PCR检测癌组织及癌旁组织SBF2-AS1的表达量。SBF2-AS1的qRT-PCR引物序列为5'-AGACCATGTGGACCTGTCACTG-3'( 正 向 引物)5'-GTTTGGAGTGGTAGAAATCTGTC-3'(反向引物);内参GAPDH(磷酸甘油醛脱氢酶)的引物序列为5'-CCACATCGCTCAGACACCAT-3'(正向引物)5'-ACCAGGCGCCCAATACG-3'(反向引物)。
1.3.2 细胞培养及质粒转染 培养A549细胞,在转染前1天铺6孔板,使细胞密度在70%左右,铺好细胞后的第2天按照Lipofectamine RNAimax reagent的说明书转染细胞。siRNA 序列为分别为si204:5'-CAG AAGGAGUCUACUGCUAAG-3';si1021:5'-GCAAGCC UGCAUGGUACAUTT-3'。
1.3.3 细胞增殖速率检测 在转染24 h后用96孔板铺细胞,每个孔铺2 000个细胞,3个复孔。然后37℃,5%二氧化碳CO2培养箱培养。按照MTT说明书分别检测24、48、72和96 h时的450 nm波长吸光度。做出细胞的增值曲线。
1.3.4 流式细胞仪检测细胞的凋亡和周期 用不含EDTA的胰酶消化转染后的细胞,检测细胞凋亡时按照PE-Annexin-V/7-AAD细胞凋亡试剂盒说明书双染细胞,流式检测,分析早期凋亡及晚期凋亡细胞比例。检测细胞周期时,细胞离心,去上清,用预冷的PBS洗2遍,然后用75%的乙醇重悬,-20℃固定过夜,离心,PBS洗涤后加10 mg/L溴化丙啶和0.1%的RNase A的PBS溶液300μl,室温避光染色10 min后,流式细胞仪检测细胞周期,分析细胞G1/G0、S期及G2期细胞比例变化。
1.4 统计学方法
数据分析采用SPSS 19.0统计软件,计量资料以均数±标准差(±s)表示,两样本均数的比较采用配对t检验或独立样本t检验,两组以上样本均数的比较采用析因设计的方差分析,分类资料采用χ2检验或Fisher精确概率法,P<0.05为差异有统计学意义。
2 结果
2.1 SBF2-AS1在非小细胞肺癌中的表达及与临床病例特征的关系
已有关于非小细胞肺癌中长链非编码RNA表达谱的数据表明,SBF2-AS1在肺癌中高表达。本研究在48例肺癌患者组织样本中检测SBF2-AS1的表达量,结果表明与正常组织比较,肺癌组织SBF2-AS1表达量升高。其相对表达量为正常组织的5.05倍(P<0.05)。并且SBF2-AS1的表达量与淋巴结转移、临床病理分期有密切关系。见附表。
2.2 SBF2-AS1在293T、H1299及A549细胞中的表达
SBF2-AS1在非小细胞肺癌细胞系A549、H1299的表达量与293T相比差异有统计学意义(P<0.05),结果见图1所示。选定A549和H1299细胞作为研究对象,进行后续实验。
2.3 SBF2-AS1对A549细胞增殖的影响
两条siRNA(si204,si1021)都能干扰SBF2-AS1的表达,导致SBF2-AS1的表达量降低,见图2所示,但si1021的效果优于si204,因此选择si1021进行后续试验。MTT细胞增殖实验结果表明,SBF2-AS1的表达被干扰降低后导致A549和H1299细胞的增殖速率减慢,见图3。空白对照组(blank组)与siRNANC组相比差异无统计学意义(P>0.05),si1021组与siRNA-NC组比较差异有统计学意义(P<0.05)。

附表 SBF2-AS1在肺癌组织样本中的表达及与临床病例特征的关系 例
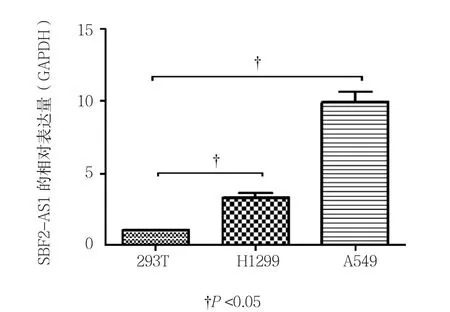
图1 SBF2-AS1在293T、H1299及A549细胞中的表达
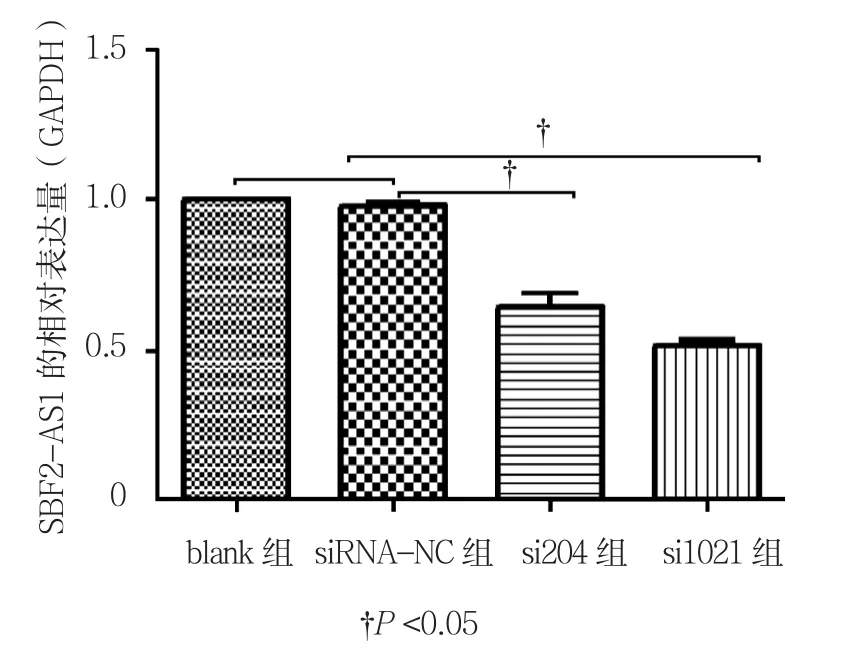
图2 si204和si1021干扰SBF2-AS1的表达
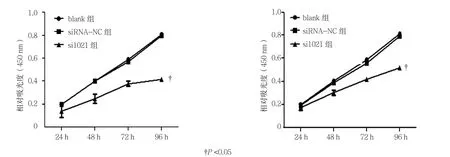
图3 SBF2-AS1对A549和H1299细胞增殖的影响
2.4 SBF2-AS1对A549和H1299细胞凋亡的影响
siRNA-NC组与blank组比较差异无统计学意义,si1021组细胞凋亡率与siRNA-NC组细胞凋亡率比较,A549为(11.31±1.792)% vs (3.35±2.014)%(P<0.05);H1299 为(17.89±1.792)% vs(9.16±2.014)%(P<0.05)。结果表明si1021干扰SBF2-AS1表达量下降后,A549及H1299细胞凋亡增加,所以SBF2-AS1促进肿瘤生存。见图4。
2.5 SBF2-AS1对A549和H1299细胞周期的影响
2种细胞各个时期细胞比例siRNA-NC组与blank组比较差异无统计学意义。对于A549细胞来说,与siRNA-NC组比较,si1021组G1/G0细胞由65.66%增至72.84%,而S期+G2期细胞由(34.34±2.32)%降至(27.16±1.59)%(P<0.05),对 H1299细胞来说,与NC组比较,si1021组G1/G0细胞由67.84%加至84.67%,而S期+G2期细胞由(32.16±2.32)%降至(15.33±1.59)%(P<0.05)。见图 5。
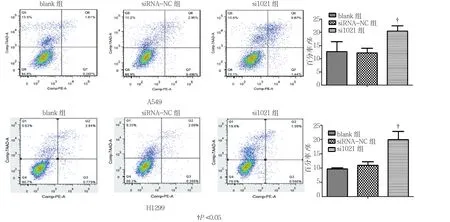
图4 SBF2-AS1对A549及H1299细胞凋亡的影响

图5 SBF2-AS1对A549和H1299细胞周期的影响
3 讨论
近年来,随着lncRNA的深入研究,大量研究结果表明lncRNA与实体瘤如脑[5-6]、肺[7-8]、乳腺[9]、胰腺[10-11]、肝[12]等有密切关系。这为癌症的研究提供了更为广阔的思路。对于肺癌来说,众多特异性的lncRNA也逐渐被发现。
SBF2-AS1作为一个新发现的lncRNA,并未有相关功能研究的报导。首先根据仅有的数据提示,检测其在肺癌及癌旁组织表达量的差异,发现其在癌组织中高表达。并由此进一步分析其表达量与临床病理特征的关系,发现其与肺癌临床病理分期及淋巴结转移关系密切。在非小细胞肺癌细胞株A549和H1299用siRNA干扰SBF2-AS1的表达,使其表达水平下降之后,分析SBF2-AS1对肺癌细胞株功能的影响。发现随着SBF2-AS1的表达下调,A549和H1299细胞增殖均减慢,流式细胞术分析发现,细胞凋亡比例增加,细胞周期出现G1/G0阻滞。已有诸多相关研究表明,lncRNA可通过调节细胞增殖与凋亡之间的平衡来调控细胞生长,这也是lncRNA发挥功能的1个机制,如 lncRNA GAS5[13]、PCGEM1[14]、PCAT-1[15]、CCAT2 等[16]功能的相关研究,SBF2-AS1亦可通过影响细胞周期调控细胞增殖。
综上所述,SBF2-AS1在肺癌组织中高表达且其表达量与患者的临床病理分期及淋巴结转移相关。该lncRNA的高表达还会通过对凋亡和细胞周期的调节加快肺癌细胞株的增殖速率。新的lncRNA SBF2-AS1功能的发现及阐明提供了新的肺癌诊断及预后判断的分子标志物,及潜在的治疗靶点。但其具体转录调控机制还有待进一步阐明。
参 考 文 献:
[1] ESTELLER M. Non-coding RNAs in human disease[J]. Nat Rev Genet, 2011, 12(12): 861-74.
[2] PONTING C P, OLIVER P L, REIK W. Evolution and functions of long noncoding RNAs[J]. Cell, 2009, 136(4): 629-641.
[3] WANG K C, CHANG H Y. Molecular mechanisms of long noncoding RNAs[J]. Mol Cell, 2011, 43(6): 904-914.
[4] WEI T Y, JUAN C C, HISA J Y, et al. Protein arginine methyltransferase 5 is a potential onco-protein that upregulates G1 cyclins/cyclin-dependent kinases and the phosphoinositide 3-kinase/AKT signaling cascade[J]. Cancer Sci, 2012, 103(9):1640-1650.
[5] ALOYZ R, XU Z Y, BELLO V, et al. Regulation of cisplatin resistance and homologous recombinational repair by the TFIIH subunit XPD[J]. Cancer Res, 2002, 62(19): 5457-5462.
[6] ZHANG X, SUN S, PU J K, et al. Long non-coding RNA expression profiles predict clinical phenotypes in glioma[J].Neurobiol Dis, 2012, 48(1): 1-8.
[7] JI P, DIEDERICHS S, WANG W, et al. MALAT-1, a novel noncoding RNA, and thymosin beta4 predict metastasis and survival in early-stage non-small cell lung cancer[J]. Oncogene,2003, 22(39): 8031-8041.
[8] YANG F, HUO X S, YUAN S X, et al. Repression of the long noncoding RNA-LET by histone deacetylase 3 contributes to hypoxiamediated metastasis[J]. Mol Cell, 2013, 49(6): 1083-1096.
[9] XING Z, LIN A, LI C, et al. lncRNA directs cooperative epigenetic regulation downstream of chemokine signals[J]. Cell, 2014,159(5): 1110-1125.
[10] BRUNNER A L, BECK A H, EDRIS B, et al. Transcriptional profiling of long non-coding RNAs and novel transcribed regions across a diverse panel of archived human cancers[J]. Genome Biol, 2012, 13(8): R75.
[11] YANG F, YI F, ZHENG Z, et al. Characterization of a carcinogenesis-associated long non-coding RNA[J]. RNA Biol,2012, 9(1): 110-116.
[12] ASKARIAN-AMIRI M E, CRAWFORD J, FRENCH J D, et al.SNORD-host RNA Zfas1 is a regulator of mammary development and a potential marker for breast cancer[J]. RNA, 2011, 17(5):878-891.
[13] MOURTADA-MAARABOUNI M, PICKARD M R, HEDGE V L, et al. GAS5, a non-protein-coding RNA, controls apoptosis and is downregulated in breast cancer[J]. Oncogene, 2009, 28(2): 195-208.
[14] SRIKANTAN V, ZOU Z, PETROVICS G, et al. PCGEM1, a prostate-specific gene, is overexpressed in prostate cancer[J]. Proc Natl Acad Sci U S A, 2000, 97(22): 12216-12221.
[15] PRENSNER J R, IYER M K, BALBIN O A, et al. Transcriptome sequencing across a prostate cancer cohort identifies PCAT-1, an unannotated lincRNA implicated in disease progression[J]. Nat Biotechnol, 2011, 29(8): 742-749.
[16] QIU M T, XU Y T, YANG X, et al. CCAT2 is a lung adenocarcinoma-specific long non-coding RNA and promotes invasion of non-small cell lung cancer[J]. Tumor Biol, 2014,35(6): 5375-5380.

