基于RFLP-PCR的猪肉溯源技术研究
吴华莉,涂尾龙,曹建国,吴 潇,常 华,都启晶,谈永松*
(1上海市农业科学院畜牧兽医研究所,上海 201106;2上海种猪工程技术研究中心,上海 201302;3上海市农业科学院生物技术研究所,上海 201106;4云南农业大学动物科学技术学院,昆明650201;5青岛农业大学食品科学与工程学院,青岛 266109)
基于RFLP-PCR的猪肉溯源技术研究
吴华莉1,2,涂尾龙1,2,曹建国1,2,吴 潇3,常 华4,都启晶5,谈永松1,2*
(1上海市农业科学院畜牧兽医研究所,上海 201106;2上海种猪工程技术研究中心,上海 201302;3上海市农业科学院生物技术研究所,上海 201106;4云南农业大学动物科学技术学院,昆明650201;5青岛农业大学食品科学与工程学院,青岛 266109)
选取杜洛克、大白猪和长白猪等3个品种共330头样品,提取基因组DNA,采用RFLP-PCR方法检测13个基因在3个猪品种群体内15个SNP位点的多态性,以期寻找到多态性信息含量丰富的SNP位点用于猪肉DNA溯源标记。结果表明:ADAMTS、DAZL、FBXO32、FUT1、MC4R、MyoG、NR4A1和 PSMB基因的9个 SNP位点可以用于杜洛克猪、大白猪和长白猪肉产品检测。根据9个SNP位点基因型对应的9个数字和字母组合形成的DNA条形码可以用于杜洛克猪、大白猪和长白猪肉产品溯源。
猪肉;DNA溯源;SNP
肉品的溯源检测在保护和提高肉类品牌过程中能起到重要作用,也可以在食品危机时有效辨别和召回污染食品。近年来,人们逐渐重视食品的质量和安全,一些学者在牛、羊和猪等肉类品种溯源方面开展了相关研究[1]。Rodríguez-Ramírez等[2]采用11个微卫星标记检测墨西哥牛肉样品,结果显示:7个微卫星标记可以用来追溯牛的屠宰和销售等各个环节采集的样品是否来源于同一个体。Goffaux等[3]研究了长白×大白×皮特兰杂交猪、纯种皮特兰和长白猪种,共检测出21个SNP位点标记可以用于品种溯源。猪肉溯源功能是指消费者向养猪场获取生猪或猪肉产品的来源,即在猪养殖、屠宰、加工和销售等环节都能追溯到产品猪的个体信息。分子溯源标记为猪溯源提供一种方便方法,由于每个猪个体的DNA序列具有唯一性和稳定性,通过鉴别个体DNA特征能够准确地识别猪种个体。本试验采用RFLP-PCR方法分别对杜洛克猪、大白猪和长白猪的相关SNP进行检测分析,以期获得可用于3个猪种猪肉产品溯源系统的SNP标记。
1 材料与方法
1.1 动物样品
三个品种的猪样品来自上海祥欣畜禽有限公司,杜洛克(100头)、大白猪(150头)、长白猪(80头)共计330头,采集猪耳组织并保存于75%酒精中,置于冰盒中运回实验室,-20℃冰箱保存备用。
1.2 方法
1.2.1 DNA提取
采用AXYGEN组织DNA提取试剂盒提取基因组DNA,并稀释到30 ng/mL,-20℃保存。
1.2.2 引物及PCR扩增
查阅NCBI数据库和已有报道,根据遗传多样性分布特点选择15个SNP作为候选标记位点(表1),包括与猪肉质性状相关的基因H-FABP(猪心脏脂肪酸结合蛋白基因)、MC4R(黑皮质素受体-4基因)和MyoG(肌细胞生成素基因),与猪繁殖性状相关的基因ADAMTS(含凝血酶敏感蛋白模体的去解联金属蛋白酶基因)、RBP4(视黄醇结合蛋白4基因),与猪病有关的基因FUT1[α-(1,2)海藻糖转移酶1基因]、DAZL(类似无精子症基因)、PSMB10(20S蛋白酶体基因beta亚基基因10)、FBXO32(泛素连接酶基因)、PSMC3(19S调节亚基 Rpt5基因)、BPI(杀菌通透性增强蛋白基因)、NR4A1(核受体4A1基因)、ALAS1(5-氨基酮戊酸合成酶基因)。随机抽取10个个体的DNA样品作为扩增模板,检测15对引物(表1)的扩增效率,引物由上海生工生物工程公司合成。PCR扩增条件为:94℃预变性4 min;94℃变性30 s,退火30 s(退火温度见表1),72℃延伸30 s,共36个循环;最后72℃延伸10 min。PCR产物由1.5%琼脂糖凝胶电泳检测,要求条带清晰无杂带。

表1 用于检测13个基因的15个SNP位点引物和内切酶Table1 Primers and endonucleases used to detect 15 SNP lociof 13 genes
1.2.3 PCR-RFLP分析
将扩增成功的片段用于酶切反应,所使用的限制性内切酶如表1所示。酶切体系由10μL PCR产物、5 U限制性内切酶和1μL Buffer组成,反应时间根据PCR产物大小调整,PCR产物片段小于500 bp为15 min,大于500 bp为30 min左右。所使用的限制性内切酶购自Fermentas、NEB和宝生物工程有限公司。
1.2.4 统计方法
计算出每个SNP的等位基因频率,杂合度的计算公式:H=1-∑pi,可用于DNA溯源标记的SNP要求H值大于0.30。
1.2.5 DNA条形码的编制方法
DNA条形码编制方法:数字条形码编制时,各SNP位点先组合形成DNA条形码。SNP位点使用阿拉伯数字标记。DNA条形码中每个SNP位点都有3种基因型,命名为A、B和C。例如第1对引物扩增SNP1位点对应的DNA条形码编号为1A、1B和1C,具体编码要根据个体检测结果加以命名。最终形成DNA条形码为数字和字母的组合,包括SNP位点信息和基因型信息。
2 结果与分析
2.1 MyoG(2FR)、PMSB(1FR)、MC4R和 DAZL基因的RFLP分析
如图1所示,PCR产物和酶切产物条带清晰明亮,可以判定基因型。根据不同基因型选取样品,一般每个基因型选择2个样品进行测序。根据测序结果在NCBI数据库进行blast比对,确定所研究的13个基因序列为猪 H-FABP、MC4R、MyoG、ADAMTS、RBP4、FUT1、DAZL、PSMB10、FBXO32、PSMC3、BPI、NR4A1和ALAS1基因(由于位点数目较多,仅提供几个RFLP示例图作为参考)。
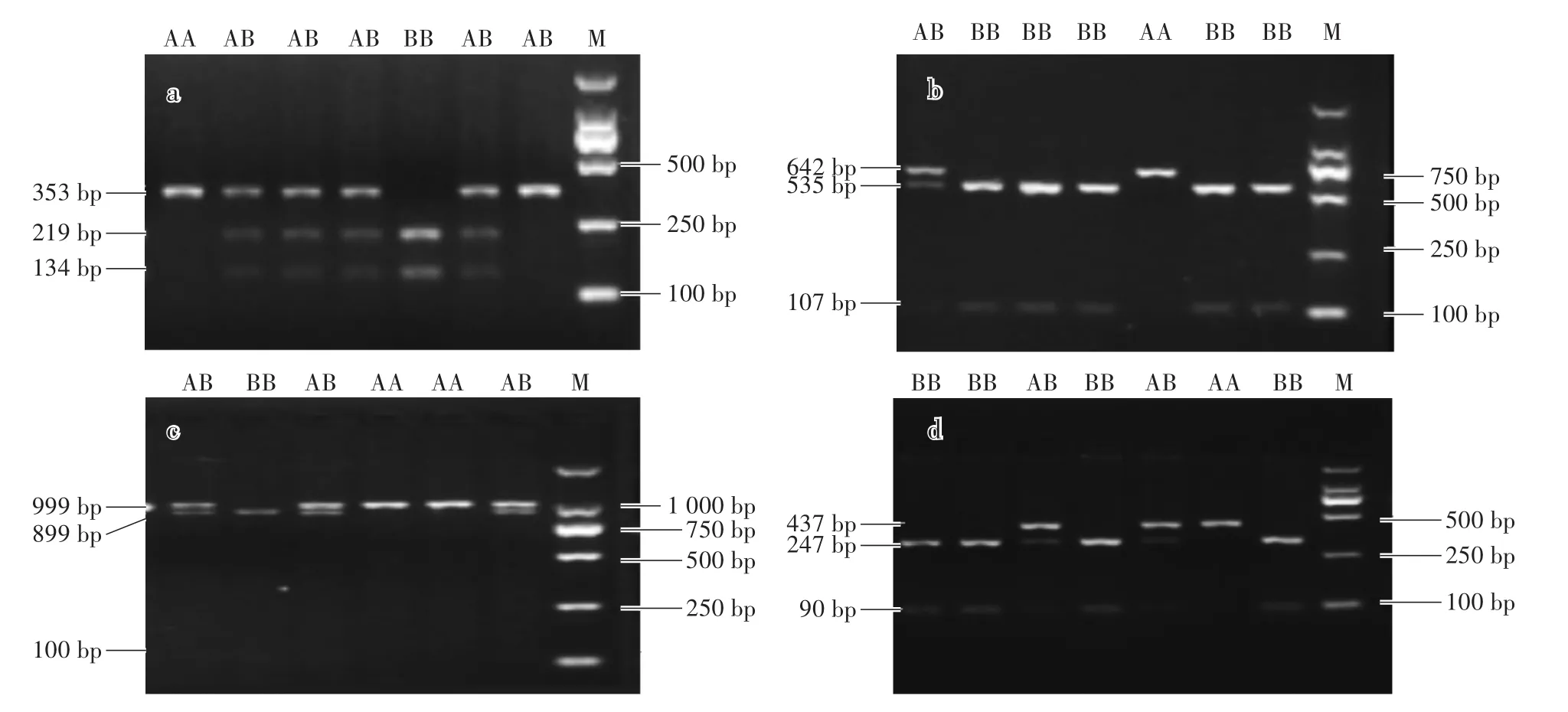
图1 猪MyoG(2FR)、PMSB(1FR)、MC4R和DAZL基因的RFLP多态位点分析Fig.1 RFLP polymorphism analysis of MyoG(2FR),PMSB(1FR),MC4R and DAZL in pig
2.2 PCR-RFLP检测结果
利用15个SNP标记引物对大白、长白及杜洛克猪进行检测,结果显示:每个引物在不同个体扩增出的片段大小是一致的,但经PCR-RFLP后电泳检测的条带大小和数目不尽相同。产生酶切位点的等位基因称为B型基因,酶切位点消失的等位基因称为A型基因。ADAMTS扩增出413 bp,经内切酶PvuⅡ分成413/316/97 bp,记为 SNP1。FBXO32扩增出1 077 bp,经内切酶 Sna BⅠ分成 1 077/608/469 bp,记为SNP5。SNP1和SNP5在3个猪种中等位基因分布较平衡。ALAS1(SNP2)、DAZL(SNP4)、MyoG(SNP9和SNP10)和 RBP4(SNP15)基因扩增出长度分别为359 bp、437 bp、959 bp、353 bp和480 bp的5个片段,5个片段分别经限制性内切酶MSPⅠ消化后产生324/277/47/35 bp、437/247/190 bp、418(264+154)/370/171 bp、353/219/134 bp和190/154/136/125/75 bp。统计结果显示:SNP2和SNP9位点等位基因频率分布不平衡,3个品种都是A型基因占优势;SNP4位点等位基因频率分布不平衡,在杜洛克猪种中B型基因占优势,在长白猪和大白猪种中A型基因占优势;SNP10位点等位基因频率分布不平衡,3个品种都是B型基因占优势;SNP15位点等位基因频率分布不平衡,在杜洛克猪和长白猪中A型基因占优势,大白猪种中B型基因占优势。H-FABP基因可由引物扩增出816 bp的片段,经HaeⅢ内切酶切分成683(405+278)/117/16 bp,记为SNP7。SNP7位点等位基因频率在大白猪种分布平衡,在杜洛克猪和长白猪种中B型基因占优势。MC4R(SNP8)基因扩增出长度为999 bp的片段,被DdeⅠ消化后所得片段长度为999/899/100 bp,这个位点的等位基因在3个猪种中分布不平衡,在杜洛克猪和大白猪中A型基因占优势,在长白猪中B型占优势。BPI(SNP3)和PSMB10(1FR)(SNP12)基因可扩增出长度为445 bp和642 bp的片段,分别被HpaⅡ和Eco81Ⅰ消化后所得片段长度为445/304/142 bp和642/535/107 bp,这两个位点的等位基因在3个猪种中分布不平衡,在杜洛克猪中A型基因占优势,在长白猪和大白猪中B型占优势。FUT1(SNP6)和PSMC3(SNP14)基因可扩增出长度为328 bp和642 bp的片段,分别被HhaⅠ和MvaⅠ消化后所得片段长度为328/241/87 bp和440(282+158)bp,这两个位点的等位基因在3个猪种中分布平衡,都是B型占优势。NR4A1(SNP11)基因扩增出长度为128 bp的片段,被DdeⅠ消化后所得片段长度为128/96/32 bp,这个位点的等位基因在3个猪种中分布平衡,都是A型占优势。PSMB10(2FR)(SNP13)基因扩增长度为802 bp,被Eco72Ⅰ消化后所得片段长度为802/550/252 bp,这个位点的等位基因在3个猪种中分布不平衡,在杜洛克猪中B型基因占优势,在长白猪和大白猪中A型占优势。
2.3 杂合度(H)分析
结果显示(表2):在不同猪种中SNP1、SNP5、SNP11和SNP13位点的H值均高于0.30,表明多态性信息含量丰富,可用于溯源标记;SNP4、SNP6、SNP8、SNP10和SNP12的H值在0.21—0.53,多态信息含量丰富,可用于溯源标记;SNP15在不同猪种显示出不同的多态性,在长白猪和大白猪中H值在0.28以上,在杜洛克猪中H值为0.18,SNP8位点能否作为溯源标记还需进一步验证;SNP2在杜洛克猪种中H值为0,不适合作为溯源标记;SNP3在长白猪和大白猪种中H值小于0.1,显示出低度多态性,不适合作为溯源标记;SNP7在长白猪种中H值为0,不符合溯源标记要求;SNP9和SNP14在杜洛克猪和长白猪中H值为0,不能选用此标记为溯源标记位点。因此,能够用于杜洛克猪、长白猪和大白猪肉DNA溯源标记的位点有 SNP1、SNP4、SNP5、SNP6、SNP8、SNP10、SNP11、SNP12和 SNP13。
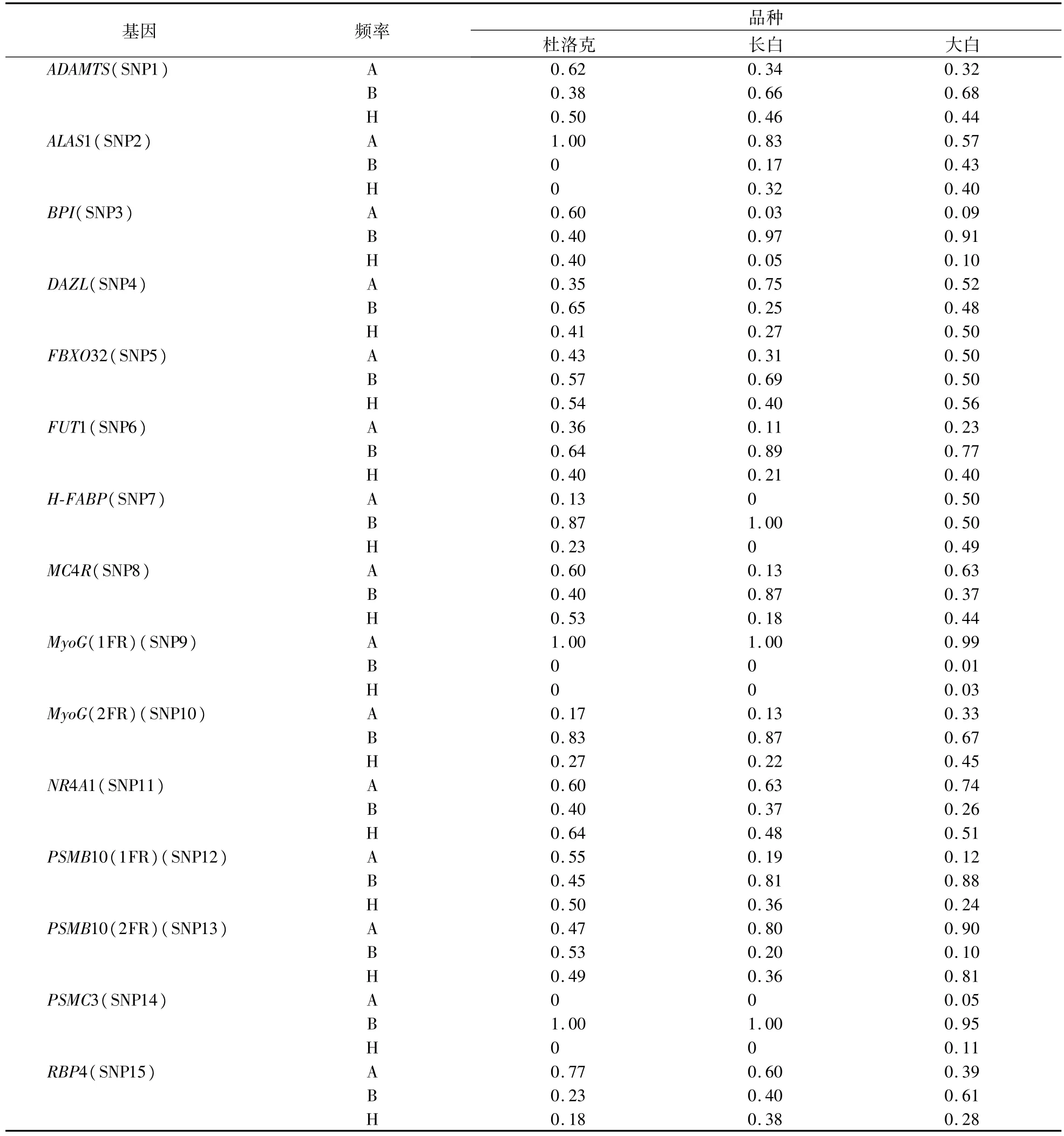
表2 15个SNP在3个猪品种中所显示的等位基因频率及杂合度Table 2 Allele frequencies and heterozygosity of the 15 SNP markers in 3 pig varieties
2.4 SNP用于肉品溯源的验证
2.4.1 9 个SNP位点在3个猪种群体中的杂合度
为进一步验证上述9个SNP在溯源标记中的可行性,在不同地点重新取样进行RFLP-PCR分析,结果显示:9个SNP位点H值均大于0.3。从杂合度上来说,这9个SNP位点符合溯源标记的要求(表3)。
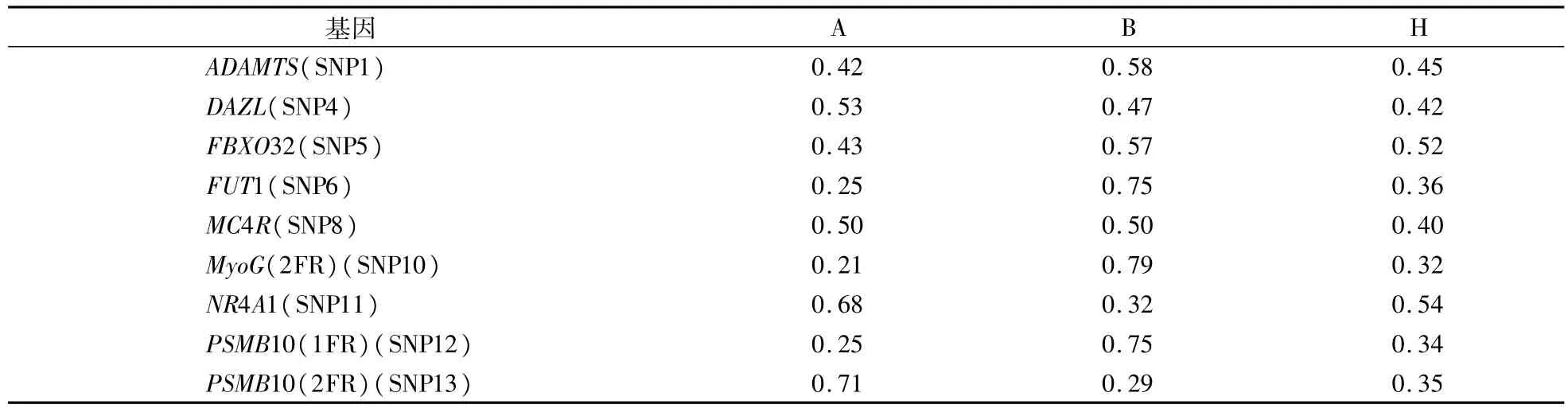
表3 9个SNP标记在330个样品中所显示的等位基因频率及杂合度Table 3 Allele frequencies and heterozygosity of the 9 SNPmarkers in 330 samples
2.4.2 根据9个SNP位点信息编制DNA条形码用于猪肉溯源的验证试验
DNA条形码中9个SNP位点的每个SNP位点都有3种基因型,命名为A、B和C(此处A、B和C表示3种基因型,与2.2处 A和 B代表含义不同)。9个 SNP位点分别为 SNP1、SNP4、SNP5、SNP6、SNP8、SNP10、SNP11、SNP12、SNP13。表4列出10个样品的检测结果和命名方法,具体DNA条形码编码要根据个体检测结果加以命名。在采集的杜洛克、长白和大白猪群体样本中,根据本试验筛选到的9个SNP位点编制个体对应DNA条形码,可以区分不同个体,每个个体对应的DNA条形码都是唯一的。杜洛克、长白和大白猪基因型也检测出差异,在编制DNA条形码时,发现同一品种内某个SNP位点检测出同种基因型,这会导致同一品种在编制DNA条形码时出现几个SNP位点相同数字和字母组合。这同时也表明DNA条形码在区别不同个体的同时,也可以区别不同的品种。表4中样品5和样品7来源于杜洛克猪,它们的条形码中出现8个SNP相同,SNP9位点检测出不同基因型。样品8、9和10来源于长白猪,分别在SNP4和SNP8位点检测出不同的基因型。
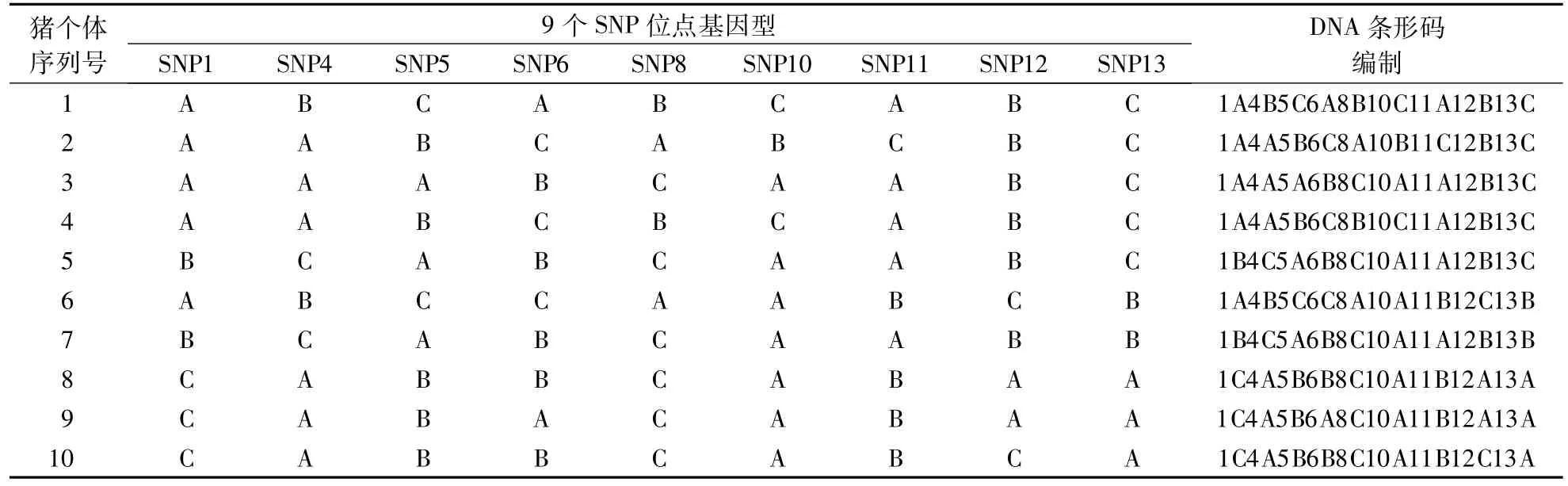
表4 根据9个SNP位点编制的DNA条形码Table 4 DNA barcode based on 9 SNP loci in 3 pig varieties
3 讨论
我国肉类溯源标记技术仍停留在传统的标签溯源技术上,致使追溯链出现漏洞,动物被屠宰分割后溯源难以继续追踪下去。国外已经开始采用DNA技术进行肉制品溯源,并建立了猪肉追踪系统[16],而我国目前还没有应用此技术。基于RFLP-PCR的DNA溯源技术易分型、重复性好、检测手段简单,但同时也要求用于检测的组织样品必须为单一纯净组织样品,因为来自不同个体组织样混合成的肉样品会混淆检测信号,从而给检测带来分析困难[3]。
本试验选择13个基因的15个SNP作为候选标记位点,采用RFLP-PCR方法对杜洛克猪、长白猪和大白猪种进行检测。结果表明:每个SNP位点在3个猪种中所显示的多态性分布是不同的,其中SNP12和SNP3虽然位于PSMB10基因上,但两个位点之间不存在遗传连锁现象,相互独立,因此二者都可作为3个猪肉产品的有效溯源标记。张小波等[17]选择12个SNP作为候选标记位点,以上海本地常用猪种为材料检测每个SNP等位基因的分布情况,研究结果证实H-FABP、MC4R、MyoG、ADAMTS和ESR基因6个SNP位点可用于猪肉产品的溯源体系。本试验筛选的MC4R、MyoG、ADAMTS与张小波等[17]研究结果一致。本试验H-FABP基因检测结果显示,在长白猪群体中只出现一种基因型,这种偏态现象的原因可能是由于长白猪经过长期选育造成某种基因型缺失,这一推测还需要扩大样品加以验证。
本试验筛选 ADAMTS、DAZL、FBXO32、FUT1、MC4R、MyoG、NR4A1和 PSMB10基因的9个 SNP可满足杜洛克猪、长白猪和大白猪肉产品的识别,理论上可用于2×104=20 000头猪个体身份DNA识别的数字条形码编制,基本可以满足500—600头种猪规模的养猪场猪的个体身份识别,如果规模更大,则可通过新增有效SNP位点来实现。基于RFLP-PCR的猪肉溯源技术应用的过程:从养殖场运到屠宰场的猪,采集血液样本或者耳组织块,进行多个SNP位点检测,建立个体“DNA条形码”。当猪肉销售时出现问题,采集猪肉样品做多个SNP位点分析,然后从数据库查询与之匹配的信息,从而快速鉴定出市场中生鲜肉类是否掺假。DNA溯源技术不受胴体被分割的限制,选择多态信息含量丰富的标记位点能够识别不同品种猪不同个体的来源,从而将每个动物个体追溯到原产地,对猪屠宰后肉制品的跟踪、追溯具有参考价值,这种技术可从源头上控制并消除食品安全隐患,
[1]CUNNINGHAM E P,MEGHEN CM.Biological identification systems:geneticmarkers[J].Revue scientifique et technique(international office of epizooties),2001,20(2):491-499.
[2]RODRÍGUEZ-RAMÍREZ R,ARANA A,ALFONSO L,et al.Molecular traceability of beef from synthetic Mexican bovine breeds[J].Genetics and Molecular Research,2011,10(4):2358-2365.
[3]GOFFAUX F,CHINA B,DAMSL,etal.Developmentof a genetic traceability test in pig based on single nucleotide polymorphism detection[J].Forensic Science International,2005(151):239-247.
[4]徐珊珊,于丽丽,乐凯.猪ADAMTS-1基因对繁殖性状的遗传效应分析[J].安徽农业科学,2008,36(24):10374-10376.
[5]刘林清.猪HSD17B1、NR4A1、ALAS1基因的分离、鉴定及功能初步研究[D].武汉:华中农业大学,2009.
[6]朱璟,潘章源,訾臣,等.11个猪种群体BPI基因第10外显子HpaII遗传变异分析[J].中国畜牧杂志,2011,47(23):14-17.
[7]张玉皓.猪H2A.Z、DAZL基因分离鉴定及遗传效应分析[D].武汉:华中农业大学,2008.
[8]于晶.猪FBXO32与其转录因子家族的基因克隆、定位、SNP检测及与生产性状的关联分析[D].武汉:华中农业大学,2006.
[9]赵金凤,张冬杰,谷溪,等.猪 GPX5基因、FUT1基因和 NCOA1基因的多态性分析[J].华北农学报,2008,23(4):69-71.
[10]GERBENS F,VANERP A JM,HARDERSF L,et al.Effect of genetic variants of the heart fatty acid-binding protein gene on intramuscular fat and performance traits content in pigs[J].Journal of Animal Science,1999,77:846-852.
[11]王文涛,杨秀芹,何鑫淼,等.五个品种猪MC4R基因的PCR-RFLP分析[J].黑龙江畜牧兽医,2008(10):29-30.
[12]SOUMILLION A,ERKENS JH,LENSTRA JA,et al.Genetic variation in the porcinemyogenin gene locus[J].Mammalian Genome,1997,8(8):564-568.
[13]吴潇.猪泛素-蛋白酶体途径定位、序列及性状关联分析[D].武汉:华中农业大学,2006.
[14]王彦芳.猪PA28和PA700基因家族相关基因的分离、定位、SNPs检测及其与性状的关联分析[D].武汉:华中农业大学,2004.
[15]赵伟.RBP4,RARG,MYOG基因多态性及其与猪繁殖性能关系的研究[D].哈尔滨:东北农业大学,2008.
[16]LOFTUSR.Traceability of biotech-derived animals:application of DNA technology[J].Revue scientifique et technique,2005,24(1):231-242.
[17]张小波,吴潇,何慧,等.基于 SNPs标记的猪肉 DNA溯源技术的研究[J].中国农业科技导报,2011,13(3):85-91.
Study on the traceability technology of pork based on RFLP-PCR
WU Hua-li1,2,TUWei-long1,2,CAO Jian-guo1,2,WU Xiao3,CHANG Hua4,DU Qi-jing5,TAN Yong-song1,2*
(1Institute of Animal Husbandry and Veterinary Science,Shanghai Academy of Agricultural Sciences,Shanghai201106,China;2Shanghai Pig Breeding and Engineering Research Center,Shanghai201302,China;3Institute of Biological Technology,Shanghai Academy of Agricultural Sciences,Shanghai201106,China;4Faculty of Animal Science and Technology,Yunnan Agricultural University,Kunming650201,China;5College of Food Science and Engineering,Qingdao Agricultural University,Qingdao266109,China)
A total of 330 samples of Duroc,Large white and Landrace pigswere selected to extract genome DNA.The RFLP-PCR method was used to detect the polymorphism of13 genes in 15 SNP loci in 3 pig varieties to find SNP lociwith rich polymorphism information for the traceabilitymarker of pork DNA.The results showed that9 SNP loci ADAMTS,DAZL,FBXO32,FUT1,MC4R,MyoG,NR4A1 and PSMB genes could be used for the detection of Duroc,Large white and Landrace pork products.DNA barcodes formed by the combination of 9 numbers and alphabetical combinations of 9 SNP loci genotypes could be used in Duroc and Large white,Landrace pork traceability.
Pork;DNA traceability;Single nucleotide polymorphism
2016-04-01
上海市科委科技支撑项目(13391900100);上海市科技兴农重点攻关项目[沪农科攻字(2013)第5-6号)、沪农青字(2014)第1-34号]
吴华莉(1975—),女,博士,助理研究员,研究方向为猪遗传育种。E-mail:hiwuhuali@163.com
*通信作者,E-mail:typine@163.com
S879.2
A
1000-3924(2017)06-057-06
闫其涛)

