植物低温胁迫响应m iRNAs在植物抗寒研究中的应用前景
王丽丽,赵统利,葛金涛,刘兴满
(连云港农业科学院,连云港 222000)
植物低温胁迫响应m iRNAs在植物抗寒研究中的应用前景
王丽丽,赵统利,葛金涛,刘兴满*
(连云港农业科学院,连云港 222000)
对植物低温胁迫调控机制、miRNAs的生物合成及作用方式,及其在低温调控网络中的相关作用进行了综述,并运用生物信息学对植物miRNAs在低温胁迫下参与的调控以及3类低温胁迫相应miRNAs进行了分析。结果表明:第一类miRNAs中,miR156家族、miR169家族、miR172家族和miR396家族成熟序列碱基保守性相对较高;第二类 miRNAs中,整体 miRNAs家族碱基保守性都较高;第三类miRNAs中,miR397家族和miR408家族成熟序列碱基保守性相对较低。以期利用miRNAs成熟序列保守性特征,从miRNA角度分析植物的耐寒机制,为进一步开展植物抗寒机理研究提供新思路。
miRNAs;碱基保守性;低温胁迫
microRNA(miRNAs)是一类长度为 21—23 nt[1]的非编码序列。1993年,Lee等[2-3]在线虫(Caenorhabditis elegansl)的研究中首次发现了lin-4 miRNA基因,2002年植物中第一个miRNA的发现使miRNA引起了极大的关注[4-5]。miRNA属于真核生物中内源性、非编码的小分子RNA,产生于具有发夹结构的前体,在植物中主要通过介导对应靶基因降解对生长发育及抗逆过程发挥作用[6],具有高度的保守性、时序性和组织特异性[7]。
早期研究认为,不同植物的miRNA间存在着保守性差异[8]。随着研究的不断深入,发现目前研究过的miRNAs中,植物的miRNA相较于动物miRNA更加保守,尤其是在其二级结构以及成熟序列方面。当前较热门的生物信息学分析软件正是基于这个原理,对miRNA及其靶基因进行成功预测,也是植物miRNA预测比动物miRNA预测更准确的原因。
低温是植物生长发育的主要限制因素之一,但目前对植物抗寒方面的研究多集中于拟南芥、水稻等模式植物生理生化检测以及耐受胁迫蛋白等方面,从植物非编码基因角度对植物抗寒机制的研究在大部分物种中还未见报道。本文基于植物miRNA在物种间具有较高的保守性的特点,对植物低温响应的3类miRNAs进行综述。
1 m iRNA的生物合成与作用方式
在植物中,II型RNA聚合酶(Pol-II)或 III型 RNA聚合酶(Pol-III)可与 miRNA基因上游启动子结合,转录出可以形成发夹结构的前体miRNAs(pri-miRNAs)[9]。miRNA合成场所包括细胞核以及细胞质。在细胞核中,pri-miRNAs在Dicer同源异性蛋白DCL1[10-11]及辅助因子HYL1[12]的作用下完成切割,形成成熟茎环结构的前体(pre-miRNA);pre-miRNAs被DCL1切割产生21—26 nt的双链miRNA,构成miRNA::miRNA*复合物[13],之后此复合物经过DCL1以及HEN1介导的甲基化后,进入AGO1中,一部分miRNA分子转移出核,并通过HASTY转运蛋白进入细胞质,从而发挥相应的功能。
miRNA主要通过剪切降解、抑制翻译和染色体重塑等方式调控靶基因的表达而发挥作用。miRNA对靶基因的作用方式因与靶标基因之间的匹配程度不同而不同[14],当miRNA与靶基因完全或者近完全互补时,miRNA主要通过降解发挥调控靶基因的作用;而当miRNA与靶基因不完全匹配时,miRNA则主要通过抑制其翻译发挥作用[15]。在植物中,miRNA与靶基因特定位点是完全或近完全互补的,且作用位点位于miRNA成熟序列的第10—11位碱基[16],因此可精确切割靶基因发挥相应的降解作用。
2 植物在低温胁迫条件下参与的网络调控机制
2.1 转录水平调控
低温胁迫条件下,植物细胞壁和质膜的结构发生变化,使细胞质内的Ca2+浓度提高,从而激发COR(Cold-response)基因的表达[17]。目前,低温信号转导途径主要包括胞外信号途径、胞内第二信使、转录因子以及功能基因等多种。在植物低温信号转导途径中起关键作用的转录因子包括bLHL(Basic helix-loophelix)、bZIP(Basic leucine zipper)和 CBF(C-repeat/dehydration-responsive element binding factor)等。植物低温信号转导途径主要包括两个途径,即ABA依赖型和非ABA依赖型[18]。
当植物处于低温条件下时,植物体内的ABA含量会有暂时的上升,研究表明这与植物抗寒因子启动有关[19]。这些基因的启动子区均包含有序列较保守的ABREs(ABA response elements)顺式作用元件。另外,bZIP类的转录因子可以和ABRE结合,激活ABA依赖基因的表达。非ABA依赖型主要包括依赖CBF途径和不依赖CBF途径。依赖CBF途径主要以ICE1-CBF3-COR的途径存在,在低温胁迫条件下,ICE1基因编码MYC类bHLH转录因子并与CBF3基因的启动子结合,从而激活CBF3基因的表达[20],之后CBF3基因与COR基因启动子区域的顺式作用元件结合,诱导其表达[21-22]。除了这种途径外,还存在MYC/MYB-COR和ROS-bZIP-COR途径,这两种途径中,MYB和bZIP类转录因子可通过结合COR基因启动子区域的CRT/DRE序列启动与抗寒相关基因的表达,从而提高植物的抗寒性[18]。
2.2 m iRNA在低温胁迫下参与的调控
在逆境胁迫条件下,植物通过调节miRNA的表达水平,进而调节对应靶基因的表达,从而引起相关代谢与信号转导途径的变化来实现对逆境的响应。因此,鉴于植物miRNA与逆境胁迫相关,越来越多的研究将miRNA加入低温胁迫调控网络中,以期更全面地解答植物应答低温胁迫的调控网络。针对水稻(Oryza sativa)[23]、拟南芥(Arabidopsis thaliana)[24]、甘蔗(Saccharum officinarum)[25]以及毛竹(Phyllostachy edulis)[26]的研究发现,miRNAs参与植物低温胁迫调控过程。如在低温胁迫下,水稻中miR156以及毛竹中miR397表达量均下降,但miR172在拟南芥中表达量升高。同一miRNA在不同物种中在低温胁迫条件下表达量变化也有所不同。如受低温胁迫影响,miR168在拟南芥中表达量上调,但在水稻中表达量却呈现下调趋势,这可能是不同植物中miRNA调控低温胁迫的方式不同所致[27]。
根据对应靶基因的不同,可将响应低温胁迫的miRNAs分为3类[24]。第一类miRNAs:间接响应低温胁迫,其对应靶标基因主要是调控基因表达、信号转导类的转录因子,这类转录因子主要调控在低温胁迫中起作用的蛋白的表达,包括 miR156、miR165、miR169、miR171、miR172、miR319和 miR396;第二类 miRNAs:其调控的靶基因可直接响应低温胁迫,包括miR160、miR167、miR168、miR393、miR394和miR395;第三类miRNAs:其对应的靶基因编码水解酶或氧化还原酶,可响应多种胁迫,包括miR397和miR408。
利用数据库 miRbase 21.0(http://www.mirbase.org/index.shtml)[28]搜索以上 3类 miRNAs各家族成员的成熟序列,下载得到响应低温胁迫的 miRNA成熟序列;利用在线平台 WebLOGO(http://weblogoberkeley.edu/logo.cgi)[29]分析3类响应低温胁迫miRNA的碱基保守性高低;通过在线平台http://plantgrn.noble.org/psRNATarget/[30]预测各家族主要的靶基因;利用美国生物信息技术中心平台(NCBI:http://www.ncbi.nlm.nih.gov/)对靶标基因进行BLAST分析,初步确定3类miRNA对应靶标基因的功能。
2.3 三类低温胁迫响应m iRNA生物信息学分析
2.3.1 成熟序列保守性分析
目前,miRbase 21.0中共收录了223个物种的35 828个miRNA成熟序列信息。本文在miRbase 21.0中下载3类miRNAs各家族成员成熟序列,并利用在线平台对其进行成熟序列保守性分析。结果表明:第一类miRNAs中,miR156家族、miR169家族、miR172家族和miR396家族成熟序列碱基保守性相对较高,而miR165家族、miR171家族和miR319家族成熟序列碱基保守性相对较低,只有个别位点碱基保守性较高,如miR165家族成熟序列中第9、第11和第12位碱基(分别为G、C、U)以及miR171家族成熟序列第4、第8、第16和第19位碱基(图1);第二类miRNAs中,整体miRNA家族碱基保守性都较高(图2);第三类miRNAs中,miR397家族和miR408家族成熟序列碱基序列保守性都较低,其中miR397家族成熟序列第6、第13和第19位碱基保守性相对较高(图3)。
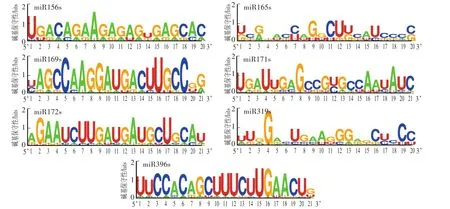
图1 第一类m iRNAs成熟序列的碱基保守性Fig.1 The site conservation of the first kind ofm iRNAsmature sequence

图2 第二类m iRNAs成熟序列的碱基保守性Fig.2 The conservation ofmature sequence of the second kind ofm iRNAs

图3 第三类m iRNAs成熟序列的碱基保守性Fig.3 The site conservation of the third kind ofm iRNAsmature sequence
2.3.2 三类miRNAs靶基因预测
通过在线平台对3类低温胁迫响应的miRNAs进行靶基因预测,结果显示(表1):miR156家族的靶基因是SBP基因,其在促进植物营养期转变和植物开花过程中起重要的作用。研究表明:miR156在低温胁迫诱导下表达量下降,导致其靶基因SBP表达量上调,开花时间延迟,植物通过生长代谢减慢来应对胁迫环境[31]。miR165和miR172的靶标基因是Class III HD-ZIP和AP2转录因子家族,调控生长素信号通路[32]。miR160、miR167的靶基因是ARF(Auxin response factors),主要通过调控生长素信号通路应对逆境。miR393的靶基因是bHLH转录因子,与ICE1编码蛋白属同一个家族,因此推断可能在植物应答低温逆境胁迫中发挥作用。miR397的靶基因是漆酶,其与植物抗病、细胞壁木质素合成代谢等过程有关[33]。
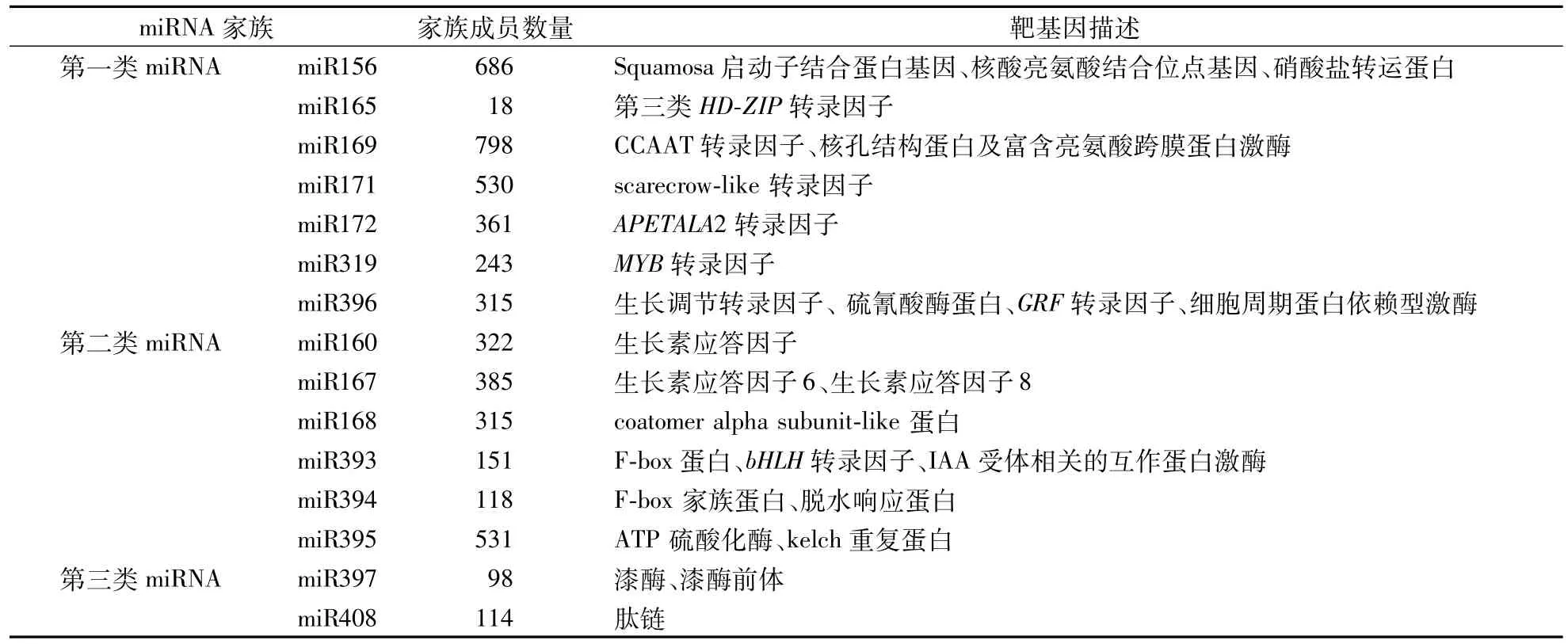
表1 植物低温胁迫相应m iRNAs信息Table 1 The corresponding m iRNAs information of cold stress in plant
3 结论与讨论
当前,对植物耐寒性研究多集中在生理生化检测以及相关功能基因方面,从非编码基因miRNA角度对植物耐寒调控机制研究较为鲜见。本文对植物耐寒网络调控,包括转录水平调控以及miRNAs水平调控进行了综述与分析,并利用生物信息学方法对低温胁迫响应3类miRNAs各家族成员成熟序列保守性进行了分析。结果表明:第一类miRNAs中,miR156家族、miR169家族、miR172家族以及miR396家族成熟序列的碱基保守性相对较高;第二类miRNAs中,整体miRNA家族碱基保守性都较高;第三类miRNAs中,miR397家族和miR408家族成熟序列的碱基序列保守性相对较低,只在个别碱基位点保守性较高。这暗示着miRNAs成熟序列在不同物种间具有一定的保守性,这与之前相关研究结果一致[7]。
随着科技水平的进一步提高,生理生化实验与新兴的分子生物学实验方法的结合为今后植物耐寒机理的研究奠定了坚实的基础。从常规育种角度,首先通过引种驯化,初步筛选出植物耐寒新品系,通过区试试验以及生产试验,逐步开展植物耐寒新品系的推广工作;从分子育种角度,通过低温胁迫试验检测差异表达基因,获得相关基因片段,克隆抗寒相关基因以及分子标记等技术是目前研究植物耐寒机制的主要方法。虽然这些研究对理解植物抗寒机制起到一定的促进作用,但大多局限在单基因功能的分析和探讨上,各基因间的相互作用及其上游调控并不明晰。因此,利用miRNAs成熟序列具有较高保守性的特点,可在植物耐寒机制的研究中引入miRNAs技术,即从3类低温胁迫响应miRNAs角度为全面地揭示低温胁迫下植物的基因表达网络提供新视角。现有与抗寒相关的miRNAs的研究都集中在miRNAs的初步鉴定方面,缺乏后续功能的研究,已鉴定的miRNAs与低温胁迫应答网络中的关键因子到底是如何互作的还不清楚。因此,可进一步结合生物信息学方法、高通量测序以及功能验证等技术研究植物低温响应miRNAs,同时利用靶基因预测软件(如psRNATarget等)和RLM-5’RACE进一步对miRNAs精确作用位点进行鉴定,进而分析植物低温胁迫条件下的低温应答网络。另外,也可利用茎环引物定量法分析低温胁迫后响应miRNA在植物中的表达先后及表达量差异等,为筛选植物耐寒新品种提供新途径。
[1]ZHANG B H,STELLWAG E J,PAN X P.Large-scale genome analysis reveals unique features ofmicroRNAs[J].Gene,2009,443(1/2):100-109.
[2]LEE R C,FEINBAUM R L,AMBROSV.The C.elegans heterochronic gene lin-4 encodes small RNAs with antisense complementary to lin-14[J].Cell,1993,75(5):843-854.
[3]WIGHTMAN B,HA I,RUVKUM G.Posttranscriptional regulation of the heterochronic gene lin-14 by lin-4 mediates temporal pattern formation in C.elegans[J].Cell,1993,75(5):855-862.
[4]LLAVE C,KASSCHAU K D,RECTOR M A,et al.Endogenous and silencing-associated small RNAs in plants[J].Plant Cell,2002,14(7):1605-1619.
[5]REINHART B J,WEINSTEIN E G,RHOADESMW,et al.MicroRNAs in plants[J].Genes Development,2002,16:1616-1626.
[6]LLAVE C,XIE Z,KASSCHAU KDM,et al.Cleavage of Scarecrow-likemRNA targets directed by a class of Arabidopsis miRNA[J].Science,2002,297(5589):2053-2056.
[7]HE L,HANNON G J.MicroRNAs:small RNAswith a big role in gene reguation[J].Nature Reviews Genetics,2004,5(7):522-531.
[8]FLOD SK,BOWMAN JL.Gene regulation:AncientmicroRNA target sequences in plants[J].Nature,2004,428:485-486.
[9]KURIHARA Y,WATANABE Y.Arabidopsis microRNA biogenesis through DICER-LIKE1 protein functions[J].PNAS,2004,101:12753-12758.
[10]VOINNETO.Origin,biogenesis,and activity of plantmicrornas[J].Cell,2009,136(4):669-687.
[11]MILLAR A A,WATERHOUSE P M.Plant and animal microRNAs:similarities and differences[J].Funct.Integr.Genomics,2005,5(3):129-135.
[12]LAGOSQM,RAUHUT R,LENDECKEL W,et al,Identification of novel genes coding for small expressed RNAs[J].Science,2001,294(5543):853-858.
[13]YU B,YANG Z Y,LIJJ,et al.Methylation as a crucial step in plantmicroRNA biogenesis[J].Science,2005,307(5711):932-935.
[14]ZHANG B H,PAN X P,ANDERSON T A.Indentification of 188 conserved maize microRNAs and the its targets[J].FEBS Lett.,2006,580(15):3752-3762.
[15]LAIE C,TOMANCAK P,WILLIAMSRW,et al.Computational identification of DrosophilamicroRNA genes[J].Genome.Biol.,2003,4(7):R42.
[16]KASSCHAU KD,XIE ZX,ALLEN E,etal.P1/HC-Pro,a viralsuppressor of RNA silencing,interfereswith Arabidopsis developmentandmiRNA function[J].Dev.Cell,2003,4(2):205-217.
[17]KNIGHTM R.Signal transduction leading to low-temperature tolerance in Arabidopsis thaliana[J].Philosophical Transactions of the Royal Society of London Series B-biological Science,2002,357(1423):871-875.
[18]计淑霞,戴绍军,刘炜.植物应答低温胁迫机制的研究发展[J].生命科学,2010,22(10):1013-1019.
[19]HEIDARVAND L,MAALI A R.What happens in plant molecular response to cold stress?[J].Acta Physiologiae Plantarum,2010,32(3):419.
[20]CHINNUSAMY V,ZHU J,SUNKAR R.Gene Regulation During Cold Stress Acclimation in Plants[J].Methods Mol Biol,2010:639(1):39.
[21]STOCKINGER E J,GILMOUR S J,THOMASHOW M F.Arabidopsis thaliana CBF1 encodes an AP2 domain-containing transcriptional activator that binds to the C-repeat/DRE,a cis-acting DNA regulatory element that stimulates transcription in response to low temperature and water deficit[J].Proceedings of The National Academy of Sciences of The United States of America,1997,94(3):1035-1040.
[22]LIU Q,KASUGA M,SAKUMA Y,etal.Two transcription factors,DREB1 and DREB2,with an EREBP/AP2 DNA binding domain separate two cellular signal transduction pathways in drought and low temperature responsive gene expression respectively in Arabidopsis[J].Plant Cell,1998,10(8):1391-1406.
[23]LY D K,BAIX,LIY,et al.Profiling of cold-stress-responsivemiRNAs in rice bymicroarrays[J].Gene,2010,459(1/2):39-47.
[24]LIU H H,TIAN X,LIY J,et al.Microarray-based analysis of stress-regulated microRNAs in Arabidopsis thaliana[J].RNA,2008,14(5):836-843.
[25]THIEBAUT F,ROJASC A,ALMEIDA K L,etal.Regulation ofmiR319 during cold stress in sugarcane[J].Plant Cell and Environment,2012,35(3):502-512.
[26]王丽丽,赵韩生,孙化雨,等.毛竹miR397和miR1432的克隆及其逆境胁迫响应表达分析[J].林业科学,2015,51(6):63-70.
[27]SUNKAR R,LIY F,JAGADEESWARAN G.Functions ofmicroRNAs in plant stress responses[J].Trends in Plant Science,2012,17(4):196-203.
[28]ANA K,SAM G J.miRBase:annotating high confidencemicroRNAs using deep sequencing data[J].Nucleic Acids Reasearch,2014,42(D1):D68-D73.
[29]GAVIN E C,GARY H,JOHN M C,et al.WebLogo:A Sequence Logo Generator[J].Genome Reseach,2004,14:1188-1190.
[30]DAIX B,ZHAO X C.psRNATarget:a plant small RNA target analysis server[J].Nucleic Acids Reasearch,2011,39(suppl2):W155-W159.
[31]WU G,POETHIG R S.Temporal regulation of shoot development in Arabidopsis thaliana by miR156 and its target SPL3[J].Development,2006,133(18):3539-3547.
[32]SUNKEAR R,LIY F,JAGADEESWARAN G.Functions ofmicroRNAs in plant stress responses[J].Trends in Plant Science,2012,17(4):196-203.
[33]张志明,宋锐,彭华,等.用生物信息学挖掘玉米中的 microRNAs及其靶基因[J].作物学报,2010,36(8):1324-1335.
App lication prospects of p lant cold-stress-responsivem iRNAs in cold resistance research of plants
WANG Li-li,ZHAO Tong-li,GE Jin-tao,LIU Xing-man*
(Lianyungang Academy of Agricultural Sciences,Lianyungang222000,China)
The plant regulation mechanism in low temperature,biosynthesis and action mode ofmiRNAs,and its role in the low temperature regulation network were summarized.The regulation of plantmiRNAs under low temperature stress and 3 kinds of low temperature stress corresponding miRNAs were analyzed by bioinformaticsmethod.The results showed that in the first kind of miRNAs,the mature sequences of miR156 family,miR169 family,miR172 family and miR396 family had relatively higher site conservation.In the second kind ofmiRNAs,the wholemiRNAs family had higher site conservation.In the third kind ofmiRNAs,themature sequences ofmiR397 family and miR408 family had relatively low site conservation.We hope to use themature sequence conservation characteristics ofmiRNAs to analyze the cold resistancemechanism of plants from miRNA point of view,so as to provide new ideas for further research on cold resistancemechanism of plants.
miRNAs;Site conservation;Cold stress
2016-06-27
精准扶贫科技行动计划[KF(17)1006]
王丽丽(1988—),女,硕士,研究实习员,从事林木遗传育种研究
*通信作者:刘兴满(1982—),男,硕士,高级农艺师,从事果树及林木耐盐研究。E-mail:wangllawj@163.com
Q945.78
A
1000-3924(2017)06-129-06
闫其涛)

