急性心肌梗死相关基因的生物信息学分析
李丹辉 朱建华 乐健伟 王志宇 叶继辉 范震
急性心肌梗死相关基因的生物信息学分析
李丹辉 朱建华 乐健伟 王志宇 叶继辉 范震
目的 从基因水平揭示心肌梗死的发病机制,为心肌梗死的基础研究和临床治疗提供新思路。方法 从基因芯片公共数据库GEO(gene expression omnibus,GEO)中下载心肌梗死相关基因芯片数据,利用GENE-E进行差异基因分析,筛选出在心肌梗死患者中具有的差异表达基因,采用Panther、String等在线分析软件对差异表达基因进行生物信息学分析。结果 在200个差异表达基因所编码的蛋白中,有168个存在蛋白与蛋白的相互作用关系;其生物学通路主要涉及细胞凋亡通路、表皮生长因子受体信号通路等。结论 通过生物信息学分析心肌梗死相关基因芯片数据,提示心肌梗死发病是多种基因作用的结果,EPHB1,GRM6,CD2BP2在心肌梗死患者外周血中高表达,对相关基因的进一步分析有利于揭示心肌梗死的发病机制,从而为心肌梗死患者的早期筛查提供新的分子标志物。
心肌梗死 基因芯片 生物信息学
心肌梗死是由于冠状动脉闭塞、血流中断所导致的心肌细胞损伤和坏死。缺血可引起活性氧积聚,细胞膜稳定性受到破坏,细胞内大量活性酶释放,最终导致心肌细胞凋亡、坏死。同时心肌梗死可致心肌细胞肥大增生、心室重构和心力衰竭而引发死亡[1-2]。
近年来,随着基因芯片技术的发展和应用,产生了大量的基因表达谱数据。如何从这些数据挖掘出有价值的信息,已成为当今研究的一个热点[3-4]。本研究通过对基因芯片公共数据库(gene expressionomnibus,GEO)中的心肌梗死患者外周血标本的芯片数据进行生物信息学分析,以期为心肌梗死患者的早期筛查提供新的分子标志物。
1 资料和方法
1.1 一般资料 在GEO Datasets搜索框中以“heart disease”为检索词,获得Suresh R等提交的GSE48060芯片数据。该研究采用的是商业化的离子通道芯片平台GPL570,共收集52例患者外周血进行基因分析,排除既往有肿瘤、肝病、脑梗死以及风湿系统等疾病,其中急性心肌梗死组来自31个急性心肌梗死患者的外周血标本,对照数据来自21个非心肌梗死患者的外周血标本。其中急性心肌梗死组共31例,分别为GSM1167072、GSM1167073、GSM1167074、GSM1167075、GSM1167076、GSM1167077、GSM1167078、GSM1167079、GSM1167080、GSM1167081、GSM1167082、GSM1167083、GSM1167084、GSM1167085、GSM1167086、GSM1167087、GSM1167088、GSM1167089、GSM1167090、GSM1167091、GSM1167092、GSM1167093、GSM1167094、GSM1167095、GSM1167096、GSM1167097、GSM1167098、GSM1167099、GSM1167100、GSM1167101、GSM1167122;对照组为21例,分别为GSM1167102、GSM1167103、GSM1167104、GSM1167105、GSM1167106、GSM1167107、GSM1167108、GSM1167109、GSM1167110、GSM1167111、GSM1167112、GSM1167113、GSM1167114、GSM1167115、GSM1167116、GSM1167117、GSM1167118、GSM1167119、GSM1167120、GSM1167121、GSM1167123。
1.2 方法
1.2.1 差异基分析 采用GENE-E软件分析GEO数据库中的数据,先将数据集导入GENE-E软件,采用两组样本t检验统计方法,对数据进行有效过滤,筛选出差异基因后进行生物信息学分析。
1.2.2 差异基因编码蛋白的相互作用分析 将上述差异性基因筛选后上传至String 10.0在线分析工具(http://string-db.org),分析其所编码的蛋白,获得差异表达基因的蛋白相互作用图,筛选出中心节点蛋白[5-6]。
1.2.3 差异基因的功能分析 将筛选出的差异基因上传至Panther数据库(http://pantherdb.org),对这些差异基因分别采用信号通路(Pathway)、生物学过程(Biological Process)及分子功能(Molecular Function)等进行分析[7-8]。
2 结果

图1 差异基因表达
2.1 两组一般资料比较 两组患者在年龄、性别、高脂血症、糖尿病、心血管家族史及合并用药方面差异比较均无统计学意义(均P>0.05)。两组高血压、吸烟史及BMI比较差异均有统计学意义(P<0.05或0.01)。
2.2 差异基因筛选结果 从GSE48060数据中筛选出的差异基因,经筛选后,最终选出了13个可能和急性心肌梗死相关的基因(图 1),其中 EPHB1、GRM6、CD2BP2在表达上调,IL-2RB、PRF1、GZMB、KLRD1、YES1、PPM1L、P2RY12,ADCY7、RBM5及 PCBP2表达下调。
2.3 差异基因编码蛋白的相互作用分析通过String 10在线分析 工具进行分析,发现168个蛋白之间存在相互作用(图2)。其中,RPL38等51个基因的蛋白与其它蛋白存在相互作用关系,为此蛋白互作网络的中心节点,删除这些节点蛋白后,网络结构涣散。
2.4 差异基因的生物学功能分析结果 通过Panther生物学过程分析表明,差异表达的基因主要涉及细胞生理过程、代谢过程、生长发育过程等(图3)。
另外,Panther分子学功能分析发现差异表达的基因主要涉及分子黏附、催化活性及受体活性等(图4)。
3 讨论
心血管疾病是当今全球人群的首要死因,急性心肌梗死是严重危害人类健康的心血管急症,目前大量研究从流行病学、病理生理学、分子生物学等方法揭示急性心肌梗死的发病机制,并取得了重大进展[9]。
基因表达谱从生物信息学方面研究急性心肌梗死的发病机制,对基因芯片数据进行后续的生物学探讨,是目前挖掘基因芯片数据在急性心肌梗死机制研究中的重要方向。本研究通过生物信息学软件对芯片数据(GSE48060)进行分析,在200个明显差异基因中筛选出168个差异表达基因,并对这些差异基因进行筛选以及蛋白相互作用分析,筛选出了13个可能和急性心肌梗死相关的核心基因。在分析过程中,我们发现相对于对照组,EPHB1、GRM6、CD2BP2在心肌梗死组表达量均上调,而 IL2RB、PRF1、GZMB、KLRD1、YES1、PPM1L、P2RY12,ADCY7、RBM5及PCBP2则总体上在心肌梗死组表达均下调。此外,对上述这些表达差异明显的基因深人研究后,发现除P2RY12与心血管疾病的关系已有文献报道外[10-11],其他基因与心血管疾病的关系未做类似的研究,它们的具体作用机制尚不明确。
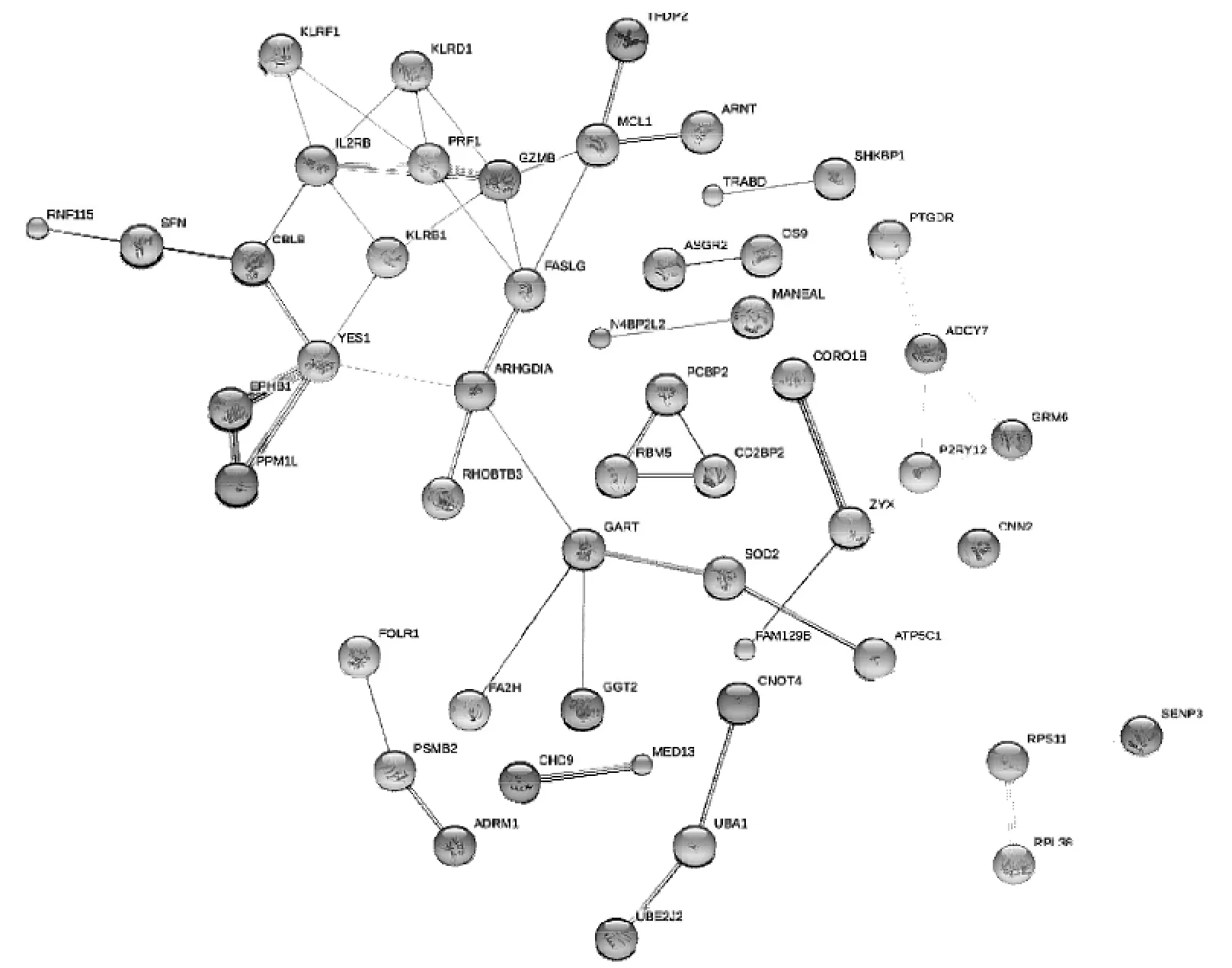
图2 差异基因编码蛋白的相互作用分析
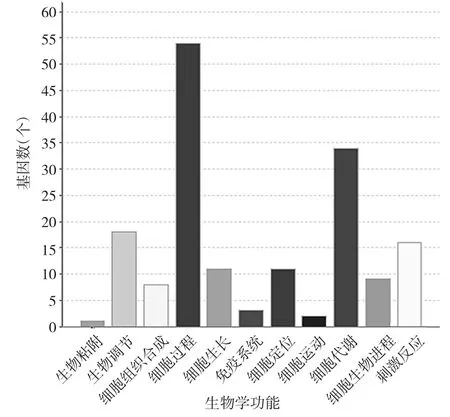
图3 差异基因生物学功能
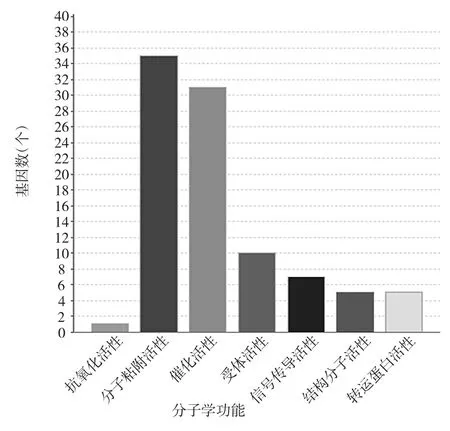
图4 差异基因分子学功能
本研究发现EPHB1、GRM6、CD2BP2在急性心肌梗死患者外周血中高表达,EPHB1主要表达于血小板上,血小板除了表达EPHB1外,还表达EphA4及配体ephrinB1。Eph/ephrin信号增强血小板激动剂引起聚集,并提高细胞内的Ca2+,通过激活RAP1、EPHB1、EphB2以及EphB4,进而导致血小板的活化、聚集,最终导致血栓形成[12-13]。GRM6是一种蛋白质编码基因,主要与夜盲症、先天性静止性夜盲症等疾病发生有关,其通过G蛋白偶联受体活性和谷氨酸受体活性发挥作用。CD2BP2(CD2胞质尾结合蛋白2)能编码双功能蛋白质,在细胞核中是U5小核糖核蛋白复合物的组成部分,参与RNA剪接,在细胞质中通过调节T淋巴细胞的活化,参与免疫调节。因此,我们推测活化的EPHB1可能通过诱导血小板的活化、聚集,最终导致心肌梗死的发生,但目前相关研究仍尚无报道,因此,需进一步临床研究对该基因进行验证。
综上所述,我们采用生物信息学方法通过对心肌梗死患者外周血基因芯片差异基因的筛选,以及对其分子功能、生物学功能分析,最终筛选出13个可能与急性心肌梗死相关的核心基因,其中EPHB1、GRM6、CD2BP2在心肌梗死组表达均上调,IL2RB、PRF1、GZMB、KLRD1、YES1、PPM1L、P2RY12,ADCY7、RBM5 及 PCBP2在心肌梗死组表达均下调,本文存在不足之处主要是缺乏体内外大量实验研究对上述基因进行验证,并且得以证实,从而为心肌梗死患者的早期筛查及发病机制提供新的思路。
[1] 侍作胜,王临光.急性冠脉综合征的诊断和治疗[M].中国矿业大学出版社,2005:2-39.
[2] Wang J C,Normand S L,Mauri LWangJ C,et al.Coronary artery spatialdistribution ofacute myocardialinfarction occlusions[J].Circulation,2004,110(3):278-284.doi:10.1161/01.CIR.0000135468.67850.F4.
[3] Turner F S,Clutterbuck D R,Semple C A.POCUS:mining genomic sequence annotation to predict disease genes[J].Genome Biol,2003,4(11):R75.doi:10.1186/gb-2003-4-11-r75.
[4] 余海浪,马文丽,郑文岭.用于基因数据挖掘的基因表达数据库GEO[J].中国生物工程杂志,2007,8:96-103.
[5] SalwinskiL,Miller C S,Smith A J,et al.The Database of Interacting Proteins:2004 update[J].Nucleic Acids Res,2004,32:D449-451.doi:10.1093/nar/gkh086.
[6] Mishra G R,Suresh M,Kumaran Ketal.Human protein reference database--2006 update[J].Nucleic Acids Res,2006,34(Database issue):D411-414.doi:10.1093/nar/gkj141.
[7] Ashburner M,BallC A,Blake J A,et al.Gene ontology:toolfor the unification of biology[J].Nat Genet,2000,25(1):25-29.doi:10.1038/75556.
[8] Carbon S,Ireland A,MungallC J,etal.AmiGO:online access to ontology and annotation data[J].Bioinformatics,2009,25(2):288-289.doi:10.1093/bioinformatics/btn615.
[9] 王泳钦,钱令嘉,姚林,等.代谢组学在心血管疾病研究中的应用[J].现代预防医学,2009,36(6):1172-1173.
[10] Kerneis M,Silvain J,Abtan J,et al.Platelet effect of prasugrel and ticagrelor in patients with ST-segment elevation myocardial infarction[J].Arch Cardiovasc Dis,2015,108(10):502-510.doi:10.1016/j.acvd.2015.04.004.
[11] Ulehlova J,Slavik L,Kucerova J,et al.Genetic polymorphisms of platelet receptors in patients with acute myocardial infarction and resistance to antiplatelet therapy[J].Genet Test Mol Biomarkers,2014,18(9):599-604.doi:10.1089/gtmb.2014.0077.
[12] Prévost N,Woulfe D S,Tognolini M,et al.Signaling by ephrinB1and Eph kinases in platelets promotes Rap1 activation,platelet adhesion,and aggregation via effector pathways that do not require phosphorylation of ephrinB1[J].Blood,2004,103(4):1348-1355.doi:10.1182/blood-2003-06-1781.
[13] Prevost N1,Woulfe D,Tanaka T,et al.Interactions between Eph kinases and ephrins provide a mechanism to support platelet aggregation once cell-to-cell contact has occurred[J].Proc Natl Acad Sci U S A,2002,99(14):9219-9224.doi:10.1073/pnas.142053899.
Bioinformatic analysis of genes associated with acute myocardial infarction
LI Danhui,ZHU Jianhua,LE Jianwei,et al.
Intensive Care Unit,Ningbo First Municipal Hospital,Ningbo 315000,China
Objective To perform bioinformatic analysis of genes associated with acute myocardial infarction. Methods The microarray data of myocardial infarction were downloaded from the Gene Expression Omnibus(GEO)database,and Qlucore Omics Explorer software was used to screen differentially expressed genes.The further analysis of differentially expressed genes were conducted by the on-line tools STRING and Panther. Results Among 200 differentially expressed genes,168 genes were related to interaction between proteins.These genes were involved in the biological process and molecular function of apoptosis signaling pathway and epithelial growth factor(EGF)receptor signaling pathway and so on.The analysis found that EPHB1,GRM6 and CD2BP2 genes were highly expressed in peripheral blood of patients with acute myocardial infarction.Conclusion Bioinformatic analysis shows that acute myocardial infarction may involve the interaction of multiple genes,which may provide useful information for studies on molecular mechanisms and potential therapeutic targets of acute myocardial infarction.
Myocardialinfarction Bioinformatics Gene microarray
10.12056/j.issn.1006-2785.2017.39.23.2016-1582
315000 宁波市第一医院重症医学科
朱建华,E-mail:19132411@qq.com
2016-10-08)
沈昱平)

