一株产几丁质脱乙酰酶海洋细菌的筛选、鉴定及发酵优化
来蒋丽 刘姝, 胡晟源 顾张慧 王淑军 房耀维,
(1. 淮海工学院海洋生命与水产学院,连云港 222005;2. 江苏省海洋资源开发研究院,连云港 222005)
一株产几丁质脱乙酰酶海洋细菌的筛选、鉴定及发酵优化
来蒋丽1刘姝1,2胡晟源1顾张慧1王淑军2房耀维1,2
(1. 淮海工学院海洋生命与水产学院,连云港 222005;2. 江苏省海洋资源开发研究院,连云港 222005)
利用对硝基-N-乙酰苯胺筛选培养基从海州湾海域海泥中筛选获得产几丁质脱乙酰酶细菌MCDA02。经过形态学、生理生化和16S rDNA序列分析初步鉴定该菌株为解角质素微杆菌。通过单因素优化和正交试验优化,得出最优发酵条件。在单因素优化基础上,利用正交试验优化获得菌株MCDA02最优发酵条件为发酵温度25℃,培养基起始pH7.0,装液量50 mL/250 mL,几丁质3%,发酵时间48 h。在此发酵条件下,菌株MCDA02发酵水平达到158.47 U/mL,是优化前的3.2倍。试验结果为菌株MCDA02几丁质脱乙酰酶的进一步研究奠定基础。
几丁质脱乙酰酶;海洋细菌;鉴定;产酶条件
几丁质普遍存在于昆虫的外骨骼,甲壳类动物的壳,大多数真菌和部分藻类的细胞壁,是仅次于纤维素居世界第二的天然有机生物大分子[1]。仅在水生生态系统,几丁质每年的产量就超过1011t[2]。几丁质难溶于水和有机溶剂,在工业上难以应用[3,4]。几丁质脱乙酰度达到55%以上得到壳聚糖。壳聚糖分子中有大量游离氨基,具有良好的生物相容性和生物可降解性,并具有抗癌、降低胆固醇、降血压等生物活性,广泛应用于食品、医药、轻工、环保和农业等领域[5,6]。
几丁质脱乙酰基生成壳聚糖有化学和生物两种方法。化学法需耗费大量的40%NaOH,产品质量不均一,且导致环境恶化[7]。生物法是利用几丁质脱乙酰酶(Chitin deacetylase,E.C. 3.5.1.41)催化几丁质脱乙酰基。对比化学方法,生物法条件温和,脱乙酰程度一致,且绿色环保。此外,将几丁质脱乙酰酶与几丁质酶联用,可以生产出化学法不能生产的具有特定乙酰化位置,以及分子质量分布范围窄的壳聚糖产品[8-10]。
目前,产几丁质脱乙酰酶的真菌菌株报道较多,有被孢霉属[11]、根霉属[12]、帚霉属[13]等。产几丁质脱乙酰酶的细菌有产碱杆菌属[14]和红球菌属[15]。相对于真菌,细菌产几丁质脱乙酰酶速率更快[5,16]。目前报道的几丁质脱乙酰酶存在产酶活力低、脱乙酰效果差,催化温度较高等问题[17]。海洋中存在大量几丁质,蕴含着大量分泌几丁质脱乙酰酶的微生物。因此,本研究从海洋样品中筛选产低温几丁质脱乙酰酶细菌,对菌株进行鉴定和发酵条件研究。
1 材料与方法
1.1 材料
1.1.1 样品采集 海泥样品采集于中国黄海海州湾燕尾港海域,采集后置于冰盒,并尽快带回实验室进行试验研究。
1.1.2 培养基 (1)2216E培养基:蛋白胨5 g/L,酵母粉1 g/L,FePO40.1 g/L,琼脂20 g/L,陈海水配制,pH7.0;(2)筛选培养基:胶体几丁质 2 g/L,K2HPO40.7 g/L,KH2PO40.3 g/L,MgSO40.5 g/L,对硝基-N-乙酰苯胺0.2 g/L,陈海水配制,pH7.0;(3)种子培养基:酵母粉1 g/L,蛋白胨5 g/L,NaCl 10 g/L,陈海水配制,pH7.0;(4)发酵培养基:玉米浆5 g/L,可溶性淀粉 3 g/L,(NH4)2SO43 g/L,KH2PO41.5 g/L,MgSO40.5 g/L,粉末几丁质10 g/L,陈海水配制,pH 8.0。
1.1.3 药品与仪器 几丁质由江苏澳新生物工程有限公司提供,其它生化试剂均为分析纯购于上海博微生物科技有限公司,引物由南京思普金生物公司合成。高速冷冻离心机购于美国SIGMA公司,分光光度计购于杭州明基科学仪器有限公司,Nikon 90i全电动显微镜购于上海普赫光电科技有限公司。
1.2 方法
1.2.1 产几丁质脱乙酰酶菌株的筛选 称取1 g海泥,加入9 mL灭菌陈海水,搅拌均匀后用灭菌陈海水进行10倍浓度梯度稀释。分别吸取10-4、10-5、10-6的稀释液100 μL涂布3个平板。平板倒置于25℃培养,观察菌落生长及变色圈产生情况。挑选产生黄色变色圈的菌株在2216E培养基三区划线纯化至单菌落后,斜面保藏。菌株接种于种子培养液培养,再以1%的接种量接种至发酵培养基,25℃,180 r/min培养60 h后,将发酵液8 000 r/min离心10 min,上清液作为粗酶液测定几丁质脱乙酰酶活性。
1.2.2 菌株MCDA02的鉴定 根据伯杰氏细菌鉴定手册,对纯化培养的菌株MCDA02进行形态学观察和生理生化鉴定[18]。
1.2.3 菌株的16S rDNA序列扩增及分析 利用基因组提取试剂盒提取菌株的基因组,进行16S rDNA的扩增。PCR通用引物为:27F:5'-AGAGTTTGATCCTGGCTCAG-3' 1492R:5'-GGTTACCTTGTTACGACTT-3',反应体系为:PCR mix(21 μL),上下游引物(各 1 μL),DNA 模板(2 μL)。反应程序 :94℃ 变性 5 min;94℃变性 30 s,54℃退火30 s,72℃延伸90 s,32个循环;72℃终延伸10 min。
扩增产物送至南京思普金生物公司进行测序,所得的序列上传GenBank。通过Blast程序与GenBank数据库中的序列进行同源性比对,并用MEGA7.0软件中的Neighbor-Joining法构建进化树。1.2.4 单因素试验优化发酵产酶条件 将种子液以1%接种量接种至起始pH为8.0的发酵培养基。以25℃、装液量30%、180 r/min发酵60 h为基本条件,分别对发酵时间、发酵温度、装液量、起始pH、诱导剂浓度进行单因素优化。
1.2.5 正交法优化发酵产酶条件 采用正交法得出菌株的最佳发酵条件。选择发酵时间、温度、起始pH、装液量、诱导剂浓度为5个单因素,每个因素取4个水平,测定几丁质脱乙酰酶的酶活,进行L16(45)正交试验,每个试验重复3次。1.2.6 几丁质脱乙酰酶酶活力的测定 试管中加入30℃预保温的0.05 mol/L pH7.0磷酸缓冲液3 mL,200 mol/L的对硝基乙酰苯胺水溶液1 mL,酶液1 mL,于30℃水浴反应15 min,沸水浴终止酶促反应,8 000 r/min离心10 min,测定上清液的吸光度。以添加1 mL同样浓度沸水浴灭活15 min的酶液作为对照。酶活单位(U)定义:在上述反应条件下每小时产生1 μg对硝基苯胺所需要的酶量定义为一个酶活力单位。
1.2.7 数据处理与分析 每组试验设3组平行,重复试验3次,试验结果用平均值±标准方差(±s)(n=3)表示,采用SPSS Statistics19.0软件对试验结果进行统计分析。
2 结果
2.1 产几丁质脱乙酰酶菌株的筛选和鉴定
2.1.1 产几丁质脱乙酰酶菌株的筛选 将稀释的土样涂布于含对硝基-N-乙酰苯胺的筛选培养基,挑选产生黄色变色圈的菌落,获得能够产生变色圈的菌株5株。菌株MCDA02产生的黄色变色圈见图1。用接种环将产生变色圈的菌株接入种子培养液培养24 h后,以1% 接种量接种至发酵培养基,25℃,180 r/min培养60 h。发酵液8 000 r/min离心10 min,测定发酵上清中几丁质脱乙酰酶的酶活。结果见表1,菌株MCDA02酶活最高,达到48.13 U/mL。
图1 菌株MCDA02在筛选平板上生成的变色圈

表1 菌株产几丁质脱乙酰酶酶活
2.1.2 菌株MCDA02的形态学特征 菌株MCDA02接种于2216E固体培养基,25℃培养48 h后,菌落形态为黄色,半透明,表面光滑湿润,圆形,边缘齐整,中心稍突起,易挑取(图2-A)。对菌株MCDA02进行革兰氏染色观察,该菌株为革兰氏阳性短杆菌,无芽孢(图2-B)。
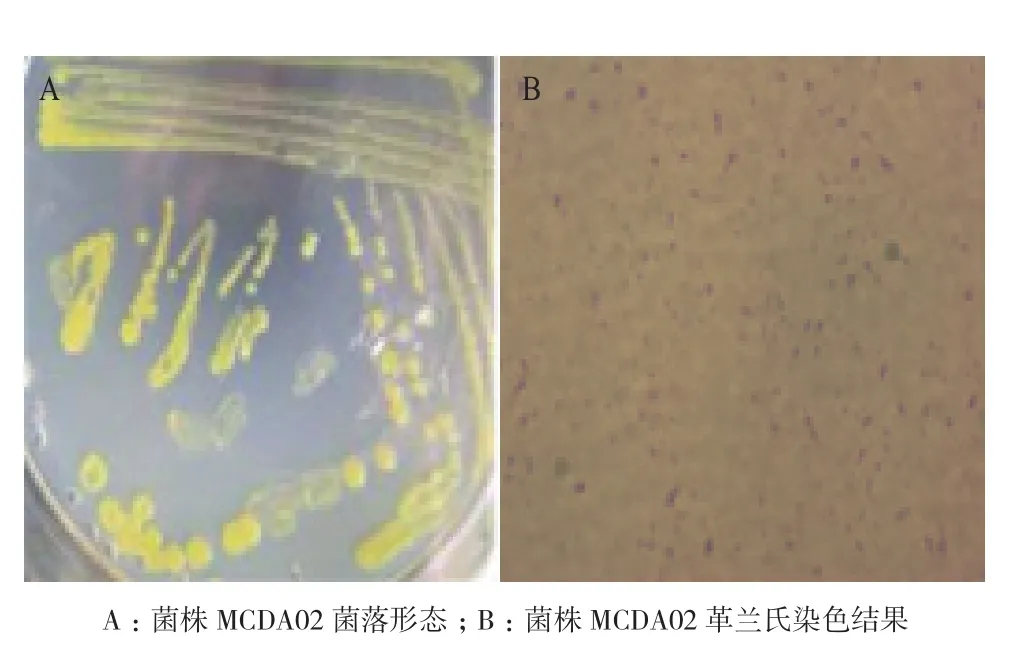
图2 菌株MCDA02的形态学特征
2.1.3 菌株MCDA02的生理生化特征 菌株MCDA02的生理生化特征见表2。该菌株淀粉水解、吲哚试验、硫化氢产生、精氨酸脱羧酶试验均呈阳性,柠檬酸盐利用、接触酶、明胶液化、VP试验、甲基红、硝酸盐还原试验均为阴性。
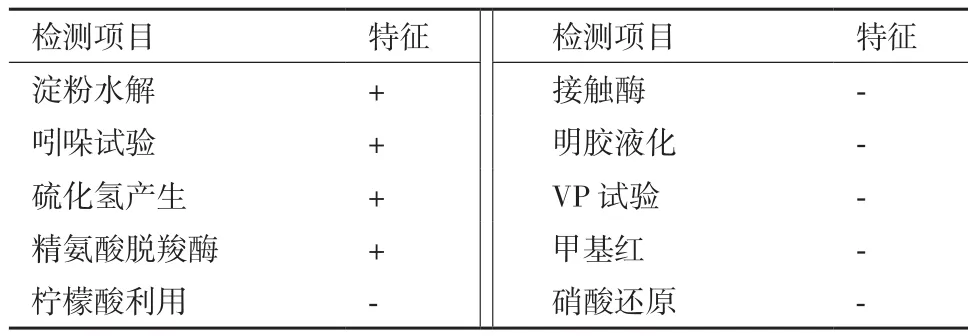
表2 MCDA02菌株的生理生化试验结果
2.2 菌株MCDA02的16S rDNA鉴定
将菌株MCDA02的16S rDNA扩增产物测序获得的1 395 bp序列提交至GenBank(登录号:KY385626),并与GenBank数据库中序列进行同源性比对,发现与菌株Microbacterium keratanolyticum OU01(登录号:DQ118082.1)16S rDNA相似性达100%。构建系统发育树见图3,菌株MCDA02与Microbacterium keratanolyticum OU01亲缘关系最近。
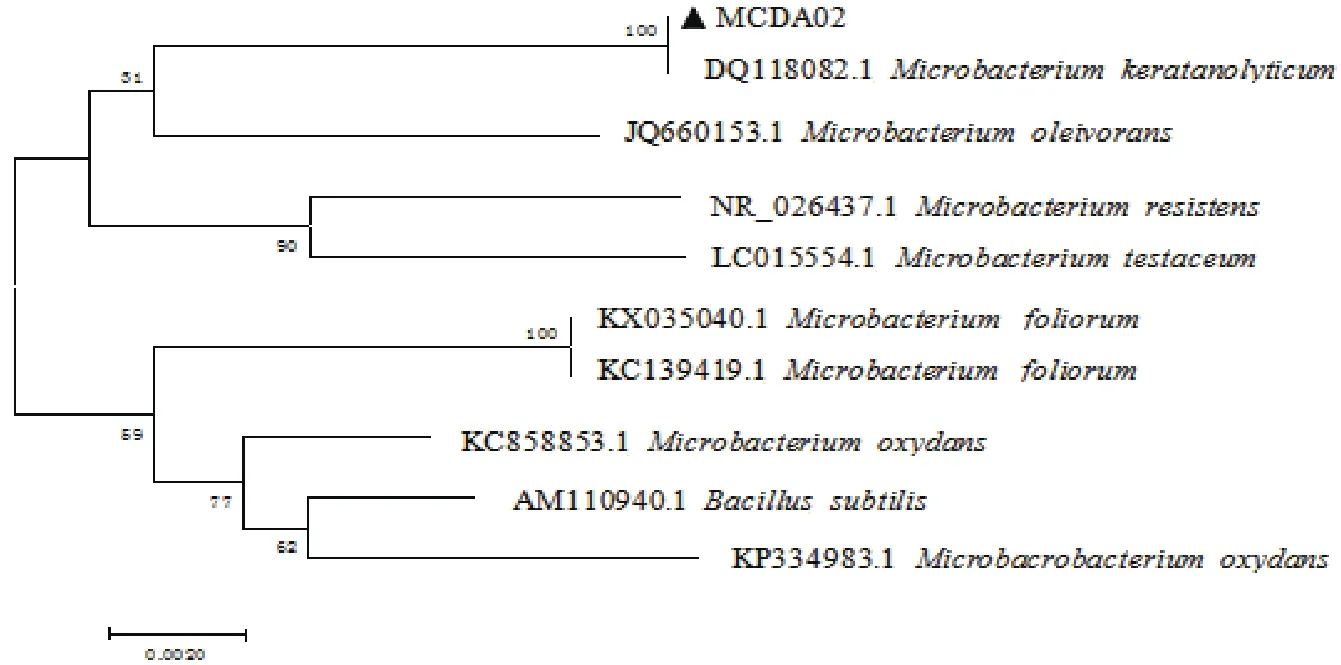
图3 基于MCDA02菌株16S r DNA序列构建的系统发育树
2.3 单因素试验优化发酵产酶条件
2.3.1 发酵时间对菌株MCDA02产几丁质脱乙酰酶的影响 发酵时间对菌株MCDA02产几丁质脱乙酰酶的影响如图4。发酵时间达到48 h时,其相对酶活达到最高。随着发酵时间的进一步增加,酶活呈下降趋势。
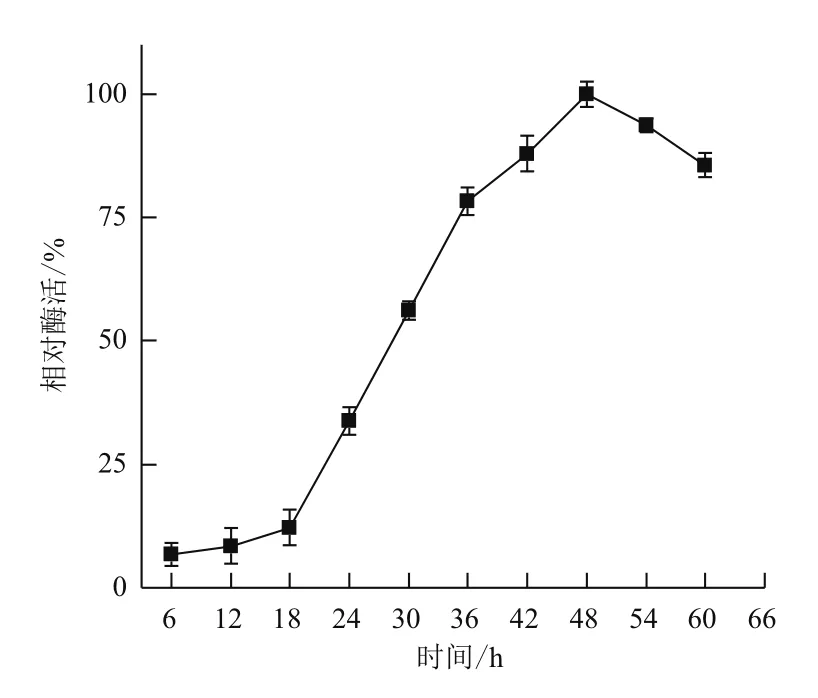
图4 发酵时间对菌株MCDA02产酶的影响
2.3.2 发酵温度对菌株MCDA02产几丁质脱乙酰酶的影响 发酵温度对菌株MCDA02产几丁质脱乙酰酶的影响如图5,几丁质脱乙酰酶酶活从10℃开始随发酵温度的升高而增大,在25-35℃范围内酶活较高,最适发酵温度为30℃,当发酵温度达到40℃以上时,酶活开始迅速下降。
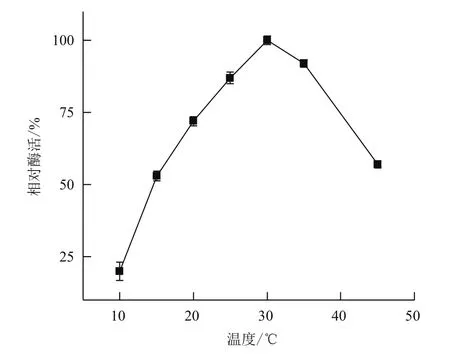
图5 发酵温度对菌株MCDA02产酶的影响
2.3.3 培养基起始pH对菌株MCDA02产几丁质脱乙酰酶的影响 培养基起始pH对菌株MCDA02产几丁质脱乙酰酶的影响如图6,发酵产酶的最适pH为8.0,培养基的起始pH为4.0或11.0时,该酶的相对酶活较低。但在pH为7.0-9.0时,酶活较高,可保持90%以上的酶活。
2.3.4 装液量对菌株MCDA02产几丁质脱乙酰酶的影响 装液量对菌株MCDA02产几丁质脱乙酰酶的影响如图7,装液量在10%-20%之间时,菌株MCDA02所产几丁质脱乙酰酶的相对酶活较高。装液量高于20%时,酶活下降显著。
2.3.5 诱导剂浓度对菌株MCDA02产几丁质脱乙酰酶的影响 诱导剂浓度对菌株MCDA02产几丁质脱乙酰酶的影响如图8。粉末几丁质浓度为0.5%-3.0%时,其相对酶活呈上升趋势。粉末几丁质浓度为3.0%-6.0%时,呈下降趋势。诱导剂的最适浓度为3.0%。
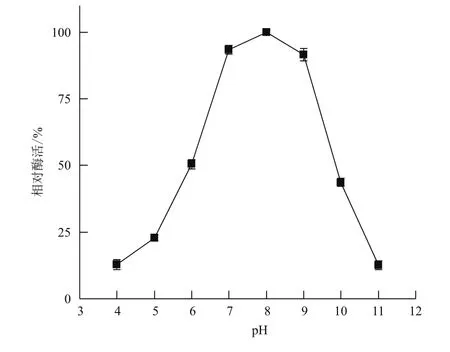
图6 起始pH对菌株MCDA02产酶的影响
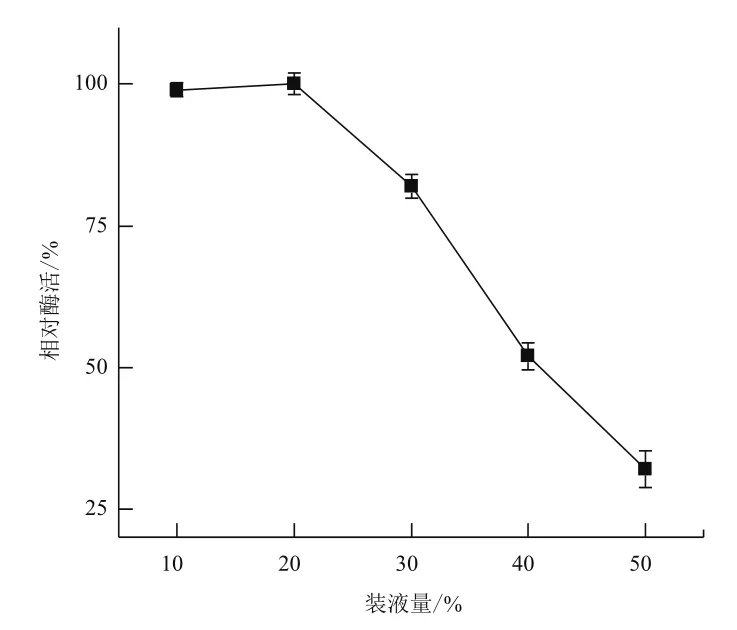
图7 装液量对菌株MCDA02产酶的影响
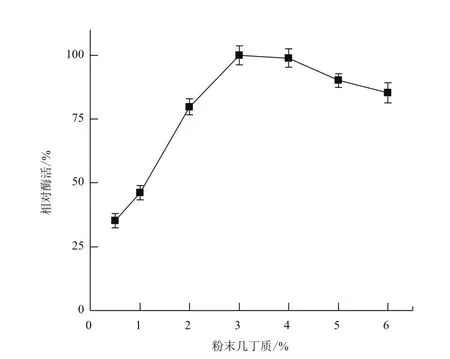
图8 几丁质浓度对菌株MCDA02产酶的影响
2.4 正交法优化发酵产酶条件
L16(45)的正交试验结果见表3。根据试验结果,计算kn值和R值,得出的最优发酵方案为:几丁质浓度为3%,培养基起始pH为7.0,装液量为20%,在温度为25℃的条件下发酵培养48 h。在此发酵条件下,菌株MCDA02发酵水平达到158.47 U/mL,是优化前的3.2倍。影响发酵结果的5个因素中,发酵温度、培养基起始pH、诱导剂浓度的R值较大,发酵时间、装液量的R值较小。对比各个因素及交互对比的极差R的大小,5个因素对发酵结果的影响力依次为:发酵温度>诱导剂浓度>培养基起始pH>发酵时间>装液量。
3 讨论
采用生物法,利用壳甲壳素制备壳聚糖较传统化学方法优势明显。筛选几丁质脱乙酰酶产生菌,发酵制备几丁质脱乙酰酶是生物法制备壳聚糖的前提条件[12]。目前几丁质脱乙酰酶产生菌以丝状真菌居多,细菌较少。研究发现细菌几丁质脱乙酰酶产酶水平较高。目前报道的多数几丁质脱乙酰酶活性较低,催化最适温度高[16]。海洋里还有大量的几丁质,且环境奇特多样,蕴含有大量的催化特性不同的几丁质脱乙酰酶产生菌,成为该酶产生菌的研究热点。
微生物发酵条件对菌株产酶有显著的影响,通过发酵优化可以显著提高酶的发酵水平。关于发酵优化提高微生物产酶的报道与日俱增,取得了显著的成果。目前尚未见解角质素微杆菌产几丁质脱乙酰酶的报道。本研究通过发酵条件优化后该菌株发酵水平达到158.47 U/mL,具有工业化应用的潜力。
微生物发酵条件对菌株产酶有显著的影响,通过发酵优化可以显著提高酶的发酵水平。应聪萍等[19]针对已筛选出的一株能生产几丁质脱乙酰酶的海洋丝状真菌并研究了其最佳发酵培养基。通过试验得出该菌的最佳发酵培养基为:7 g/L葡萄糖,7 g/L酵母浸膏,0.75 g/L氯化钙,1.25 g/L几丁质和1.4%NaCl,发酵108 h。目前对几丁质脱乙酰酶理化性质的研究主要集中于温度、pH 值、金属离子及反应底物对酶催化的影响,有的研究者也考虑了在培养基中添加生长激素、胰岛素等因子提高酶活。
同时,不同性质的底物也影响几丁质脱乙酰酶的催化活性。本研究将几丁质通过破碎、高温等处理后将其作为催化反应的底物。结果表明,经过处理的几丁质作底物,酶的脱乙酰度显著提高。多项研究表明乙二醇几丁质是几丁质脱乙酰酶作用较为理想的反应底物[12]。
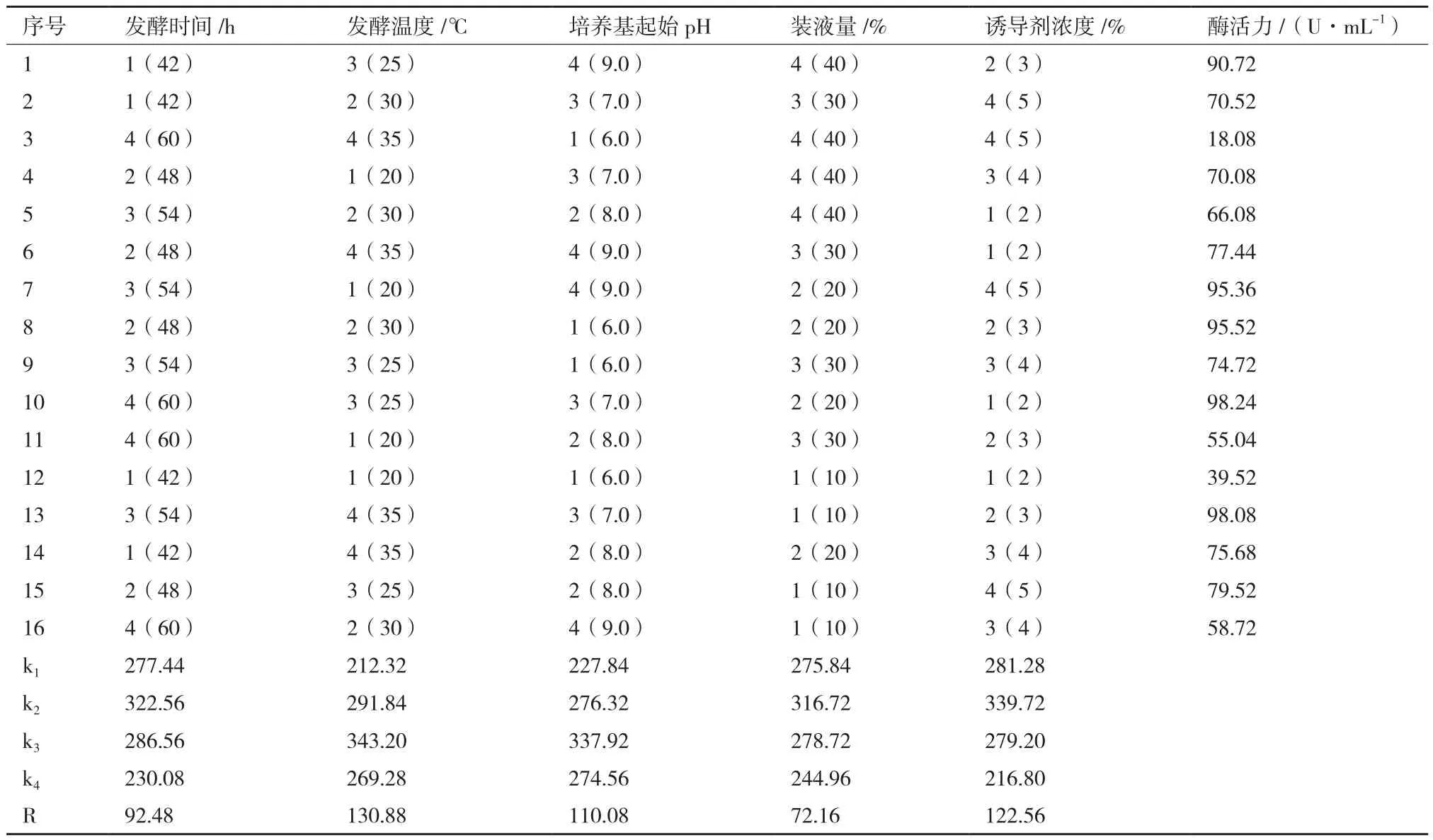
表3 L16(45)正交试验结果分析
随着研究者对几丁质脱乙酰酶研究的逐渐深入。其研究的范围不仅是从自然界分离纯化到几丁质脱乙酰酶,还可通过基因工程的手段对该酶的基因进行研究。贾志娟[20]通过对北极深海沉积物的宏基因组测序,获得了一种新的几丁质脱乙酰酶基因。闫晓萍等[21]利用RACE-PCR的方法获得了编码美国白蛾几丁质脱乙酰酶2基因全长cDNA序列,在大肠杆菌中成功表达61 kD目的蛋白。
今后还可通过定点突变、射线诱变等方法对几丁质脱乙酰酶进行改造,进一步优化其酶学性质、提高酶的耐受性等,为几丁质脱乙酰酶实现大规模工业应用奠定基础。
4 结论
本研究分离得到一株产几丁质脱乙酰酶海洋细菌MCDA02,鉴定为解角质素微杆菌。通过单因素和正交优化发酵条件,得到最优发酵条件为:诱导剂浓度为3%,培养基起始pH为7.0,装液量为20%,在温度为25℃的条件下发酵培养48 h。在此条件下,产酶量达到158.47 U/mL,是优化前产酶量的3.2倍。
[1]Khor E, Lim LY. Implantable applications of chitin and chitosan[J]. Biomaterials, 2003, 24(13):2339-2349.
[2]Hou J, Han J, Cai L, et al. Characterization of genes for chitin catabolism in Haloferax mediterranei[J]. Appl Microbiol Biotechnol, 2014, 98(3):1185-1194.
[3]Win N, Stevens W. Shrimp chitin as substrate for fungal chitin deacetylase[J]. Appl Microbiol Biotechnol, 2001, 57(3):334-341.
[4]Bartnicki-Garcia S, Bracker CE, Reyes E, et al. Isolation of chitosomes from taxonomically diverse fungi and synthesis of chitin microfibrils in vitro[J]. Experimental Mycology, 1978, 2(2):173-192.
[5]张菁菁, 董文宾, 缑敬轩. 几丁质脱乙酰酶菌株的筛选鉴定及酶学性质[J]. 食品工业科技, 2012, 33(6):244-246.
[6]Songsiriritthigul C, Lapboonrueng S, Pechsrichuang P, et al.Expression and characterization of Bacillus licheniformis chitinase(ChiA), suitable for bioconversion of chitin waste[J].Bioresource Technology, 2010, 101(11):4096-4103.
[7]Methacanon P, Prasitsilp M, Pothsree T, et al. Heterogeneous N-deacetylation of squid chitin in alkaline solution[J].Carbohydrate Polymers, 2003, 52(2):119-123.
[8]王皓, 吴丽, 朱小花. 甲壳素脱乙酰酶的研究概况及展望[J].中国生物工程杂志, 2015, 35(1):96-103.
[9]Wang SL, Peng JH, Liang TW, et al. Purification and characterization of a chitosanase from Serratia marcescens TKU011[J].Carbohydrate Research, 2008, 343(8):1316-1323.
[10]Kuroiwa T, Noguchi Y, Nakajima M, et al. Production of chitosan oligosaccharides using chitosanase immobilized on amylose-coated magnetic nanoparticles[J]. Process Biochemistry, 2008, 43(1):62-69.
[11]Zhao Y, Nguyen V, Jo G, et al. Purification and characterization of chitin deacetylase from Mortierella sp. DY-52[J]. Journal of Biotechnology, 2008, 136:S304.
[12]Chatterjee S, Chatterjee S, Chatterjee BP, et al. Enhancement of growth and chitosan production by Rhizopus oryzae in whey medium by plant growth hormones[J]. International Journal of Biological Macromolecules, 2008, 42(2):120-126.
[13]Cai J, Yang J, Du Y, et al. Purification and characterization of chitin deacetylase from Scopulariopsis brevicaulis[J]. Carbohydrate Polymers, 2006, 65(2):211-217.
[14]Li H, Xu H, Li S, et al. Strain improvement and metabolic flux modeling of wild-type and mutant Alcaligenes sp. NX-3 for synthesis of exopolysaccharide welan gum[J]. Biotechnology and Bioprocess Engineering, 2010, 15(5):777-784.
[15]Sun Y, Zhang J, Wu S, et al. Statistical optimization for production of chitin deacetylase from Rhodococcus erythropolis HG05[J].Carbohydrate Polymers, 2014, 102:649-652.
[16]Wang Y, Xia G, Wu C, et al. Porous chitosan doped with graphene oxide as highly effective adsorbent for methyl orange and amido black 10B[J]. Carbohydrate Polymers, 2015, 115:686-693.
[17]刘建军, 赵祥颖, 刘丽萍. 几丁质脱乙酰酶(CDA)的研究进展[J]. 山东食品发酵, 2007(4):40-46.
[18]Buchana RE, Gibbons NE. Bergey’s Manual of Determinative Bacteriology[M]. 8th Edition. Beijing:Science Press, 1984:382-533.
[19]应聪萍, 王瑶, 李永成. 一株产几丁质脱乙酰酶丝状真菌的发酵条件优化研究[J]. 食品工业科技, 2016(1):170-174.
[20]贾志娟. 北极深海沉积物宏基因组文库构建及一种新的几丁质脱乙酰酶基因鉴定[D]. 武汉:华中师范大学, 2012.
[21]闫晓平, 赵丹, 郭巍, 等. 美国白蛾几丁质脱乙酰酶的克隆,表达及酶学性质[J]. 中国农业科学, 2017, 50(5):849-858.
Screening and Identification of a Marine Bacterium Producing Chitin Deacetylase and Optimization of Its Fermentation Condition
LAI Jiang-li1LIU Shu1,2HU Sheng-yuan1GU Zhang-hui1WANG Shu-jun2FANG Yao-wei1,2
(1. College of Fisheries and Marine Life,Huaihai Institute of Technology,Lianyungang 222005;2. Jiangsu Marine Resources Development Research Institute,Lianyungang 222005)
A strain MCDA02 producing chitin deacetylase was screened with the media added nitro-N-acetyl aniline from the marine mud of Haizhou bay. Then,it was identified as Microbacterium keratanolyticum through morphological,biochemical characteristics,and 16S rDNA sequence analysis. With single-factor strategy and orthogonal experiments,the fermentation condition was optimized as followings:fermentation period of 48 h,initial pH of 7.0,culture temperature of 30℃,50 mL of liquid medium in a 250 mL flask,and 3% chitin. Under the optimized conditions,the highest chitin deacetylase activity of strain MCDA02 reached 158.47 U/mL,which was about 3.2 times of that before optimization. The results laid a foundation for further study of the chitin deacetylase from the strain MCDA02.
chitin deacetylase;marine bacteria;identification;fermentation condition
10.13560/j.cnki.biotech.bull.1985.2017-0413
2017-05-21
江苏省自然科学基金面上项目(BK20151282),江苏省高校“青蓝工程”,江苏省“六大人才高峰”第十二批高层次人才项目(swyy-195),中国博士后科学基金(160034),青岛博士后基金,第48批“留学归国人员”科研启动基金,淮海工学院科研创新基金(Z2014016)
来蒋丽,女,硕士研究生,研究方向:海洋微生物生物技术;E-mail:laijiangli199310@163.com
房耀维,男,副教授,研究方向:海洋微生物生物技术;E-mail:foroei@163.com
(责任编辑 李楠)

