国产水痘减毒活疫苗关键基因的序列分析和比较
权娅茹,陈 震,邱 平,崔晓雨,李长贵,袁力勇
中国食品药品检定研究院生物制品检定所呼吸道病毒疫苗室,北京 100050
·论著·
国产水痘减毒活疫苗关键基因的序列分析和比较
权娅茹,陈 震,邱 平,崔晓雨,李长贵,袁力勇
中国食品药品检定研究院生物制品检定所呼吸道病毒疫苗室,北京 100050
目的从基因水平分析国产水痘减毒活疫苗的安全性和一致性。方法对国内4家企业生产的水痘减毒活疫苗的关键基因(ORF38、ORF54和ORF62)进行PCR扩增和序列测定,并与Genbank公布的Dumas、P-Oka、V-Oka及国外上市疫苗Varilrix(GSK)、Varivax(Merck)的序列进行比对。结果4家国产水痘减毒活疫苗的ORF38和ORF54区基因序列一致,并且同V-Oka、Varilrix完全一致,与Varivax(Merck)有1处差异。4家国产水痘减毒活疫苗的ORF62区基因序列基本一致,与V-Oka、Varilrix(GSK)和Varivax(Merck)均存在个别核苷酸差异。结论4家国产水痘减毒活疫苗的ORF38、ORF54和ORF62区基因序列基本一致,稳定性较好,与V-Oka、Varilrix(GSK)和Varivax(Merck)存在较小差异,没有引入新的突变位点。
水痘疫苗;基因变异;ORF38;ORF54;ORF62
水痘是由水痘-带状疱疹病毒引起的一种高度传染病,常见于儿童。接种水痘疫苗是预防和控制水痘发生的最有效手段。Oka水痘病毒株(V-Oka)是世界卫生组织认可的唯一可用于水痘疫苗生产的减毒株,也是我国水痘减毒活疫苗的生产用毒株[1- 2]。V-Oka是将典型水痘患者疱疹液中分离得到的亲本野毒型Oka株(P-Oka)分别在人胚肺成纤维细胞、豚鼠胚胎成纤维细胞、人成纤维细胞株(WI- 38、MRC- 5)中多次传代获得,为未经纯化的混合株,在基因组中存在着多个混合位点[3- 4]。企业在生产水痘疫苗时因采用的毒种代次、传代用细胞基质等不同,疫苗成品的基因序列会存在差异[2,5]。研究显示,这种序列差异可能和水痘减毒活疫苗的毒力和其诱导的体液免疫反应有关[6- 7]。ORF38、ORF54和ORF62是流行病学和疫苗学分析常用的3个区域,因此,本研究对国内4家企业生产的水痘减毒活疫苗样本进行了ORF38、ORF54和ORF62区的基因序列测定,并与Dumas(NC001348)、P-Oka(AB097933)、V-Oka(AB097932)、英国葛兰素史克(GSK)生产的水痘减毒活疫苗(Varilrix)和美国默克(Merck)生产的水痘减毒活疫苗(Varivax)基因序列进行了比对,从而确定我国国内4家企业生产的水痘减毒活疫苗在这3个编码区的基因变异情况。
材料和方法
材料和试剂A、B、C、D 4家企业生产的水痘减毒活疫苗各1批,批号分别为201510031- 2、201510036- 1、201511073- 2和201602007。DNA提取试剂盒为德国QIAGEN公司的DNeasy®Blood & Tissue kit,核酸扩增为BioLabs的Q5热启动超保真2×Master Mix,DL2000 DNA分子量标准购自中国Takara公司。
引物及合成根据GenBank公布的VZV Dumas株(NC001348)基因序列设计引物,ORF38分3段扩增,ORF54分6段扩增,ORF62分9段扩增,共18对引物(表1),委托英潍捷基公司合成。
基因组DNA提取每批水痘减毒活疫苗取4瓶,分别用0.5 ml注射用水溶解,合并混匀后按照DNA提取试剂盒说明书提取水痘病毒基因组DNA。
目的DNA片段扩增及分析以提取的DNA为模板进行PCR扩增。25 μl反应体系:2 μl模板DNA,上下游引物(20 μmol/L)各1 μl,12.5 μl 2×Master Mix,8.5 μl去离子水。反应条件:95℃5 min;95℃ 1 min,55℃ 30 s,72℃ 1 min,共30个循环;72℃延伸10 min。PCR产物经1.5%琼脂糖凝胶电泳鉴定,委托天一辉远(北京)生物科技有限公司进行DNA序列测定。

表 1 PCR引物Table 1 PCR primers used in the analysis
以上引物用NCBI网站的primer-BLAST软件自行设计
The above primers were designed by primer-BLAST software of the NCBI website
结 果
目的基因扩增产物鉴定1.5%琼脂糖凝胶电泳显示,4家企业生产的水痘减毒活疫苗均可扩增出18条特异性条带,大小与预期相符。以A企业的水痘减毒活疫苗为例,结果见图1。
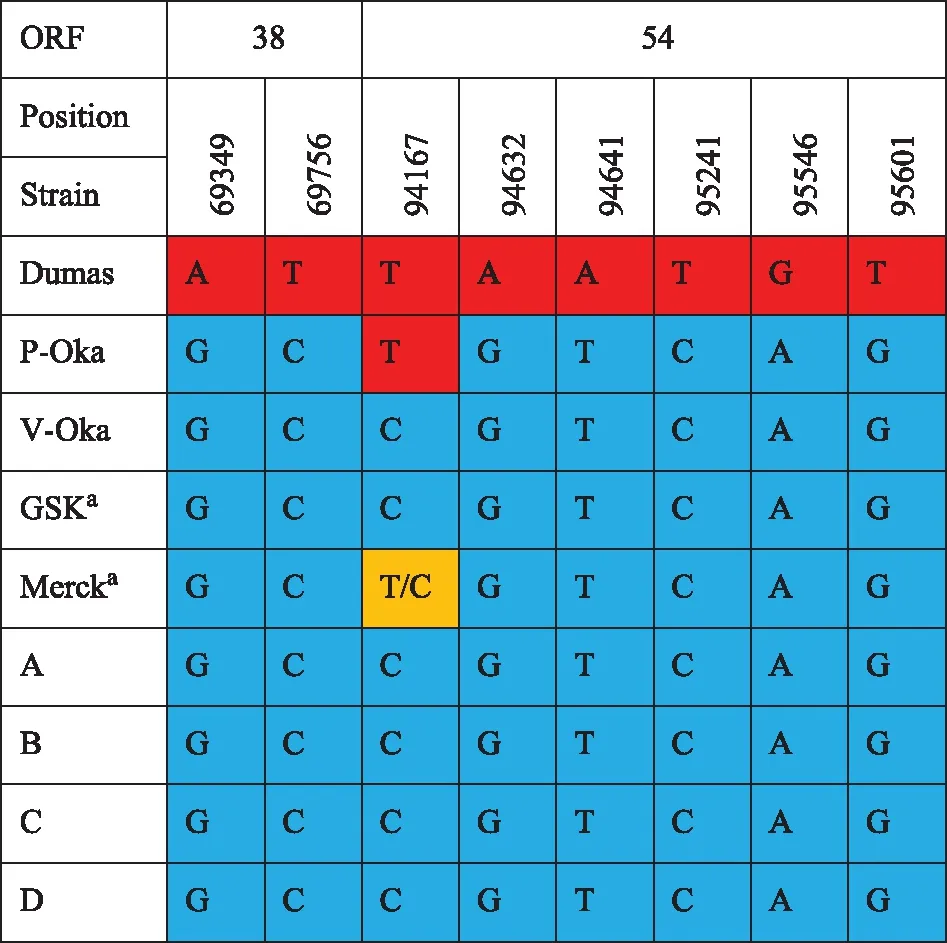
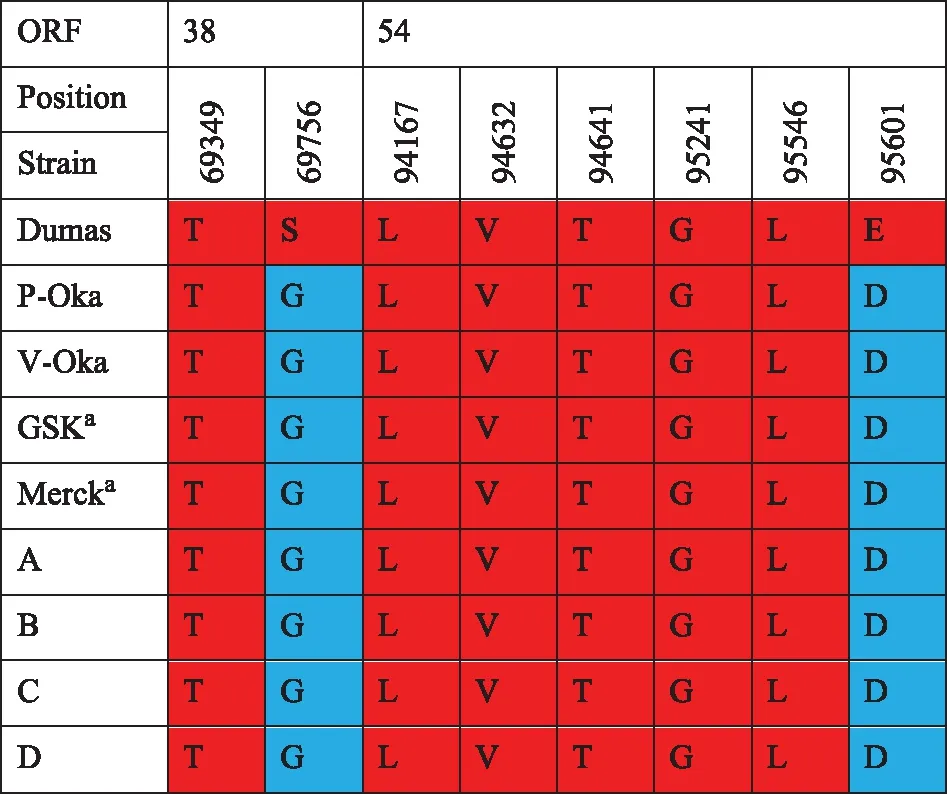
ORF38基因序列分析ORF38全基因序列测定和分析结果表明:国内不同企业的水痘减毒活疫苗,其ORF38的核苷酸和氨基酸序列完全一致,并且与GenBbank公布的p-OKA、V-Oka、Varilrix(GSK)和Varivax(Merck)同源性100%。与野生型Dumas相比,仅有2个位点的碱基发生突变,即69349位点由A突变为T,69756位点由T突变为C。前者为沉默突变,但使疫苗株PstⅠ酶切位点消失,后者则使氨基酸发生突变,由丝氨酸(S)突变为甘氨酸(G)(图2、3)。
ORF54基因序列分析与ORF38相似,国内4家企业生产的水痘减毒活疫苗ORF54基因序列完全一致,并且与GenBank公布的V-Oka和Varilrix序列同源性100%,与Varivax有1个碱基的差异,即国产水痘减毒活疫苗在94167位点是单碱基C,而Varivax是T/C同时存在,但未引起氨基酸突变。国产水痘疫苗株在ORF54与Dumas有6个碱基的差异(图2、3),其中95241位由野生型毒株的T突变为C,95601由T突变为G,前者未引起氨基酸变异,但导致疫苗株增加了1个BglⅠ酶切位点,后者则使谷氨酸(E)突变为天冬氨酸(D)。
M:DNA分子量标记;1:ORF381产物;2:ORF382产物;3:ORF383产物;4:ORF541产物;5:ORF542产物;6:ORF543产物;7:ORF544产物;8:ORF545产物;9:ORF546产物;10:ORF621产物;11:ORF622产物;12:ORF623产物;13:ORF624产物;14:ORF625产物;15:ORF626产物;16:ORF627产物;17:ORF628产物;18:ORF629产物
M:DNA Marker DL2000;1:product of ORF381;2:product of ORF382;3:product of ORF383;4:product of ORF541;5:product of ORF542;6:product of ORF543;7:product of ORF544;8:product of ORF545;9:product of ORF546;10:product of ORF621;11:product of ORF622;12:product of ORF623;13:product of ORF624;14:product of ORF625;15:product of ORF626;16:product of 627;17:product of ORF628;18:product of ORF629
图1 水痘减毒活疫苗ORF38、ORF54和ORF62扩增结果(A企业水痘减毒活疫苗)
Fig1 Amplification results of ORF38,ORF54,and ORF62 sequences of a live attenuated varicella vaccine (live attenuated varicella vaccine produced in A company)
a:Tillieux SL et al,2008
图2 不同来源的水痘毒株ORF38和54核苷酸序列比较
Fig2 Comparison of nucleotide sequences of ORF38 and 54 of varicella virus from different sources
a:Tillieux SL et al,2008
图3 不同来源的水痘毒株ORF38和54氨基酸序列比较
Fig3 Comparison of amid acid sequences of ORF38 and 54 of varicella virus from different sources
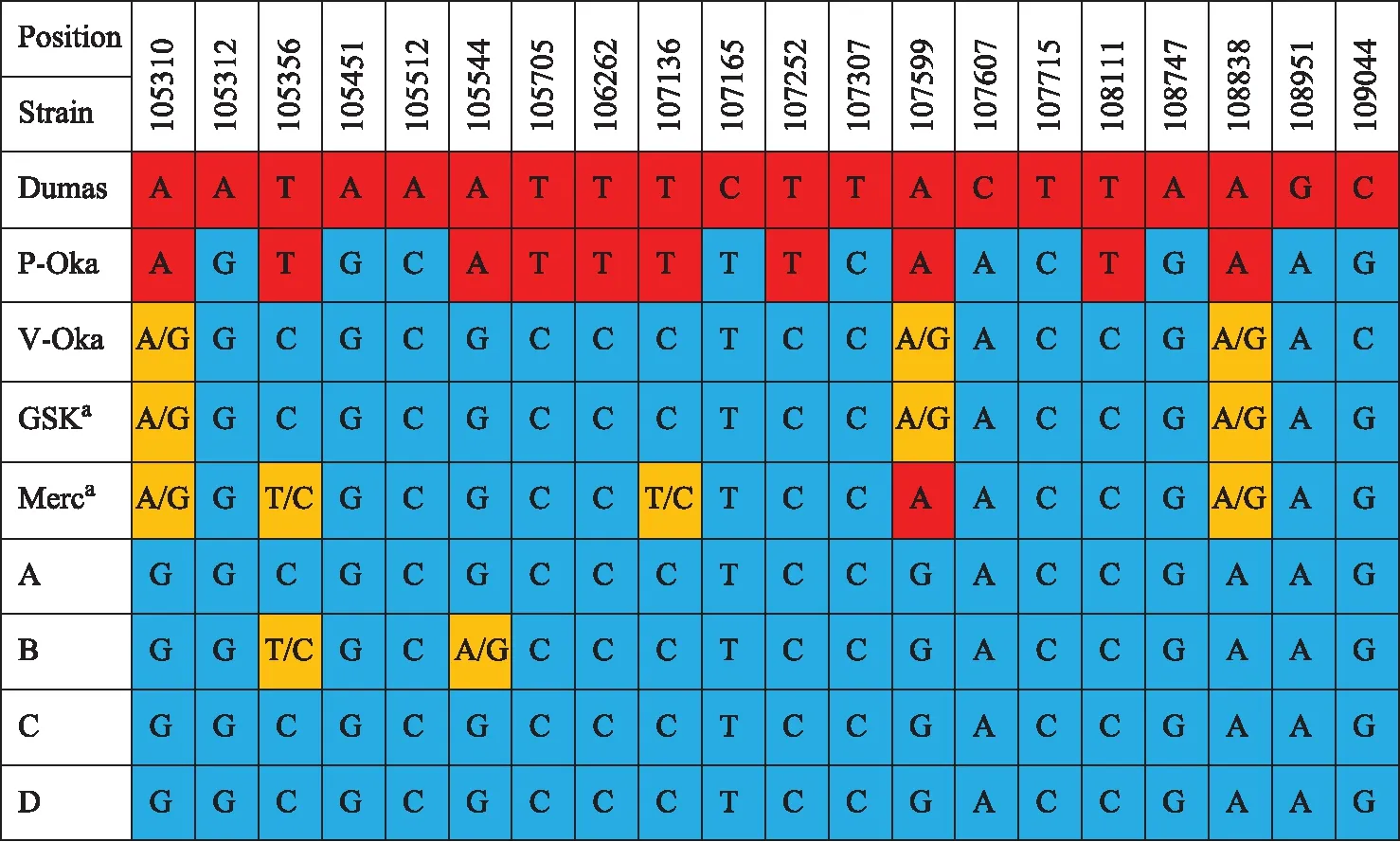
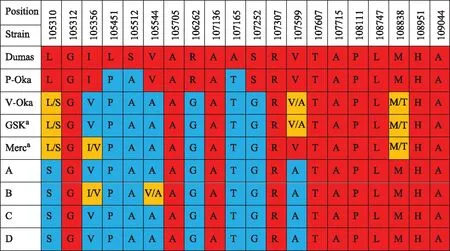
ORF62基因序列分析序列测定结果显示,与Dumes株和P-OKA株相比,疫苗株在ORF62突变较多,但国内4家企业的水痘减毒活疫苗基因序列基本一致,与Genbank公布的V-Oka、Varilrix、Varivax序列存在个别核苷酸差异。4家企业生产的水痘减毒活疫苗、V-Oka、Varilrix(GSK)和Varivax(Merck)的ORF62基因序列同Dumas相比,均有20个突变位点。B企业生产的水痘减毒活疫苗在其中一些突变位点以野毒型和减毒型碱基共存,同V-Oka、Varilrix(GSK)和Varivax(Merck)。而A、C和D 3家企业生产的水痘减毒活疫苗在这20个位点均以一种碱基存在。其中,106262、107136和107252位点突变分别在4家企业生产的水痘减毒活疫苗中引入了SmaⅠ、BssHⅡ和NaeⅠ酶切位点。另外,20个突变位点中10个位点的碱基突变导致了氨基酸的突变,其余10个位点为沉默突变(图4、5)。
a:Tillieux SL et al,2008
图4 不同来源的水痘毒株ORF62核苷酸序列比较
Fig4 Comparison of nucleotide sequences of ORF62 of varicella virus from different sources
a:Tillieux SL et al,2008
图5 不同来源的水痘毒株ORF62氨基酸序列比较
Fig5 Comparison of amid acid sequences of ORF62 of varicella virus from different sources
讨 论
水痘减毒活疫苗V-OKA株是由日本科学家在20世纪70年代获得的疫苗株病毒,用其生产的疫苗为接种者提供了持续性保护,显著降低了水痘病毒的感染率。目前,国内外上市的水痘疫苗均是由V-Oka制备的,包括Merck生产的Varivax、GSK生产的Varilrix和我国4家企业生产的水痘减毒活疫苗。V-Oka是通过将P-Oka在组织细胞中系列传代来减毒的,未克隆化,加上不同企业使用的病毒代次不同、细胞基质(包括人二倍体细胞MRC- 5和2BS)和生产工艺也有差异,这使得不同企业生产的水痘减毒活疫苗基因也存在差异。为了考察我国企业生产的水痘减毒活疫苗中病毒基因的变异情况,本研究对4家国产水痘减毒活疫苗中的ORF38、54和62区基因进行了序列测定和分析。
ORF38、ORF54、ORF62是水痘减毒活疫苗监测和流行病学研究选用的3个基因区域,并被用来区分水痘病毒野毒株和疫苗株。研究显示,4家企业生产的水痘减毒活疫苗的ORF38、54区基因序列完全相同,并且与V-Oka和Varilrix(GSK)序列保持一致,与Varivax(Merck)在ORF54区有1处差异。A、C和D企业生产的水痘减毒活疫苗的ORF62区序列完全相同,与B企业有2处差异,与V-Oka、Varilrix(GSK)和Varivax(Merck)也均有几处差异,但没有引入新的突变位点。
ORF38区的69349(PstⅠ)、ORF54区的95241(BglⅠ)和ORF62区的106262(SmaⅠ)、107136(BssHⅡ)、107252(NaeⅠ)是区分水痘病毒野毒株和疫苗株常用酶切位点[8- 13]。本研究显示我国4家企业生产的水痘减毒活疫苗在这5个位置均出现突变,产生了疫苗株特有的SmaⅠ、BssHⅡ、NaeⅠ和BglⅠ酶切位点,缺失了野毒株特有的PstⅠ酶切位点。结果同V-Oka和Varilrix(GSK)相吻合,Varivax(Merck)在107136位点以T/C混合碱基存在,未产生疫苗株特有的BssHⅡ酶切位点。
V-Oka全基因组中1/3以上碱基突变落在ORF62区域,ORF62编码即刻早期蛋白(IE62),IE62是水痘病毒的主要转录激活因子,直接参与病毒复制。研究显示ORF62在V-Oka减毒过程中可能起着关键的作用[14- 20]。Gomi等[16]发现ORF62区中105310、105356、105544、106262、107252、107599、107797和108838位点碱基突变导致的氨基酸突变可阻碍水痘病毒自身复制和在感染细胞中的扩散。Quinlivan等[6]发现水痘疫苗中37个SNP位点中的4个最可能和皮疹爆发有关,分别为ORF51区的89734、105169,ORF62区的105356和107797。本研究显示,我国4家企业生产的水痘减毒活疫苗在105310、105356、105544、106262、107252和107599位点均发生了碱基突变和由其导致的氨基酸突变,同V-Oka和Varilrix(GSK)一致,Varivax(Merck)在107599位点以野毒型碱基存在;在108838位点未发生碱基突变,以野毒型碱基A存在,而V-Oka、Varilrix(GSK)和Varivax(Merck)以A/G存在。107797位点在本研究中并未被确认为SNP位点,同Tillieux等[5]和Gomi等[16]报道的一致。
综上,本研究结果显示,我国4家企业虽然用毒株代次、细胞基质和生产工艺不同,但生产的水痘减毒活疫苗的ORF38、54、62区基因序列基本一致,与V-Oka、Varilrix(GSK)和Varivax(Merck)基因突变位点一致,没有引入新的突变位点,说明国产水痘减毒活疫苗的ORF38、54、62区基因序列稳定性很好。
[1] 梁慧颖,邹勇.冻干水痘减毒活疫苗发展现状及趋势[J].中国新药杂志,2012,21(10):1076- 1161.
[2] Sauerbrei A,Zell R,Harder M,et al. Genotyping of different varicella vaccine strains [J].J Clin Virol,2006,37(2):109- 117.doi:10.1016/j.jcv.2006.07.002.
[3] Takahashi M,Okuno Y,Otsuka T,et al. Development of a live attenuated varicella vaccine [J].Biken J,1975,18(1):25- 33.
[4] Cohrs RJ,Gilden DH,Gomi Y,et al. Comparison of virus transcription during lytic infection of the oka parental and vaccine strains of varicella-zoster virus [J].J Virol,2006,80(5):2076- 2082.doi:10.1128/JVI.80.5.2076- 2082.2006.
[5] Tillieux SL,Halsey WS,Thomas ES,et al. Complete DNA sequence of two oka strain varicella-zoster virus genomes [J].J Virol,2008,82(22):11023- 11044.doi:10.1128/JVI00777- 08.
[6] Quinlivan ML,Gershon AA,AlBassam MM,et al. Natural selection for rash-forming genotypes of the varicella-zoster vaccine virus detected within immunized human hosts [J].PNAS,2007,104(1):208- 212.doi:10.1073/pnas. 0605688104.
[7] Lau YL,Vessey SJ,Chan IS,et al. A comparison of safety,tolerability and immunogenicity of oka/merck varicella vaccine and VARILRIX in healthy children [J].Vaccine,2002,20(23- 24):2942- 2949.doi:10.1016/S0264- 410X(02)00245- 1.
[8] LaRussa P,Lungu O,Hardy I,et al. Restriction fragment length polymorphism of polymerase chain reaction products from vaccine and wild-type varicella-zoster virus isolates [J].J Virol,1992,66(2):1016- 1020.
[9] LaRussa P,Steinberg S,Arvin A,et al. Polymerase chain reaction and restriction fragment length polymorphism analysis of varicella-zoster virus isolates from the United States and other parts of the world [J].J Infect Dis,1998,178(1):S64-S66.
[10] Loparev VN,Argaw T,Krause PR,et al. Improved identification and differentiation of varicella-zoster virus (VZV) wild-type strains and an attenuated varicella vaccine strain using a VZV open reading frame 62-based PCR [J].J Clin Microbiol,2000,38(9):3156- 3160.
[11] Hondo R,Yogo Y,Yoshida M,et al. Distribution of varicella-zoster virus strains carrying a PstⅠ-site-less mutation in Japan and DNA change responsible for the mutation [J].Jpn J Exp Med,1989,59(6):233- 237.
[12] Quinlivan ML,Jensen NJ,Radford KW,et al. Novel genetic variation identified at fixed loci in ORF62 of the oka varicella vaccine and in a case of vaccine-associated herpes zoster [J].J Clin Microbiol,2012,50(5):1533- 1538.doi:10.1128/JCM.06630- 11.
[13] Quinlivan M,Gershon AA,Steinberg SP,et al. An evaluation of single nucleotide polymorphisms used to differentiate vaccine and wild type strains of varicella-zoster virus [J].J Med Virol,2005,75(1):174- 180.doi:10.1002/jmv.20253.
[14] Yamanishi K.Molecular analysis of the oka vaccine strain of varicella-zoster virus[J].J Infect Dis,2008,197(s2):S45-S48.doi:10.1086/522122.
[15] Gomi Y,Ozaki T,Nishimura N,et al. DNA sequence analysis of varicella-zoster virus gene 62 from subclinical infections in healthy children immunized with the oka varicella vaccine[J].Vaccine,2008,26(44):5627- 5632.doi:10.1016/j. vaccine.2008.07.069.
[16] Gomi Y,Sunamachi H,Mori Y,et al. Comparison of the complete DNA sequences of the oka varicella vaccine and its parental virus [J].J Virol,2002,76(22):11447- 11459.doi:10.1128/JVI.76.22.11447- 11459.2002.
[17] Kinchington PR,Hougland JK,Arvin AM,et al. The varicella-zoster virus immediate-early protein IE62 is a major component of virus particles [J].J Virol,1992,66(1):359- 366.
[18] Perera LP,Mosca JD,Ruyechan WT,et al. A major transactivator of varicella-zoster virus,the immediate-early protein IE62,contains a potent N-terminal activation domain [J].J Virol,1993,67(8):4474- 4483.
[19] Sadzot-Delvaux C,Rentier B.The role of varicella zoster virus immediate-early proteins in latency and their potential use as components of vaccines [J].Arch Virol Suppl,2001,17(17):81- 89.doi:10.1007/978- 3- 7091- 6259- 0_9.
[20] Gomi Y,Imagawa T,Takahashi M,et al. Comparison of DNA sequence and transactivation activity of open reading frame 62 of oka varicella vaccine and its parental viruses [J].Arch Virol Suppl,2001,17(17):49- 56.doi:10.1007/978- 3- 7091- 6259- 0_6.
SequenceAnalysisandComparisonoftheKeyGenesofDomesticLiveAttenuatedVaricellaVaccine
QUAN Yaru,CHEN Zhen,QIU Ping,CUI Xiaoyu,LI Changgui,YUAN Liyong
Division of Respiratory Virus Vaccines,Institute for Biological Product Control,National Institutes for Food and Drug Control,Beijing 100050,China
YUAN Liyong Tel:010- 67095423,E-mail:yuanly@nifdc.org.cn
ObjectiveTo analyze the safety and consistency of domestic live attenuated varicella vaccines (LAVVs) at gene level.MethodsThe key genes (ORF38,ORF54,and ORF62) of LAVVs produced by four Chinese manufacturers were amplified by polymerase chain reaction (PCR) and sequenced.The sequencing results were compared with the sequences of Dumas,P-Oka,and V-Oka strains in GenBank and with the sequences of Varilrix (GSK) and Varivax (Merck).ResultsThe ORF38 and ORF54 gene sequences of four domestic LAVVs were the same as each other and completely consistent with the sequences of V-Oka and Varilrix;however,it was different from Varivax (Merck) at one site.The ORF62 gene sequences of four domestic LAVVs were similar,and had individual nucleotide differences with V-Oka,Varilrix(GSK),and Varivax (Merck).ConclusionsThe sequences of ORF38,ORF54,and ORF62 of four domestic LAVVs are almost the same,showing good stability.They have small differences with V-Oka,Varilrix(GSK),and Varivax (Merck),without introducing new mutations.
varicella vaccine;gene mutation;ORF38;ORF54;ORF62
袁力勇 电话:010- 67095423,电子邮件:yuanly@nifdc.org.cn
R373.1+1
A
1000- 503X(2017)05- 0637- 06
10.3881/j.issn.1000- 503X.2017.05.007
ActaAcadMedSin,2017,39(5):637-642
2016- 12- 01)

