α-变形菌纲新的保守特征性插入缺失
吕志堂, 王 静, 周燕霞, 张维维
(1.河北大学 生命科学学院 河北省微生物多样性研究与应用实验室,河北 保定 071002;2.药物化学与分子诊断教育部重点实验室(河北大学),河北 保定 071002)
·大家专版·
α-变形菌纲新的保守特征性插入缺失
吕志堂1,2, 王 静1, 周燕霞1, 张维维1
(1.河北大学 生命科学学院 河北省微生物多样性研究与应用实验室,河北 保定 071002;2.药物化学与分子诊断教育部重点实验室(河北大学),河北 保定 071002)
在基于16S rRNA基因的分类系统中α-变形菌被定为变形菌门的一个纲。本文根据系统发育基因组学分析,首次发现并鉴定了α-变形菌纲普遍存在的5个保守特征性插入缺失(CSIs)。23S rRNA基因具有1个98~105 nt的特征性缺失和1个5~7 nt的特征性插入,DNA复制解旋酶(DnaB)具有1个17~29 aa的特征性插入,精氨酰-tRNA合成酶(ArgRS)具有1个15 aa的特征性插入,DNA促旋酶A亚基(GyrA)具有1个27~63 aa的特征性插入。α-变形菌纲的这些CSIs使得该纲与包括变形菌门其他纲在内的其他细菌显著区分开来,为该群细菌系统发育及系统分类研究提供了可靠手段。
α-变形菌纲;保守特征性插入缺失;系统发育基因组学
α-变形菌纲(α-Proteobacteria)是变形菌门(Proteobacteria)7个纲之一,占据了包括多种陆地和海洋环境的广泛的生态位,构成了细菌域一个非常大且多样性丰富的类群[1-3]。在《伯杰氏系统细菌学手册》(第2版)中,该纲被分为7个目,即红螺菌目(Rhodospirillales)、立克次氏体菌(Rickettsiales)、红细菌目(Rhodobacterales)、鞘氨醇单胞菌目(Sphingomonadales)、柄杆菌目(Caulobacterales)、根瘤菌目(Rhizobiales)和短小盒菌目(Parvularculales)[4]。该纲细菌在形态、生理、代谢和细胞分裂机制方面呈现出极其丰富的多样性特征,与真核生物密切相关,在工业、农业和医学领域都极为重要[1,5-7]。目前观点认为,α-变形菌纲起源于ε和δ-变形菌纲之后,β和γ-变形菌纲之前[1,8-9]。尽管这个类群在16S rRNA和其他基因系统发育上与其他主要细菌类群显著分开,但是没有一套确切的分子标准可以给予该群明确的界定[1,5,8-11]。因此,如何从分子特征将α-变形菌纲与其他细菌类群予以区别成为亟待解决的问题。加拿大学者Gupta及其同事建立了一种基于保守特征性插入缺失(conserved signature indels, CSIs)研究细菌系统发育和系统分类的有效方法,可以在分子水平区分细菌域的不同类群,并已经鉴定了放线细菌门(Actinobacteria)、衣原体门(Chlamydiae)、蓝细菌门(Cyanobacteria)、奇异球菌-栖热菌门(Deinococcus-Thermus)、产液菌门(Aquificae)等许多细菌类群特有的CSIs[8,12-16]。目前,该方法已经广泛用于从种到门各个分类等级细菌的系统发育和系统分类研究,他们发现的放线菌23S rRNA基因特有的CSI甚至成为放线细菌门建立的主要依据之一[12-20]。Gupta研究组已经描述了α-变形菌纲一些普遍存在的蛋白和几个推测的蛋白中特有的CSIs,可以作为该类群的独有分子特征,认为α-变形菌纲与细菌域其他类群显著不同,建议给予其细菌域门的地位[9,21-22]。随着越来越多的微生物基因组被测序和生物信息学技术的不断发展,在更大范围内通过比较基因组学发现不同微生物类群的分子特征,尤其是CSIs已经成为可能。本文报道α-变形菌纲5个新发现的CSIs。
1 材料与方法
1.1 材料
用于CSIs筛选的代表菌株涵盖细菌域的25个门的所有主要类群,包括变形菌门α-变形菌纲60属、β-变形菌纲8属、γ-变形菌纲28属、δ-变形菌纲18属、ε-变形菌纲4属、ξ-变形菌纲1属和酸硫杆菌纲(Acidithiobacillia) 1属,以及其他类群228属。所有菌株的基因信息取自NCBI Reference Sequence数据库[23](表1,由于篇幅所限,未列出各基因组序列号)。
表1 本文所用基因组数据在细菌域各门分布情况
Table 1 The distribution of genome sequences used in this study within the domain Bacteria
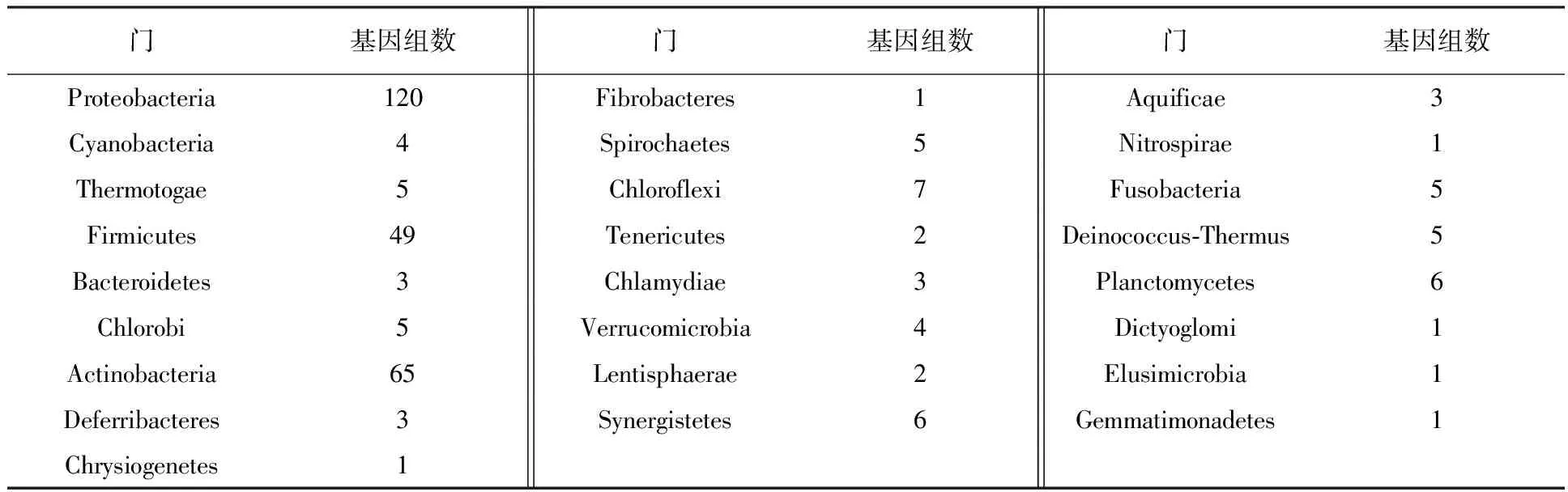
门基因组数门基因组数门基因组数Proteobacteria120Fibrobacteres1Aquificae3Cyanobacteria4Spirochaetes5Nitrospirae1Thermotogae5Chloroflexi7Fusobacteria5Firmicutes49Tenericutes2Deinococcus⁃Thermus5Bacteroidetes3Chlamydiae3Planctomycetes6Chlorobi5Verrucomicrobia4Dictyoglomi1Actinobacteria65Lentisphaerae2Elusimicrobia1Deferribacteres3Synergistetes6Gemmatimonadetes1Chrysiogenetes1
1.2 方法
首先确定筛选CSIs的基本原则为该基因普遍存在于细菌域各类群中,功能保守且多为单拷贝基因(直系同源基因),即CSIs应从核心基因中筛选。为避免基因组错误注释或假基因的影响,核心蛋白及rRNA基因序列信息分别用MUSCLE软件[24]和Infernal软件[25]进行多重序列比对。某些代表菌株部分核心蛋白序列无法找到,可利用TBLASTN在未进行注释的基因组中检索,得到由基因组错误注释或漏注释导致的核心蛋白序列信息的缺失。比对后寻找潜在的α-变形菌纲的CSIs。CSIs的位置根据大肠埃希菌(Escherichiacoli)相关序列确定。为确保CSIs的可靠性,所有潜在的CSIs两侧均有保守区存在[8,12,16,26]。分别选择含有及不含有CSIs的同源蛋白,截取包括两侧保守区在内的插入缺失所在区域,在NCBI数据库中进行Blast分析,根据分析结果最终确定特征序列。
2 结果与分析
2.1 α-变形菌纲23S rRNA基因的CSIs
结果表明,α-变形菌纲菌株23S rRNA基因中存在98~105 nt缺失,该缺失未在其他细菌类群中发现(部分比对结果见图1)。α-变形菌纲23S rRNA基因中的特异缺失可将其与变形杆菌门其他纲明显区别。值得注意的是,α-变形菌纲23S rRNA中这段特异缺失与之前介绍的放线菌纲100 nt特异插入序列毗邻。因此,含有该缺失的片段可利用放线菌纲23S rRNA基因特异引物扩增得到,通过琼脂糖凝胶电泳即可将α-变形杆菌纲菌株与其他细菌类群分开[15]。


图1 α-变形菌纲23S rRNA基因中特有的98~105 nt插入缺失序列Fig.1 The 98~105 nt characteristicindels of α-Proteobacteria within 23S rRNA gene sequence
此外,除柄杆菌目(Caulobacterales)和生丝单胞菌科(Hyphomonadaceae)外,其他α-变形菌23S rRNA基因序列中还存在5~7 nt 的插入(篇幅所限,本文仅给出部分比对结果,见图2)。这段特征插入的缺失,显示柄杆菌目和生丝单胞菌科之间在进化上存在共性。
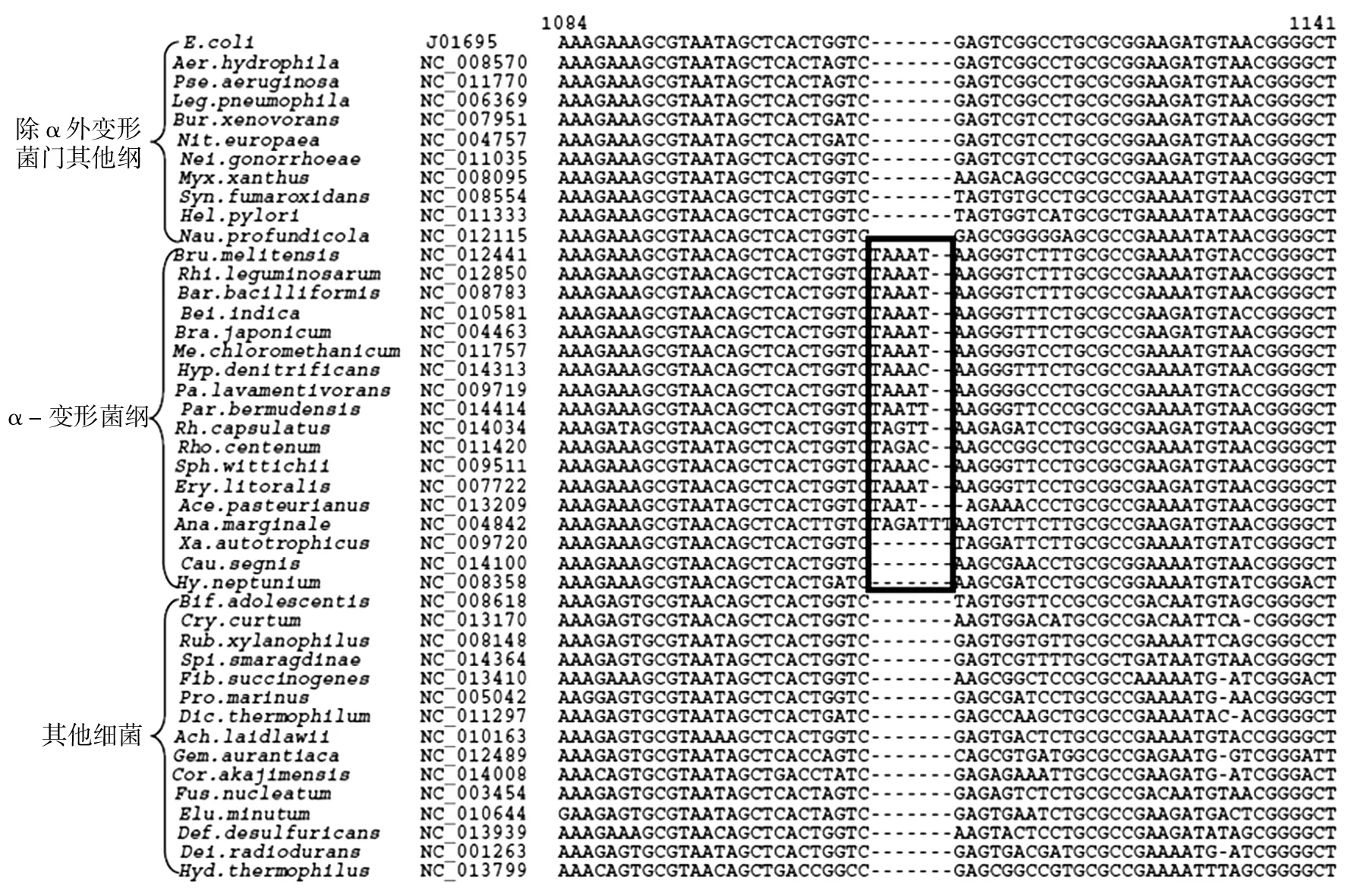
图2 α-变形菌纲23S rRNA基因中特有的5~7 nt插入缺失序列Fig.2 The 5~7 nt characteristicindels of α-Proteobacteria within 23S rRNA gene sequences
2.2 α-变形菌纲DnaB中的CSIs
2005年,Gupta[9]发现在α-变形菌纲DNA复制解旋酶蛋白(DnaB)保守区存在8~14 aa插入,这段插入为α-变形菌纲所特有。本研究发现,在DnaB碳末端,α-变形菌纲存在另一段17~29 aa保守特征性插入,部分比对序列见图3。除鞘氨醇单胞菌目(Sphingomonadales)和红螺菌目(Rhodospirillales)该段特征插入为25~29 aa外,其余α-变形菌纲成员特异插入长度为17/18 aa。另外,在α-变形菌纲特异插入序列相同位置,ε-变形菌纲存在一段不同长度的插入序列,除幽门螺杆菌(Helicobacterpylori)该段插入为34 aa,包括该属其他菌株在内的ε-变形菌均含有21 aa插入。由于α-变形菌纲和ε-变形菌纲中存在的插入序列长度不同且序列相似性低,因此基本可以判断这两段序列是独立起源的。
2.3 α-变形菌纲ArgRS中的CSIs
根据HIGH-loop赖氨酸的有无,Sekine等[27]将精氨酰-tRNA合酶(ArgRS)分为两个主要类群:KMSK群和HIGH群,且这两类同源蛋白序列相似性极低。除丙杆菌目(Caulobacterales)、细小棒菌目(Parvularculales)和生丝单胞菌科(Hyphomonadaceae)外,α-变形菌纲代表菌株所含ArgRS均属于HIGH群,并且在该同源蛋白中均存在15 aa的插入。值得指出的是,这段15 aa的插入序列同样存在于ζ-变形菌纲唯一已知种氧化亚铁深海菌(Mariprofundusferrooxydans)中,而细菌域其他类群菌株内同源蛋白中未发现存在这段CSIs序列(部分代表菌株比对序列信息见图4)。同时,在该插入序列中存在保守肽段Pro-Lys-Gly (PKG),因此,进一步探究α-变形菌纲同源蛋白中存在的大片段的氨基酸插入及保守肽段的功能可能对了解该蛋白的作用机理具有重要作用。

图4 α-变形菌纲精氨酰-tRNA合酶中保守特征性插入缺失序列Fig.4 The CSIs of α-Proteobacteria within amino acids sequencesof ArgRS
2.4 α-变形菌纲GryA中的CSIs
Gupta[8]曾报道在α、β、γ-变形菌纲DNA促旋酶A亚基(GyrA)同源蛋白中存在大片段的氨基酸插入,其中在α-变形杆菌纲中存在30 aa插入,而在β、γ-变形菌纲中存在34 aa插入。本研究结果显示,除γ-变形菌纲中存在插入序列与报道一致外,α、β-变形菌纲中存在的插入序列长度与报道不符。其中在α-变形杆菌纲同源蛋白相同位置处,仅存在26 aa的插入。绝大多数β-变形菌纲代表菌株插入片段长度为34或36 aa,而伯克霍尔德氏菌目(Burkholderiales)代表菌株同源蛋白相同位置处存在41或42 aa插入。另外,在酸杆菌门(Acidobacteria)和浮霉状菌门(Planctomycetes)代表菌株同源蛋白相同位置处分别存在34个和28 aa插入(部分序列比对信息见图5)。

图5 变形菌门各纲DNA促旋酶A亚基中保守特征性插入缺失序列Fig.5 The CSIs of each class of the phylum Proteobacteria within amino acids sequencesof GyrA
3 讨 论
1992年Roller等[28]首次报道,与大肠埃希菌相比荚膜红细菌(Rhodobactercapsulatus)23S rRNA基因存在103 nt序列缺失。之后,Ludwig等[29]提出23S rRNA中80 nt的缺失未在其他类群中发现,因此,可能是α-变形菌纲代表菌株的特征序列。随着测序技术的发展,23S rRNA序列信息骤增,可以利用更多数据信息确定该缺失是否为α-变形菌纲菌株独有特征。本研究通过对迄今为止数量最多、覆盖最全的细菌基因组比对,证实α-变形菌纲23S rRNA中存在98~105 nt缺失这一典型的CSIs,不仅可以区别于变形菌门其他纲,也与细菌域其他门显著区别。这一发现支持将α-变形菌纲提升至细菌域一门水平的建议[9,21-22],同时也为这一类群菌株系统发育及系统分类提供了典型的分子特征。
本研究还首次发现了α-变形菌纲DnaB、ArgRS和GyrA各含有1个CSIs。根据聚类结果,α-变形菌纲柄杆菌目(Caulobacterales)、细小棒菌目(Parvularculales)和生丝单胞菌科(Hyphomonadaceae)ArgRS属于KMSK群,其余属于HIGH群;加之生丝单胞菌科和柄杆菌目成员23S rRNA基因均缺少5~7 nt的特征性插入,说明生丝单胞菌科与柄杆菌目比与红杆菌目(Rhodobacterales)关系更近。
新发现的α-变形菌纲这些CSIs使得该纲与包括变形菌门其他纲在内的其他细菌显著区分开来,为该群细菌系统发育及系统分类研究提供了可靠手段。
同时,研究表明,幽门螺杆菌的DNA复制解旋酶(HpDnaB)碳末端34个插入氨基酸对维持该蛋白结构完整性和活性都是必须的[30]。然而,去除大肠埃希菌DNA复制解旋酶(EcDnaB)碳末端55个氨基酸并不会影响该蛋白体外ATP酶活和解旋酶活[31]。我们发现α-变形菌纲和ε-变形菌纲DnaB中存在独立起源、长度不等、总体较长但序列相似性很低的CSIs,它们可能对该蛋白结构及酶活也具有独特作用。同样,研究在α-变形菌纲ArgRS、GyrA中存在的大片段的氨基酸插入及保守肽段的功能,可能对了解这些蛋白的作用机理具有重要作用。
[1] Kersters K, De Vos P, Gillis M.Introductionto theProteobacteria. InTheProkaryotes: An EvolvingElectronic Resource for the Microbiological Community 3rd edition. Editedby: DworkinM[M]. New York, Springer-Verlag; 2003. Release 3.12 http//141.150.157.117:8080/prokPUB/chaprender/jsp/showchap.jsp?chapnum=379
[2] Giovannoni SJ, Tripp HJ, Givan S, et al. Genome streamlining in a cosmopolitan oceanic bacterium[J]. Science, 2005, 309:1242-1245.
[3] Williams KP, Kelly DP. Proposal for a new class within the phylumProteobacteria,Acidithiobacilliaclassis nov., with the type orderAcidithiobacillales, and emended description of the classGammaproteobacteria[J].Int J SystEvolMicrobiol, 2013, 63 (8): 2901-2906.
[4] Krieg NR, Brenner DJ, Staley JT.TheProteobacteria. In: Bergey’sManual of Systematic Bacteriology(2nd edn), Volume 2[M]. New York: Springer, 2005: 16.
[5] Stackebrandt E, Murray RGE,Trüper HG.Proteobacteriaclassisnov., a Name for the phylogenetic taxon that includes the "purple bacteria and their relatives"[J].IntJSystBacteriol, 1988, 38:321-325.
[6] Dworkin M, Falkow S, Rosenberg E, et al.Fredricks DN: Introduction to the Rickettsiales and other intracellular prokaryotes. In The Prokaryotes: A Handbook on theBiology of Bacteria 3rd edition edition.[M]. New York:Springer,2006:457-466.
[7] Skorpil P, Broughton WJ. Molecular interactions between Rhizobium and legumes[J].Prog Mol Subcell Biol,2006, 41:143-164.
[8] Gupta RS. The phylogeny of Proteobacteria: relationships toother eubacterial phyla and eukaryotes[J]. FEMS Microbiol Rev, 2000, 24:367-402.
[9] Gupta RS. Protein signatures distinctive of Alpha proteobacteriaand its subgroups and a model for Alpha proteobacterialevolution[J]. Crit Rev Microbiol, 2005, 31:101-135.
[10]Battistuzzi FU, Feijao A, Hedges SB. A genomic timescale ofprokaryote evolution: Insights into the origin of methanogenesis, phototrophy,and the colonization of land[J].BMC.Evol.Biol, 2004, 4: 44.
[11]Stepkowski T, Czaplinska M, Miedzinska K, et al. The variable part of the dnaK gene as an alternative marker for phylogenetic studies of rhizobia and related alpha Proteobacteria[J]. Syst Appl Microbiol,2003, 26: 483-494.
[12]Gupta RS. Protein phylogenies and signature sequences: areappraisal of evolutionary relationships among archaebacteria,eubacteria, and eukaryotes[J]. Microbiol Mol Biol Rev, 1998, 62: 1435-1491.
[13]Gupta RS, Griffiths E. Critical Issues in Bacterial Phylogenies[J]. Theor Popul Biol, 2002, 61:423-434.
[14]Gupta RS, Pereira M, Chandrasekera C, et al. Molecular signatures in protein sequences that are characteristic of Cyanobacteria and plastid homologues[J]. Int J Syst Evol Microbiol, 2003, 53:1833-1842.
[15]Gao B, Gupta R S. Conserved indels in protein sequences that arecharacteristic of the phylum Actinobacteria[J].Int J Syst Evol Microbiol, 2005,55: 2401-2412.
[16]Griffiths E, Gupta RS. Distinctive protein signatures providemolecular markers and evidence for the monophyleticnature of the Deinococcus-Thermusphylum[J]. J Bacteriol, 2004, 186:3097-3107.
[17]Bhandari V,Ahmod NZ, Shah HN, et al. Molecular signatures for Bacillus species: demarcationof theBacillussubtilisandBacilluscereuscladesin molecular terms and proposal to limit theplacement of new species into the genusBacillus[J].Int J Syst Evol Microbiol, 2013, 63: 2712-2726.
[18]Adeolu M, Gupta RS. Phylogenomics and molecular signatures for the order Neisseriales: proposal for division of the order Neisseriales into the emended family Neisseriaceae and Chromobacteriaceae fam. Nov[J].Antonie Van Leeuwenhoek, 2013,104(1):1-24.
[19]Griffiths E, Gupta RS.Signature sequences in diverse proteinsprovide evidence for the late divergence of the order Aquificales[J].Int Microbiol, 2004, 7: 41-52.
[20]Bhandari V,Gupta RS.Molecular signatures for the phylum (class) Thermotogae and a proposal for its division into three orders (Thermotogales, Kosmotogales ord. nov. and Petrotogales ord. nov.) containing four families (Thermotogaceae, Fervidobacteriaceae fam. nov., Kosmotogaceae fam. nov. and Petrotogaceae fam. nov.) and a new genusPseudothermotogagen. nov.with five new combinations[J].Antonie Van Leeuwenhoek, 2014, 105(1):143-68.
[21]Gupta RS, Mok A. Phylogenomics and signature proteins for thealphaProteobacteriaand itsmain groups [J]. BMC Microbiol, 2007, 7: 106-125.
[22]Kainth P, Gupta RS. Signature proteins that are distinctive ofAlphaproteobacteria[J]. BMCGenomics, 2005, 6: 94-109.
[23]Pruitt KD, Tatusova T, Klimke W, et al. NCBI Reference Sequences: current status, policy, andnew initiatives[J]. Nucleic Acids Research, 2009, 37 (Database issue): D32-D36.
[24]Edgar RC. MUSCLE: a multiple sequence alignment method with reduced time and spacecomplexity [J]. BMC Bioinformatics, 2004, 5: 113.
[25]Nawrocki EP, Eddy SR. Query-Dependent Banding (QDB) for Faster RNA Similarity Searches[J]. PLoS Comput Biol, 2007, 3: e56.
[26]Ajawatanawong P, Baldauf S L. Evolution of proteinindels in plants, animals and fungi[J]. BMC Evol Biol,2013, 13: 140.
[27]Sekine S, Shimada A, Nureki O, et al. Crucial role of the HIGH-loop lysine for the catalytic activity ofarginyl-tRNA synthetase[J]. Journal of Biological Chemistry, 2001, 276(6):3723-3726.
[28]Roller C, Ludwig W, Schleifer K H. Gram-positive bacteria with a high DNA G+C content are characterized by a common insertion within their 23S rRNA genes[J]. J Gen Microbiol, 1992,138:1167-1175.
[29]Ludwig W, Schleifer K H.Bacterial phylogeny based on 16S and 23S rRNA sequence analysis[J]. FEMS Microbiol Rev, 1994, 15: 155-173.
[30]Nitharwal RG,Paul S,Dar A, et al. The domain structure ofHelicobacterpyloriDnaBhelicase: the N-terminal domain can be dispensablefor helicase activity whereas the extreme C-terminalregion is essential for its function[J].Nucleic Acids Res, 2007, 35:2861-2874.
[31]Biswas EE, Biswas SB. Mechanism of DNA bindingby the DnaB helicase ofEscherichiacoli: analysis of the roles ofdomain gamma in DNA binding[J].Biochemistry, 1999, 38:10929-10939.
Novel Conserved Signature Indels of the Class α-Proteobacteria
LYU Zhi-tang1,2, WANG Jing1, ZHOU Yan-xia1, ZHANG Wei-wei1
(1.CollegeofLifeSciences,HebeiUniversityandKeyLaboratoryofMicrobialDiversityResearchandApplicationofHebeiProvince,Baoding, 071002; 2.KeyLaboratoryofMedicinalChemistryandMolecularDiagnosisoftheMinistryofEducation(HebeiUniversity),Baoding071002)
α-Proteobacteriaare currently recognized as a class within the Proteobacteria phylum on the basis of 16S rRNA trees. In this work, five conserved signature indels (CSIs)widely distributed within the class α-Proteobacteria are found and described based on phylogenomics analysis, which are distinctive characteristics of the α-Proteobacterial species and are not found in any other groups of bacteria. The identified signatures include a 98 to 105 nt deletion and a 5-7 nt insertion in 23S rRNA, a 17-29 aa insert in replicative DNA helicase (DnaB), a 15 aa inserts in arginyl-tRNA synthase(ArgRS), and a 27-63 aa insert in DNA gyrase A subunit(GyrA).The presence of these characteristic sequences within α-Proteobacteria shows that this group can be distinguished from all other bacteria including other classes in the Proteobacteria thus provide an powerful approaches for better understand of phylogeny and systematics of this group.
α-Proteobacteria; conserved signature indels (CSIs); phylogenomics
吕志堂,博士,河北大学生命科学学院教授、硕士生导师,生物技术系主任。兼任河北省微生物学会副理事长、河北省生物工程学会常务理事、中国微生物学会永久会员、BISMiS创始会员、《微生物学杂志》编委和河北大学教指委委员等。长期从事微生物资源与系统学研究及相关课程的教学,省级精品课《微生物学》负责人;主持和参加完成国家自然科学基金、河北省自然科学基金和载人航天预研项目等14项,目前承担国家科技重大专项、河北省自然科学基金和企业合作项目等4项;发表论文40余篇,其中SCI收录论文10余篇;在国际上首先将包括核糖体蛋白在内的核心蛋白序列用于微生物系统学研究,证明串联核糖体蛋白序列和核心蛋白序列较之16S rRNA基因序列能更好的揭示微生物的进化关系,相关研究论文被BioMedLib评为2012年以来Top 20 Articles第5。获河北省科技进步三等奖和河北省自然科学三等奖各1项,发明专利1项,出版专著1部,教材2部。
河北省生物工程重点学科建设项目(1050-5030023);河北省生物学强势特色学科建设项目(1050-5030004)
吕志堂 男,教授,硕士生导师。研究方向为微生物资源与系统学。Tel:0312-5079364, E-mail:lzt325@126.com
2016-01-12
Q933
A
1005-7021(2016)02-0001-07
10.3969/j.issn.1005-7021.2016.02.001

