RBD+ODC及其突变体融合表达质粒的构建和表达验证
马怡晖,庞 霞,许晶晶,夏培苡,高汉青
(郑州大学第一附属医院病理科、河南省肿瘤病理重点实验室,河南 郑州 450052)
RBD+ODC及其突变体融合表达质粒的构建和表达验证
马怡晖,庞霞,许晶晶,夏培苡,高汉青
(郑州大学第一附属医院病理科、河南省肿瘤病理重点实验室,河南 郑州 450052)
目的构建RBD+ODC及其突变体融合表达质粒,并进行验证。方法选取Ras下游效应分子Raf-1中能够与K-Ras蛋白相互作用的RBD+CRD氨基酸片段作为结合结构域,以ODC蛋白分子全长作为功能结构域。以正常人胰腺组织的cDNA为模板扩增相应的目的基因片段,应用分子克隆的方法通过在引物中引入GS片段使结合结构域和功能结构域相连并通过双轮PCR法在ODC功能位点引入突变,所获得的核苷酸片段均连入pcDNA3.1真核表达载体中。结果与结论经测序和Western Bolt验证,成功构建了RBD+ODC及突变体RBD+ODC的重组质粒,2种质粒均能够在人HEK293T细胞中正常表达。
结合结构域;功能结构域;RBD;ODC;AZ
[Abstract]ObjectiveTo construct RBD+CRD and mutant RBD+CRD expressing vectors.MethodsThe RBD+CRD was chosen as the binding domain,and full length ODC as function domain,and the two domain were connected by molecular cloning.The mutant “RBD+ODC” was constructed by two step PCR.All the nucleotide fragments were connected into pcDNA3.1 vector.Results and ConclusionRBD+CRD and mutant RBD+CRD were constructed successfully by validation of sequencing and Western blot.
[Key words]binding domain; function domain; RBD; ODC; AZ
生物体内蛋白分子降解的途径有2种,第1种是泛素-蛋白酶体途径(ubiquitin-proteasome pathway,UPP),该途径控制着生物体内绝大多数蛋白质的降解,主要由泛素分子、泛素激活酶E1、泛素转移酶E2、泛素连接酶E3及26S蛋白酶体构成,通过对底物的泛素化修饰完成对底物蛋白的降解[1]。第2种是鸟甘酸脱羧酶/抗酶系统(ODC/AZ),其中ODC直接与底物蛋白结合,自身形成二聚体,进一步与抗酶AZ结合后蛋白构像被改变而直接被26S蛋白酶体识别降解[2-4]。相比较于第1种途径,ODC/AZ系统对蛋白的降解不需要泛素化修饰,因此作用更直接快速。我们拟在前期靶向泛素化降解K-Ras癌蛋白研究的基础上[5],尝试应用ODC/AZ系统实现敲减胰腺癌细胞中K-Ras癌蛋白,而研究的关键就是构建能够与K-Ras癌蛋白相互作用的融合蛋白,该融合蛋白需同时能够与K-Ras癌蛋白相互结合,又要保留ODC分子的活性作用。
1 材料与方法
1.1试剂与仪器基因组DNA提取试剂盒、琼脂糖凝胶DNA回收试剂盒(北京天根生物有限公司),DNA聚合酶、dNTP、各种核酸内切酶、T4连接酶(日本Takara公司),质粒提取试剂盒、lipofection2000转染试剂、高溶点琼脂糖(美国lnvitrogen公司),鼠抗人β-actin单克隆抗体、c-Myc标签单克隆抗体(美国Santa Cruz公司),HPR标记二抗(北京中杉金桥生物有限公司),丙烯酰胺、N’N’-亚甲基双丙烯酰胺、过硫酸胺、SDS(美国Pierce公司),DMEM培养基(美国Gibco公司),胎牛血清(天津灏阳公司),ECL发光液(北京普利莱公司)。
1.2细胞培养人HEK293T细胞系用DMEM培养基培养于37 ℃、体积分数5% CO2孵箱中。
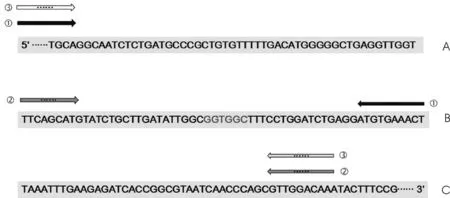
图1 双轮PCR定点突变ODC活性位点示意图
以mRBD+CRD作为模板,分别应用双轮引物(A和B)扩增出相应的核苷酸片断;再以第1轮上游引物和第2轮下游引物作为引物(C)扩增含有突变位点的核苷酸片断
表1各扩增片段的引物序列

引物名称引物序列RBD正向5’-CGGGATCCATGGGCAAACTCACAGATCCTTCTAAGACAAG-3’RBD反向5’-TAAAGCGGCCGCGAATTCAGGCATCCTGGAAACAGACTC-3’ODC正向5’-ATTTGCGGCCGCGGTTCCGGTTCCGGTTCCGGTTCCATGAACAACTTTGGTAATGAAGAG-3’ODC反向5’-CTACACATTAATACTAGCCGAAGCACAGGCTGCTCTGTGG-3’mRBD+ODC第1轮正向5’-CGGGATCCATGGGCAAACTCACAGATCCTTCTAAGACAAG-3’mRBD+ODC第1轮反向5’-CACATCCTCAGATCCAGGAAAGGTGGTGCCAATATCAAGC-3’mRBD+ODC第2轮正向5’-GCATGTATCTGCTTGATATTGGCACCACCTTTCCTGGATC-3’mRBD+ODC第2轮反向5’-CTACACATTAATACTAGCCGAAGCACAGGCTGCTCTGTGG-3’
1.4RNA的提取及逆转录参照Trizol Reagent试剂盒说明书,提取正常人胰腺组织中的总RNA,采用逆转录反应体系合成cDNA,以cDNA为模板扩增相应的目的基因或片段。表2列出了各表达载体中连入核苷酸的理论数目。
1.5测序所有测序工作均委托上海生工生物工程技术服务有限公司完成。
1.6融合蛋白表达验证用RIPA裂解液处理瞬时转染重组质粒48 h后的人HEK293T细胞,提取相应的蛋白,以β-actin作为内参,采用Western blot方法对不同处理组中携带有Myc标签的融合蛋白表达进行检测。表2列出了各融合蛋白分子氨基酸理论数目及相应的蛋白相对分子质量。
2 结果
2.1质粒测序验证所有构建的质粒经测序证实,连入的核苷酸序列是正确的。见图2。
表2多肽片段在原蛋白分子中的氨基酸位点及选用的酶切位点

片段名称理论氨基酸残基位数/aa实际连入氨基酸残基位数/aa目的片段核苷酸数目/bp融合蛋白相对分子质量(×103)选用酶切位点RBD+CRD/mRBD+CRD51~22046~225195776.0上游BamHI/下游NotIODC1~461(全长)1~461(全长)138354.5上游NotI/下游XhoI

图2 测序结果(A:RBD+ODC;B:mRBD+ODC)
2.2质粒表达验证将所构建的2个融合蛋白表达质粒瞬时转染人HEK293T细胞,于转染后48 h收集细胞,裂解后上样检测各重组质粒Myc标签表达情况。设立pcDNA3.1空载体转染组作为对照,以管家基因β-actin作为内参,结果显示2个融合蛋白表达质粒均可正常表达,蛋白相对分子质量与理论值基本一致。见图3。
3 讨论
蛋白敲减技术是依据泛素-蛋白酶体途径特异性降解蛋白分子的原理发展而来,通过构建能与底物蛋白特异性结合的融合型泛素连接酶E3蛋白[7-10]或化学合成PROTACS小分子[11-14],直接在蛋白水平(即翻译后水平)实现对底物蛋白的降解(即敲减)。因此与传统的基因沉默技术相比,该技术具有更大的灵活性。我们前期研究应用该策略,构建了能够与Ras癌蛋白相互结合的融合型E3,该E3通过泛素化途径靶向敲减了胰腺癌细胞中的K-Ras癌蛋白并抑制了胰腺癌细胞的生长活性。ODC/AZ系统是不同于UPP的另一条蛋白降解途径,该蛋白降解过程不依赖泛素化修饰,因此受影响因素少,作用更直接快速。理论上,将能够与底物结合的结合结构域同ODC融合表达,在AZ的催化下,应该可以实现不依赖泛素化修饰的蛋白降解。因次,本研究在前期靶向泛素化降解K-Ras癌蛋白的基础上,尝试通过构建能与KRAS相互作用的RBD+ODC融合蛋白,实现对K-Ras不依赖泛素化修饰的降解。实验结果表明,应用分子克隆手段,我们成功构建了RBD+ODC以及突变体RBD+ODC的重组质粒,2种质粒均能够在人HEK293T细胞中正常表达。本实验为下一步深入研究并评价RBD+ODC作为敲减底物蛋白工具的作用有效性奠定了基础。

图3 Western Blot实验结果
[1]Hershko A,Ciechanover A.The ubiquitin system[J].Annu Rev Biochem,1998,67:425-479.
[2]Kahana C,Asher G,Shaul Y.Mechanisms of Protein Degradation An Odyssey with ODC[J].Cell Cycle,2005,4(11):1461-1464.
[3]Elias S,Bercovich B,Kahana C,et al.Degradation of ornithine decarboxylase by the mammalian and yeast 26S proteasome complexes requires all the components of the protease[J].Eur J Biochem,1995,229(1):276-283.
[4]Gandre S,Kahana C.Degradation of ornithine decarboxylase in Saccharomyces cerevisiae is ubiquitin independent[J].Biochem Biophys Res Commun,2002,293(1):139-144.
[5]Ma Y,Gu Y,Zhang Q,et al.Targeted destruction of KRAS oncoprotein by an engineered ubiquitin ligase suppresses the proliferation of pancreatic adenocarcinoma cell PANC-1[J].Mol Cancer Ther,2013,12(3):286-294.
[6]Matsuzawa S,Cuddy M,Fukushima T,et al.Method for targeting protein destruction by using a ubiquitin-independent,proteasome-mediated degradation pathway[J].Proc Natl Acad Sci U S A,2005,102(42):14982-14987.
[7]Zhou P,Bogacki R,McReynolds L,et al.Harnessing the ubiquitination machinery to target the degradation of specific cellular proteins[J].Mol Cell,2000,6(3):751-756.
[8]Zhang J,Zheng N,Zhou P.Exploring the functional complexity of cellular proteins by protein knockout[J].Proc Natl Acad Sci U S A,2003,100(24):14127-14132.
[9]Hatakeyama S,Watanabe M,Fujii Y,et al.Targeted destruction of c-Myc by an engineered ubiquitin ligase suppresses cell transformation and tumor formation[J].Cancer Res,2005,65(17):7874-7879.
[10]Li X,Shen L,Zhang J,et al.Degradation of HER2 by Cbl-based chimeric ubiquitin ligases[J].Cancer Res,2007,67(18):8716-8724.
[11]Sakamoto KM,Kim KB,Kumagai A,et al.Protacs:Chimeric molecules that target proteins to the Skp1-Cullin-F box complex for ubiquitination and degradation[J].Proc Nat Acad Sci U S A,2001,98(15):8554-8559.
[12]Sakamoto KM,Kim KB,Verma R,et al.Development of Protacs to target cancer-promoting proteins for ubiquitination and degradation[J].Mol Cell Proteomics,2003,2(12):1350-1358.
[13]Carmony KC,Kim KB.PROTAC-Induced Proteolytic Targeting[J].Methods Mol Biol,2012,832:627-638.
[14]Cyrus K,Wehenkel M,Choi EY,et al.Jostling for position:optimizing linker location in the design of estrogen receptor-targeting PROTACs[J].ChemMedChem,2010,5(7):979-985.
Construction and Validation of RBD+CRD and Mutant RBD+CRD Expressing Vectors
Ma Yihui,Pang Xia,Xu Jingjing,Xia Peiyi,Gao Hanqing
(DepartmentofPathology,theFirstAffiliatedHospital,ZhengzhouUniversity,Zhengzhou450052,China)
国家自然科学基金青年项目(编号:81401936)
马怡晖(1978- ),女,博士,副主任医师,主要从事胰腺癌发病机制研究。E-mail:allsunshine123@126.com
10.3969/j.issn.1673-5412.2016.04.001
R735.9;R730.23
A
1673-5412(2016)04-0277-04
2016-05-10)

