绍兴黄酒酒曲总DNA的提取及其真菌多样性
王洋怡舟,李柏林,欧 杰,赵 勇,刘海泉
(上海海洋大学 食品学院,上海 201306)
绍兴黄酒酒曲总DNA的提取及其真菌多样性
王洋怡舟,李柏林,欧杰*,赵勇,刘海泉
(上海海洋大学 食品学院,上海 201306)
目的:对于黄酒酒曲样品,选择一种合适的总DNA提取方法,并对其中真菌群落结构进行多样性分析。方法:因其质地,结合以往DNA提取方法,比较分析不同方法所获得的总DNA纯度、得率及凝胶电泳图,对实验结果较好的DNA样本进行真菌引物PCR扩增后纯化定量,再经Miseq高通量测序后进行生物信息分析。结果:实验结果表明,使用试剂盒+物理化学酶法获得了较高质量的样品总DNA,A260/A280比值也接近理想水平,PCR扩增后凝胶电泳图能够得到目的条带,OTU分类、Shannon曲线和系统进化树图显示了其真菌多样性。结论:通过数据的比较和分析,确立了选择的方法是一种较理想的酒曲总DNA的提取方法,对其真菌群落的测定及分析,也为后期开拓黄酒微生物资源提供了有力工具。
酒曲,DNA的提取,真菌,PCR,多样性
黄酒是我国特有民族传统特产,是利用麦曲中微生物共同糖化发酵制成,在发酵过程中,酒曲具有复合糖化发酵粗酶制剂的功能,给黄酒的酿造提供多种需要的酶,积累的微生物代谢产物赋予了黄酒独特的色、香、味[1]。
根据微生物细胞壁裂解方法的不同,常用提取方法可分为3类:物理方法,即通过外力作用破坏细胞结构[2];化学法,例如SDS法或者CTAB法,其它试剂会与螯合剂(如EDTA)共同使用;酶法,使用溶菌酶、溶壁酶等[3]。
黄酒制曲过程是开放的,其微生物种类也是极其复杂的[4]。目前研究微生物多样性的方法可分为两类:一是利用传统分离培养技术分析菌群相关性和差异性。二是利用现代生物技术提高研究的准确性[5],更加快速准确检测微生物群落结构和功能[6]。方华[7]采用3种培养基分离麦曲中微生物,得其真菌菌株。李旺军[8]对酒曲真菌进行研究补充。曹钰[9]等采用传统分离培养方法和RISA技术对其多样性进行研究。近年来,分子生物学在真菌分类学中发挥了越来越大的作用[10]。陈建尧[11]等对不同酒曲进行传统培养和免培养分子鉴定研究所含真菌。陆健等[12]采用ITS测序方法对黄酒麦曲真菌组成和特性及过程中的变化进行了研究。
OTU(Operational Taxonomic Units)是微生物遗传学的概念,为了便于进行分析,给某一个分类单元设置的同一标志,在微生物多样性中,OTU分类是根据基因测序结果对微生物进行分类。通过OTU分析,可以知道其微生物多样性和不同微生物的丰度;往往会和已知菌一起做聚类,从而完成微生物的种属归属鉴定。基于OTU的结果,计算在OTU相似水平(94%~99%)的Alpha多样性指数,包括稀释曲线、Shannon-Wiener曲线等。根据已有微生物物种的分类学信息数据库,将测序得到的物种丰度信息回归至数据库的分类学系统关系树中,从整个分类系统上全面了解样品中所有微生物的进化关系和丰度差异[13-14]。
本文选择建立了一种免培养式黄酒酒曲总DNA提取方法,对不同方法得出的DNA得率、纯度以及凝胶电泳图进行比较分析后选择较好数据结果的总DNA样本,对其进行真菌区PCR扩增和二次纯化,应用Miseq高通量测序后,通过OTU分类和Alpha评估得到其真菌多样性,了解其主要微生物构成,对以往的黄酒酒曲真菌群落结构研究结果补充,为进一步探讨真菌群落与产酶的功能关系奠定基础。
1 材料与方法
1.1材料与仪器
绍兴某黄酒厂酒曲样品,装入无菌袋中,常温保存。
D2000 MD114、GMO Food DNA Extraction Kit 深加工食品DNA提取试剂盒(非离心柱型)DP326 N3020、50 mg/mL Lysozyme、10 U/μL Lyticase、蛋白酶K天根TIANGEN公司;Premix Taq(Ex TaqTMVersion 2.0 plus dye)、10×Loading Buffer CA1701ATaKaRa宝生物工程有限公司;λ-Hind Ⅲ DNA Marker宝生物工程(大连)有限公司;50×TAE;Acetic Acid;EDTA生工生物工程(上海)股份有限公司;异丙醇、十二烷基硫酸钠国药集团化学试剂有限公司;Agarose MBBI Life Sciences;Extraction Buffer:0.1 mol/L磷酸盐,0.1 mol/L EDTA(pH8.0),0.1 mol/L Tris-HCl(pH7.0),1.5 mol/L NaCl,1% CTAB。
AG离心机EPPENDORF艾本德公司;SLFPDAD多功能酶标仪GENE Company Limited基因有限公司;AG 22331 Hamburg PCR仪EPPENDORF艾本德公司;BIO-RAD Powerpac Basic美国伯乐公司;Molecular Image GelDocTMXR+with Image LabTMSoftware电泳凝胶成像系统GelDoc XR 美国伯乐公司。
1.2样品总DNA提取方法
1.2.1样品预处理将酒曲用研钵粉碎均匀,取约0.10 g置于2 mL离心管中,加入1 mL无菌水,振荡1 min,12000 r/min离心5 min,去掉液体,重复步骤1至2次。
1.2.2酒曲DNA提取方法1试剂盒法。参照GMO Food DNA Extraction Kit 深加工食品DNA提取试剂盒(非离心柱型)DP326 N3020的操作步骤。
1.2.3酒曲DNA提取方法2物理化学法。参照文献[15]中1.2.2的“液氮研磨法+化学法”的方法。
1.2.4酒曲DNA提取优化方法3取酒曲样品置于研钵中,加入液氮覆盖样品,在其解冻前用力研磨,重复3次,倒液氮时注意粉末溅出。取研磨好的样品0.150 g,加入2 mL离心管中。加入1 mL无菌水,于振荡器振荡1 min,随后12500 r/min离心5 min,去掉液体,重复1至2次。加入500 μL试剂盒中缓冲液GMO1、20 μL 50 mg/mL Lysozyme、20 μL 10 U/μL Lyticase,旋涡振荡1 min后放入预热37 ℃水浴锅,水浴30 min。加入20 μL的Proteinase K(20mg/mL),旋涡振荡1 min后,放入预热56 ℃水浴锅水浴45 min,每隔15 min振荡一次。液氮冻融三次。56 ℃水浴15 min。加入200 μL缓冲液GMO2,充分混匀,涡旋振荡1min。37 ℃水浴锅水浴10 min。12500 r/min离心5 min,将上清转移至新的离心管中。向上清液中加入0.7倍体积的异丙醇,充分混匀。12500 r/min离心3 min,弃上清,保留沉淀。加入700 μL 70%乙醇,涡旋振荡5 s,12500 r/min离心2 min,弃上清。重复操作加入700 μL 70%乙醇,涡旋振荡5 s,12500 r/min离心2 min,弃上清。小心倒掉液体,65 ℃烘干30 min,至烘干为止,防止残余的乙醇影响后续的酶反应。加入20~50 μL洗脱缓冲液TE,旋涡振荡1 min,最终得到DNA溶液,放入-20 ℃冰箱保存。
1.3DNA提取质量检测
在PCR扩增过程中,模板DNA的提取质量是决定能否扩增出目标条带的限制因素[16]。DNA的提取效率,尤其是DNA的纯度严重影响实验结果[17]。
采用GENE Company Limited 基因有限公司的多功能酶标仪SLFPDAD测定三种方法提取样品的DNA含量、260、280以及A260/A280的比值,使用Gen5 1.11 Software读取酶标仪数据。
1.4琼脂糖凝胶电泳检测
电压120 V,电泳30 min,使用EB染色,用电泳凝胶成像系统拍照得到电泳图。
1.5真菌18S rDNA PCR扩增
PCR现已成为功能基因组学研究广泛应用的技术[18]。PCR分子技术分析真菌群落的流程与分析细菌群落[19]是相似的,其关键在于真菌特异性DNA 扩增引物的选择或设计。
PCR扩增引物见表1,引物由生工生物工程(上海)股份有限公司合成。
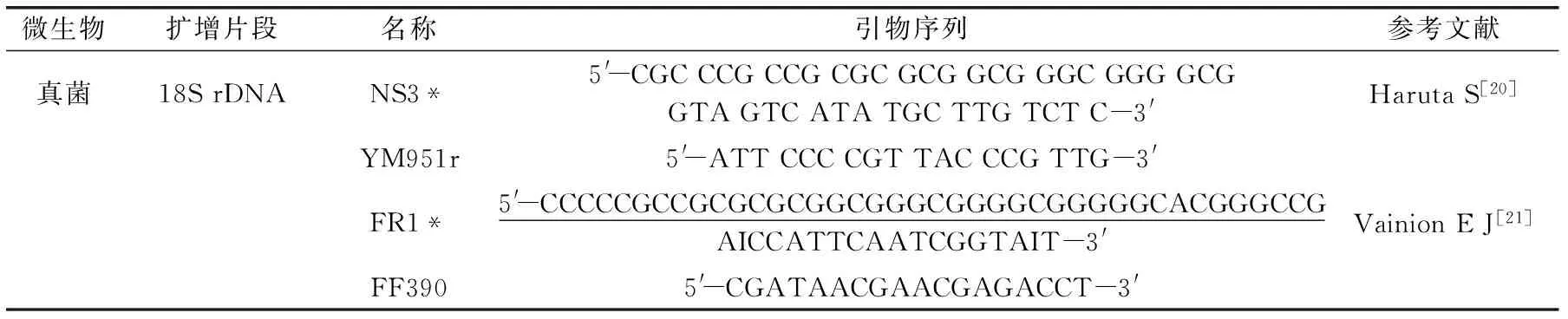
表1 引物一览表Table 1 List of PCR primers
注:*为带“GC”夹。PCR反应体系:Premix Taq 12.5 μL,正反引物各1 μL,模板(酒曲DNA)2 μL,加dd H2O(8.5 μL)至总体积25 μL;同样,以dd H2O代替酒曲DNA模板配置阴性对照。
PCR反应条件:95 ℃变性5 min,94 ℃变性30 s,51 ℃退火30 s,72 ℃延伸45 s,共30个循环。随后在72 ℃延伸10 min,于4 ℃结束。PCR产物保存于-20 ℃备用,PCR结果用琼脂糖电泳检测。
1.6纯化PCR产物及Miseq高通量测序
对于目的条带进行割胶回收,作为第二次PCR扩增DNA模板。选取第二对PCR引物FR1-GC/FF390进行扩增,将PCR产物纯化后交由上海翰宇生物科技有限公司人类基因组研究中心基因组测序部应用MiSeq Reagent Kit v3在Illumina MiSeqTM测序平台进行双向测序。使用mothur v.1.32.1[22]对原始数据进行处理,进行过滤处理后,对得到的优质序列进行OTU聚类分析。基于OTU聚类分析结果,可以进行Alpha多样性分析以及进行群落结构的统计分析。使用MEGAN5 v5.7.1[23]来绘制分类学系统组成树。
2 结果与分析
2.1酒曲中总DNA得率
评判不同方法提取的样品DNA结果质量有两个硬性的指标,即得率和纯度。将酶标仪数据进行整合后(见表2),可以看出使用方法1的平均DNA得率最低,每μL样品提取出的浓度均在200 ng以下;而方法2中的DNA得率相对于方法1来说,平均DNA浓度值增加了83%,平均值在328 ng/μL,证明了使用方法2提取酒曲中总DNA效果比方法1效果好;而方法3提取的DNA得率相较于方法1和方法2大幅度提升,可得800 ng/μL 以上DNA含量,得率平均达到方法1得率的4.7倍,方法2得率的2.56倍。
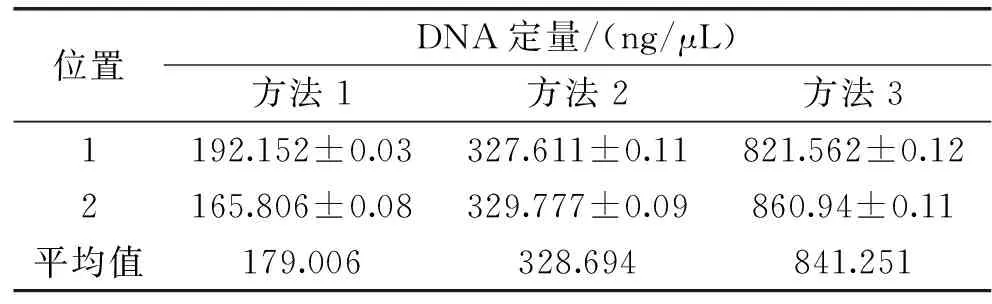
表2 三种方法提取酒曲样品的DNA得率Table 2 DNA quantity analyses isolated from sample using three methods
2.2酒曲中总DNA纯度
在酒曲长期贮藏的过程中很容易受到自身因素和环境因素的影响,比如温度、蛋白质、糖分以及酚类物质等污染而影响酒曲中自身DNA纯度,从而会进一步影响后期实验结果。因此,在样本DNA提取过程中,纯度的保证也格外重要。一般情况下,检测DNA样品纯度指标有A260/A230和A260/A280比值,其中,A260/A280的比值主要用来检测蛋白质和酚类物质的污染情况,理想值在1.8~2.0之间[24]。由于酒曲样品DNA纯度的影响因素主要取决于蛋白质污染以及一些酚类物质,因此本文选取A260/A280比值来考量不同方法结果的纯度。表3综合了三种提取方法的数据结果,从方法1和方法3的结果数值来看,A260/A280比值都在1.8~2.0之间,方法1的比值平均值为1.8005,方法3的比值平均值为1.8100,表示该两种方法提取的DNA纯度在理想范围内。使用方法2得到的A260/A280比值低于1.8,表明该方法在提取DNA纯度上效果不佳,缺少采取去除蛋白质或者酚类物质的步骤,使得提取的总DNA中存在这些因素的影响。

表3 三种方法提取样品DNA纯度数据Table 3 DNA quality data extracted from sample using three methods
2.3凝胶电泳图验证提取结果
通过对比三种提取方法的样品总DNA凝胶电泳图,发现使用方法3“酶法+试剂盒法+物理化学法”对于酒曲样品中DNA的提取相对比较成功。
试剂盒法(方法1)中未采用任何破壁的步骤(物理方法和酶解破壁都未使用),得到的结果如图1,在7~9.4 kb之间出现条带,表明使用方法1提取的DNA片段较小,从图中可以看出点样孔较亮。一般情况下,点样孔滞留的是蛋白质,说明方法1的实验步骤中未较好的去除一些蛋白质和多糖杂质的污染。
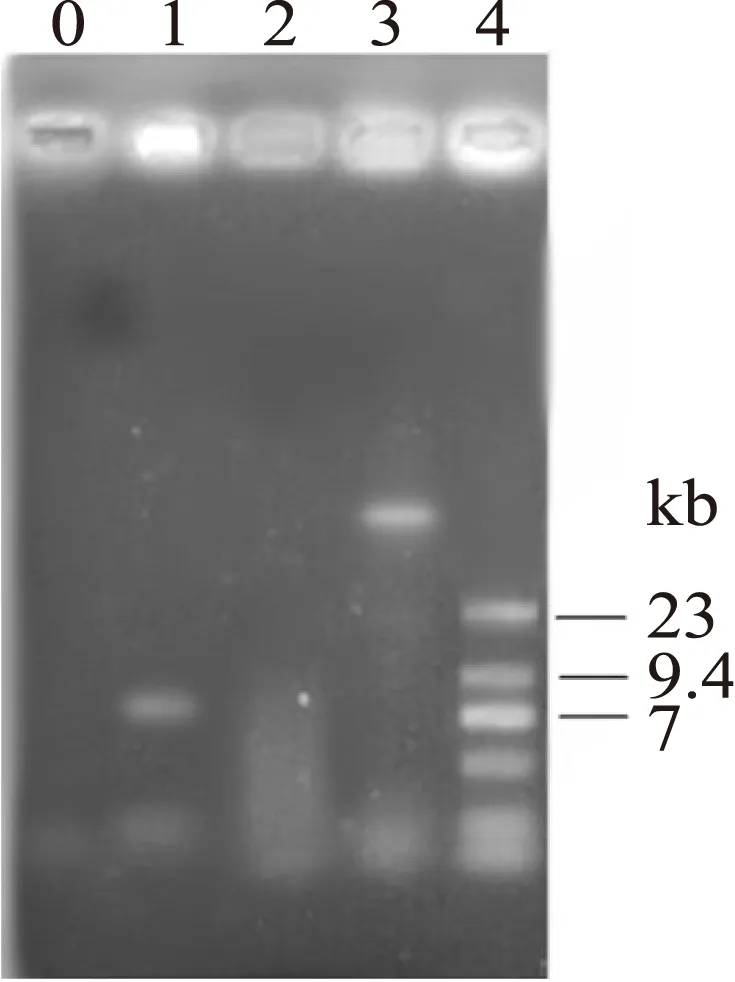
图1 三种方法所获得的酒曲样品总DNA的凝胶电泳图Fig.1 Electrophoresis of total DNA extracted from sample注:0为阴性对照,1、2、3相对应方法123, M为λ-HindⅢ DNA marker。
物理化学法(方法2)在整个提取DNA的步骤中使用了液氮破壁的物理方法,未使用酶解法来进行破壁处理,提取的总DNA得率(见表2)相对方法1较好,方法2的电泳条带(见图1,2泳道)有明显的拖带现象,看不清目的条带,和DNA提取过程中未去除杂质以及细胞壁破壁不够彻底有关。
试剂盒+物理化学+酶法(方法3)的结果数据(见表2、表3)与前两个方法的数据相比结果较好,表2证明了结合了酶解和物理破壁法处理样品,得到了较高的DNA浓度,得到的电泳图(见图1)在23 kb上方显示出条带,提取出的DNA片段相对较完整。无论从DNA得率、纯度还是凝胶电泳图的实验结果来看,方法3提取的酒曲样品DNA与方法1、2相比结果较好,可以直接用于后续的分子生物学实验。
2.4PCR扩增结果
为了进一步验证方法3对于酒曲样品的DNA提取效果,本文选取引物NS3-GC/YM951r对方法3获得的DNA模板进行四组平行PCR扩增,从PCR产物凝胶电泳结果图分析其提取效果,结果如图2,证明提取后的DNA经真菌引物PCR扩增结果较成功,在正确的位置出现了目标片段。分子量大约在250~500 bp。

图2 PCR扩增后凝胶电泳图Fig.2 Gel electrophoresis after fungi zone PCR amplification注:1、2、3、4为四组平行实验。
2.5OTU划分与Alpha评估
2.5.1OTU分类使用 PyNAST和数据库比对后用 UCHIME[25]方法检测并去除Chimeric序列。非嵌合体序列使用 Silva/GreenGene等核糖体数据库中的aligned(16S/18S,SSU)核糖体序列数据比对,去除非目的物种序列污染。统计样本的优质序列及长度分布(结果如表4和图3),从结果中可以看出使用方法3提取的样品DNA,经PCR扩增后获得的平均片段长度在真菌(ITS/18S,28S~5.8S)核苷酸片段长度区间,在300~320 bp之间的序列数达到140000左右,样本2平行实验提取DNA数量较小,后续多样性分析将该数据剔除。

表4 序列统计情况Table 4 Sequence statistics

图3 序列长度分布图Fig.3 Sequence diagram
2.5.2多样性评估(community diversity)本文Alpha多样性评估采用Shannon-Wiener曲线,经PCR扩增后割胶纯化后定量进行评估。Shannon-Wiener是反映样品中微生物多样性的指数,利用测序量在不同测序深度时的微生物多样性指数构建曲线,以此反映各样本在不同测序数量时的微生物多样性。当曲线趋向平坦时,说明测序数据量足够大,可以反映样品中绝大多数的微生物物种信息[26]。图4为cutoff=0.03水平下的Shannon曲线。剔除数据2,134三组平行实验得到的曲线结果都可以趋向平坦,可以较好的获取样品中微生物情况。

图4 Shannon多样性曲线 Fig.4 Shannon diversity curve注:cutoff=0.03,图5同。
2.6系统进化树分析
对所有样本做系统进化分析,根据OTU来区分样品内及多个样本间不同物种并构建进化树,通过分析 OTU 组分丰度来计算不用物种在样品内的丰度。根据分化时间来判断物种间的相似度及进化关系,处在同一分支上的物种说明进化关系较近。默认使用数据为代表性序列,分析水平为目。结果如图5。在以往对黄酒酒曲真菌多样性的研究表明,其生长最多的真菌有Absidiacorymbifera(伞枝犁头霉)、Rhi-zomucorPusillus(微小毛霉)、Aspergillusorgyze(米曲霉)、Aspergillusfumigatus(烟曲霉)和Rhizopusoryzae(米根霉)。Aspergillusfumigatus(烟曲霉)是麦曲中的主要丝状真菌。图5进化树显示本研究提取的酒曲DNA中真菌有如下几个菌属:Saccharomyceta(酵母属)、Basidiomycota(担子菌类)和一些早期其他分支的真菌属目,如Harpellales(钩孢毛菌目)和Mucorales(毛霉菌目)。酒曲样品中优势菌为Eurotiales(散囊菌目),其次是Saccharomycetales(酵母菌目)、Mucorales(毛霉菌目)、Harpellales(钩孢毛菌目)和Capnodiales(煤炱目)。
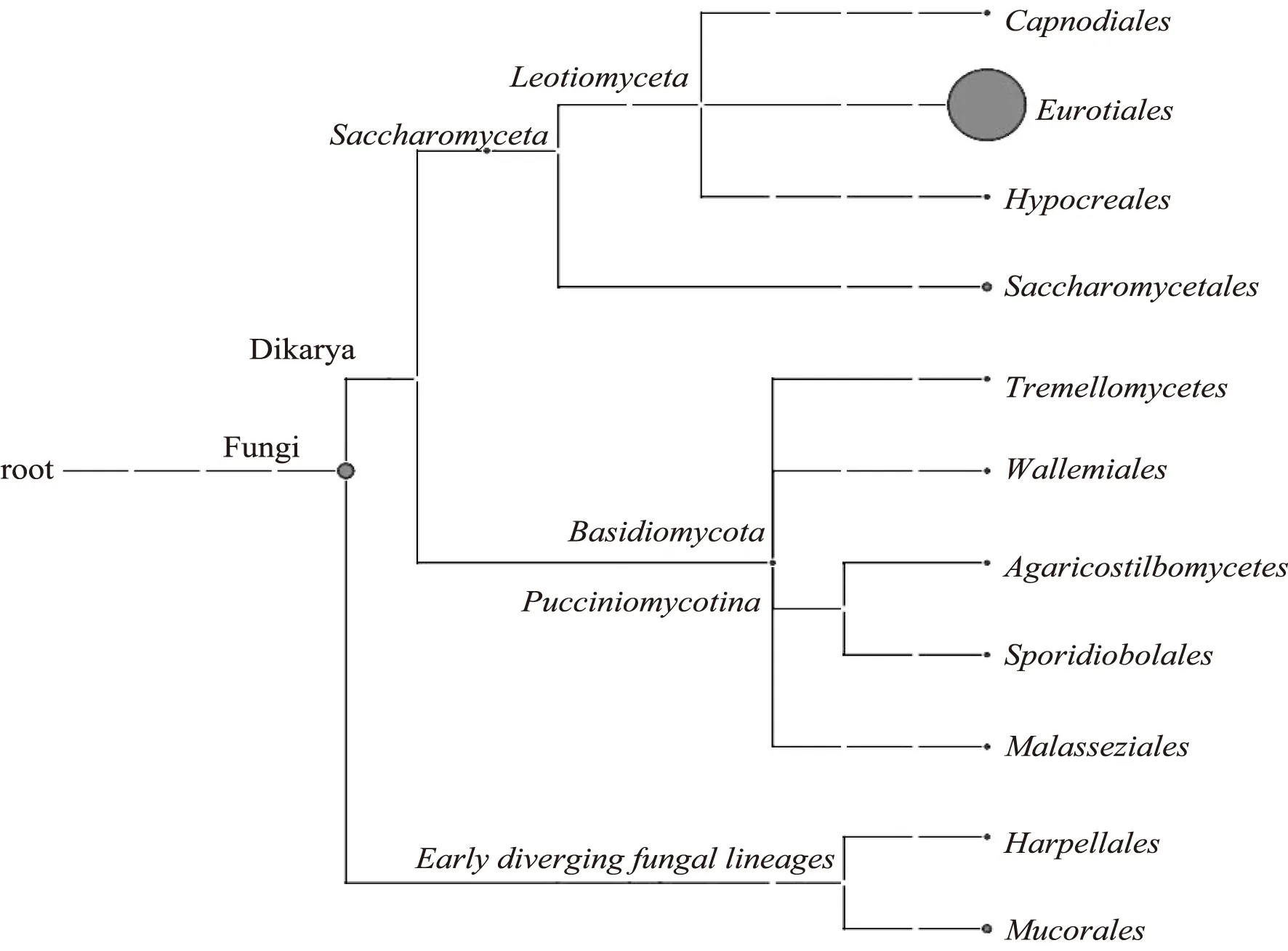
图5 系统进化树图 Fig.5 Phylogenetic tree graph
3 结论与讨论
微生物DNA的提取大多根据微生物其自身不同特点或者具体的实验要求去选择合适的提取方法。张宁等[27]对DNA提取方法进行研究总结,综合了各类各样DNA的提取方法,在设计实验方案之前可相互借鉴,总结出一个适合自己样品的优势方法,进行多次尝试和数据整理后们可以找到较适合的DNA提取方法。与方法1、2相比,方法3的实验结果表现出了一定的优势,总体来说,方法3(酶法+试剂盒法+物理化学法)在以下几个关键步骤进行了优化处理:增加了样品预处理步骤。因为酒曲样品曲块硬,水分含量少,吸水性较强,如不进行前处理步骤,直接加入缓冲液、溶菌酶、溶壁酶以及蛋白酶K等,样品会将这些物质全部吸收,从而使得加入的试剂物质起不到酶解和去除蛋白质的作用;对于真菌DNA的提取关键在于破碎其细胞壁,本文在方法1中未使用任何破壁的步骤;方法2使用液氮研磨物理破壁法;方法3在酒曲样品前处理之前,先使用液氮研磨处理样品,随后在加入的缓冲液中结合了酶裂解的破壁法(溶菌酶、溶壁酶),在加入蛋白酶K后,再使用了液氮冻融的物理破壁法,保证了样品中DNA的高质量提取。从总DNA凝胶电泳图和酶标仪数据得出方法3较为适合黄酒酒曲样品。后续分子实验采用该方法提取出的DNA样本,PCR扩增电泳条带以及纯化后PCR产物高通量分析数据也较好的证实了使用该DNA提取方法可较好获取黄酒酒曲中微生物多样性情况。本研究通过分子生物手段,得到黄酒酒曲真菌多样性,测出其真菌序列长度、构建出Shannon多样性曲线,得出真菌优势菌种,鉴定的结果认为Saccharomyceta(酵母属)、Basidiomycota(担子菌类)是黄酒酒曲中的主要真菌目。酒曲样品中优势菌为Eurotiales(散囊菌目),其次是Saccharomycetales(酵母菌目)、Harpellales(钩孢毛菌)、Mucorales(毛霉目)和Capnodiales(煤炱目)。
与以往传统方式不同,本文减少了传统培养菌种以及筛选过程,大大减少时间和成本从而得出黄酒酒曲的真菌多样性,得出的优势菌种等数据结果对以往的研究结果进行了补充,这也反映出对于一个微生物群落组成的体系,使用单一的方法都不能完整的揭示其复杂构成。深入认识其微生物组成结构,也为后续研究黄酒整个发酵过程中微生物多样性提供必要的实验依据。
[1]陆健,曹钰,方华,等.绍兴黄酒麦曲中真菌的初步研究[J]. 食品与生物技术学报,2008,27(2):78-83.
[2]李旺军,方华,曹钰,等.不同破壁方法提取绍兴黄酒麦曲中的微生物总DNA[J].中国酿造,2007(4):17-20.
[3]Robe P,Nalin R,Capellano C,et al. Extraction of DNA from soil[J]. European Journal of Soil Biology,2003,39:183-190.
[4]寿虹志,凌志勇,杨旭,等.浅析黄酒麦曲中的微生物与黄酒风味的关系[J].中国酿造,2007(8):55-57.
[5]谭映月,胡萍,谢和.我国白酒酿造微生物多样性的研究现状及展望[J].酿酒科技,2011,(11):100-105.
[6]许爱清,李宗军,王远亮,等.应用PCR-DGGE技术检测发酵食品和饲料中真菌菌群[J].食品科学,2010,31(7):317-322.
[7]方华.绍兴黄酒麦曲中微生物的初步研究[D]. 无锡:江南大学,2006.
[8]李旺军.绍兴黄酒麦曲中真菌资源的初步研究[D].无锡:江南大学,2007.
[9]曹钰,陆健,方华,等.绍兴黄酒麦曲中真菌多样性的研究[J].食品科学,2008,29(3):277-282.
[10]燕平梅,马雁飞,倪玲.发酵食品中微生物多样性研究方法进展[J].中国酿造,2011,(2):12-14.
[11]曹钰,陈建尧,谢广发,等.黄酒麦曲天然发酵中真菌群落的成因初探[J].食品与生物技术学报,2008,27(5):95-101.
[12]陈建尧,曹钰,谢广发,等.黄酒机械成型麦曲制曲过程中真菌动态变化的研究[J].微生物学报,2008,34(8):42-50.
[13]Edgar R C,Haas B J,Clemente J C,et al. UCHIME improves sensitivity and speed of chimera detection[J]. Bioinformatics,2011,27(16):2194-2200.
[14]DeSantis T Z,Hugenholtz P,Larsen N,et al. Greengenes,a chimera-checked 16S rRNA gene database and workbench compatible with ARB. Applied and environmental microbiology,2006,72(7):5069-5072.
[15]闫亮珍,李晓然,全哲学,等.汾酒大曲和酒醅样品DNA提取方法的优化[J].食品与发酵工业,2011,37(3):32-36.
[16]Manuel Porcar,Silvia Ramos A,Mparo Latorre.A simple DNA extraction method suitable for PCR detection of genetically modified maize[J]. Journal of the Science of Food and Agriculture,2007,87:2728-2731.
[17]杨艳秋,王丽,贺丹,等.真菌DNA提取方法的建立和比较[J].中国组织工程研究与临床康复,2007,11(50):10093-10096.
[18]Ray Chaudhuri S,Pattanayak AK,ThakurAR.Microbial DNA extraction from samples of varied origin[J].Current Science,2006,91(12):1697-1700.
[19]ERCOLINI D. PCR-DGGE fingerprinting:novel strategies for detection of microbes in food[J]. Journal of Microbiological Methods,2004,56(3):297-314.
[20]HARUTA S,SHINTARO U,EGAWA I,et al. Succession of bacterial and fungal communities during a traditional pot fermentation of rice vinegar assessed by PCR-mediated denaturing gradient gel electrophoresis[J]. International Journal of Food Microbiology,2006,109(1/2):79-87.
[21]Vainio E J,Hantula J. Direct analysis of wood-inhabiting fungi using denaturing gradient gel electrophoresis of amplified ribosomal DNA[J]. Mycological Research,2000,104:927-936.
[22]Schloss P D,Westcott S L,Ryabin T,et al. Introducing mothur:open-source,platform-independent,community-supported software for describing and comparing microbial communities[J]. Applied and environmental microbiology,2009,75(23):7537-7541.
[23]El Hadidi M,Ruscheweyh H J,Huson D. Improved metagenome analysis using MEGAN5. In Joint 21st Annual International Conference on Intelligent Systems for Molecular Biology(ISMB)and 12th European Conference on Computational Biology(ECCB)2013,8.
[24]赵裕栋,周俊,何璟.土壤微生物总DNA提取方法的优化[J].微生物学报,2012,52(9):1143-1149.
[25]Robert C. Edgar. UCHIME improves sensitivity and speed of chimera detection Bioinformatics,2011,27(16):2194-2200.
[26]王瑞风,雷海燕,臧璞,等.大黄酸对糖尿病小鼠肠道菌群影响的初步研究[J].中国微生态学杂,2016,28(1):21-24.
[27]张宁,王凤山.DNA提取方法进展[J].中国海洋药物,2004,(2):40-46.
Extracting total DNA in Shaoxing rice wine wheat starter and its fungal diversity
WANG Yang-yi-zhou,LI Bai-lin,OU Jie*,ZHAO Yong,LIU Hai-quan
(College of Food Science and Technology,Shanghai Ocean University,Shanghai 201306,China)
Objective:To choose appropriate extraction method of total DNA of yellow rice wine and analyze its fugal diversity. Methods:Because of its character,different methods detection were compared and analyzed in this study,including total DNA purity,yield and gel electrophoresis combining with previous DNA extraction method. Objective bands were purified and quantified after PCR amplification of fungi primer,and conducted biological information analysis after Miseq high throughput sequencing. Results:Experimental result showed that kit+Physical+enzyme method could get better quality and concentration fungal DNA,A260/A280ratio was close to the ideal level,and could get obvious target band figure after PCR amplification gel electrophoresis. OTU classification,Shannon diversity curve and phylogenetic tree graph showed fungal diversity. Conclusion:Through the comparison and analysis of data,established method in this paper was an ideal method to extract total DNA in the rice wine wheat starter,determination of the fungal community also provided a powerful tool for later developing rice wine microbial resources.
wheat starter;DNA extraction;fungal;PCR;diversity
2016-02-26
王洋怡舟(1991-),女,硕士研究生,研究方向:食品微生物学,E-mail:422621249@qq.com。
欧杰(1964-),男,硕士,副教授,研究方向:食品生物技术,E-mail:jou@shou.edu.cn。
TS201.3
A
1002-0306(2016)15-0176-06
10.13386/j.issn1002-0306.2016.15.026

