球毛壳菌降解天然木质纤维素能力差异及酶系基因分析
郝晓冉,牛学良,李强,潘皎,朱旭东
1.北京师范大学 生命科学学院,北京 100875;2.南开大学 生命科学学院,天津 300071
木质纤维素是植物体利用太阳能合成的常见的可再生高能聚合物,主要由纤维素、半纤维素和木质素组成,是地球上分布最广泛、含量最为丰富的生物质[1]。将木质纤维素酶解为单糖,在生物燃料及生物材料行业有很好的应用前景,对于减缓全球变暖、能源危机和环境污染具有十分重要的意义。木质纤维素物质转化为乙醇的主要工艺流程包括:预处理分离木质素,将纤维素和半纤维素暴露出来;酶解纤维素和半纤维素;微生物发酵单糖物质合成乙醇[2]。木质纤维素结构复杂,往往需要通过预处理才可以使得多糖物质降解为可发酵单糖。传统的物理化学预处理方法条件要求严格、能耗大、成本高,新型的生物预处理方法不仅摒弃了上述缺陷,而且温和环保,因此受到越来越多的关注。新方法主要利用自然界中广泛存在的微生物(主要是白腐真菌、木腐真菌、放线菌和细菌等[2-3]),通过分泌漆酶(laccase)、木质素过氧化物酶(lignin peroxidase,LiP)、锰过氧化物酶(manganese peroxidase,MnP)等攻击、解聚生物质中的木质素以达到预处理的效果[2]。生物预处理还被应用于造纸行业中,不仅可大大提高纸张的亮度和张力,而且还节省了37%~47%的能耗[4]。高效降解木质纤维素的白腐真菌还曾被用来对木质纤维素物质的生物降解[5]及对环境的生物修复[6-7]。木质纤维素材料中的纤维素成分的生物降解至少需要3类酶的参与,即内切葡聚糖酶(endoglucanase,EG,水解β-1,4糖苷键)、外切葡聚糖酶(exoglucanase)[包括β-1,4-D-葡聚糖水解酶(1,4-β-D-glucan glucano⁃hydrolase)和纤维二糖水解酶(cellobiohydrolase,CBH),作用于纤维素线状分子的末端,连续水解β-1,4糖苷键,生成纤维二糖或葡萄糖分子]和β-葡聚糖苷酶(β-glucosidase,BGL,可将纤维糊精或纤维二糖水解成葡萄糖分子[8])。此外,纤维二糖脱氢酶(cellobiose dehydrogenase,CDH)和糖苷水解酶(glu⁃coside hydrolase,GH)等协同作用,还可促进纤维素的酶解糖化[9]。半纤维素的主要成分是木聚糖,通过木聚糖酶(xylanases)加以分解,包括内切木聚糖酶(endoxylanases)、β-木聚糖苷酶(β-xylosidase)、木聚糖乙酰酯酶(xylan acetyl esterase)和α-葡萄糖醛酸酶(α-glucuronidase)[10]。
毛壳属(Chaetomium)真菌是一类分布仅次于青霉(Penicillium)和曲霉(Aspergillus)的常见真菌[11],在分解利用植物体及其他纤维素物质的过程中起重要作用。迄今,陆续从该属真菌中分离鉴定出内切葡聚糖酶、外切葡聚糖酶、热稳定性纤维二糖水解酶、纤维二糖脱氢酶、木聚糖酶等一系列参与纤维素半纤维素降解的酶[12-16]。该属的某些种类如嗜热毛壳菌(C.thermophilium)还能够分泌漆酶[17]。毛壳属的常见种类球毛壳菌(C.globosum)广泛分布于土壤、堆肥、植物种子、盐沼、木材及含纤维素的制品中,研究者发现球毛壳菌还具有分解棉纤维、麦秸秆、咖啡豆及油棕榈树叶的作用[18-20]。由于球毛壳菌在生长过程中能产生球毛壳甲素(chaetoglobosin A,ChA)等具有抗癌抗线虫活性的多种次级代谢产物[21-23],目前的研究热点主要集中于球毛壳菌次级代谢产物方面,而有关球毛壳菌降解木质纤维素的报道还很少。我们从自然界中分离筛选出4株能分解利用木质纤维素的内共生真菌,通过18S rDNA及形态学特征分析将其鉴定为球毛壳菌,并将4株菌培养在分别以微晶纤维素、杨树叶和木屑为惟一碳源的培养基上,比较分析球毛壳菌菌株间木质纤维素酶活力差异及球毛壳甲素合成情况。结合已测序成功的球毛壳菌CBS148.51的基因组信息,对球毛壳菌基因组中分布的木质纤维素降解酶类进行了归类分析,为球毛壳菌木质纤维素降解过程的研究及该菌种的开发利用奠定了基础。
1 材料和方法
1.1 材料
球毛壳菌NK102(柏树)、NK103(日本油松)、NK104(红皮云杉)、NK105(水杉)为本实验室自行分离的植物内共生真菌,纯化的菌株在土豆培养基(potato dextrose agar,PDA)上于28℃连续培养7 d,用于后续试验。用于木质纤维素酶活力检测及发酵合成ChA的3种基物培养基参考Umikalsom等报道的配方[18],1 L培养基中包括2 g KH2PO4、0.3 g Mg⁃SO4·7H2O、0.2 g CaCl2·2H2O、2 mg CoCl2、1.6 mg MnSO4·H2O、1.4 mg ZnSO4·H2O、2 mL Tween-80、1 mL微量元素溶液[1 g/L FeSO4·7H2O,1 g/L Mn⁃SO4·H2O,0.25 g/L Na2MoO4·2H2O,0.1 g/L H3BO4,0.25 g/L CuCl2· 2H2O,0.25 g/L ZnCl2,0.1 g/L NH4NO3,0.25 g/L Co(NO3)2·6H2O,0.1 g/L NiSO4·6H2O,5 mL/L H2SO4]、9 g KNO3,以及10 g基物碳源[微晶纤维素(microcrystalline cellulose)、杨树叶(Populus sp.leaves)或木屑(wood powder)]。
1.2 真菌鉴定
各菌株接种于PDA平板上,于28℃培养数天,观察菌落形态;连续培养7 d后收集菌丝、孢子并涂布于载玻片上进行显微观察。参照Raeder等[24]的方法提取各菌株的基因组DNA,以UK4F(5'-CYGGTT⁃GATCCTGCCRG-3')、UREV(5'-GYTACCTTGTTAC⁃GACTT-3')[25]为引物进行PCR反应,扩增各菌株的18S rDNA序列,并将目的片段交华大基因公司进行测序,将测序结果上传至GenBank,利用NCBI Blast在数据库中对测序结果进行匹配,用CLUSTAL_X2软件对各序列及与其同源性较高的序列进行Align⁃ment,用MEGA4.0软件的邻接法(Neighbor-joining method)构建系统进化树。
1.3 木质纤维素利用能力检测
用菌种尖端接种法分别接种菌丝块于羧甲基纤维素(carboxymethylcellulose,CMC)和纤维素刚果红(cellulose-Congo red agar)[26]平板,检测菌株的纤维素降解能力。木质素降解能力的检测在Bavendamm平板上进行,具有木质素降解能力的真菌在生长过程中可分泌木质素降解酶类(漆酶、木质素过氧化物酶、锰过氧化物酶),能使愈创木酚等化合物聚合,在菌落周围形成红棕色轮环,呈阳性反应,不能利用木质素、仅利用纤维素的菌株呈阴性反应。记录菌落生长直径d1和变色圈直径d2,计算d1/d2,比较各菌株的木质素降解能力。
1.4 酶活力检测
用菌丝尖端接种法分别将各菌株接种于含200 mL液体培养基的500 mL锥形瓶中,30℃恒温振荡(120 r/min)连续培养4 d,发酵产物于4500 r/min离心5 min,上清液作为粗酶液进行后续反应。
纤维素酶活力通过降解纤维素物质产生的还原糖量来测定。酶解反应于0.1 mol/L、pH4.8的醋酸盐缓冲液中进行,于50℃水浴中反应30 min。1个酶活力单位(U)定义为酶促反应中每分钟生成1 μmol葡萄糖(还原糖)所需的酶量[27]。漆酶活力通过氧化2,2'-azmobis-(3-ethylbenzthiazoline-6-sulpho⁃nate)(ABTS)来测定[28]。
1.5 HPLC检测发酵液中的ChA
用菌丝尖端接种法分别将各菌株接种于含200 mL液体培养基的500 mL锥形瓶中,28℃恒温振荡(120 r/min)连续培养12 d,用布氏漏斗分离菌丝和发酵液。发酵液用等体积的氯仿/甲醇(10∶1)萃取,有机相于45℃低压旋蒸浓缩至干,甲醇重悬沉淀物,混合均匀后12 000 r/min离心10 min。上清液经0.45 μm微孔滤膜过滤后进行HPLC检测(C18 ODS柱,4.6 mm×250 mm;柱温箱调节为30℃,流动相为70%甲醇水溶液,流速1 mL/min,检测波长227 nm)。
2 结果
2.1 NK103、NK104和NK105菌种鉴定及与NK102亲缘关系分析
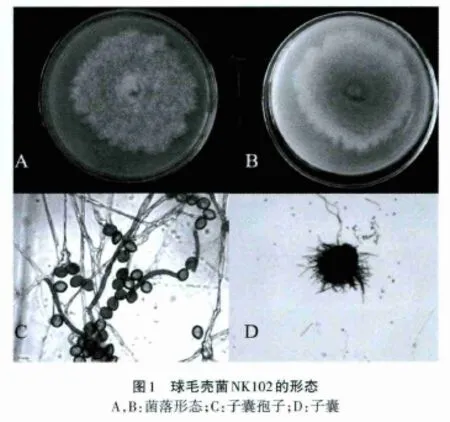
NK103、NK104和NK105于PDA培养基上生长良好,28℃培养4 d,肉眼观察4株菌形态相似,菌落初期为白色,后为棕褐色,菌落背面呈圆环状;菌丝有色,分支有隔;子囊孢子为单细胞、柠檬状、深褐色;子囊壳球形,孔口有深褐色的缘丝。NK102的形态学特征见图1。
利用真菌18S rDNA通用引物分别扩增NK103、NK104和NK105的18S rDNA序列,测序后将序列上传至GenBank,登录号分别为JN394588、JN639019和JN639021。通过NCBI Blast,发现这3株菌与球毛壳菌18S rDNA序列(GenBank:AB048285)的同源性均最高,达99%。结合NK102的18S rDNA序列(HQ529774),利用邻接法构建了这4株菌的系统进化树(图2),表明NK102、NK103、NK104、NK105与球毛壳菌、Humicola grisea var.grisea都处于同一生态位分支上。鉴于上述4株菌的形态学特征与球毛壳菌更为相近,确定该4株菌为球毛壳菌。并且,NK104与NK105的亲缘关系最为接近;NK103与NK102的亲缘关系接近,与NK104、NK105的亲缘关系相对较远。

2.2 NK102、NK103、NK104和NK105的木质纤维素降解能力差异
采用CMC培养基、纤维素刚果红培养基及常用的Baverndamm变色反应对NK102、NK103、NK104和NK105的木质纤维素降解能力进行比较,结果如图3。4株菌在CMC平板上生长旺盛,28℃培养4 d后菌落直径可达30~75 mm,革兰氏碘液(Gram's io⁃dine)[26]浸泡后形成明显的水解圈,直径约3.0~3.2 cm,菌株间差异不显著。4株菌生长在纤维素刚果红培养基上,28℃培养4 d后菌落变红,在菌落周围形成可见的水解圈,直径依次为1.8、1.4、1.9和1.9 cm。NK103所形成的水解圈略小,推测其在这4株菌中降解羧甲基纤维素的能力稍弱。NK102、NK103、NK104和NK105在Bavendamm变色反应实验中都呈阳性反应。在Bavendamm培养基Ⅰ上生长良好,并且与愈创木酚发生反应形成红棕色变色圈。在Bavendamm培养基Ⅱ上生长缓慢,28℃培养15 d后都能形成棕色圈,变色圈直径d2明显大于菌落直径d1。根据d1/d2,这4株菌降解木质素的能力由强到弱依次为NK103、NK102、NK105、NK104。

2.3 NK102、NK103、NK104和NK105的木质纤维素酶和漆酶酶活力比较
为初步比较球毛壳菌NK102、NK103、NK104和NK105在分解利用天然木质纤维素材料过程中纤维素酶和漆酶的表达情况,我们将这4株菌分别接种于以微晶纤维素、杨树叶和木屑为惟一基物碳源的液体培养基中,于30℃培养箱中120 r/min连续培养4 d后取样,直接检测发酵产物中纤维素酶和漆酶的酶活力。培养物目测发现,4株菌都可以有效分解利用微晶纤维素,并在培养5~7 d时分解完全,但所产生的纤维素酶活力都低于或等于0.1 U/mL发酵液,远远低于利用其他2种基物碳源产生的纤维素酶活力。如图4A所示,NK102、NK103、NK104和NK105在以杨树叶为基物碳源的培养基中产生的纤维素酶活力依次为0.35、0.26、0.18和0.20 U/mL,是以微晶纤维素为基物碳源时的2~4倍。4株菌在以木屑为惟一碳源的培养基中生长非常缓慢,添加氮源蛋白胨之后,菌丝生长旺盛,纤维素酶活力大大提高,依次为0.76、0.70、0.73和0.66 U/mL,是以微晶纤维素为基物碳源时的6~19倍。4株真菌中以NK102分泌的纤维素酶活力最高,可达0.76 U/mL发酵液。我们还检测了发酵液中漆酶的酶活力,各菌株的漆酶活力较低,以反应液吸光度值与对照吸光度值的差来表示各菌株的漆酶活力大小。如图4B所示,在以微晶纤维素为唯一碳源时,4株菌都能分泌漆酶,但差异显著,NK103的漆酶活力最高,这一结果与平板试验一致。在以杨树叶为唯一碳源底物时,NK103的漆酶活力升高,而其他3株菌的漆酶活力下降甚至为零。在添加蛋白胨的木屑培养基中,各菌株都不分泌漆酶,说明蛋白胨成分对漆酶产生抑制作用,仅提供碳源促进球毛壳菌的生长。

2.4 NK102、NK103、NK104和NK105合成ChA的能力差异
前期工作发现NK102、NK103、NK104和NK105在生长过程中都能够合成ChA。为检测4株菌在分解利用微晶纤维素、杨树叶及木屑等基物时ChA的合成情况,分别接种 NK102、NK103、NK104和NK105于上述3种基物碳源培养基中,在28℃培养箱中以120 r/min的转速连续培养12 d取样,发酵产物萃取纯化后进行HPLC检测。依照ChA回归曲线,通过峰面积计算发酵液中的ChA含量。结果如图5,4株菌在3种基物培养基上生长都能合成ChA,但ChA产量受到基物碳源的影响。在木屑培养基中,4株菌虽然生长良好,但HPLC检测到的发酵产物量都很低,ChA合成量均≤1.0 mg/L。NK102、NK103、NK104、NK105在3种基物碳源培养基中生长所合成的ChA量都远远低于生长在PDA培养基中所得的量。NK102在微晶纤维素液体培养基生长12 d后ChA合成量为4.47 mg/L发酵液,在杨树叶液体培养基中,其ChA产量是微晶纤维素的2倍。球毛壳菌合成ChA的能力还表现出菌株间的差异。NK103在微晶纤维素培养基中合成的ChA更多(5.11 mg/L发酵液),是杨树叶培养基中的3.6倍,与NK102不同。另外,NK104和NK105无论是培养在以微晶纤维素还是木屑为惟一碳源的培养基中,其ChA产量均≤1.0 mg/L,变化不大,但在以杨树叶为惟一碳源时,ChA产量大大升高至原来的20倍左右(≥11 mg/L)。总的来说,3种基物碳源中,杨树叶最适合培养球毛壳菌合成ChA。

2.5 球毛壳菌CBS148.51基因组中木质纤维素降解酶基因的分布
纤维素酶多为糖蛋白,酶分子的一级结构由核心催化域(catalytic domain,CD)、碳水化合物结合结构域1(carbohydrate binding domain 1,CBM1)和将这2部分相连的链接区(linker)组成。CBM1的氨基酸序列高度保守[29],不仅在纤维素酶中存在,在降解半纤维素和其他一些细胞壁结构成分的酶中也发挥功能[30]。Longoni[31]以真菌 CBM1(Cdd:smart00236)为参考序列,在球毛壳菌CBS148.51基因组中寻找到30个纤维素代谢相关基因;以糖水解酶GH7保守的催化结构域(Cdd:cd07999)为参考序列,在球毛壳菌基因组中找到7个可能编码GH7的基因。他们没有对编码参与半纤维素和木质素降解的酶的基因进行归纳,并且寻找到的参与纤维素降解的酶也不完善。鉴于此,我们在其工作基础上,以CAZy数据库(http://www.cazy.org/)中可能参与纤维素降解的GH家族蛋白的氨基酸序列作为参考序列,通过IMGBlastp(E值<1e-5)对球毛壳菌CBS148.51基因组中可能编码纤维素半纤维素降解酶的基因进行了归纳。同时,以黄孢原毛平革菌(Phanerochaete chryso⁃sporium)的纤维二糖脱氢酶(cellobiose dehydroge⁃nase,CDH)基因(GenBank:Q01738.1)为参考序列,在球毛壳菌CBS148.51基因组中找到11个同源相关序列。以白腐真菌变色栓菌(Trametes versicolor)漆酶基因(GenBank:BAA23284.1)和粗糙脉孢菌(Neu⁃rospora crassa)漆酶基因(GenBank:AAA33592.1)为参考序列,通过NCBI blastp在球毛壳菌CBS148.51基因组中寻找编码漆酶的基因。
如表1所示,球毛壳菌CBS148.51基因组中分布有119个纤维素半纤维素酶降解相关基因。其中有54个可能编码内切葡聚糖酶的基因(GH5、GH6、GH7、GH12、GH45、GH51、GH61家族)、8个可能编码外切葡聚糖酶的基因(GH7家族)、1个可能编码β-葡聚糖苷酶的基因(GH1家族)、11个可能编码纤维二糖脱氢酶的基因、18个可能编码β-木糖苷酶的基因(GH3和GH43家族)、7个可能编码内切木聚糖酶的基因(GH10家族)、12个可能编码木聚糖酶的基因(GH11、XynB)、1个可能编码木聚糖乙酰酯酶的基因、1个可能编码α-葡萄糖醛酸酶的基因。也就是说,球毛壳菌CBS148.51拥有完整的降解纤维素半纤维素酶系。此外,CBS148.51基因组中还分布有8个可能编码漆酶的基因,分别为CHGG_00543、CHGG_02290、CHGG_03552、CHGG_06172、CHGG_08215、CHGG_08781、CHGG_10025和CHGG_11082。参考黄孢原毛平革菌编码锰过氧化物酶的序列(GenBank:AAA33743.1),在CBS148.51基因组中找到2个(CHGG_01567和CHGG_02518)可能编码MnP的基因。没有找到可能编码木质素过氧化物酶的基因。上述基因组学基础表明球毛壳菌具有分解利用木质纤维素的能力,在开发真菌分解利用木质纤维素的进程中具有举足轻重的地位。

表1 球毛壳菌CBS148.51基因组木质纤维素降解酶类基因功能预测
3 讨论
木质纤维素降解为小分子单糖的过程非常复杂,至少需要几十种酶协同作用才能破坏植物细胞壁的复杂结构,降解木质素、纤维素和半纤维素。白腐真菌因具有能彻底降解木质素的功能[32],在自然界能有效降解木质纤维素的微生物中研究得最为深入。土壤、空气和植物体内广泛存在的球毛壳菌在生长过程中可产生丰富的次生代谢产物,如具有杀线虫、抗癌抗真菌活性的ChA[21-22],近年来引起研究者广泛的关注。除了作为重要的生防真菌,球毛壳菌在生态系统中还扮演着分解者的角色,具有分解利用木质纤维素的能力[18-20]。我们利用已测序的球毛壳菌代表菌株CBS148.51的基因组信息,寻找到119个编码纤维素半纤维素降解酶的基因、8个编码漆酶的基因和2个编码锰过氧化物酶的基因,证明球毛壳菌具有完整的纤维素半纤维素降解酶系统。这可为后续球毛壳菌木质纤维素降解过程的研究及开发利用提供参考。
本研究的另一个目的是检测分离自不同植物体的内共生球毛壳菌NK102、NK103、NK104、NK105的木质纤维素降解能力,并进一步研究不同菌株间的差异。本研究用到的碳源基物杨树叶为作者校园中的普通行道和绿化树种,在我国北方十分常见,秋季落叶处理是很大的问题。另外一种基质木屑来自普通的家具厂,也是需要处理的富含木质纤维素的废弃物。我们将分离到的4株球毛壳菌进行了初步分类,对其利用纤维木质素的能力进行了分析,发现它们都能很好地以微晶纤维素(纯纤维素)为惟一碳源,特别是在不加碳氮源的杨树叶和木屑培养基上,这4株菌均能生长,在杨树叶培养基上生长良好,能够将完整的树叶基质完全降解(目测消失),说明球毛壳菌可独自处理杨树叶。而木屑(我们并未有意限定木质种类,仅为了观察这些菌降解木质纤维素的一般能力)培养基中加入适当氮源后,菌株的降解能力大大提高,这可能是因为木屑中的氮源含量较低。我们还比较了NK102、NK103、NK104和NK105发酵粗酶液中纤维素酶和漆酶的分泌情况。这4株菌可以分泌纤维素酶和漆酶,酶活力受到碳源的影响;蛋白胨的添加可大大提高纤维素酶活力,但不会诱导漆酶的合成。以微晶纤维素为碳源时,4株菌都产漆酶,产量有所不同;而以木屑为碳源时,即使添加漆酶最适氮源蛋白胨,在4株菌的培养液中也几乎都检测不到漆酶;菌株间的差异比营养条件对漆酶合成的影响更大。针对所测的3种碳源,NK103在4株菌中漆酶的合成量始终最高,在木质素利用方面有很大的潜力。同时,由于球毛壳菌可分泌热稳定性木聚糖酶及大量的β-葡糖苷酶,在分解利用纤维素方面优于瑞氏木霉(Trichoderma reesei)[33]。因此,球毛壳菌可作为利用天然木质纤维素等植物生物质,开发有用产品的良好基盘生物。同时具有分解木质素和纤维素能力的微生物并不多见,球毛壳菌具有这种特性,因而这类真菌用来开发降解农业、环境中废弃纤维木质素物质具有一定的优势。
前期工作首次发现ChA具有高效杀死根结线虫的作用,因为ChA是稳定的天然产物,开发成为抗线虫的农用化合物有自身优点,使得利用球毛壳菌防治线虫成为可能。高效液相色谱分析显示,4株菌在分解利用微晶纤维素、杨树叶和木屑的过程中都能够稳定合成ChA,值得一提的是,NK104在以杨树叶为基质时产生的ChA最高,达到14.88 mg/L。各菌株在3种碳源基物培养基上生长,其ChA合成能力受到碳源的影响,菌株间表现出显著差异,与亲缘关系有关。如亲缘关系近的NK104和NK105在微晶纤维素和木屑培养基中都只能合成微量ChA(≤1.0 mg/L),但培养在杨树叶培养基中时,ChA产量大幅增长,达11 mg/L以上。这个结果表明,利用自然界中大量存在的废弃物木质纤维素生物质工业化生产天然生防制剂ChA,不仅可以大大节省成本,对于解决三农问题,减少环境污染也是完全可能的。
[1]Holtzapple M T.Cellulose,hemicelluloses,and lignin[M]//Mac⁃rae R,Robinson R K,Sadler M J.Encyclopedia of food sci⁃ence,food technology,and nutrition.London:Academic Press,1993.
[2]Saritha M,Lata A A.Biological pretreatment of lignocellulos⁃ic substrates for enhanced delignification and enzymatic digest⁃ibility[J].Indian J Microbiol,2012,52(2):122-130.
[3]Leonowicz A,Matuszewska A,Luterek J,et al.Biodegradation of lignin by white rot fungi[J].Fungal Genet Biol,1999,27(2-3):175-185.
[4]Breen A,Singleton F L.Fungi in lignocellulose breakdown and biopulping[J].Curr Opin Biotechnol,1999,10(3):252-258.
[5]Dashtban M,Schraft H,Syed T A,et al.Fungal biodegrada⁃tion and enzymatic modification of lignin[J].Int J Biochem Mol Biol,2010,1(1):36-50.
[6]Chairin T,Nitheranont T,Watanabe A,et al.Biodegradation of bisphenol A and decolorization of synthetic dyes by lac⁃case from white-rot fungus,Trametes polyzona[J].Appl Bio⁃chem Biotechnol,2013,169(2):539-545.
[7]Lee H,Jang Y,Kim J M,et al.White-rot fungus Merulius tremellosus KUC9161 identified as an effective degrader of polycyclic aromatic hydrocarbons[J].J Basic Microbiol,2013,53(2):195-199.
[8]Hasunuma T,Okazaki F,Okai N,et al.A review of enzymes and microbes for lignocellulosic biorefinery and the possibility of their application to consolidated bioprocessing technology[J].Bioresour Technol,2013,135:513-522.
[9]Langston J A,Shaghasi T,Abbate E,et al.Oxidoreductive cellulose depolymerization by the enzymes cellobiose dehydro⁃genase and glycoside hydrolase 61[J].Appl Environ Microbi⁃ol,2011,77(19):7007-7015.
[10]Podkaminer K K,Guss A M,Trajano H L,et al.Characteriza⁃tion of xylan utilization and discovery of a new endoxylanase in Thermoanaerobacterium saccharolyticum through targeted gene deletions[J].Appl Environ Microbiol,2012,78(23):8441-8447.
[11]Pangallo D,Chovanová K,Simonovicová A,et al.Investiga⁃tion ofmicrobialcommunity isolated from indoorartworks and airenvironment:identification,biodegradative abilities,and DNA typing[J].Can J Microbiol,2009,55(3):277-287.
[12]El-Gindy A A,Saad R R,Fawzi E.Purification and some properties of exo-1,4-beta-glucanase from Chaetomium oliva⁃ceum[J].Acta Microbiol Pol,2003,52(1):35-44.
[13]Thompson A J,Heu T,Shaghasi T,et al.Structure of the cat⁃alytic core module ofthe Chaetomium thermophilum family GH6 cellobiohydrolase Cel6A[J].Acta Crystallogr D Biol Crys⁃tallogr,2012,68(Pt 8):875-882.
[14]Harreither W,Sygmund C,Augustin M,et al.Catalytic proper⁃ties and classification of cellobiose dehydrogenases from asco⁃mycetes[J].Appl Environ Microbiol,2011,77(5):1804-1815.
[15]Li Y L,Li H,Li A N,et al.Cloning of a gene encoding thermostable cellobiohydrolase from the thermophilic fungus Chaetomium thermophilum and its expression in Pichia pasto⁃ris[J].J Appl Microbiol,2009,106(6):1867-1875.
[16]Latif F,Asgher M,Saleem R,et al.Purification and character⁃ization of a xylanase produced by Chaetomium thermophile NIBGE[J].World J Microbiol Biotechnol,2006,22(1):45-50.
[17]Chefetz B,Chen Y,Hadar Y.Purification and characteriza⁃tion of laccase from Chaetomium thermophilium and its role in humification[J].Appl Environ Microbiol,1998,64(9):3175-3179.
[18]Umikalsom M S,Ariff A B,Shamsuddin Z H,et al.Produc⁃tion of cellulase by a wild strain of Chaetomium globosum us⁃ing delignified oil palm empty-fruit-bunch fibre as substrate[J].Appl Microbiol Biotechnol,1997,47(5):590-595.
[19]Varnaité R,Paškevicius A,Raudoniené V.Cellulose degrada⁃tion in rye straw by micromycetes and their complexes[J].Ekologija,2008,54(1):29-31.
[20]Eyini K Pa M.Effect of co-fungal treatment on biodegrada⁃tion of coffee pulp waste in solid state fermentation[J].Asian J Exp Biol Sci,2010,1(2):352-359.
[21]Sekita S,Yoshihira K,Natori S,et al.Chaetoglobosins,cyto⁃toxic 10-(indol-3-yl)-[13]cytochalasans from Chaetomium spp.I.Production,isolation and some cytological effects of chaeto⁃globosins A-J[J].Chem Pharm Bull(Tokyo),1982,30(5):1609-1617.
[22]Hu Y,Zhang W,Zhang P,et al.Nematicidal activity of chae⁃toglobosin A poduced by Chaetomium globosum NK102 against Meloidogyne incognita[J].J Agric Food Chem,2013,61(1):41-46.
[23]Li X,Tian Y,Yang S X,et al.Cytotoxic azaphilone alka⁃loids from Chaetomium globosum TY1[J].Bioorg Med Chem Lett,2013,23(10):2945-2947.
[24]Raeder U,Broda P.Rapid preparation of DNA from filamen⁃tous fungi[J].Lett Appl Microbiol,1985,1(1):17-20.
[25]Metz A M,Haddad A,Worapong J,et al.Induction of the sexual stage of Pestalotiopsis microspora,a taxol-producing fungus[J].Microbiology,2000,146(8):2079-2089.
[26]Kasana R C,Salwan R,Dhar H,et al.A rapid and easy method forthe detection ofmicrobialcellulases on agar plates using gram's iodine[J].Curr Microbiol,2008,57(5):503-507.
[27]Umikalsom M S,Ariff A B,Zulkifli H S,et al.The treat⁃ment of oil palm empty fruit bunch fibre for subsequent use as substrate for cellulase production by Chaetomium globosum Kunze[J].Bioresour Technol,1997,62(1-2):1-9.
[28]Bourbonnais R,Paice M G.Oxidation of non-phenolic sub⁃strates.An expanded role for laccase in lignin biodegradation[J].FEBS Lett,1990,267(1):99-102.
[29]Rabinovich M L,Melnick M S,Bolobova A V.The structure and mechanism of action of cellulolytic enzymes[J].Biochemis⁃try(Mosc),2002,67(8):850-871.
[30]Hervé C,Rogowski A,Blake A W,et al.Carbohydrate-bind⁃ing modules promote the enzymatic deconstruction of intact plant cell walls by targeting and proximity effects[J].Proc Natl Acad Sci USA,2010,107(34):15293-15298.
[31]Longoni P,Rodolfi M,Pantaleoni L,et al.Functional analysis of the degradation of cellulosic substrates by a Chaetomium globosum endophytic isolate[J].Appl Environ Microb,2012,78(10):3693-3705.
[32]Gold M H,Alic M.Molecular biology of the lignin-degrading basidiomycete Phanerochaete chrysosporium[J].Microbiol Rev,1993,57(3):605-622.
[33]Gandhi J P,Rao K K,Dave P J.Characterization of extracel⁃lular thermostable xylanase from Chaetomium globosum[J].J Chem Technol Biotechnol,1994,60(1):55-60.

