DNA序列在蚌科分子系统学研究中的应用*
欧阳解秀,吴小平,欧阳珊,李绍波
(南昌大学生命科学与食品工程学院,江西南昌 330031)
DNA序列在蚌科分子系统学研究中的应用*
欧阳解秀,吴小平*,欧阳珊,李绍波
(南昌大学生命科学与食品工程学院,江西南昌 330031)
蚌科动物是世界上濒危的动物类群之一,DNA作为遗传信息的载体,直接对DNA序列进行分析和比较是研究蚌科分子系统学最理想的方法,也是研究最佳保种计划最关键的资料。论文从研究方法(包括基因组DNA的提取、目的基因的选择、PCR引物和扩增条件的选用)和各亚科分子系统学的研究结果等方面,总结了蚌科分子系统学的研究进展,并展望了中国蚌科分子系统学的研究方向。
DNA序列;蚌科;分子系统学
蚌科动物是淡水底栖动物的重要类群,由于其生态学意义、经济上的重要性、资源丰富、复杂的生活史和最近数量上的急剧下降,已经引起了很多研究者的关注[1]。蚌科动物是淡水贝类最大的一个科,包括674个物种[2-3]。由于蚌科动物存在趋同现象,且目前大多数种和属的分类都是以历史上形态特征为依据,种和属的分类都是在18世纪末和19世纪初定义的,使得蚌类保护工作的困难就在于对物种进行准确的分类[4]。随着研究的深入,以形态特征为基准的系统发育分析常常遇到困难,近代生物化学和分子生物技术的发展,特别是聚合酶链反应(PCR)技术的广泛应用,孕育了分子系统学,DNA序列分析为生物系统学的研究提供了分子水平上的证据。以分子数据来描述蚌科各个类群之间关系的研究,已经引起国内外学者的重视和兴趣[5-6]。本文对近年来DNA序列在蚌科分子系统学方面的研究进行较为系统的总结,以期为后来的研究者提供一定的借鉴。
1 研究方法
1.1 蚌科基因组DNA提取法
高质量的基因组DNA是进行分子系统研究的基础,苯酚法和CTAB法是提取蚌基因组DNA常用的两种方法(950 mL/L乙醇中的浸泡标本提取基因组DNA)。
1.1.1 苯酚法 取0.1 g闭壳肌或外套膜用ddH2O洗净,加液氮碾碎→转移至1.5 mL离心管加入 500 μ L组织匀浆缓冲液(10 mmol/L Tris-HCl,pH 8.0;50 mmol/L EDTA,pH 8.0)→混匀后加入200 μ L 100 g/L 的SDS 和 5 μ L 的蛋白酶K,50℃~55℃裂解 6 h→加入等体积苯酚,5 000 r/min离心5 min→取上清转移至一新离心管加3 μ L RNaseA,37℃水浴1 h→加入等体积苯酚,5 000 r/min离心5 min→取上清,移至一新离心管加入等体积苯酚,5 000 r/min离心5 min→取上清加1/10体积的NaAc 3 μ L和2倍体积的无水乙醇,混匀,于-20℃沉淀30 min→10 000 r/min离心10 min,去液体 →加入 750 mL/L乙醇洗涤,6 000 r/min离心5 min,去液体→干燥2 h~3 h→再加入50 μ L TE,使DNA 充分溶解-20 ℃保存[7]。
1.1.2 CTAB法 取0.1 g闭壳肌放于消毒过的玻片上,用小刀将组织切碎,转移至2 mL的预冷离心管中→加入 600 μ L CTAB提取液,混匀→65℃中水浴3 h,水浴期间每隔30 min将离心管颠倒混匀→加入等体积苯酚,参照苯酚法抽提→取上清用预冷的异丙醇-20℃沉淀3 h→10 000 r/min离心10 min,去液体→加入75%乙醇洗涤,6 000 r/min离心5 min,去液体→干燥2 h~3 h→再加入 50 μ L TE,使DNA充分溶解,置-20℃保存[8]。
苯酚法提取的DNA纯度高、电泳条带完整、清晰,所得DNA质量有可能优于CTAB法,但其提取过程较为复杂,耗时长、成本较高。CTAB法所提取的DNA能适用于一般分子生物学研究,且该方法在实验操作中不用液氮碾磨,耗时短、降低了成本。对于分子系统学研究来说,两种方法都是可行的。
1.2 目的基因、研究类群、PCR引物的选择及其扩增条件
1.2.1 目的基因、PCR引物的选择 不同目的基因,所选用的引物不同,蚌科动物分子系统发育研究选用的目的基因引物序列都比较多,详见表1。
1.2.2 不同基因片段的PCR扩增反应条件
(1)扩增16 S rRNA片段所用引物为16 S 3L和 16 S 4H(表 1)。
94℃,2 min→35个循环(94℃,1 min→57℃,30s→72℃,1 min)→72℃延伸8 min[5]。
(2)COI片段扩增用引物为COI-22me和COI-700dy(表1)。
94℃,2 min→35个循环(94℃,30 s→42℃,30 s→72℃,1.5 min)→72℃延伸3 min[9]。
(3)ND1片段扩增用引物为Leu-uurF和NIJ-12073(表1)。
94℃,2 min→34个循环(94℃~98℃,40 s→50℃~58℃,1 min→68-72℃,1.5 min)→72℃延伸3 min[10]。
(4)Cytb片段扩增用引物为Cytb-F和Cytb-R(见表1)。
94℃,2 min→40个循环(94℃,1 min→50℃,1 min→72℃,2 min)→72℃延伸3 min[11]。
(5)ITS1片段扩增用引物为18 S和5.8 S(见表1)。
95℃,5 min→30个 循环(95℃,30s→48℃~55℃,1 min→72℃,1 min)→72℃延伸3 min[12]。
(6)ITS2片段扩增用引物为ITS2-F和ITS2-R(见表1)。
95℃,5 min→30个循环(95℃,30s→48℃~55℃,30 s~60 s→72℃,1 min)→72℃延伸3 min[13]。
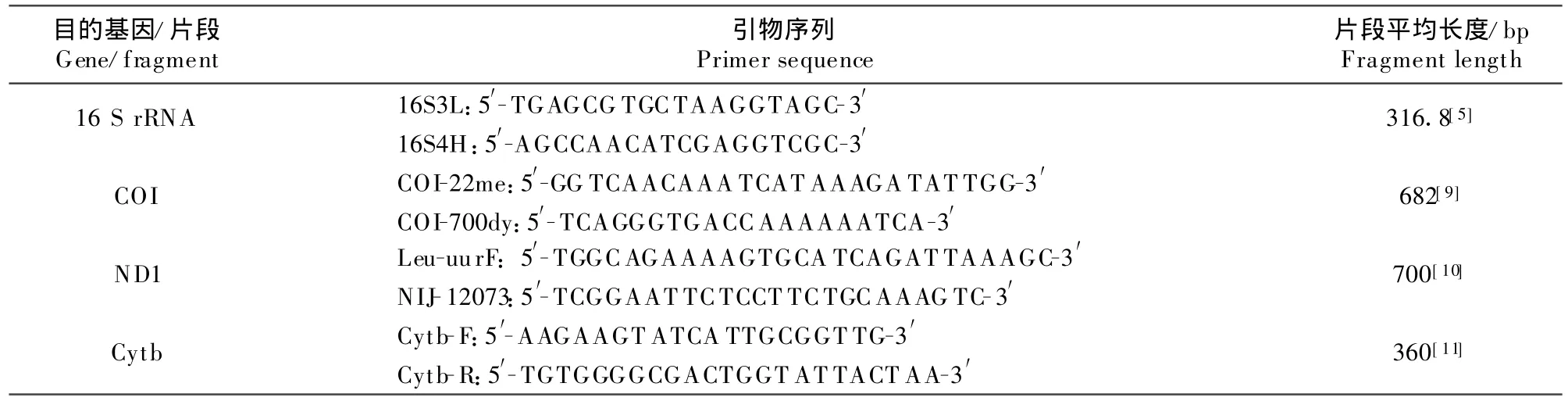
表1 蚌科动物扩增不同DNA片段/基因所用引物序列Table 1 Primers of different DNA/gene in Unionidae for PCR
2 主要研究结果
Graf Daniel L等[2]将蚌科分为两个亚科,即Unioninae和 Ambleminae,其中 Unioninae包括Unionini和Anodontini两大类群,Ambleminae包括 Amblemini、Quadrulini、Pleurobemini、和 Lampsilini共四大类群。
而根据 NCBI(http://www.ncbi.nlm.nih.gov/Taxonomy/Browser/wwwtax.cgi?mode=Undef&id=47526&lvl=3&keep=1&srchmode=1&unlock)的分类信息来看,全世界所有蚌科动物可 分 为 Unioninae、Anodontinae、Ambleminae、Lampsilinae 4个亚科。我国的蚌科动物主要属于Unioninae和Anodontinae,有部分种类可归为Ambleminae[2],Lampsilinae大多分布在为北美地区(参考:http://www.inhs.uiuc.edu/%7eksc/Mussel-Genera.html)。
2.1 Unioninae(珠蚌亚科)的系统发育研究
Unioninae珠蚌亚科蚌类包括Acuticosta(尖嵴蚌属)、Arconaia(扭蚌属)、Cuneopsis(楔蚌属)、Hyriopsis(帆蚌属)、Lanceolaria(矛蚌属)、Schistodesmus(裂脊蚌属)、Solenaia(蛏蚌属)、Unio(珠蚌属)、Ptychorhynchus(尖锄蚌属)、Inversidens、Villosa共 11个属。除Inversidens分布在日本,Villosa分布在北美,其他9个属均为我国特有蚌类[2]。
Huang Y Y等[5]以线粒体16S rRNA部分序列为基础,以贻贝为外类群,结果表明:帆蚌属、蛏蚌属、尖锄蚌属应归为小方蚌亚科。魏开建[12]以IST1部分序列为基础,长耳珠母贝(Pinctada chemnitzi)和合浦珠母贝(Pinctada martensi)2个种类为外类群,结果表明,我国珠蚌亚科的扭蚌、剑状矛蚌、短褶矛蚌、圆头楔蚌、绢丝丽蚌、圆顶珠蚌、中国尖峭蚌聚为一支,置信度为100%。周春花等根据16 S rRNA基因和ND1基因的部分核苷酸序列构建了中国蚌科丽蚌属的分子系统发育,并建立中国蚌科的系统发育树。结果显示:丽蚌属形成两个明显的类群,构成尖丽蚌属和丽蚌属。尖丽蚌属属于珠蚌亚科,丽蚌属属于缓行蚌亚科(小方蚌亚科)[14]。
2.2 无齿蚌亚科的系统发育研究
Anodontinae(无齿蚌亚科)包括Anodonta(无齿蚌属)、Cristaria(冠蚌属)、Lepidodesma(鳞皮蚌属)、Alasmidonta(Say,1818-North America)、Anodontoides(Simpson,1898-North America)、Gonidea(Conrad, 1857-North America)、Lasmigona(Rafinesque,1931-North America)、Pilsbryoconcha(Simpson 1900-Southeast Asia)、Pseudanodonta(Simpson, 1900-Europe)、Pyganodon(Crosse&Fischer,1894-North America)、Strophitus(Rafinesque,1820-North America)、Utterbackia(Baker 1928-North America)13 个属。
Mock K E等[15]以COI序列为基础,对Bonneville Basin流域和北美西部其他几个流域的Anodonta(无齿蚌属)群体进行了比较,结果表明,Bonneville Basin流域的无齿蚌属群体与相邻Nevada的Lahontan Basin关系较近,但总的系统发育树差异较小,因此,影响无齿蚌的数量减少的因素需要对人为因素进行更多的探讨,同时要更深的综合分析北美西部无齿蚌的遗传与形态变异。
Chong Jer Pin等[16]以COI序列为基础分析了北美西部无齿蚌属群体相互关系,表明了存在着3个差异明显 的 类 群,即A.californiensis/nuttalliana、A.oregonensis/kennerlyi和A.beringiana,A.californiensis和A.oregonensis距离较远,而A.b eringiana和亚洲的A.woodiana关系则比其他两个类群都要近。King Tim L等[17]以COI和ITS1序列为基础分析了Lasmigona subviridis种内的系统发育关系,表明Susquehanna和Potomac Rivers很多南部群体存在种内遗传分离,不同河流之间基因流动少,南北群体生殖隔离,在保种时应该作为两个不同的单元进行保种,也许Lasmigona属的4个代表种还可以进一步分类。
2.3 Ambleminae(小方蚌亚科)的系统发育研究
Ambleminae(小方蚌亚科)包括Lamprotula(Simpson,1900-Asia,丽蚌属)、Cyprogenia(Agassiz,1852-North America,膨蚌属)、Dromus(Simpson,1900-North America,走蚌属)、Fusconaia(Simpson,1900-North America,楔状水蚌属)、Plethobasus(Simpson,1900-North America,丰底蚌属)、Pleurobema(Frierson,1927-North America,侧底蚌属)、Quadrula(Rafinesque,1820-North America,方蚌属)、Amblema(Rafinesque,1820-North America)、Caelatura(Egypt)、Ellipsaria(Rafinesque,1820-North America)、Elliptio(Rafinesque,1819-North America)、Elliptoideus(Frierson,1927-North America)、Hemistena(Rafinesque,1820-North America)、Lexingtonia(Ortmann,1914-North America)、Megalonaias(Utterback,1915-North America)、Plectomerus(Conrad,1853-North America)、Potamida(Swainson,1840-European)、Pseudodon(Gould 1844-Singapore)、Quincuncina(Ortmann,1922-North A-merica)、Tritogonia(Agassiz,1852-North America)、Uniomerus(Conrad,1853-North America)共21个属。
Elderkin C L等[9]以COI序列为基础分析了Ambelma plicata的群体遗传结构,结果表明,从北到南群体内的遗传变异不断增大,Strawberry河流和Lake Erie流域群体的遗传变异较低Lake Erie流域和Ohio河流有着相似的遗传结构,对Ambelma plicata来说保护物种的多样性要注重群体各个体的数量。
Sleem Setaita H等[18]用分子标记RAPD技术研究表明Caelatura属Nile河的两个有疑问种C.companyoi和C.prasidens按遗传距离的标准来评价也是完全两个不一样的物种。
Elderkin C L等[19]以COI序列为基础,分析了北美 Lake Erie和Ohio River水域,沿着Ouachita和Strawberry河的东南方Elliptio dilatata的种群结构,结果表明,种群内遗传分化明显,不同水域差异明显,由北到南,种群内变异不断增加,Strawberry河的遗传分化较低,同时发现在Ouachita河的种群有着明显的生殖隔离现象,北部的基因流动较少。
Serb Jeanne M 等[10]以ND1序列为基础,分析了北美 Quadrula属的系统发育关系,结果表明Quadrula可分为3大类群,即Quadrula,Metanvera和Pustulosa,与以往的分类不同的是pustulosaquadrula类群还包含 Fusconaia和 Quincuncina属,Quadrula还包括T ritogonia属。
Grobler Paul J等[20]以ND1和IST1序列为基础,分析了美国东南部 Tennessee River水域Dola-belloides分类情况,结果表明该区域各流域的类群距离差异小,按分子信息的标准属于一类,但在Tennessee Rive水域可以划为两个管理单元。
Burdick Ryan C等[21]以COI序列为基础,分析了Mississippi River,加拿大,Great Lakes和Gulf Coast水域Fusconaia f lava的系统发育情况,表明改物种可以分为两大类,即F.f lava和F.cerina,而不是通常认为的单个类群,但两大类群的关系需要结合遗传和形态特征进行进一步研究。
2.4 亚科的系统发育研究
Lampsilinae亚科包括Actinonaias(Crosse&Fischer,1886-North America)、Cyrtonaias(Crosse&Fischer,1894-Central America)、Epioblasma(Simpson,1900-North America)、Glebula(Conrad,1853-North America)、Hamiota(Roe and Hartfield(2005)-North America)、Lamellidens(Simpson, 1900-Asia-India)、Lampsilis(Rafinesque, 1820-North America)、Leptodea(Rafinesque,1920-North America)、Medionidus(Simpson,1900-North America)、Obliquaria(Rafinesque,1820-North America)、Obovaria(Conrad, 1853-North America)、Potamilus(Rafinesque, 1818-North America )、Ptychobranchus(Simpson,1900-North America)、Toxolasma(Rafinesque,1831-North America)、Truncilla(Rafinesque,1820-NorthAmerica)、Paraleptodea共 16个属,除Lamellidens(Simpson,1900-Asia-India)、Paraleptodea外其他14个属都分布在北美。
Zanatta David T等[22]用微卫星标记技术和以COI序列为基础,分析了北美流域Epioblasma triquetra的系统地理学和群体遗传结构,结果表明,Clinch和St Francis河流的mtDNA差异显著,微卫星标记结果也表明,群体之间差异显著,并有着明显的遗传结构,这表明Epioblasma triquetra群体可分为3大地理类群,即Tennessee River,Ozark Crest河南部和与Mississippi相邻的Ohio River的下游。Zanatta David T等[23]用微卫星标记技术研究表明,加拿大Ontario流域Lampsilis f asciola群体不是随机交配的,而有着显著的群体遗传结构,且基因流动明显。
3 中国蚌科分子系统学研究现状和展望
我国蚌科有一百多种,是蚌科物种多样性丰富的地区之一[24]。近年来随着湖泊、河流等内陆水域污染程度加剧、对蚌科栖息地的人为干预和资源的过度开发利用,在一定程度上对蚌类的种质资源造成破坏,一些经济蚌类的自然资源日益减少甚至濒临灭绝。目前中国的分类体系是基于传统的形态学分类[25],分子上的证据还很匮乏。通过DNA序列变化的研究,来探讨中国蚌科的系统发育问题,可为进一步研究中国淡水贝类的系统发育研究提供遗传物质本身——DNA依据。结合形态学、解剖学、生理学各方面的特征,将是我国蚌科分子系统学未来发展的主要研究手段。
[1]Stray er D L,Downing J A,Haag W R,et al.Changing perspectives on pearly mussels,orth America's most imperiled animals[J].Bioscience,2004,54:429-439.
[2]G raf D L,Cummings K S.Review of the systematics and global diversity of freshwater mussel species(Bivalvia:Unionoida)[J].J Molluscan Studies,2007,73(4):291-314.
[3]Bogan A E.Global diversity of freshwater mussels(Mollusca,Bivalvia)in freshwater[J].Hydrobiologia,2008,595:139-147.
[4]Strayer D L.Challenges for freshwater invertebrate conservation[J].J North American Benthological Society,2006,25:271-287.
[5]Huang Y Y,Liu H Z,Wu X P,et al.Testing the relationships of chinese freshwater unionidae(Bivalvia)based on analysis of partial mitochondrial 16 S rRNA sequences[J].J Molluscan Studies,2002,68:359-363.
[6]Walker J M,Curole J P,Wade D E,et al.Taxonomic distribution and phylogenetic utility of genderassociated mitochondrial genomes in the Unionoida(Bivalvia)[J].Malacologia,2006,48:265-282.
[7]贾名静,李家乐,牛东红,等.长江中下游褶纹冠蚌10个群体COⅠ基因序列变异分析[J].动物学杂志,2009,44(1):1-8.
[8]Shahjahan R M,Hughes K J,Leopold R A,et al.Lower incubation temperature increases yield of insect genomic DNA isolated by the CTAB method[J].Biotechniques,1995,19:332-334.
[9]Elderkin C L,Christian A D,Vaughn C C,et al.Population genetics of the freshwater mussel,Amblema plicata(Say 1817)(Bivalvia:Unionidae):Evidence of high dispersal and post-glacial colonization[J].Conserv Genet,2007,8:355-372.
[10]Serb J M,Buhay J E.M olecular systematics of the North A-merican freshwater bivalve genusQuadrula(Unionidae:Ambleminae)based on mitochondrial ND1 sequences[J].Molecular Phylogenetics and Evolution,2003,28:1-11.
[11]Zanatta D T,Murphy R W.Range-wide population genetic analysis of the endangered northern riffleshell mussel,Epioblasma torulosa rangiana(Bivalvia:Unionoida)[J].Conserv Genet,2007,8:1393-1404.
[12]魏开建.中国蚌科的遗传多样性与系统发育的研究[D].湖北武汉:华中农业大学,2004.
[13]Cheng Han-Liang,Xia De-Quan,Wu Ting-Ting,et al.Study on sequences of ribosomal DNA internal transcribed spacers of clams belonging to the veneridae family(mollusca:bivalvia)[J].Acta Genetica Sinica,2006,33(8):702-710.
[14]周春花,欧阳珊,吴小平,等.基于16S rRNA和ND1基因序列的中国蚌科丽蚌属的系统发育[J].动物学报,2007,53(6):1024-1030.
[15]M ock K E,Brim box J C,Miller M P,et al.Genetic diversity and divergence among freshwater mussel(Anodonta)populations in the Bonneville Basin of Utah[J].Mol Ecol,2004,13:1085-1098.
[16]Chong J P,Brim Box J C,Howard J K,et al.Three deeply divided lineages of the freshwater mussel genusAnodontain western North America[J].Conserv Genet,2008,9:1303-1309.
[17]King T L,Eackles M S,Gjetvaj B,et al.Intraspecific phylogeography ofLasmigona subviridis(Bivalvia:Unionidae):conservation implications of range discontinuity[J].Mol Ecol,1999,8:S65-S78.
[18]Sleem S H,Ali T G.Application of RAPD-PCR in tax onomy of certain freshwater bivalves of genusCaelatura[J].Global J M ol Sci,2008,3(1):27-31.
[19]Elderkin C L,Christian A D,Metcalfe-smith J L,et al.Population genetics and phylogeog raphy of freshwater mussels in No rth America,Elliptio dilatataandActinonaias ligamentina(Bivalvia:Unionidae)[J].Mol Ecol,2008,17,(9)2149-2163.
[20]Grobler Paul J,Jones W,Johnson N A,et al.Patterns of genetic differentiation and conservation of the Slabside pearl ymussel,lexingtonia dolabelloides(LEA,1840)in the tennessee river drainag[J].J Molluscan Studies,2006,72(1):65-75.
[21]Burdick Ryan C,White M M.Phy logeography of the wabash pig toe,Fusconaia f lava(rafinesque,1820)(Bivalvia:uninidae)[J].J Molluscan Studies,2007,73:367-375.
[22]Zanatta D T,Fraley S J,Murphy R W.Phylogeography of the wabash pigtoe,Fusconaia Flava(Rafinesque,1820)(Bivalvia:unionidae)[J].Can J Zool,2007,85(11):1169-1181.
[23]Zanatta D T,Murphy R W.The phylogeographical and management implications of genetic population structure in the imperiled snuffboxmussel,Epioblasma triquetra(Bivalvia:Unionidae)[J].Bio J Linnean Society,2008,93:371-384.
[24]Prozorova L A,Sayenko E M,Bogatov V V,et al.Bivalves of the Yangtze River drainage[J].Byulleten'Dal'nevostochnogo M alakologicheskogo Obshchestva,2005,9:46-58.
[25]刘月英,张文珍,王跃先,等.中国经济动物志(淡水软体动物)[M].北京:科学出版社,1979:66-116.
Application of DNA Sequences in Phylogenetic Study of Unionidae
OUYANG Jie-xiu,WU Xiao-ping,OUYANG Shan,LI Shao-bo
(College of Li fe Sciences and Food Engineering,Nanchang University,Nanchang,J iangxi,330031,China)
Unionids are among the most imperiled organisms in the world.DNA is the carrier of genetic information,and sequencing and comparison of DNA are the ideal methods for unionidae molecular phylogenesis,is and the the most critical informations for making conservation plan.From two aspcets of methods and results,the paper summarized the advances in molecular systematics of unionidae.The methods including DNA extraction,gene sampling,primers choosing,gene amplification conditions.The paper also pointed out the status and prospects in molecular systematics research for unionidae in China.
DNA sequence;Unionidae;molecular phylogeny
Q789
A
1007-5038(2010)08-0069-05
2010-01-20
资金项目:国家自然科学基金项目(30860045)资助
欧阳解秀(1978-),女,湖南邵阳人,博士研究生,主要从事蚌类分子系统学研究。*通讯作者