通过加权基因共表达网络和LASSO分析识别儿童呼吸道合胞病毒的诊断基因
摘 要 目的:运用生物信息学方法挖掘儿童呼吸道合胞病毒(RSV)的诊断标志物,为RSV的靶向治疗提供依据。方法:从GEO中获得RSV的基因表达数据集。运用差异表达分析、免疫浸润分析、WGCNA和LASSO确定RSV的关键生物标志物。同时,绘制ROC曲线对关键基因的诊断性能进行评估。结果:共识别出592个差异基因和10种差异浸润的免疫细胞,例如中性粒细胞。WGCNA和LASSO分析筛选出7个关键的生物标志物,包括ADM、LTF、UBE2J1、PDK4、S100A4、BST1、CAMP。此外,ROC曲线分析发现这些基因具有较高的诊断价值。结论:筛选的7个基因可作为RSV的诊断生物标志物。
关键词 呼吸道合胞病毒 诊断基因 中性粒细胞 加权基因共表达网络分析
中图分类号:R446.9; R725.1 文献标志码:A 文章编号:1006-1533(2024)19-0063-09
引用本文 董海丽, 王国胜, 谷双龙, 等. 通过加权基因共表达网络和LASSO分析识别儿童呼吸道合胞病毒的诊断基因[J]. 上海医药, 2024, 45(19): 63-71.
Identification of diagnostic genes related to respiratory syncytial virus in children based on WGCNA and LASSO algorithms
DONG Haili, WANG Guosheng, GU Shuanglong, SONG Nanping
(Department of Pediatrics, Zhengzhou 460 Hospital, Zhengzhou 450000, China)
ABSTRACT Objective: The diagnostic biomarkers of respiratory syncytial virus (RSV) in children were explored by bioinformatics analysis to provide basis for targeted therapy of RSV. Methods: Gene expression profiles of RSV were obtained from the GEO database. Differential expression analysis, immune infiltration analysis, WGCNA and LASSO were applied to identify the key biomarkers of RSV. Meanwhile, ROC curve was utilized to evaluate the diagnostic performance of key genes. Results: A total of 592 differentially expressed genes and 10 differential immune cells (such as neutrophils) were identified between RSV and healthy samples. Further, WGCNA and LASSO analysis screened out seven key biomarkers, including ADM, LTF, UBE2J1, PDK4, S100A4, BST1, and CAMP. In addition, ROC curve showed that these genes had high diagnostic value. Conclusion: The identified seven biomarkers may serve as diagnostic biomarkers for RSV.
KEY WORDS respiratory syncytial virus; diagnostic genes; neutrophils; weighted gene co-expression network analysis
呼吸道合胞病毒(respiratory syncytial virus,RSV)是全球儿童呼吸道感染的主要原因[1]。据统计,每年有210万5岁以下的儿童因RSV感染需要门诊治疗或住院,并且在全世界28 d至6个月的婴儿中,RSV疾病的总死亡率负担为1/28[2-3]。因此,迫切需要有效的RSV预防策略来解决这一重大公共卫生问题和负担。帕利珠单抗广泛的用于预防婴儿RSV疾病[4]。尽管其药效已经得到证实,但是尚未引进国内临床应用[5]。迄今为止,RSV疫苗仍然处于开发阶段。此外,诊断RSV感染的有效方法也十分有限。所以,了解RSV感染的分子发病机制对于指导未来的诊断和治疗至关重要。目前,已有研究旨在寻找RSV的诊断标志物,揭示其潜在的分子机制[6-7]。但是,识别RSV诊断生物标志物的相关研究仍然缺乏。本研究利用一系列生物信息学分析旨在探讨与RSV中免疫浸润细胞相关的关键生物标志物。本工作将有望为提高RSV患者的诊断以及治疗靶点的开发提供帮助。
1 材料和方法
1.1 表达谱数据的收集和预处理
从NCBI GEO数据库中下载6个儿童RSV芯片数据集,包括GSE188427、GSE105450、GSE103842、GSE103119、GSE80179、GSE77087。其中,GSE188427包括353个样本,来自于51个健康志愿者和302个RSV患者的全血样本,在本研究中作为训练集。其余数据集作为模型验证数据集。另外,使用R语言中的affy包对下载的数据进行预处理,并使用rma函数进行归一化处理。
1.2 基因差异表达分析
通过limma包对健康和RSV患者中的基因进行差异表达分析,计算基因相应的P值和logFC值。同时,利用FDR方法对P值进行多重检验校正,得到P-adjust(Benjamini-Hochberg校正)。差异表达基因(differential expressed genes,DEGs)的阈值为:P-adjust<0.05和|logFC|>1.2。
1.3 GSEA功能分析
为了探索疾病组和正常组之间的差异调节通路,使用GSEA(V4.3.2)对DEGs进行功能富集分析,包括GO-BP、KEGG和Reactome。显著富集的阈值为P-adjust<0.05。
1.4 免疫微环境分析
基于患者和对照样本的基因表达数据,利用CIBERSORT对样本中22种免疫细胞的比例进行计算。随后,运用Wilcox检验比较了两组间免疫细胞浸润水平的差异。
1.5 WGCNA分析
使用WGCNA筛选与RSV有关的关键基因。通过筛选中位数绝对偏差(median absolute deviation,MAD)排名前75%且MAD值>0.01的基因进行共表达分析(13 963个基因)。随后,计算每个模块与差异的免疫细胞浸润程度之间的相关性。获得与免疫细胞最相关的模块后(又称为hub模块),将模块基因与DEGs取交集,从而得到与免疫细胞浸润相关的关键基因用于后续分析。
1.6 关键基因的功能富集分析
利用DAVID 2021对这些基因进行GO功能和KEGG通路富集分析。选取P-adjust<0.05作为显著富集的阈值。
1.7 诊断基因筛选
运用R4.3.1语言中的glmnet包(V4.1-7)从关键基因中筛选诊断基因。首先,进行单因素logistic回归分析,选取P<0.05的基因进行后续分析。接下来,利用LASSO分析来筛选RSV的诊断基因。LASSO回归分析在R软件中运行10次K交叉验证,用于对包含的变量进行居中和归一化处理,然后选择最佳的l值。
1.8 列线图的构建
通过rms包绘制包括诊断基因的列线图来判断RSV患病的风险率,并且绘制校正曲线来验证模型的准确性。
1.9 外部数据集验证
在5个验证队列中,对诊断基因在健康对照和RSV样本中的表达水平进行比较。随后,利用pROC包绘制诊断基因的ROC曲线,通过计算AUC值评估基因的预测性能。
2 结果
2.1 DEGs筛选和GSEA富集分析
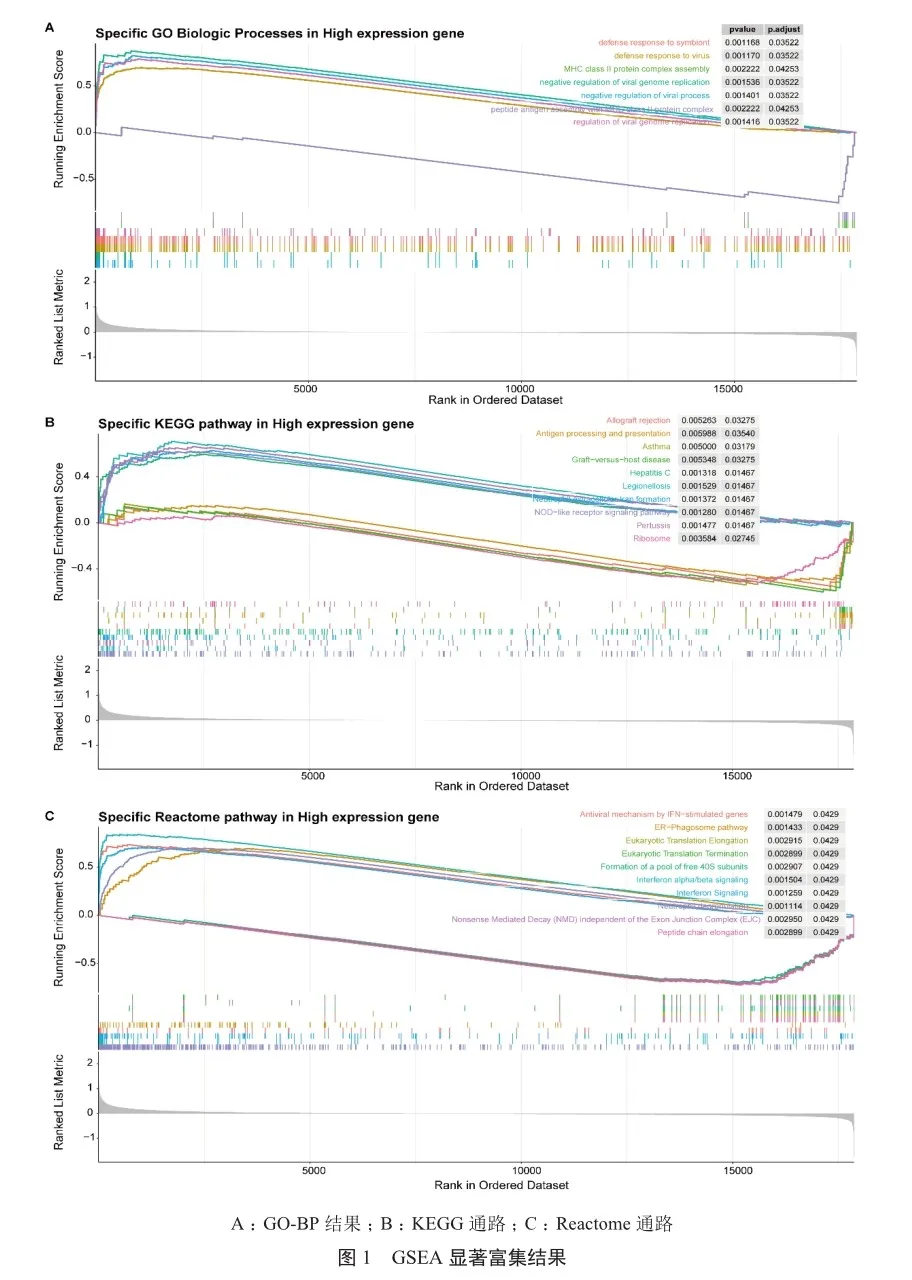
在RSV和对照中共筛选到592个DEGs,包括453个上调和139个下调基因。GSEA结果揭示这些基因主要富集在380个GO-BP类目,57个KEGG通路和149个Reactome通路。图1展示了NES排名前5的结果。
2.2 RSV和健康对照患者呈现不同的免疫景观
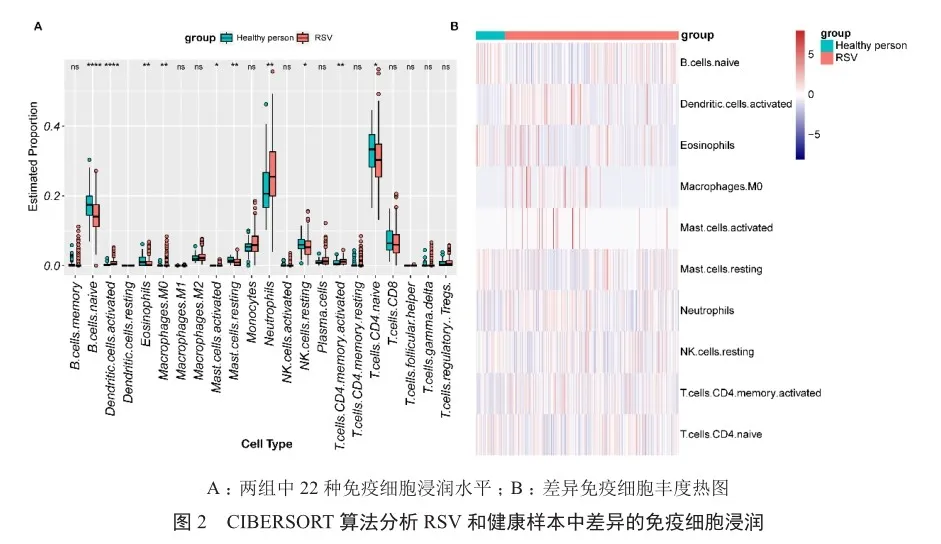
10种免疫细胞的浸润水平在两组中有显著差异(图2A和2B)。在RSV样本中,活化树突状细胞(activated dendritic cells)、M0型巨噬细胞(M0 macrophages)、活化肥大细胞(activated mast cells)、中性粒细胞(neutrophils)、活化CD4记忆T细胞(activated T cells CD4 memory)的丰度远高于对照样本,而native B细胞(native B cells)、嗜酸性粒细胞(eosinophils)、静息肥大细胞(resting mast cells)、静息自然杀伤细胞(resting NK cells)、天然CD4 T细胞(native T CD4 cells)的丰度低于对照样本。
2.3 利用WGCNA识别关键模块
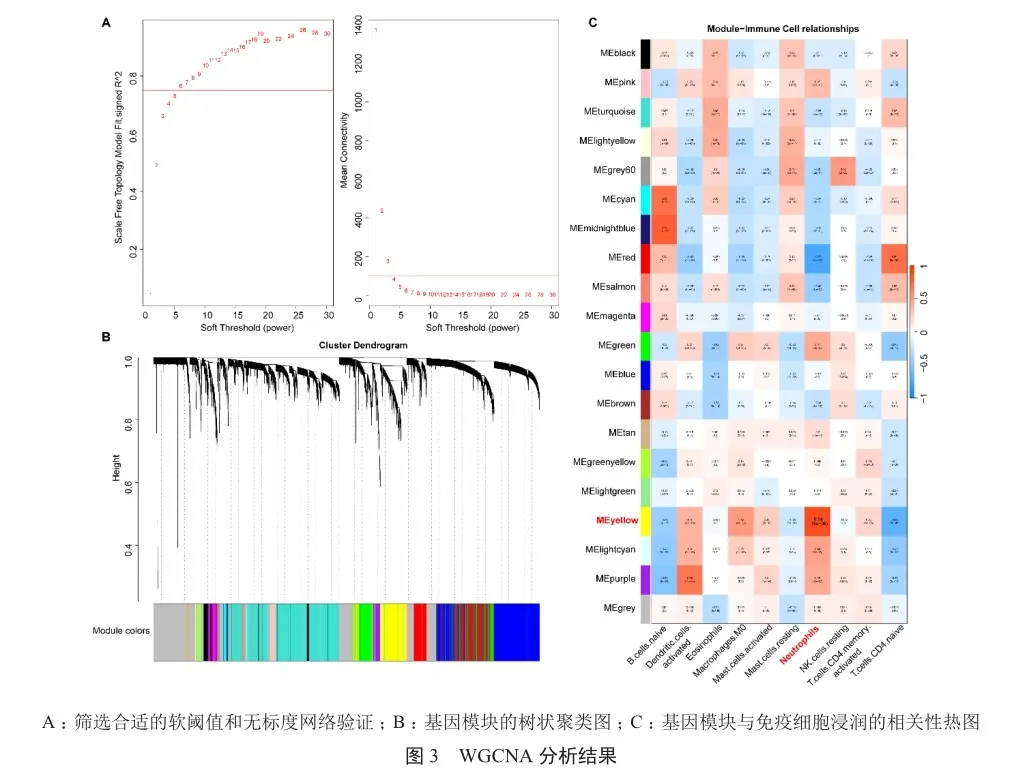
如图3A所示,当软阈值为6时(对应的R2为0.75),满足网络的无标度拓扑,且平均连通性高。通过基因相关性构建层次聚类树状图,共识别出20个相似的基因模块(图3B)。随后,以免疫浸润程度作为表型,分析模块特征值与10种差异免疫细胞之间的相关性。包含991个基因的“MEYellow”模块与中性粒细胞具有较强的相关性,皮尔森相关系数为0.84(图3C)。因此认为“MEYellow”模块为hub模块,其中包含的基因用于后续分析。
2.4 RSV中关键差异基因的筛选和功能富集分析
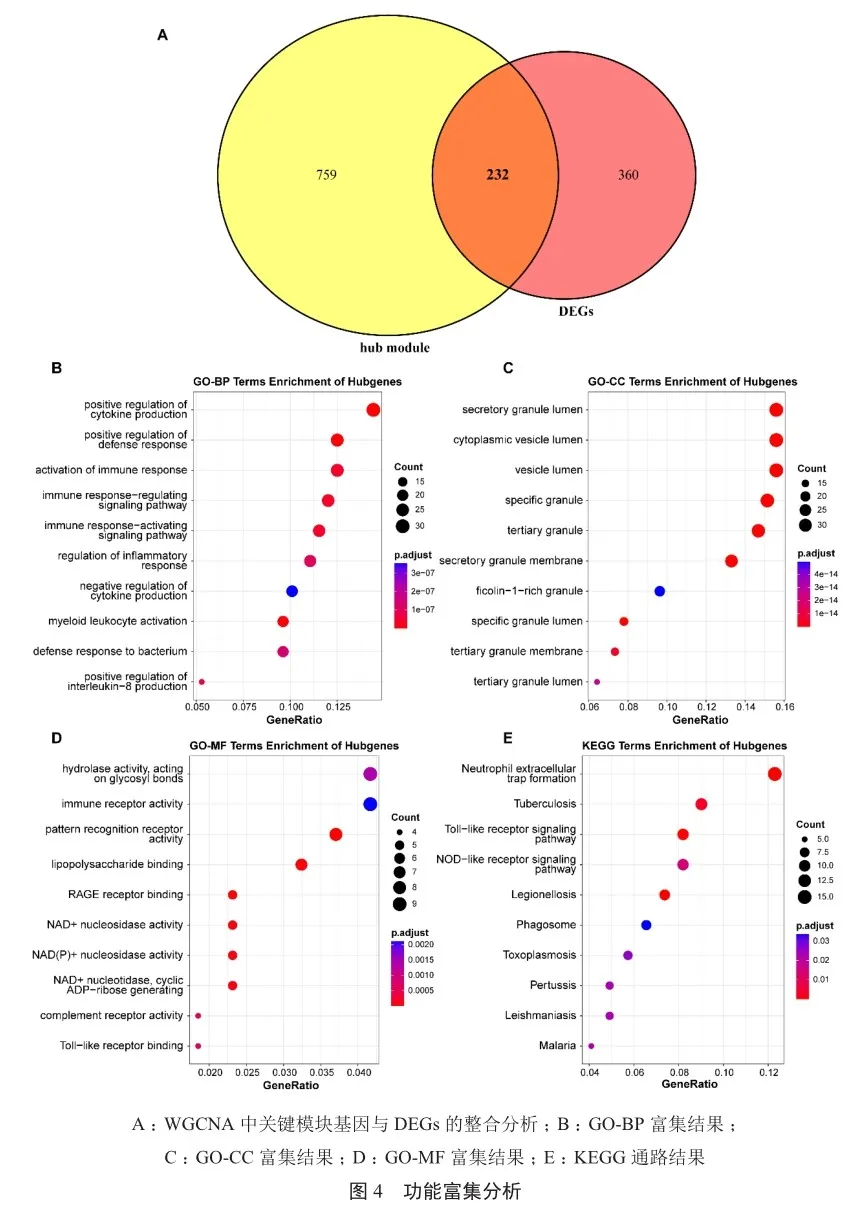
将DEGs和“MEYellow”模块基因整合,得到232个重叠的基因(图4A)。对这些关键基因进行功能富集分析。GO富集分析结果显示,基因主要影响细胞因子产生的正向调节(positive regulation of cytokine production)(GO-BP)、分泌颗粒腔(secretory granule lumen)(GO-CC)、模式识别受体活性(pattern recognition receptor activity)(GO-MF)(图4B~D)。KEGG通路分析发现基因在中性粒细胞胞外陷阱的形成(neutrophil extracellular trap formation)等通路中显著富集(图4E)。
2.5 RSV中诊断基因的鉴定
为了进一步识别核心的诊断基因,基于lambda值进行了LASSO回归分析。共识别出7个RSV中最具代表性的诊断基因,包括ADM、LTF、UBE2J1、PDK4、S100A4、 BST1、CAMP(图5A~C)。随后,基于这7个诊断生物标志物,建立了预测RSV的列线图模型(图5D)。校准曲线显示疾病的预测概率和实际发生概率之间的误差很小,表明列线图对RSV患者的预测准确性较高(图5E)。

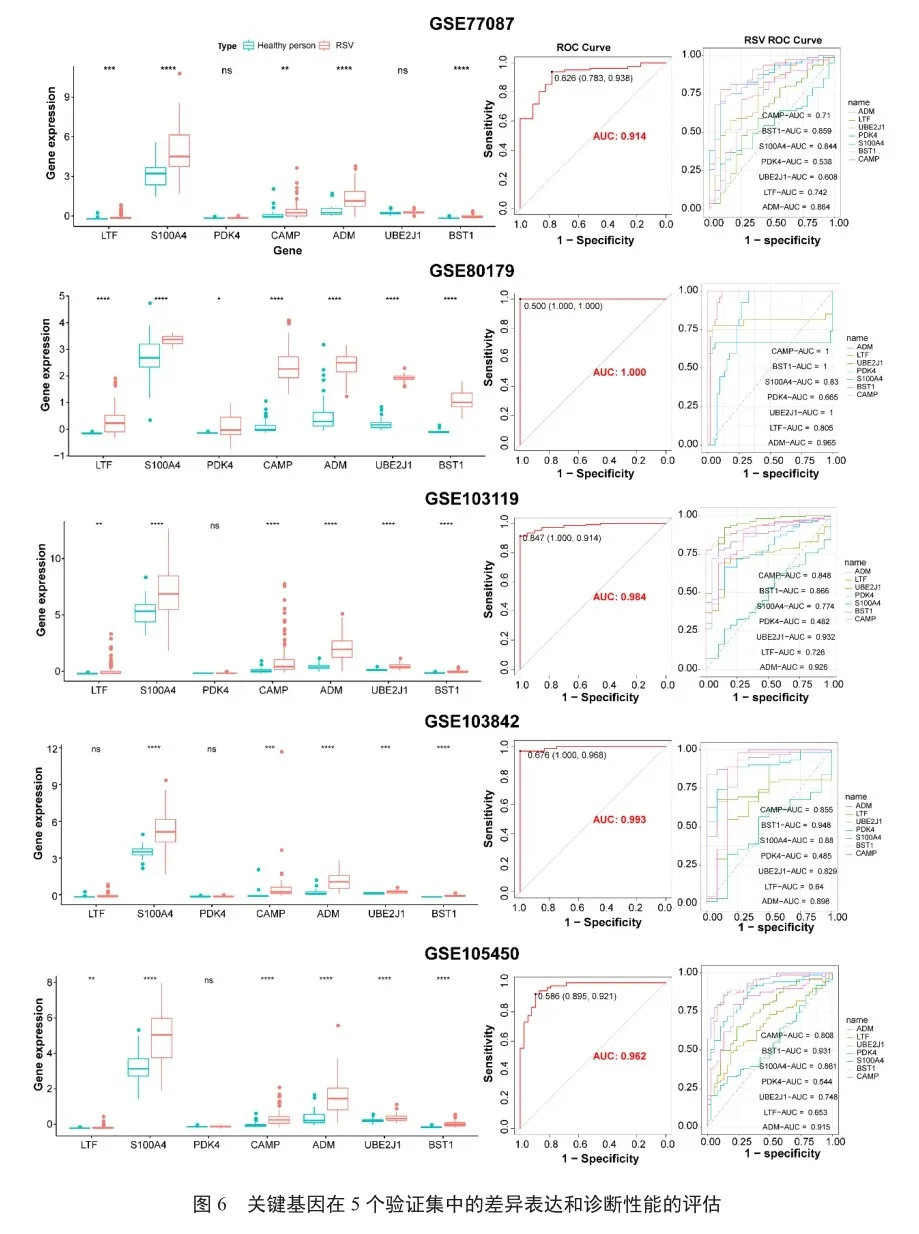
2.6 生物标志物的验证
利用5个外部验证数据集对7个特征基因的表达和诊断效能进行验证(图6)。与对照组相比,RSV组中7个基因的mRNA水平呈现上调趋势,尤其是S100A4、CAMP、ADM、BST1(均P<0.05)。在5个数据集中,诊断基因联合模型的AUC值均大于0.9,表明模型具有较高的分类性能。此外,还对每个基因进行了ROC分析。在5个验证集中,ADM、UBE2J1、S100A4、BST1、CAMP的AUC值均大于0.7。这些结果表明筛选的特征标志物在RSV中显示出良好的诊断效能。
3 讨论
有证据表明天然免疫应答的不同组成部分参与RSV感染的控制,且适当的固有免疫应答在RSV感染的消退中具有重要的作用[8-9]。所以,RSV的免疫病理学机制的研究有助于疫苗的研发。
本研究发现中性粒细胞在RSV中的浸润水平显著增加。已有研究报道了RSV感染与中性粒细胞之间的相关性。临床研究发现,RSV感染后,约80%浸润的细胞是中性粒细胞,并且其强烈的内流是RSV诱导的急性下呼吸道感染的气道炎症的常见特征[10-11]。另外,RSV感染中的中性粒细胞气道炎症会导致黏液生成过多、上皮损伤和气道重塑,进而引起肺功能损伤和肺部疾病[12]。因此,在RSV感染期间靶向中性粒细胞可能是改善患儿的不良肺部预后的潜在治疗方法。后续的分析中,本研究重点关注了中性粒细胞相关的关键基因。
结合WGCNA和LASSO回归分析,最终确定了7个具有诊断性能的生物标志物,包括ADM、LTF、UBE2J1、PDK4、S100A4、BST1、CAMP。ROC分析发现这些基因均具有良好的诊断性能。气道ADM的异常表达可能导致上皮和黏膜功能失调,与哮喘的发生和发展有关,并可能是预测社区获得性肺炎死亡率的一个关键的生物标志物[13-14]。UBE2J1在内质网上可降解错误折叠的蛋白质,其过表达可增强登革热病毒感染,并且该基因可通过影响泛素化过程在重症COVID-19患者中起关键作用[15-16]。S100A4在炎症过程中发挥重要作用。已有证据表明其与淋巴细胞计数呈正相关,因此该基因参与哮喘气道炎症的发病机制[17]。BST1可抑制中性粒细胞通过内皮层的浸润,发挥抗炎特性[18]。CAMP(又称为LL-37)具有抗菌、细胞趋化和炎症反应调节功能[19]。RSV感染后可诱导气道上皮细胞表达LL-37,并且该基因可能是先天性宿主抵抗RSV感染的重要靶点[20]。LTF编码的蛋白在中性粒细胞的次级颗粒中发现,具有已知的抗病毒活性,包括RSV[21]。儿童脓毒症心肌病患者的血清中PDK4水平显著升高,并且与疾病的严重程度和器官损伤密切相关[22]。总而言之,这些研究证实筛选出来的基因均与炎症相关的疾病有关,可间接或直接地参与RVS的发生。
本研究还发现基因主要富集在“中性粒细胞胞外陷阱的形成”通路中。在RSV患儿的气道中观察到大量的中性粒细胞募集,而激活的中性粒细胞可释放中性粒细胞胞外诱捕网(neutrophil extracellular trap,NETs)[23]。研究发现NETs能够捕获RSV,从而阻止病毒颗粒与靶细胞结合,防止感染。然而,NETs的大量产生会阻塞气道并损害肺功能,从而加重婴幼儿感染的炎症症状[24]。因此,必须严格调控NETs的形成[25]。然而,NETs在RVS感染下的有益和有害影响之间的界限仍未揭示,所以寻找一种靶向NETs及其相关蛋白的理想疗法是亟待解决的一个挑战。
我们的工作首次利用WGCNA结合LASSO方法识别了RSV的特异性诊断生物标志物,加深了对其发病机制的认识。然而,这些发现均是通过生物信息学分析得到的,还需要通过体内和体外实验来探索这些基因在RSV中的具体作用。
参考文献
[1] Weinberg GA. Respiratory syncytial virus mortality among young children[J]. Lancet Glob Health, 2017, 5(10): e951-e952.
[2] Li Y, Wang X, Blau DM, et al. Global, regional, and national disease burden estimates of acute lower respiratory infections due to respiratory syncytial virus in children younger than 5 years in 2019: a systematic analysis[J]. Lancet, 2022, 399(10340): 2047-2064.
[3] Hall CB, Weinberg GA, Blumkin AK, et al. Respiratory syncytial virus-associated hospitalizations among children less than 24 months of age[J]. Pediatrics, 2013, 132(2): e341-e348.
[4] Garegnani L, Styrmisdóttir L, Roson Rodriguez P, et al. Palivizumab for preventing severe respiratory syncytial virus(RSV) infection in children[J]. Cochrane Database Syst Rev, 2021, 11(11): CD013757.
[5] 李秀东. 小儿呼吸道合胞病毒感染性肺炎防治研究进展[J]. 齐齐哈尔医学院学报, 2015, 36(26): 3990-3991.
[6] Dapat C, Kumaki S, Sakurai H, et al. Gene signature of children with severe respiratory syncytial virus infection[J]. Pediatr Res, 2021, 89(7): 1664-1672.
[7] Li L, Ni YA, Song Z, et al. Identification of pathogenic genes and transcription factors in respiratory syncytial virus[J]. BMC Pediatr, 2021, 21(1): 27.
[8] Kim HY, Yun KW, Cheong HJ, et al. Respiratory syncytial virus infection and the need for immunization in Korea[J]. Expert Rev Vaccines, 2023, 22(1): 327-340.
[9] Luo J, Qin H, Lei L, et al. Virus-like particles containing a prefusion-stabilized F protein induce a balanced immune response and confer protection against respiratory syncytial virus infection in mice[J]. Front Immunol, 2022, 13: 1054005.
[10] Jones AC, Anderson D, Galbraith S, et al. Personalized transcriptomics reveals heterogeneous immunophenotypes in children with viral bronchiolitis[J]. Am J Respir Crit Care Med, 2019, 199(12): 1537-1549.
[11] 刘金玲, 陈志敏. 呼吸道合胞病毒毛细支气管炎患儿中性粒细胞活化及蛋白酶平衡的研究[J]. 中华医学杂志, 2009, 89(6): 397-399.
[12] Tahamtan A, Besteman S, Samadizadeh S, et al. Neutrophils in respiratory syncytial virus infection: from harmful effects to therapeutic opportunities[J]. Br J Pharmacol, 2021, 178(3): 515-530.
[13] Hagner S, Welz H, Kicic A, et al. Suppression of adrenomedullin contributes to vascular leakage and altered epithelial repair during asthma[J]. Allergy, 2012, 67(8): 998-1006.
[14] Torres A, Ramirez P, Montull B, et al. Biomarkers and community-acquired pneumonia: tailoring management with biological data[J]. Semin Respir Crit Care Med, 2012, 33(3): 266-271.
[15] Feng T, Deng L, Lu X, et al. Ubiquitin-conjugating enzyme UBE2J1 negatively modulates interferon pathway and promotes RNA virus infection[J]. Virol J, 2018, 15(1): 132.
[16] Huang J, Li R, Cheng J, et al. Transcriptome analysis of peripheral blood mononuclear cells response in patients with severe COVID-19 reveals crucial genes regulating protein ubiquitination[J]. Med Sci Monit, 2022, 28: e937532.
[17] Huang X, Qu D, Liang Y, et al. Elevated S100A4 in asthmatics and an allergen-induced mouse asthma model[J]. J Cell Biochem, 2019, 120(6): 9667-9676.
[18] Wang LJ, Huang YC, Lin PY, et al. BST-1 as a serum protein biomarker involved in neutrophil infiltration in schizophrenia[J]. World J Biol Psychiatry, 2022, 23(7): 537-547.
[19] Zhang L, Wu WKK, Gallo RL, et al. Critical role of antimicrobial peptide cathelicidin for controlling Helicobacter pylori survival and infection[J]. J Immunol, 2016, 196(4): 1799-1809.
[20] Currie SM, Findlay EG, Mchugh BJ, et al. The human cathelicidin LL-37 has antiviral activity against respiratory syncytial virus[J]. PLoS One, 2013, 8(8): e73659.
[21] Van Der Strate BW, Beljaars L, Molema G, et al. Antiviral activities of lactoferrin[J]. Antiviral Res, 2001, 52(3): 225-239.
[22] Chen T, Ye L, Zhu J, et al. Inhibition of pyruvate dehydrogenase kinase 4 attenuates myocardial and mitochondrial injury in sepsis-induced cardiomyopathy[J]. J Infect Dis, 2023, 25: jiad365.
[23] Funchal GA, Jaeger N, Czepielewski RS, et al. Respiratory syncytial virus fusion protein promotes TLR-4-dependent neutrophil extracellular trap formation by human neutrophils[J]. PLoS One, 2015, 10(4): e0124082.
[24] Cortjens B, De Boer OJ, De Jong R, et al. Neutrophil extracellular traps cause airway obstruction during respiratory syncytial virus disease[J]. J Pathol, 2015, 238(3): 401-411.
[25] Porto BN, Stein RT. Neutrophil extracellular traps in pulmonary diseases: too much of a good thing?[J]. Front Immunol, 2016, 7: 311.

