基因芯片初步探讨乳腺癌转移相关蛋白Nucleobindin- 2介导的信号通路
符青云 欧阳小明 郅 程 曾 亮
1 广东省江门市五邑中医院病理科(江门 529031)
2 广州医科大学附属第二医院病理科(广州 510260)
3 广州市妇女儿童医疗中心病理科(广州 510623)
本研究前期结果发现核结合蛋白2(nucleobindin- 2,NUCB2)表达与乳腺癌转移和预后有关[1]。关于NUCB2介导的信号通路有一些报道,如在结肠癌及子宫内膜癌的研究中发现NUCB2通过LKB1/AMPK/TORC1/ZEB1途径增强癌细胞的迁移、侵袭和上皮间充质转化[2,11- 12]。NUCB2通过雷帕霉素靶向途径抑制卵巢癌细胞的增殖[3]。在本研究中,通过基因芯片方法研究乳腺癌细胞中NUCB2介导的信号通路及相关信号分子,为进一步阐明NUCB2乳腺癌中促转移的功能提供思路。
1 材料与方法
1.1 材料
本研究使用乳腺癌细胞株MDA-MB- 231,培养基为90%高糖DMEM+10%胎牛血清。 生长条件为95%空气和5%CO2,37 ℃。qPCR所使用的引物序列见表1。

表1 qPCR的逆转录引物
1.2 RNAi 慢病毒载体构建、包装、感染细胞并检测感染效率
目的基因及工具载体信息,载体酶切:配制 50 μL 酶切体系。对载体酶切产物进行琼脂糖凝胶电泳,回收目的条带。目的基因片段的获取:核心序列、病毒载体构建框架、合成单链引物、引物退火形成双链 DNA。退火产物与载体进行连接。转化。菌落 PCR 鉴定。测序。质粒抽提。最终用于实验的分组包括对照组(无病毒感染的细胞组)、NC组(阴性对照病毒感染的细胞组)、KD组(感染NUCB2基因shRNA病毒的细胞组)。
1.3 总RNA 抽提及实时定量PCR
总RNA 抽提(上海普飞公司Trizol 试剂盒)抽提,收集细胞(6 孔板 80%细胞密度),2 000 r/min离心 5 min,去上清,细胞沉淀中加入 1 mL Trizol,充分混匀后室温静置 5 min,然后转至新的 1.5 mL EP 管中;每管加入 200 μL 氯仿,用手上下颠倒 EP 管 15 s,室温静置 10 min。4 ℃、12 800 r/min,离心 15 min。吸取上层液体转至新的 1.5 mL EP 管,加入等体积预冷的异丙醇,混匀后 4 ℃静置 10 min。4 ℃、12 800 r/min 离心 12 min 后,弃上清。加入 1 mL 、75%乙醇(用 DEPC 水新鲜配制),洗涤沉淀。4 ℃、11 800 r/min离心5 min,弃去大部分上清。 4 ℃、11 800 r/min 再次离心5 min,弃去上清,室温干燥。待 RNA 沉淀基本透明时,加入 RNase-free 水,至完全溶解,Nanodrop 2000/2000C 分光光度计分析测定所抽提 RNA 的浓度及质量。反转录获得cDNA(使用Promega M-MLV 试剂盒)。
Real-time PCR 检测按下列比例配置反应体系(12 μL体系):MicroRNA PCR引物来自广州市锐博生物科技有限公司。试剂每管加入量SYBR premix ex taq 6.0 μL,上游引物(5 μmol) 0.5 μL,下游引物(5 μmol) 0.5 μL,模板(反转录产物) 1.0 μL,RNase-Free H2O 4.0 μL。② RNA PCR试剂 每管加入量SYBR premix ex taq 6.0 μL,引物mix(5 μmol) 0.3 μL,模板(反转录产物) 0.6 μL,RNase-Free H2O 5.1 μL。相对定量分析F=2-ΔΔCt,ΔCt=目的基因Ct值-内参基因Ct值;-ΔΔCt=NC组ΔCt平均值-各样品ΔCt值;ΔΔCt反映各样品相对NC组样品目的基因的相对表达水平,引物序列见表1。
1.4 Affymetrix基因表达谱芯片筛选下游通路相关基因
RNA 质检使用 NanoDrop2000 与 Agilent 2100 检测。3′IVT 反应:总 RNA 起始量范围:50~500 ng。RNA 扩增程序: First-Strand cDNA Synthesis 42 ℃,2 hr → 4 ℃保持。Second-Strand cDNA Synthesis 16 ℃,1 hr → 65 ℃,10 min → 4 ℃保持。IVT 40 ℃,16 hr → 4 ℃保持。Fragmentation 94 ℃,35 min → 4 ℃保持。poly-A RNA 对照准备:将稀释好的 poly-A RNA 与总 RNA 混合,poly-A RNA 作为整个操作流程的内部对照。一链 cDNA 合成:配制一链合成反应液,加入 poly-A RNA/总 RNA mix。使用“First-Strand cDNA Synthesis”程序孵育,合成 cDNA。二链 cDNA 合成:配制二链合成反应液,加入一链合成产物。使用“Second-Strand cDNA Synthesis”程序孵育,合成双链的 cDNA 模板。IVT 反应:配制 IVT(体外反转)反应液,加入二链合成产物。使用“IVT”程序孵育,反转获得带生物素标签的 aRNA。aRNA 纯化:用 GeneChip 3′IVT Express Kit 内的纯化试剂纯化 aRNA。纯化的 aRNA 用 nanodrop 2000 测定浓度。 aRNA 片段化:配制 aRNA 片段化反应液,加入已纯化的 aRNA。Hybridization 98 ℃,10 min→ 45 ℃,3 min→ 45 ℃保持。使用“Fragmentation”程序孵育,获得片段化产物。杂交: 配制杂交反应液。使用“Hybridization”程序加热杂交液。同时,向芯片注入 130 μL 预杂交液,45 ℃杂交炉预杂交 10 min。吸去芯片的预杂交液,注入 130 μL 杂交液。放于杂交炉中,45 ℃,60 r/min 杂交 16 min。杂交完成后取出芯片,用 GeneChip Fluidics Station 450 仪器进行自动洗染。洗染完成后进行扫描,获得数据。
1.5 统计学分析
使用SPSS 20.0软件进行统计。需要分析的2组通过F检验(方差同质性检验)进行检验。当F检验值大于0.05时,采用等方差双样本检验得到t检验值。当F检验值小于0.05时,采用异方差双样本检验得到t检验值。t检验值<0.05,说明两个实验组之间差异有统计学意义,t检验值>0.05,说明2组之间差异没有统计学意义。
2 结果
2.1 qPCR检测乳腺癌细胞中NUCB2的表达
qPCR结果显示与NC组比较,KD组NUCB2基因敲减效率达到90.6%,见图1和表2。
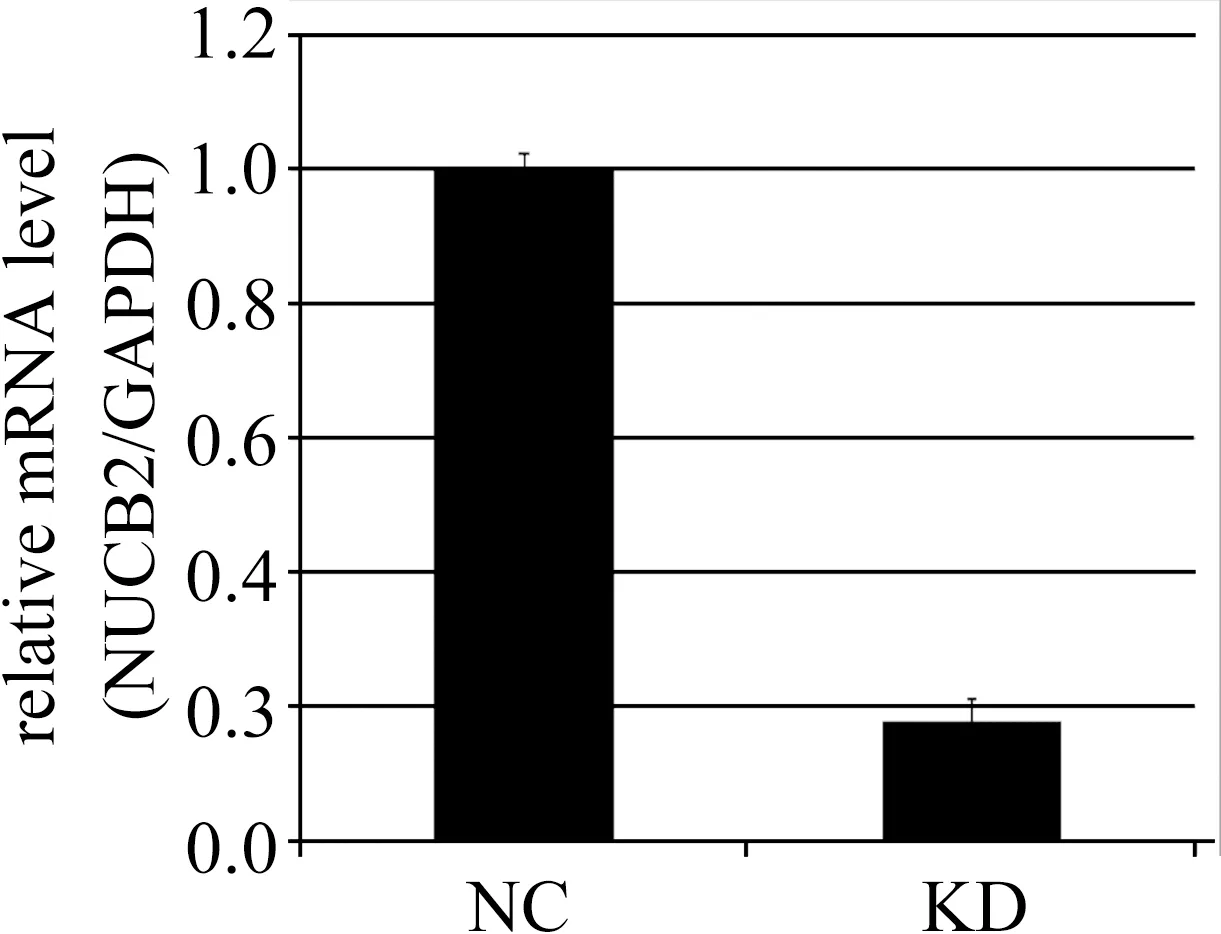
图1 qPCR显示siRNA敲除后 MDA-MB- 231细胞株中NUCB2的mRNA水平

表2 qPCR显示siRNA敲除后 MDA-MB- 231细胞株中NUCB2mRNA水平
2.2 Path-Array 芯片研究NUCB2介导的差异表达基因
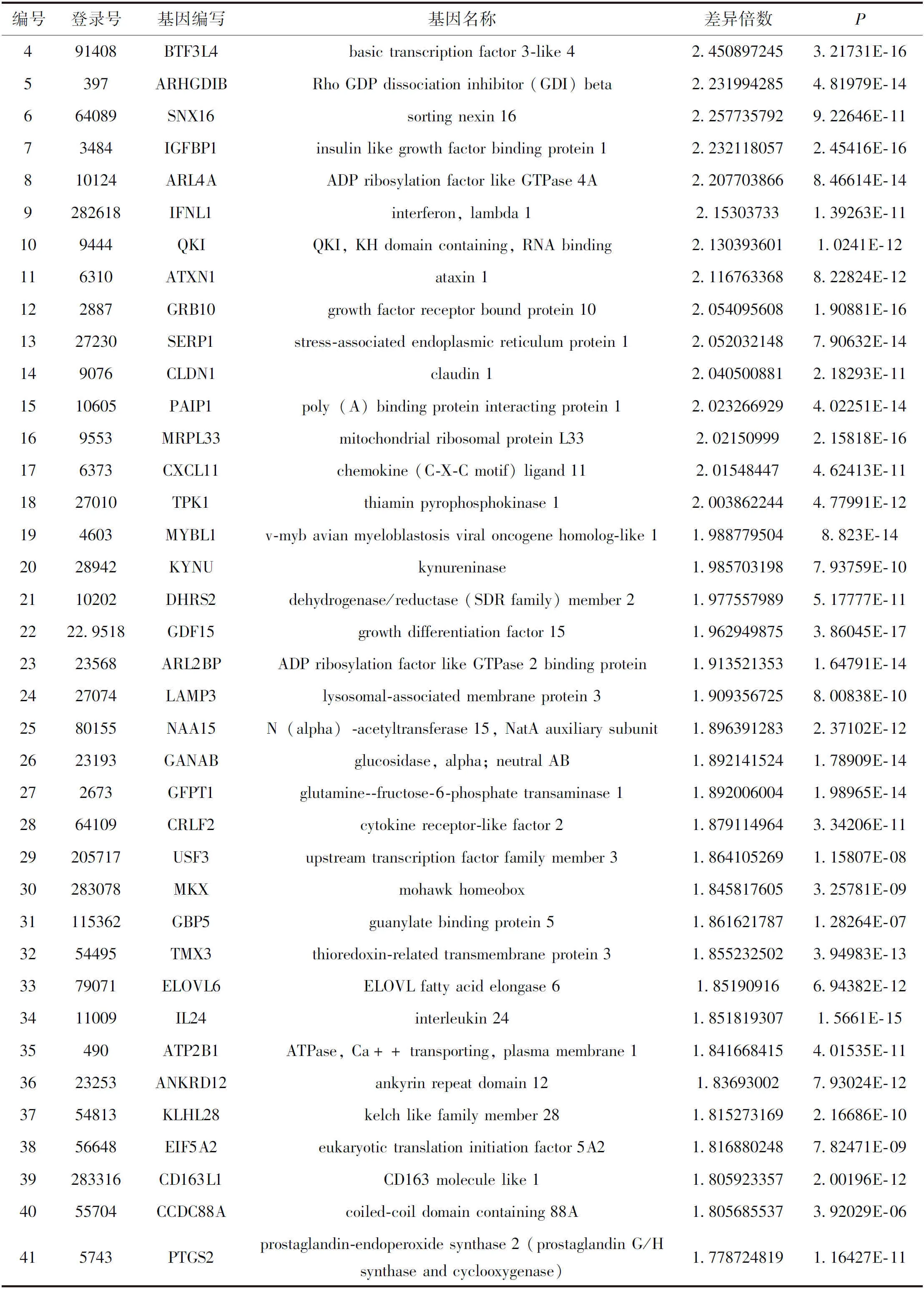
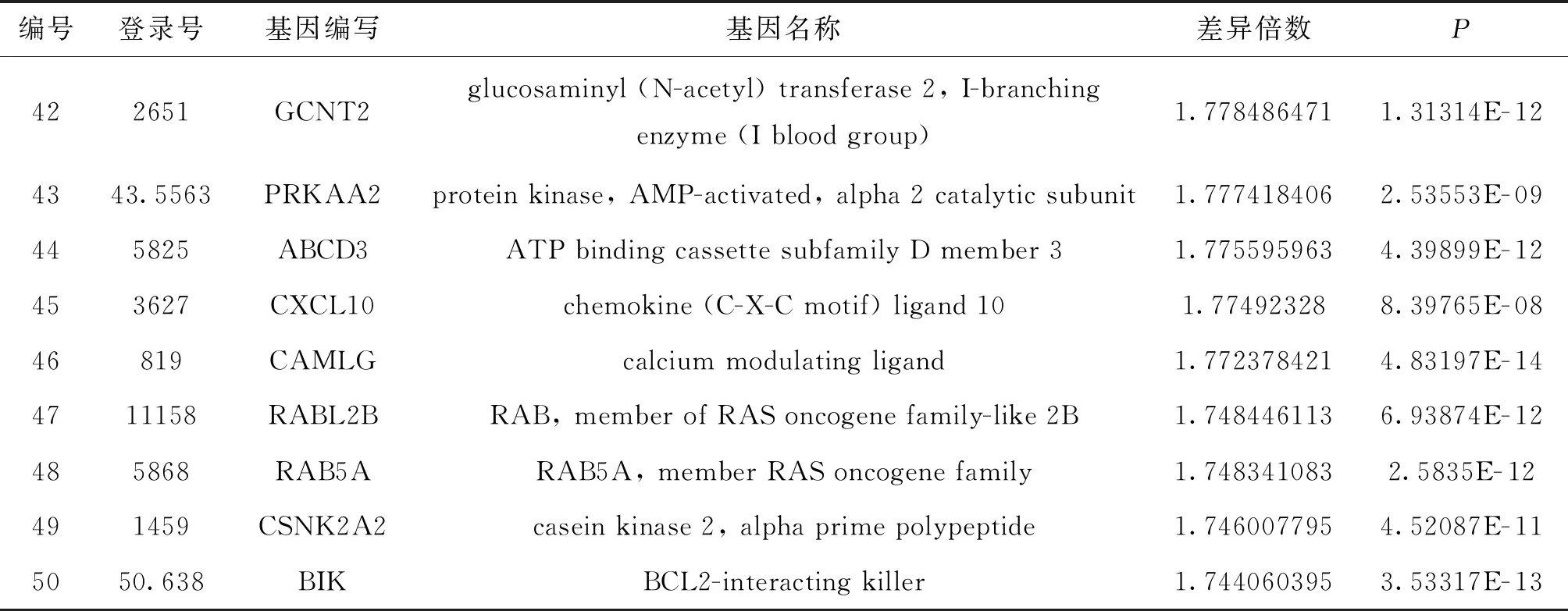
在本项目中,使用基于经验偏差分布的线性模型来计算显著性差异水平P值,并使用Benjamini-Hochberg方法来校正显著性差异水平(false discovery rate,FDR)。显著差异表达基因的筛选标准为|Fold Change|>1.5和FDR<0.05。与NC组比较,KD组上调基因186个,下调基因356个,部分差异表达基因见表3、表4。

表3 阻断nucb2后MDA-MB- 231细胞部分上调基因
续表
续表
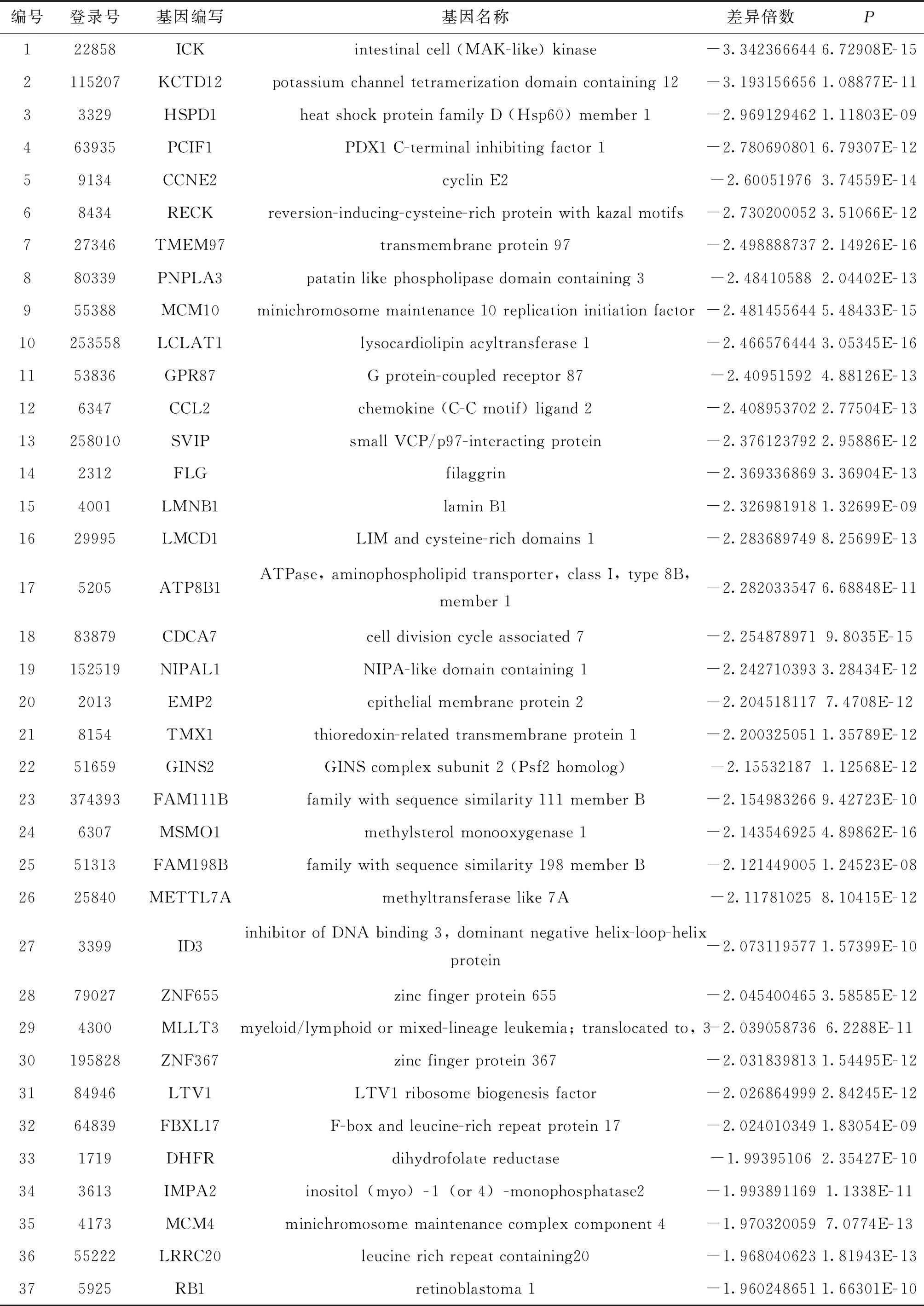
表4 阻断NUCB2表达后MDA-MB- 231细胞部分下调基因
2.3 用qPCR验证部分差异表达基因的表达情况
在MDA-MB- 231细胞株中,qPCR检测阻断NUCB2后部分上调基因,即与NC组比较KD组上调基因,包括即刻早期反应3相互作用蛋白1(immediate early response 3 interacting protein 1,IER3IP1)、基本转录因子3样4(basic transcription factor 3-like 4, BTF3L4)、Rho-GDP解离抑制因子β(Rho GDP dissociation inhibitor beta,ARHGDIB)、胰岛素样生长因子结合蛋白1(insulin like growth factor binding protein 1,IGFBP1),共济失调蛋白1(ataxin, ATXN1)。结果显示KD组IER3IP1、BTF3L4、ARHGDIB、IGFBP1、ATXN1的mRNA水平高于NC组,见图2A-E。
在MDA-MB- 231中,qPCR检测阻断NUCB2后下调基因,即与NC组比较KD组下调基因,包括肠细胞激酶(intestinal cell kinase,ICK)、钾通道四聚体化结构域12(potassium channel tetramerization domain containing 12, KCTD12)、热休克蛋白家族D成员1(heat shock protein family D member 1, HSPD1)、PDX1 C末端抑制因子1(PDX1 C-terminal inhibiting factor 1,PCIF1)、细胞周期蛋白E2(cyclin E2, CCNE2)。结果显示KD组的ICK、KCTD12、HSPD1、PCIF1、CCNE2的mRNA水平低于NC组,见图2F-J。
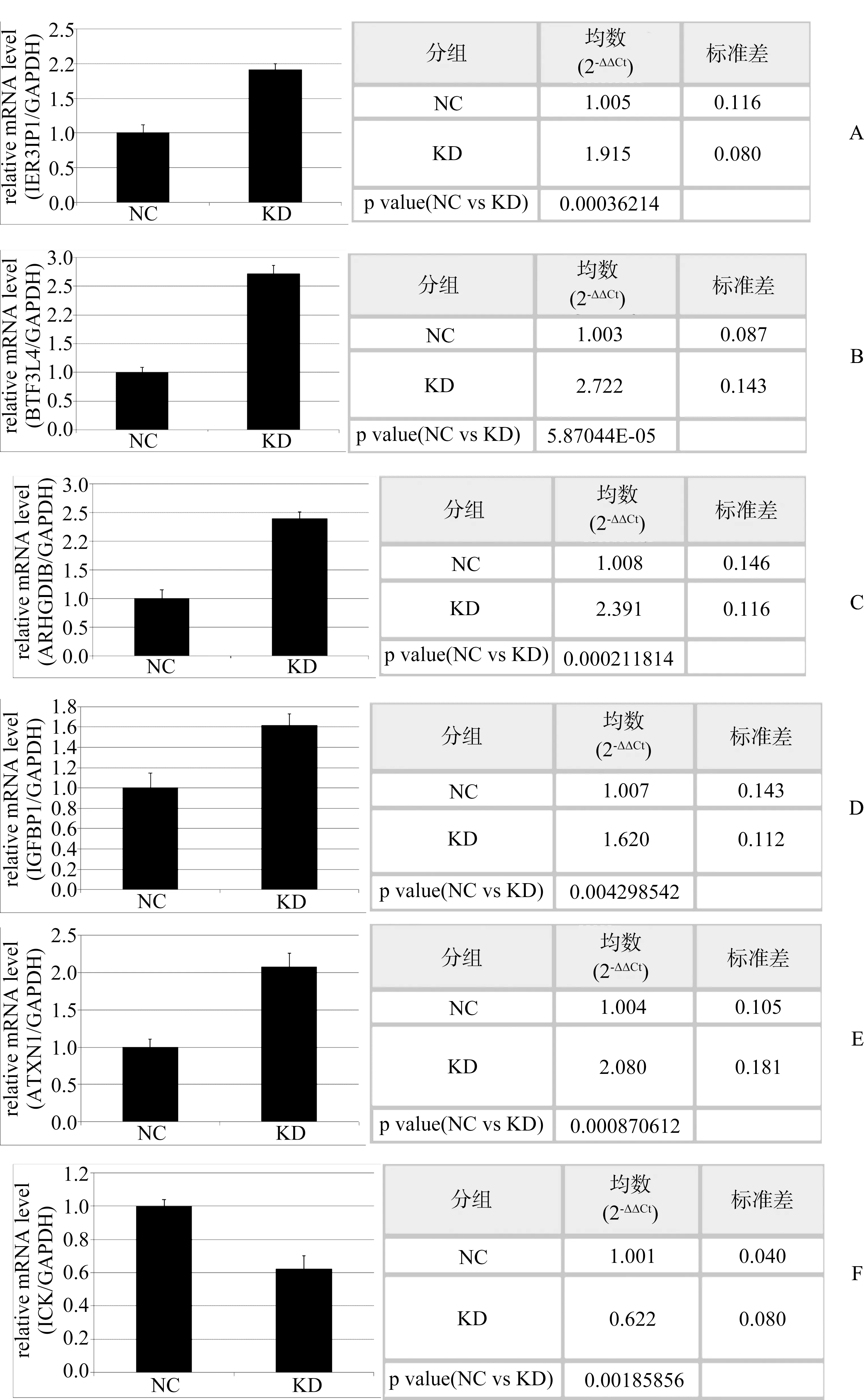
图3 qPCR对kd和nc部分差异表达基因的表达水平
2.4 NUCB2介导的差异表达基因的生物信息学分析
2.4.1 基于独创性通路分析(ingenuity pathway analysis,IPA)的经典通路分析 图4显示差异基因在经典通路中的显著性富集情况。在图4中,横坐标为通路名称,纵坐标为富集的显著性水平(以10为底的负对数变换);其中,橙色标注表示通路被激活(Z-score>0),蓝色标注表示通路被抑制(Z-score<0),橙色和蓝色的深浅(或Z-score的绝对值)代表激活或抑制的程度(依照IPA的内部算法和标准,认为Z-score>2代表该通路被显著激活,Z-score<- 2代表该通路被显著抑制);Ratio表示在此信号通路中差异基因数与该通路中包含的所有基因数的比值。在本项目中,胆固醇生物合成的超途径(Superpathway of Cholesterol Biosynthesis)被显著抑制,Z-score为- 3.162。
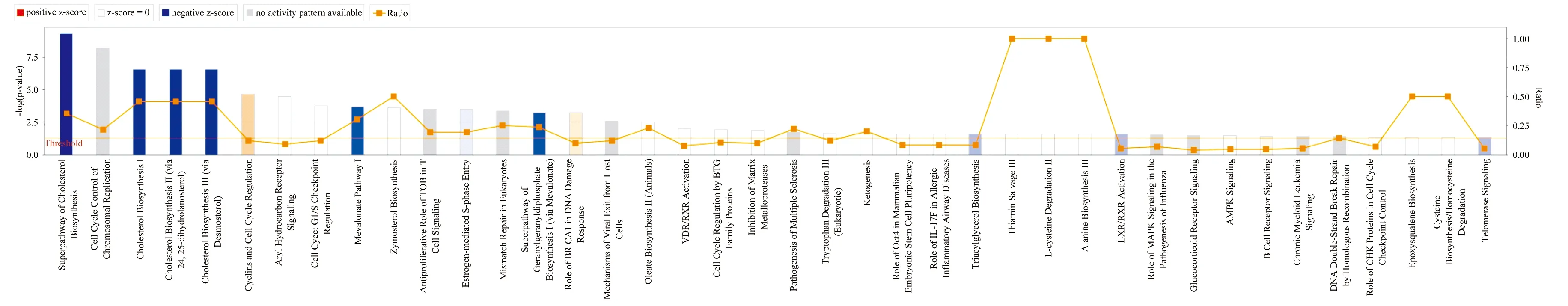
图4 通过PathArray进行的NUCB2介导的经典路径分析
2.4.2 基于IPA的上游控制分析 上游调控因子分析表展示了本项目中所有差异基因的上游调控因子。上游调控因子可以是任何能够影响基因表达的分子,它覆盖了全部的分子类型,包括转录因子,细胞活素,小RNA,受体,激酶,化学分子和药物。IPA使用了Activation Z-score算法,对上游调控子的激活或者抑制进行预测,并降低了由于随机数据造成的显著预测。在本项目中NUPR1被预测为强烈激活,该基因存在45个一致激活的基因,E2F1被预测为强烈抑制,该基因存在21个一致抑制的基因。
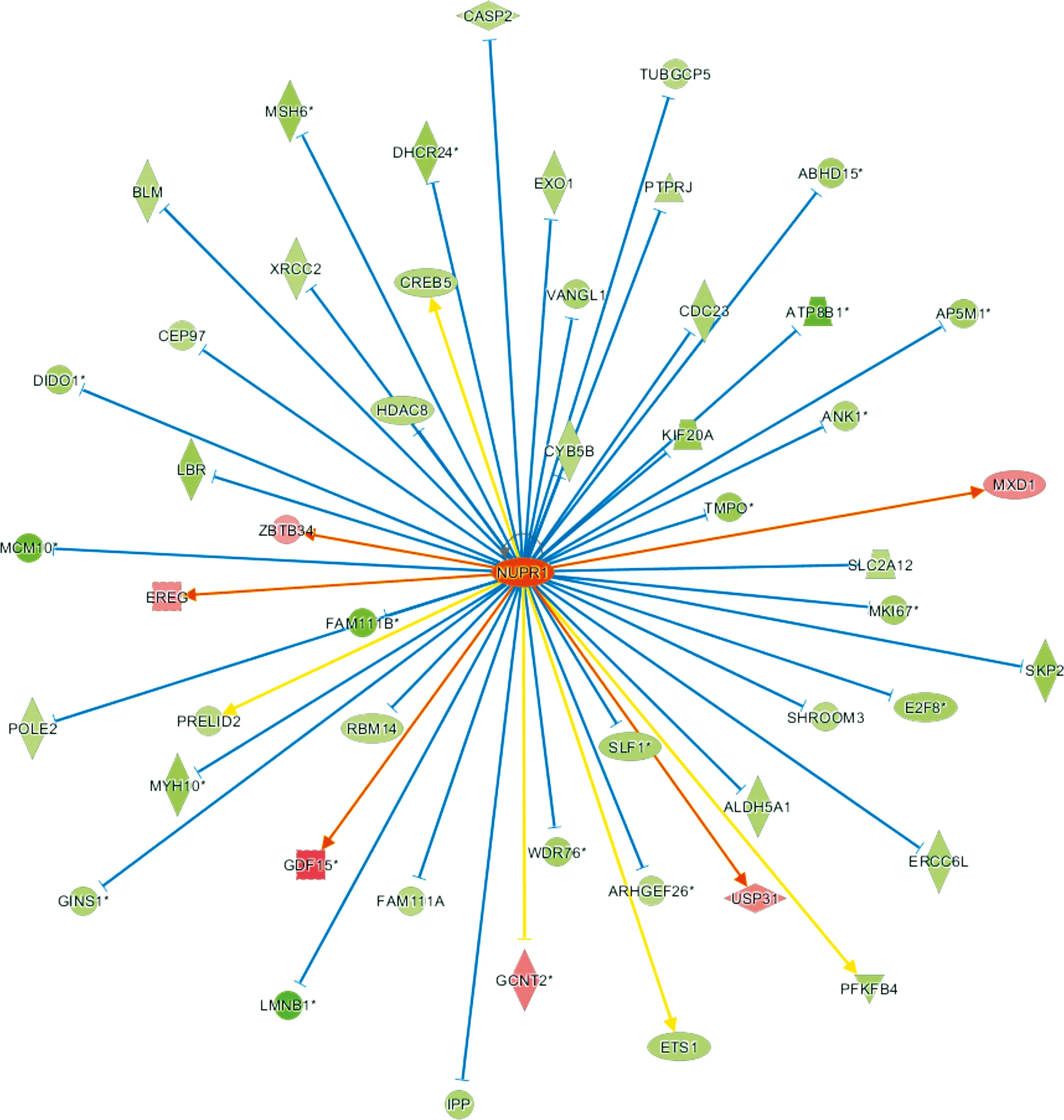
图5 NUCB2上游调控因子网络图
2.4.3 基于IPA的疾病和功能分析 图6显示差异基因在疾病与功能中的显著性富集情况。在图6中,横坐标为通路名称,纵坐标为富集的显著性水平(以10为底的负对数变换)。柱状图显示出明显的激活相关功能,包括金属释放、脂质积聚和明显的抑制相关功能如细胞死亡、肌肉萎缩。
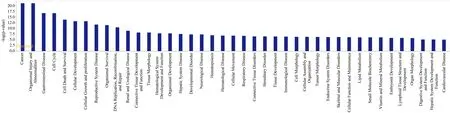
图6 NUCB2介导的疾病功能分析柱状图
图7显示差异基因表达水平的变化对疾病与功能的激活抑制关系。橙色代表疾病或功能状态被激活(Z-score>0),蓝色代表疾病或功能状态被抑制(Z-score<0),灰色表示疾病或功能状态未确定(Z-score无法计算)。依照IPA的内部算法和标准,认为Z-score>2代表该疾病或功能被显著激活,Z-score<- 2代表该疾病或功能被显著抑制。显著激活的疾病或功能包括:发病率或死亡率(Z-score=4.080),生物的死亡(3.921)等;显著抑制的疾病或功能包括:成纤维细胞株凋亡(- 2.481),葡萄糖代谢障碍(- 2.380)等。
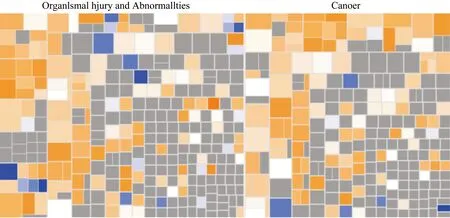
图7 NucB2介导的疾病和功能的热图
图8显示基因与疾病或功能之间的激活与抑制关系。网络中包含与指定的疾病或功能相关的所有差异基因,并展示了它们之间基于Ingenuity知识库的可能的互作关系和表达变化趋势。其中橙色线表示基因表达水平变化对该功能具有激活作用,蓝色线表示基因表达水平变化对该功能具有抑制作用,黄色线表示基因表达水平变化对该功能的影响与现有文献报道不一致,灰色线表示调控关系未知。
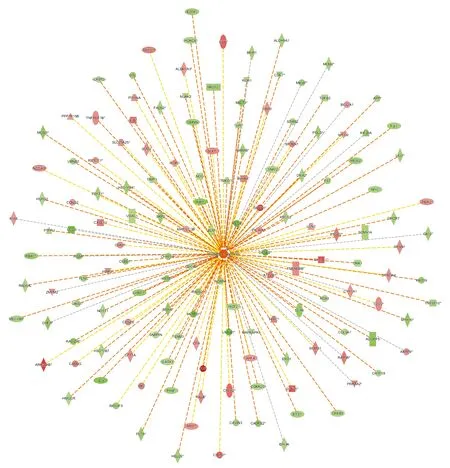
图8 基因疾病或功能网络图
2.4.4 基于IPA的调控效应分析 图9显示差异基因参与的上游调控网络与下游功能的可能的作用途径。Consistency Score是网络中上游调控因子、数据集和疾病与功能之间的因果关系一致性和密集连接的度量标准,Consistency Score越高,表示调控效应结果越准确。在本项目中DLL4等调控子通过CCNE2等基因对Benign lesion具有激活作用。

图9 调控效应分析表
图10展示了数据集中的基因与调控子和功能之间的相互作用关系。此调控效应分析中排名第一的调控网络展示了数据集可能由于调控子DLL4, HDL-cholesterol, mir- 8通过CCNE2, DHCR7, EGR1, ETS1, FDFT1, FHOD1, GATA3, MCM3, MCM7, MYH10, NR2F2, SKP2, TGFB2等基因对良性病变(benign lesion),先心病(congenital heart disease),生长阻滞(growth failure),围产期死亡(perinatal death)具有激活作用,对细胞运动(cell movement), 体质量(size of body)具有抑制作用。
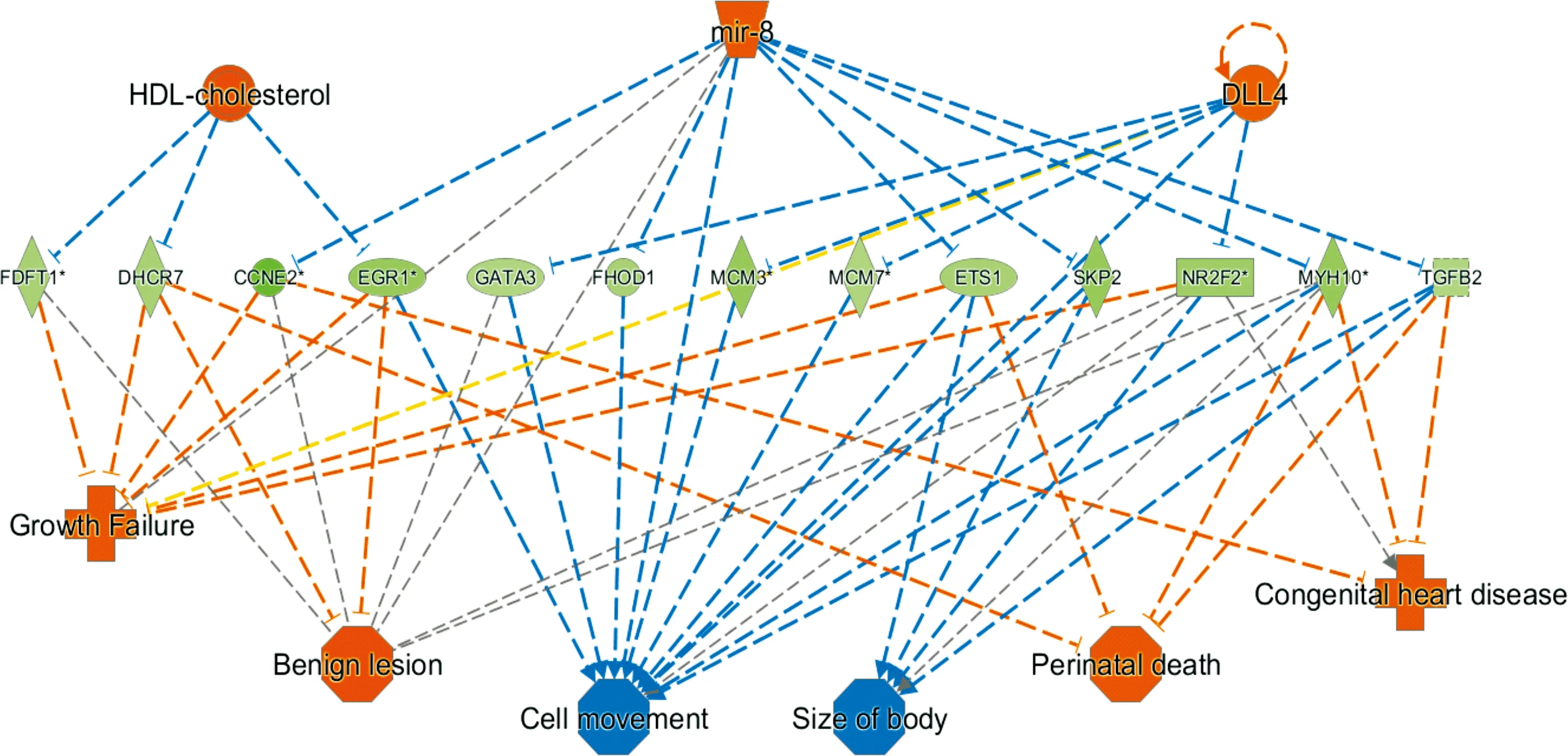
图10 调控效应网络图
3 讨 论
本研究在以往对乳腺癌中NUCB2作用的蛋白质组学和临床病理学研究基础上,进一步利用Path-Array芯片筛选了NUCB2介导的下游和信号通路。乳腺癌细胞中阻断NUCB2后,发现共有186个上调基因和356个下调基因,其中一些差异表达基因经过mRNA检测进行验证,验证结果和Path-Array结果的高度一致。
应用IPA分析不同表达基因。首先,经典的通路分析表明,阻断NUCB2可抑制一系列通路包括胆固醇生物合成超级通路、胆固醇生物合成通路I、胆固醇生物合成通路II、胆固醇生物合成通路III、ERK/MAPK信号,其中胆固醇生物合成超级通路受到最显著的抑制。据报道 Nesfatin- 1通过其受体诱导cAMP反应元件结合蛋白CREB的磷酸化,从而激活神经元细胞内信号级联反应的[4]。NUCB2调控EGF刺激的MAPK激酶/ERK信号传导、细胞增殖和脂肪细胞分化,NUCB2敲除细胞的细胞增殖显著降低[5]。
但是,NUCB2与胆固醇生物合成关系的研究还没有直接报道。其次,上游调控因子分析表明,上游调控因子可以是影响基因表达的任何分子,它涵盖了所有的分子类型,包括转录因子、细胞因子、小RNA、受体、激酶、化学分子和药物。本研究发现调控NUCB2的分子中,NUPR1被预测为强烈激活,E2F1被预测为强烈抑制,该基因有21个一致的基因。进一步的上游调控子网络图显示了与上游调控因子直接相关的数据集中上游调控因子与下游分子共存的相互作用。例如,NUPR1可以提高Ets1的mRNA水平,但在实验数据中Ets1明显下调。NUPR1和IPP的表达趋势一致。此外,IPA的疾病和功能柱状图显示出明显的激活相关功能,包括金属释放、脂质积聚和明显的抑制相关功能如细胞死亡、肌肉萎缩。
这些生物信息学分析结果表明,一方面是参与脂质合成和代谢的途径和分子可能在NUCB2功能中发挥重要作用,值得进一步研究。另一方面是释放与NUCB2功能相关的金属,NUCB2与G蛋白偶联受体相互作用,导致钙离子增加,这与培养大鼠神经元中蛋白激酶C的激活有关[6]。NUCB2通过N型通道刺激钙离子内流激活迷走神经传入神经元,证实NUCB2可将信号传到脑组织[7]。NUCB2也参与高尔基体内钙离子储存,以及涉及DNA结合和蛋白质相互作用的生物学过程。在没有钙离子的情况下,NUCB的钙离子结合域处于无结构状态,而加上Ca2+则会折叠[8,4]。据报道,NUCB2可抑制核因子kappa-B依赖性炎症反应,减轻大鼠创伤性脑损伤后caspase- 3介导的神经元细胞凋亡[9]。NUCB2通过参与Bax、Bcl-Xl和Bcl- 2基因以及ERK1/2, p38 和JNK1/2 信号级联,抑制人肾上腺皮质细胞模型(H295R)细胞的生长并促进凋亡。这提示NUCB2在肾上腺皮质区发育中发挥作用,也可能是肾上腺癌的治疗靶点[10,12]。据报道,NUCB2通过mTOR和RhoA/ROCK信号通路诱导凋亡,抑制人卵巢上皮癌HO- 8910细胞系的增殖[3]。NUCB2调控EGF刺激的MAPK激酶/ERK信号传导、细胞增殖和脂肪细胞分化[5]。
关于NUCB2在疾病特别是肿瘤发生和进展中信号通路的研究报道较少,对于本研究发现的一些NUCB2相关信号通路,进一步的研究重点是阐明NUCB2功能与信号转导途径的关系。

