长链非编码RNA LNC01309对人肝癌细胞增殖和迁移能力的影响及作用机制
刘红燕,李婧姣,路泽军,刘咏真,温居一,
1 安徽医科大学 海军临床学院,合肥 230032;2 解放军总医院第六医学中心 肿瘤内科,北京 100048
肝细胞癌(以下简称肝癌)是全球发病率和病死率均较高的恶性肿瘤。近年来肝癌的诊断和治疗取得了较大进展,但晚期肝癌患者预后仍较差。目前肝癌的发病机制尚未完全阐明,深入分析肝癌的发生、发展机制,将有助于发现新的肝癌诊断标志物和治疗靶点。长链非编码RNA(long non-coding RNA,lncRNA)在肿瘤形成过程中的表达机制颇受关注,已被公认参与多种肿瘤的发生、发展及预后,与肝癌亦关系密切[1-2]。
在人类基因组中,参与编码蛋白质的基因仅占2%,超过98%基因组不编码蛋白质,其基因转录产物被称为非编码RNA(non-coding RNA,ncRNA)[3]。根据其长度,可将ncRNA分为miRNA、snoRNA、piRNA和lncRNA等[4]。ncRNA中大多数为lncRNA,lncRNA的基因长度往往>200 bp,其开放阅读框一般短于50~100 bp。根据lncRNA在基因组中与编码蛋白质基因的相对位置,可将其分成5类,即反义lncRNA、正义lncRNA、双向lncRNA、基因内lncRNA和基因间lncRNA[5]。随着二代测序技术的快速发展,成千上万的lncRNA在脊椎动物和非脊椎动物体内被快速鉴别出来。但是,二代测序技术在实际应用中也存在很多缺陷,如文库制备过程中PCR扩增很大程度会使后续结果出现误差,而且尽管已经开发出各种算法来提高测序结果的质量,但其对于组装一个横跨整个基因组的长片段仍然是一项挑战。lncRNA通常长度较长且存在多种剪接形式,单纯的RNA-Seq并不能分辨各种异构体,因此第三代测序技术的诞生就显得尤为重要[6]。
PacBio作为最新一代的测序技术,是由美国Pacific Bioscience开发的单分子实时测序(molecule real time sequencing,SMRT)技术,测序过程无需PCR扩增。其在读取长度方面远超于二代测序技术,并且无GC偏好性,能够大大提升基因组组装的质量[7]。PacBio在研究转录组学方面最大的优势在于它能够获得全长或接近全长的转录本,这对可变剪切的鉴定、lncRNA的分析[8-9]以及新基因的发现等研究有着重要意义;且能在原有研究基础上进行补充甚至发现新的机制。
本研究主要利用PacBio三代测序技术在肝癌组织中鉴定新的lncRNA,并在肝癌细胞中进行功能机制的初步探索。
1 材料与方法
1.1 研究材料 收集2018年2月—2019年6月解放军总医院第六医学中心12例肝癌患者的肿瘤组织和对应的癌旁组织。人肝癌细胞系(Hep G2、SNU-398和Hep 3B)购自北京协和医学院基础学院基础医学细胞中心;人永生化正常肝细胞(THLE-2)购自上海泽叶生物科技有限公司;RPMI-1640及胎牛血清购自美国Thermo Fisher Scientific公司;Smarter PCR cDNA合成试剂盒购自美国Clontech公司;SYBR Green染料购自日本Takara公司;所有引物购自生工生物工程(上海)股份有限公司;CNOT4、EIF4G2、HNRNPA1、MBNL1、RBM24、RBM6、SRSF10抗体购自美国Santa Cruz Biotechnology公司;RBM38、N-cadherin、Vimentin、E-cadherin和GAPDH抗体及辣根过氧化物酶标记的二抗均购自英国Abcam公司;CCK-8试剂盒购自日本同仁化学公司;Transwell小室购自康宁公司;RNA荧光原位杂交(RNA fluorescence in situ hybridization,RNA FISH)和EdU检测试剂盒购自广州锐博生物科技有限公司;靶向lncRNA MIR205HG的siRNA由上海吉玛制药技术有限公司设计与合成;pCMV-RBM38购自北京义翘神州生物技术有限公司;pEXP-LNC01309野生型和突变型表达载体由广州锐博生物科技有限公司构建完成。
1.2 细胞培养与细胞转染 按照培养基和血清配比为9∶1配置完全培养基;所有细胞均采用RPMI-1640完全培养基培养;5% CO2,37 ℃恒温培养箱为细胞培养环境。细胞转染采用Lipofectamine 3000试剂。在6孔板中,接种细胞浓度为1×106/孔,培养基体积2 mL,其中siRNA用量为20 nmol/L/孔,质粒用量为8 μg/孔,共孵育24 h后,进行后续试验。
1.3 RNA提取 采用Trizol提取组织或细胞中的RNA。按100 mg组织加1 mL Trizol研磨,制备组织悬液;5×106细胞加入1 mL Trizol吹打,收获细胞悬液;且冰上裂解5~10 min后,按200 μL氯仿﹕1 mL Trizol的比例加入氯仿,混匀液体,静置15 min。然后4 ℃ 12 000 r/min离心分离RNA,吸取上清液至新的EP管中并弃去沉淀。加入等体积异丙醇,4 ℃静置10 min,12 000 r/min离心10 min,弃去上清液。加入75%酒精充分洗涤沉淀,离心去除上清液。最后将RNA样品沉淀置于超净台干燥5 min,加入DEPC水溶解,Nanodrop测量总RNA浓度,并置于-80 ℃保存。
1.4 PacBio三代测序 利用Trizol提取组织RNA。根据产品说明书,共使用4 μg混合RNA与Pacbio Sequel系统(Pacific Biosciences,CA,USA)进行全长转录序列测序。使用Clontech Smarter PCR cDNA合成试剂盒和Pacific Biosciences的luePippin Size Selection系统(PN100-092-800-03),根据异构体测序(Iso-Seq)协议制备Iso-Seq文库。后续测序与分析由北京基石生命科技有限公司完成。
1.5 荧光定量PCR 按照试剂盒说明书使用寡(dT)引物在总体积为20 μL的条件下进行cDNA合成;采用染料法,使用最终体积为25 μL的500 ng cDNA模板进行荧光定量PCR。重复3次试验,数据采用2-ΔΔCt方法处理,以GAPDH为内参。引物序列如下(5′-3′),lncRNA MIR205HG:正向引物AGCAGCAGCAGCAAGAGTAA,反向引物GAAAGATTAAGGTTCCCATC;LNC01309:正向引物AGCAGCAGCAGCAAGAGTAACT,反向引物GAAAGATTAAGGTTCCCATC;RBM38:正向引物GCTACGGCTTCGTGACCAT,反向引物GTAAGTCCGCTGGATCAAGGT;GAPDH:正向引物AGAAGGCTGGGGCTCATTTG,反向引物AGGGGCCATCCACAGTCTTC。
1.6 Western Blot 冰上裂解细胞并提取细胞中的总蛋白,BCA法检测总蛋白浓度,向50 μg蛋白样品中加入等体积的2×Laemmli上样缓冲液,总上样体积为20 μL,进行SDS-PAGE电泳。接着进行恒流(200 mA)转膜,脱脂奶粉封闭,4 ℃孵一抗过夜。次日进行二抗常温孵育,二抗孵育完成后加入化学发光液与蛋白条带反应,凝胶成像仪观察结果。
1.7 细胞增殖检测 使用CCK-8和EdU检测细胞增殖。CCK-8检测:按照1×104/孔细胞数接种至96孔细胞培养板,待细胞贴壁生长覆盖率达到50%左右,Lipofectamine 3000转染细胞并持续培养4 d。分别于不同时间点(0、24、48、72、96 h)弃去培养基,加入无血清培养基配置的10% CCK-8反应液,37 ℃恒温培养箱培养40 min,酶标仪检测波长在450 nm处的吸光度值。EdU检测:细胞接种于96孔板中,经转染处理24 h。然后将50 μmol/L EdU加入培养孔,37 ℃孵育2 h后,用4%多聚甲醛固定细胞。按照标准程序,anti-EdU工作液和DAPI染色孵育细胞。于荧光显微镜(奥林巴斯IX71)下观察,随机选取不同视野记录试验结果。
1.8 细胞划痕愈合试验 将各组Hep G2细胞按1×106/孔接种于6孔板中,置于37 ℃,5% CO2培养箱培养过夜。第2天用10 μL枪头划两条平行线,形成划痕。使用PBS轻轻漂洗2~3遍,加人无血清培养基。放入37 ℃,5% CO2培养箱继续培养,分别于0、12 h观察,拍照。用Image J分析迁移面积并计算迁移率。
1.9 Transwell迁移试验 将转染后24 h的各组细胞按照5×104细胞数分别接种于Transwell的上室,下室中加入800 μL含20%胎牛血清的培养基。继续培养24 h,取出小室,采用PBS清洗3次,甲醇固定15 min,用DAPI染色后在荧光显微镜下观察并随机选取5个以上视野拍照计数。
1.10 RNA FISH 使用预冷的Triton X-100(0.1%)使细胞膜通透,并用多聚甲醛(4%)处理载玻片。然后用LNC01309探针杂交过夜。SCC缓冲液清洗细胞。采用DAPI染细胞核后,荧光显微镜观察结果。
1.11 蛋白质的稳定性测定 用终浓度100 μg/mL的放线菌酮处理细胞,并在指定时间点(0、2、4 h)收取细胞。从细胞中提取蛋白质并进行蛋白质印迹分析。β-肌动蛋白用作参照。
2 结果
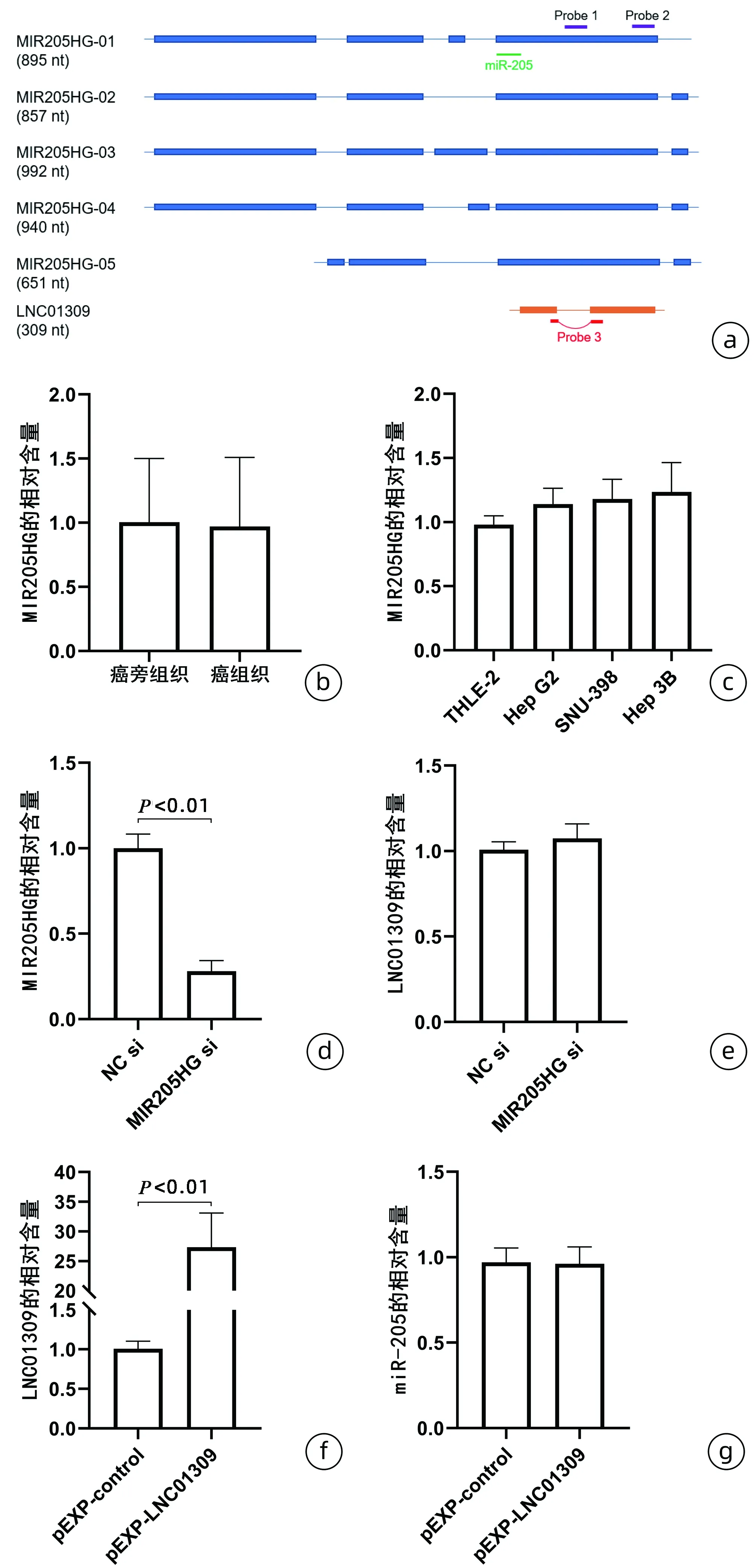
2.1 lncRNA LNC01309在肝癌组织和细胞中的表达 在肝癌和对应的癌旁组织中利用PacBio测序分析发现1条高丰度且未见报道的lncRNA(图1a),长度为309 nt,暂命名为LNC01309。利用荧光定量PCR检测分析,结果显示肝癌组织和对应的癌旁组织中LNC01309水平分别为4.225±2.285与1.541±0.530,差异有统计学意义(t=3.618,P=0.004)(图1b)。将永生化正常肝细胞(THLE-2)和多种肝癌细胞(Hep G2、SNU-398和Hep 3B)中的LNC01309表达情况进行比较,结果显示,LNC01309在肝癌细胞中的相对含量分别为1.860±0.126、2.313±0.383和3.917±0.393,均明显高于THLE-2细胞(1.011±0.074)(t值分别为4.231、6.489、14.480,P值分别为0.004、<0.001、<0.001)(图1c)。

注:a,PacBio测序分析获得的LNC01309序列;b,利用荧光定量PCR分析LNC01309在肝癌组织与癌旁组织中的表达差异;c,利用荧光定量PCR分析LNC01309在肝癌细胞和正常肝细胞中的表达差异。
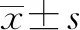
2.2 LNC01309与lncRNA MIR205HG的关系分析 利用NCBI BLAST进行序列比对分析,LNC01309定位于1号染色体(209432225-209432545),与lncRNA MIR205HG存在较高的序列相似性(表1)。lncRNA MIR205HG亦定位于1号染色体(209428820-209432549),其目前存在5种剪接体,通过序列比对分析,笔者设计了特异的PCR引物用于区分检测lncRNA MIR205HG和LNC01309(图2a)。结果显示,lncRNA MIR205HG无论是在肝癌组织还是在肝癌细胞中均不存在异常表达现象(图2b、c)。随后,在Hep 3B肝癌细胞中利用siRNA敲降lncRNA MIR205HG,分为NC siRNA对照组(NC si)和MIR205HG siRNA处理组(MIR205HG si)。荧光定量PCR结果显示,随着lncRNA MIR205HG的下调(1.001±0.082 vs 0.281±0.062,t=15.690,P<0.001)(图2d),LNC01309并未明显下降(1.008±0.046 vs 1.075±0.085,t=1.534,P=0.164)(图2e)。值得注意的是,LNC01309本身序列中并不含有miR-205序列,因此在肝癌细胞中过表达LNC01309,分为质粒对照组(pEXP-control)和过表达组(pEXP-LNC01309)(1.006±0.099 vs 27.350±5.744,t=9.172,P<0.001)并没有导致miR-205相对含量的变化(0.971±0.083 vs 0.962±0.100,t=0.142,P=0.892)(图2f、g)。
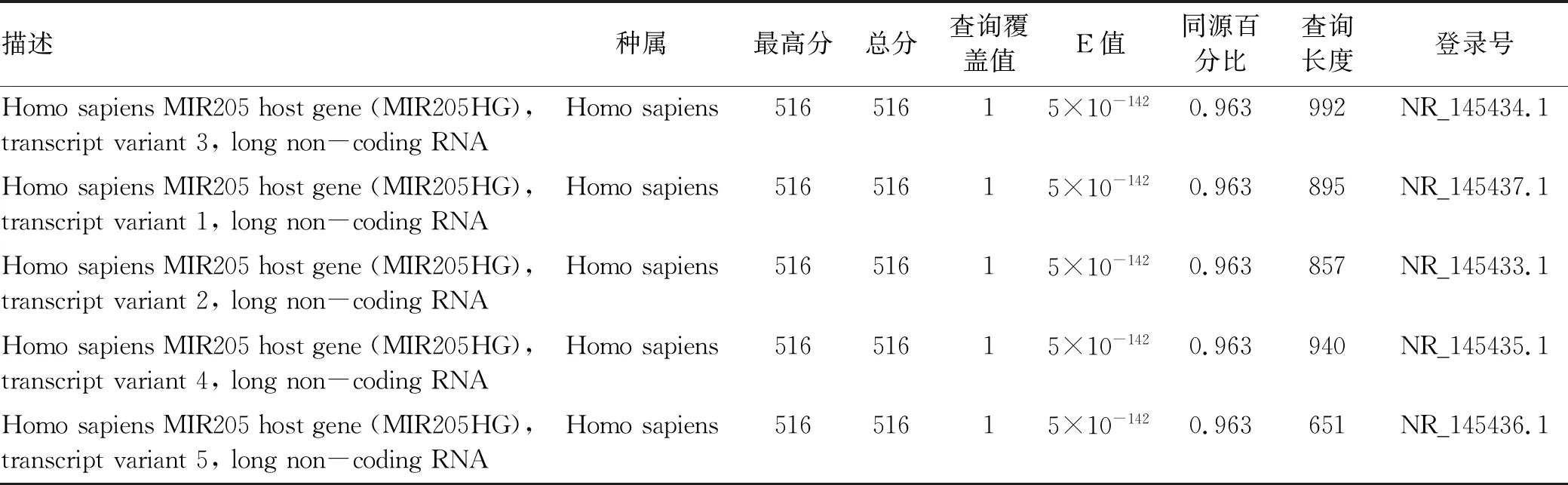
表1 利用NCBI BLAST比较LNC01309与lncRNA MIR205HG序列相似性
2.3 LNC01309促进肝癌细胞增殖与迁移 笔者尝试在含有较低丰度LNC01309的Hep G2细胞中过表达LNC01309(1.006±0.099 vs 27.350±5.744,t=9.172,P<0.001)。RNA FISH试验结果与在线工具Locate-R(http://locate-r.azurewebsites.net/)预测结果相一致,LNC01309主要定位于细胞质中(图3、4)。随着LNC01309水平的升高,肝癌细胞的增殖能力(第96小时OD450值:1.885±0.107 vs 2.527±0.234,t=4.330,P=0.012)显著上调(图5、6)。同时,划痕愈合试验(11.65%±2.40% vs 35.66%±4.90%,t=9.837,P<0.001)(图7)和Transwell试验结果(100.00%±3.11% vs 161.00%±35.93%,t=4.399,P=0.005)(图8)均显示肝癌细胞迁移能力增强。Western Blot结果显示,过表达LNC01309会促进上皮间质转化(EMT)的标志蛋白N-cadherin(0.466±0.067 vs 1.253±0.046,t=5.322,P=0.009)和Vimentin(0.372±0.126 vs 2.638±0.413,t=7.292,P=0.018)的表达,同时下调E-cadherin的表达(0.909±0.032 vs 0.151±0.014,t=50.450,P<0.001)(图9)。
注:a,LNC01309与lncRNA MIR205HG序列相似性,并根据序列差异性设计特异性检测引物;b,利用荧光定量PCR分析lncRNA MIR205HG在肝癌组织与癌旁组织的表达差异;c,利用荧光定量PCR分析lncRNA MIR205HG在肝癌细胞和正常肝细胞中的表达差异;d、e,Hep 3B细胞经转染siRNA(20 nmol/L)处理24 h后,利用荧光定量PCR分别检测lncRNA MIR205HG和LNC01309的相对表达含量;f、g,Hep G2细胞中转染过表达质粒24 h后,利用荧光定量PCR分别检测LNC01309和miR-205的相对表达含量。

图3 在线工具Locate-R预测LNC01309的细胞定位
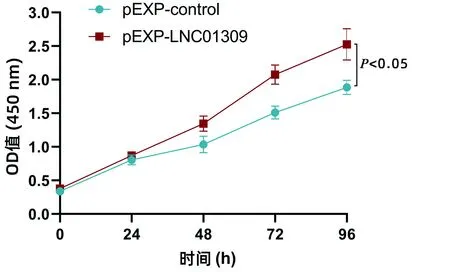
注:Hep G2细胞中转染LNC01309表达质粒以及对照质粒(24孔板中使用量为1 μg/孔)处理,于不同时间点利用CCK-8检测细胞活性。
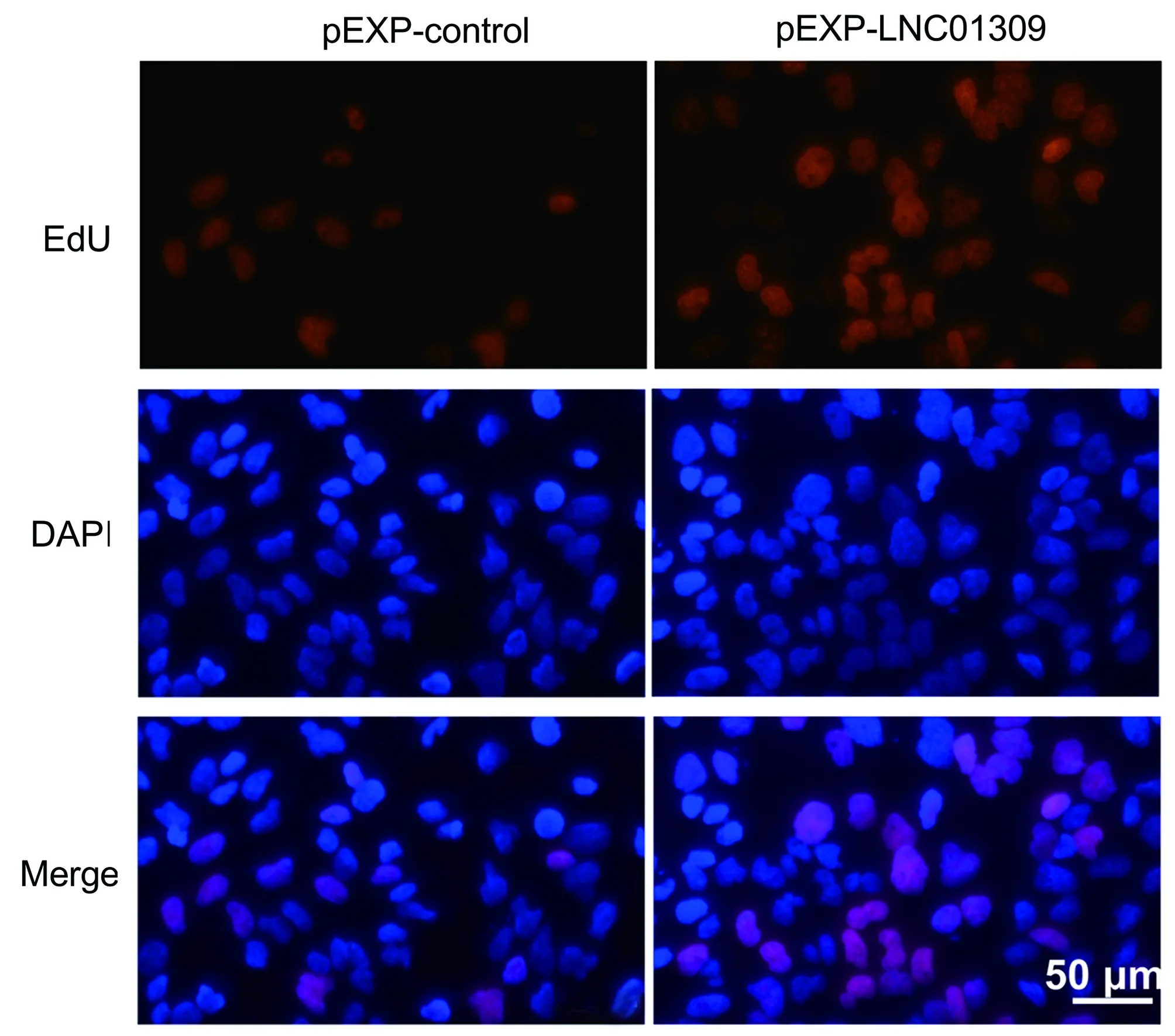
图6 EdU染色试验检测转染LNC01309表达质粒的Hep G2细胞增殖
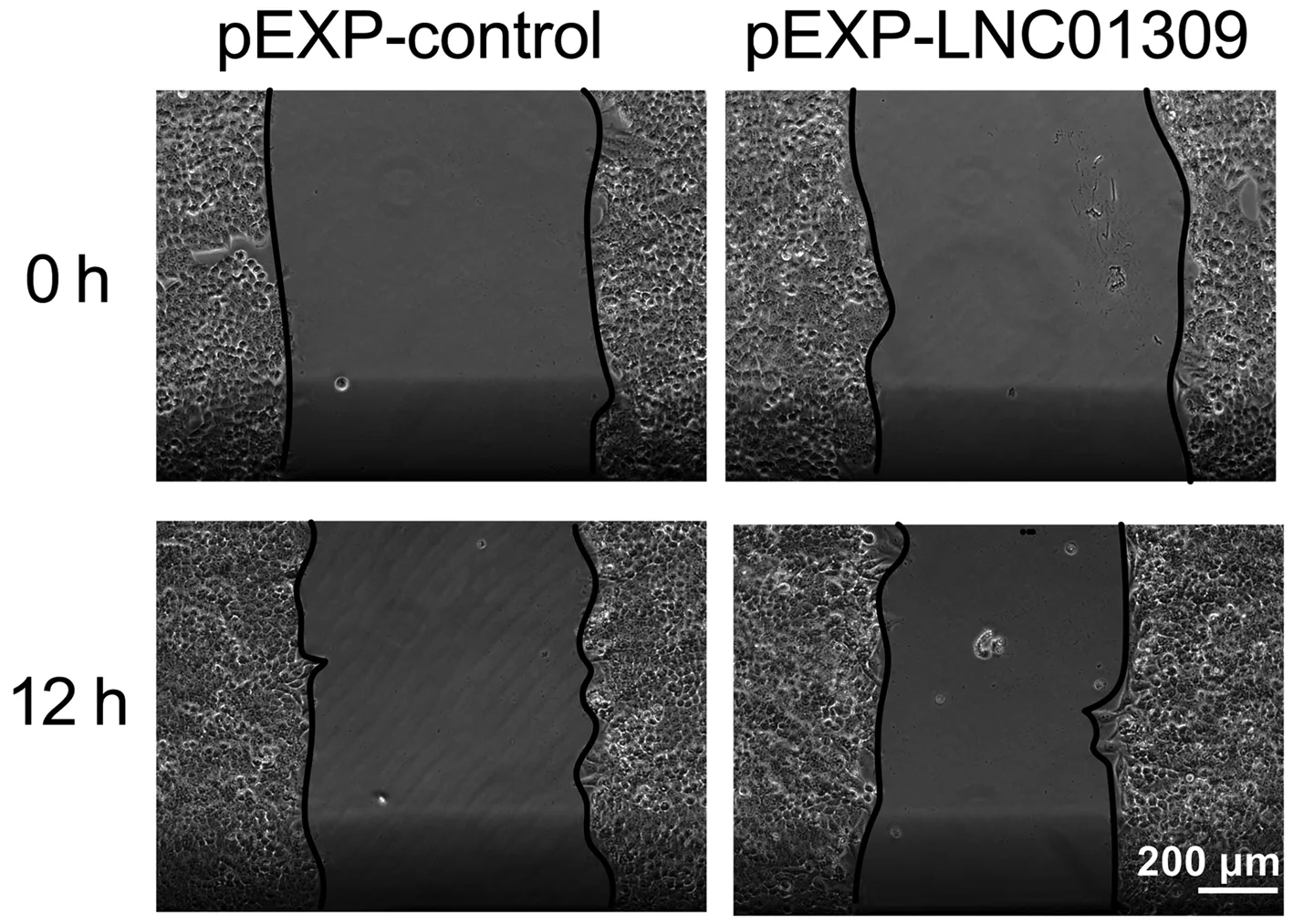
图7 细胞划痕试验检测转染LNC01309表达质粒的Hep G2细胞的迁移能力
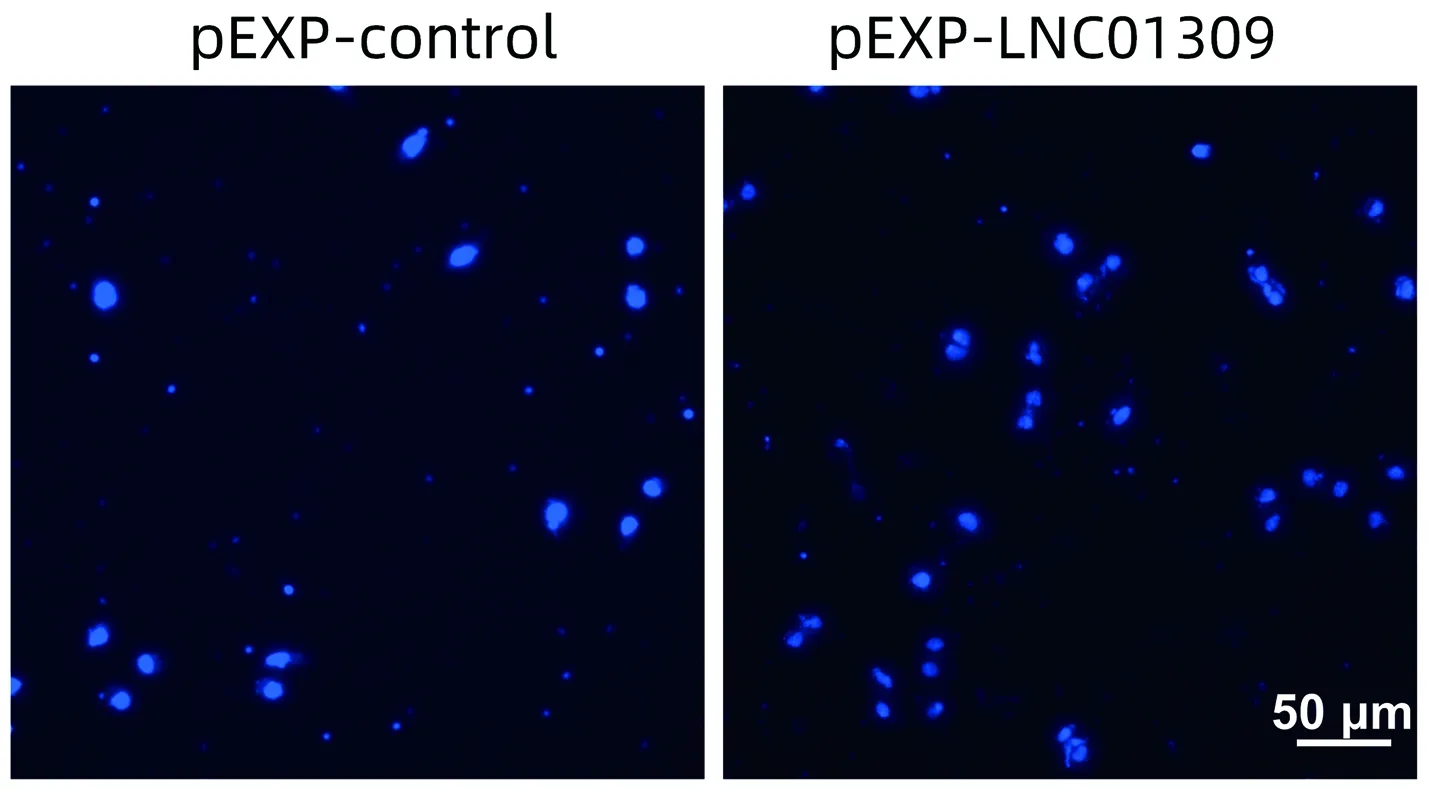
图8 Transwell检测转染LNC01309表达质粒的Hep G2细胞的迁移能力
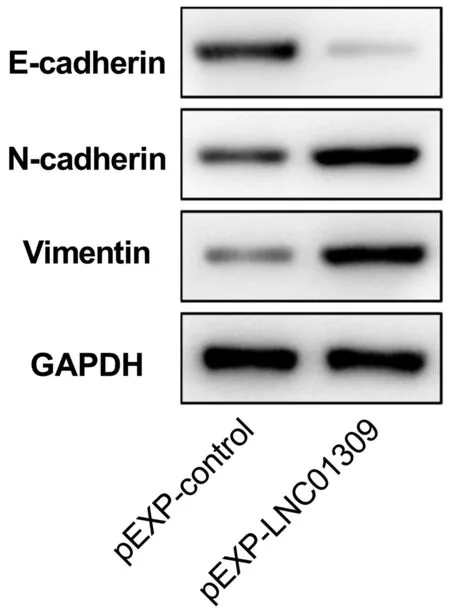
图9 Western Blot检测各组细胞中EMT标志蛋白的相对含量
2.4 LNC01309与RNA结合蛋白的相互作用 利用在线工具RBPmap(http://rbpmap.technion.ac.il/index.html)和catRAPID(http://service.tartaglialab.com/page/catrapid_group)分析LNC01309的潜在结合蛋白,获得了CNOT4、EIF4G2、HNRNPA1、MBNL1、RBM24、RBM38、RBM6和SRSF10共8个排名靠前的候选蛋白(表2)。随后,在Hep G2细胞中过表达LNC01309,利用候选蛋白的特异抗体进行RNA免疫共沉淀试验,富集结果分别为0.087±0.044、0.120±0.107、0.192±0.105、0.164±0.093、0.153±0.108、0.502±0.213、0.124±0.085和0.168±0.068;与IgG对照组比较,仅RBM38抗体组差异有统计学意义(0.502±0.213 vs 0.060±0.029,t=3.846,P=0.031)(图10a)。将RBM38潜在结合位点(gugugcg)

表2 利用RBPmap和catRAPID在线预测能够与LNC01309结合的潜在靶标蛋白
缺失突变后构建突变型LNC01309。RNA免疫共沉淀实验结果显示,与IgG对照组比较,RBM38抗体组沉淀富集的野生型LNC01309相对含量显著升高(0.434±0.122 vs 0.034±0.017,t=5.123,P=0.036),而突变型LNC01309在两组中的沉淀富集丰度差异无统计学意义(0.077±0.051 vs 0.031±0.006,P=0.201)(图10b)。
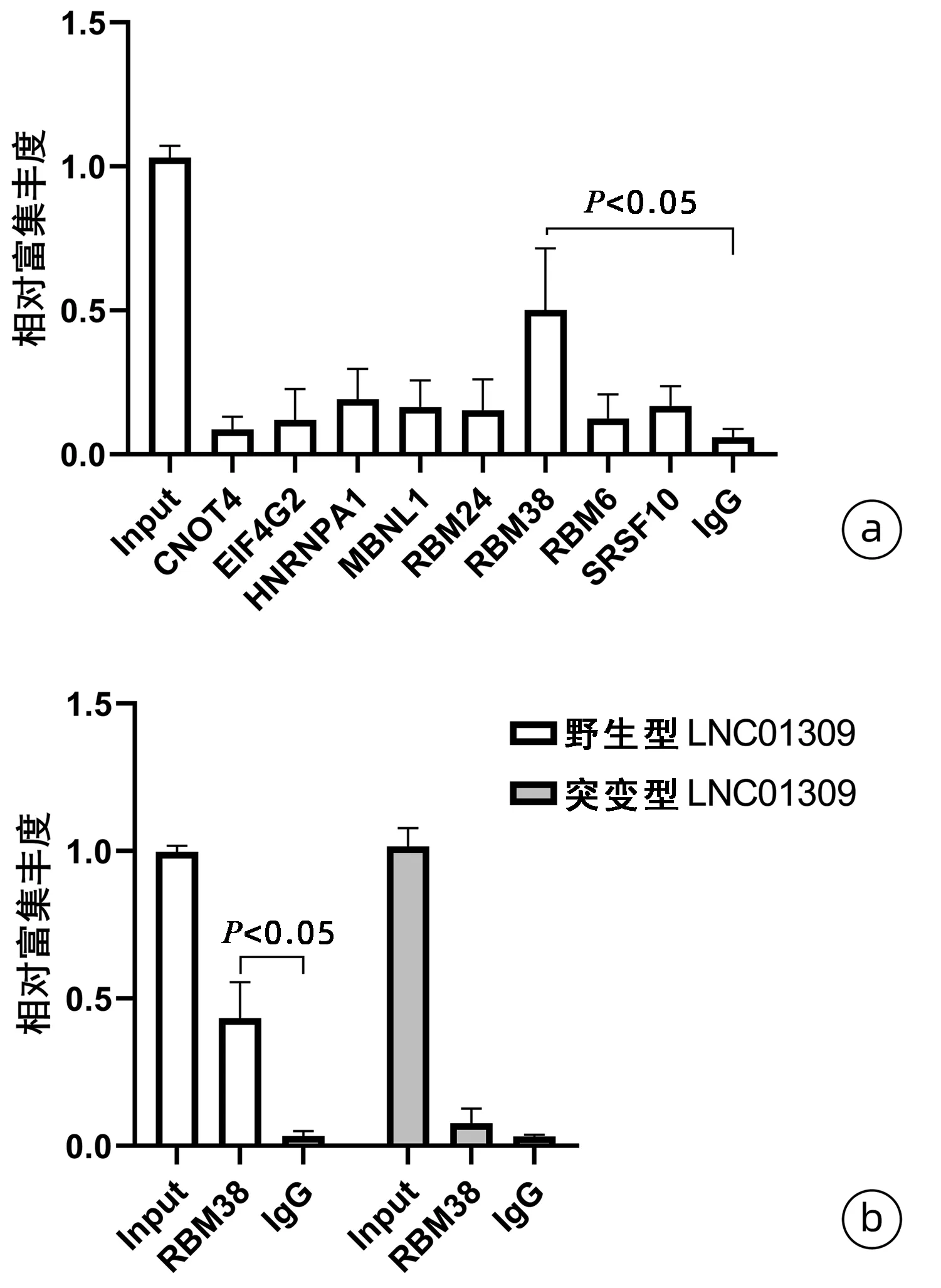
注:a,利用潜在RNA结合蛋白的特异性抗体进行RNA-IP试验,荧光定量PCR检测LNC01309的富集丰度;b,荧光定量PCR比较RBM38的抗体对野生型和突变型LNC01309的富集丰度差异。
2.5 LNC01309影响RBM38蛋白稳定性 在相对低表达LNC01309的Hep G2细胞中过表达LNC01309时,并不会影响RBM38 mRNA的相对表达量(1.022±0.074 vs 0.953±0.076,P=0.185),但会降低RBM38蛋白的相对含量(0.960±0.057 vs 0.228±0.044,t=5.526,P=0.008)(图11)。利用放线菌酮处理Hep G2细胞,LNC01309过表达组细胞中RBM38蛋白的稳定性显著低于对照质粒组(第4小时相对蛋白含量:0.610±0.075 vs 0.179±0.054,t=8.038,P=0.001)(图12、13)。此外,过表达的RBM38能够有效抑制LNC01309的促细胞增殖能力(第96小时OD450值:2.500±0.227 vs 1.913±0.282,t=2.812,P=0.048)(图14)。细胞划痕愈合试验结果(30.69%±5.80% vs 16.37%±1.66%,t=6.218,P<0.001)(图15)和Transwell试验结果(168.00%±9.43% vs 117.20%±18.03%,t=6.622,P<0.001)(图16)均显示过表达的RBM38能够有效抑制LNC01309诱导的肝癌细胞迁移能力的上调。

图11 Western Blot检测转染LNC01309表达质粒的Hep G2细胞24 h后RBM38蛋白的相对表达含量
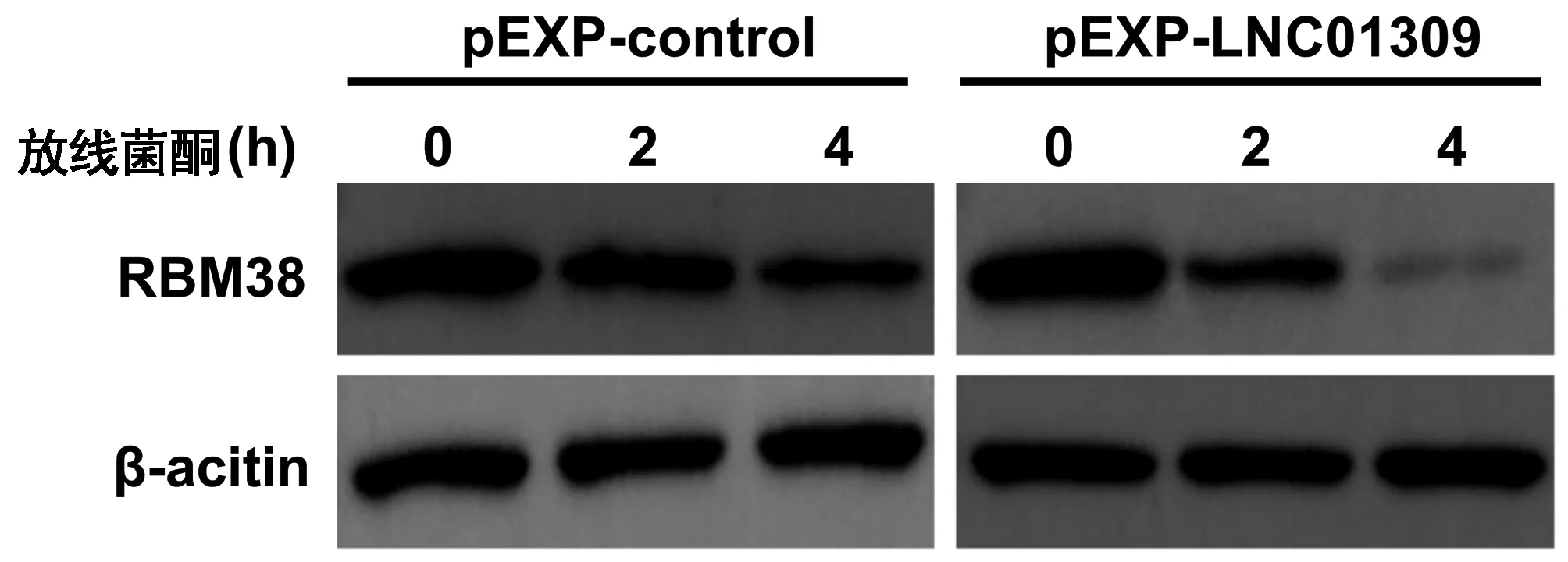
图12 Western Blot检测不同时间点经放线菌酮处理转染LNC01309表达质粒的Hep G2细胞中的RBM38蛋白相对含量
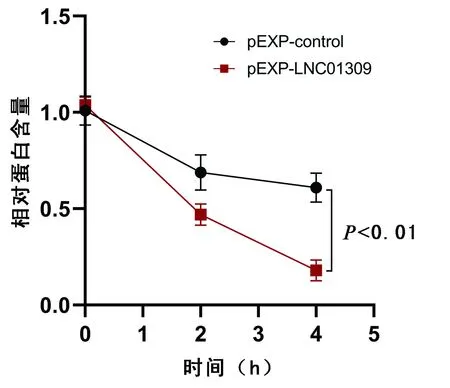
图13 不同时间点检测经放线菌酮处理转染LNC01309表达质粒的Hep G2细胞中RBM38蛋白相对含量

注:Hep G2细胞中单独转染pEXP-LNC01309或者共转染pCMV-RBM38质粒24 h后,于不同时间点利用CCK-8检测各组细胞活性。

图15 细胞划痕愈合试验检测各组转染HepG2细胞的迁移能力
图16 Transwell检测各组转染Hep G2细胞的迁移能力
3 讨论
大量的研究[10-12]工作已经证实lncRNA与肝癌的发生、发展及预后密切相关。例如,lncRNA ATB竞争性地结合miR-200,进而激活ZEB1和ZEB2的表达;ZEB1和ZEB2与IL-11 mRNA的相互作用增强了Stat3信号,促进肝癌转移,加速器官受累[13]。lncRNA HULC在肝癌中异常高表达[14],能够介导肿瘤抑制因子P18的下调,可促进肝癌的细胞增殖[15]。lncRNA HOTAIR降低
了CREB、P300、RNApoⅢ在SETD2启动子区域的重新分配,从而抑制了SETD2的表达和磷酸化,借此促进人类肝癌干细胞的恶性生长[16-17]。目前发现lncRNA主要有以下几个方面的调控功能[18-19]。(1)表观遗传学:通过干扰转录因子与启动子结合、诱导蛋白质修饰和染色质发生重构等方式影响编码基因表达;(2)转录调控:与靶蛋白结合后影响基因转录;(3)转录后调控:能调控mRNA稳定性和翻译进程,作用蛋白结合伴侣或调节蛋白的亚细胞定位影响蛋白活性等,进而影响肿瘤进展[20];(4)编码微肽:lncRNA重新获得编码能力,产生具有功能的微肽[21]。笔者通过PacBio测序分析LNC01309显示,在肝癌组织和肝癌细胞中均呈现高表达状态。后续的试验结果发现LNC01309能够与RBM38蛋白发生相互作用,并借此下调RBM38蛋白稳定性。
RBM38是一种RNA结合蛋白,在正常细胞中通过调控RNA参与基因转录后调控,从而影响细胞增殖、细胞周期循环、肌源性分化等多种生物学过程。目前已知RBM38可与p53、mdm2、p63、p73、p21、c-Myc和MIC-1等多个目的基因的3′非翻译区结合,调节相应mRNA的稳定性,从而在肿瘤进展中发挥关键作用[22-27]。现有研究[28]显示,RBM38可能通过稳定p53-mdm2环抑制肝癌的发生。p53-mdm2通路在肝癌中经常发生改变,mdm2和p53基因突变可增加mdm2的稳定性,加速p53降解,破坏p53-mdm2平衡,从而诱导癌变,最终促进肝癌发生。Ye等[28]报道RBM38作为稳定p53-mdm2环的重要分子,在肝癌组织中RBM38失活,而mdm2表达增加,野生型p53表达随之降低,最终破坏p53-mdm2环的平衡;相反,若增加RBM38表达则可抑制肝癌细胞mdm2的表达,恢复野生型p53的表达,从而在体外抑制肝癌细胞迁移和侵袭;裸鼠体内实验也显示RBM38对肝癌的体内致瘤性具有一定抑制作用。以上体内外研究结果表明,上调RBM38可能通过抑制mdm2从而拯救野生型p53,进而改变肝癌的生物学功能和进展。本研究结果表明RBM38能够与野生型LNC01309发生结合作用,而不能够结合突变型LNC01309。LNC01309通过与RBM38的相互作用降低RBM38的蛋白稳定性,促进肝癌细胞的恶性表型特征。
LNC01309与lncRNA MIR205HG具有较高的序列相似性。然而敲降已知的lncRNA MIR205HG不同剪接体,并不会改变LNC01309的相对含量。这些结果提示,LNC01309并不是已知lncRNA MIR205HG的再加工形式或者降解片段,其可能是MIR205HG基因初始转录本的另一种剪接体形式。已知的lncRNA MIR205HG在肝癌组织和肝癌细胞中并不存在异常表达现象,这也是与LNC01309最大的区别。
LNC01309在肝癌细胞中主要定位于细胞质。由于lncRNA通常会作为内源性RNA海绵与miRNA作用并影响miRNA的表达与功能,而lncRNA定位于细胞质是这一功能发挥的前提条件,因此LNC01309理论上存在参与调控miRNA表达与功能的潜质,这也是后续应开展的研究方向。
综上,本研究团队依赖于肝癌组织的PacBio测序分析获得一条全新lncRNA LNC01309,在肝癌细胞中进行了相关功能探索,初步证明LNC01309通过与RBM38相互作用降低了RBM38的蛋白稳定性,促进肝癌细胞的增殖与迁移。
利益冲突声明:本研究不存在研究者、伦理委员会成员、受试者监护人以及与公开研究成果有关的利益冲突。
作者贡献声明:刘红燕参与课题设计,完成试验,文章写作;李婧姣、路泽军、刘咏真参与试验、收集和分析数据及修改文章;温居一负责研究方案总体规划设计,指导撰写文章及论文修改。
——一道江苏高考题的奥秘解读和拓展

