MICA基因标签SNP检测用于结直肠癌的关联分析*
孙长江,丁伟峰
1.南通大学公共卫生学院,江苏南通 226001;2.南通大学附属医院医学检验科,江苏南通 226001
结直肠癌(CRC)是常见的消化系统恶性肿瘤[1],美国等国家CRC死亡人数占癌症死亡人数的10%,仅次于肺癌,位居第2位[2]。2017年中国肿瘤登记年报显示,我国城市CRC的发病率居恶性肿瘤第3位,并且与城市化程度相关,中国疾病预防控制中心统计的死亡人数与因癌死亡人数的比例超过10%,CRC防治形势依旧严峻[3]。
有研究发现,MLH1、MSH2、MSH6、PMS2、APC、SMAD4、BMPR1A、PTEN、STK11/LKB1、MYH等基因的单核苷酸多态性(SNP)及位点突变等遗传变异可导致CRC[4-7]。然而,这些位于人类主要组织相容性抗原以外的基因变异,不足以完全解释CRC的发病机制。主要组织相容性复合体Ⅰ类相关基因(MIC)家族包括MICA、MICB、MICC、MICD、MICE、MICF和MICG共7个成员,其中仅MICA和MICB基因编码蛋白质。MICA基因位于人第6号染色体6p21.31,全长11 772 bp,cDNA长1 382 bp,编码383个氨基酸残基[8-9]。MICA与其受体自然杀伤细胞的活化型受体(NKG2D)相互作用,共同组成免疫监视系统[8,10]。MICA基因具有高度多态性,目前已发现224种MICA等位基因(截至2020年9月的数据),共编码104种不同的蛋白质[11-13]。MICA基因的高度多态性及其在不同种族、不同人群的遗传异质性决定了其具有不同的标签单核苷酸多态性(TagSNP)。
有研究发现,MICA基因多态性与强直性脊柱炎、贝赫切特综合征、干燥综合征等自身免疫性疾病密切相关[8,11-12]。但MICA基因多态性与CRC等实体瘤的遗传免疫因素是否相关尚不清楚。因此,本文旨在研究CRC与MICA基因的TagSNP遗传免疫相关因素,探讨MICA基因的TagSNP用于连锁分析CRC的可行性。现报道如下。
1 资料与方法
1.1一般资料 选取2016年7月至2017年6月南通大学附属医院胃肠外科收治的104例散发性CRC患者作为CRC组,其中,男81例,女32例,平均年龄(66.1±10.1)岁。术中留取肿瘤组织及癌旁组织标本,采用无菌生理盐水清洗后于-80 ℃冰箱保存。536例健康对照(健康对照组)标本来自复旦大学遗传学研究项目队列[8,13]。本研究由南通大学附属医院伦理委员会审核并全程监督。
1.2Sanger测序法检测CRC组织中MICA外显子2~5区基因多态性 设计MICA外显子2~3区和4~5区的PCR扩增引物,采用Gel Extraction Kit(TianGen公司)抽提外周血或组织DNA,以全基因组DNA为模板,高保真扩增MICA外显子2~5区序列,并对PCR产物进行Sanger测序。25 μL PCR反应体系(TaKaRa公司):0.2 μg DNA模板,1×PCR反应缓冲液,200 μmol/L dNTPs,上、下游引物各0.2 μmol/L,Hotstar DNA聚合酶2.5 U。95 ℃预变性5 min,95 ℃变性30 s,56 ℃退火40 s,72 ℃延伸1 min,共35个循环。收集PCR产物。上机测序:采用ABI PRISM®BigDye®Termintor v3.1 cycle Sequencing Kit(Illumina公司)试剂及ABI 3730测序仪(ABI公司)进行测序。
1.3SNP数据分析与处理 通过数据库筛选候选MICA基因的TagSNP位点,使用Haploview软件进行TagSNP数据的鉴定与分析,根据block选择TagSNP,设置条件为少见等位基因频率(MAF)≥0.05,r2>0.5。采用自动分析系统比对SNP数据,再采用Chromas分析软件进行SNP数据的二次人工比对以保证杂合子结果的准确性。
1.4统计学处理 采用SPSS20.0统计学软件对数据进行处理。以P<0.1为差异有临界统计学意义,以P<0.05为差异有统计学意义。
2 结 果
2.1筛选候选MICA的TagSNP位点 通过人类主要组织相容性抗原网站(http://www.ebi.au.uk/imgt/hla),筛选MICA的SNP位点,结果发现,MICA基因外显子2~5区分布有83种SNP位点。其中后续分析中筛选出的29种MICA基因的SNP位点为候选TagSNP。
2.2候选MICA基因TagSNP在CRC组中的连锁性 根据104例CRC组的SNP情况,从MICA基因外显子2~5区筛选出29种TagSNP,位于1个block中。29种TagSNP的连锁不平衡性(LD)=1的TagSNP占95.3%,LD>0.8的TagSNP占96.5%;无连锁TagSNP仅占2.9%,包括rs9380254、rs1051785、rs1063632和rs1051790;说明MICA基因外显子2~5区各TagSNP间强连锁。
2.3候选MICA基因TagSNP在健康对照组中的连锁性 通过对536例健康对照组标本的Haploview检测,发现29种TagSNP的LD=1的SNP占90.6%,LD>0.8的SNP占91.8%,0 2.4候选MICA基因TagSNP在千人基因组的连锁性 从HapMap数据库(map.org)下载千人基因组MICA基因的SNP数据,使用Haploview软件,根据Hardy-Weinberge平衡,计算出MICA基因的TagSNP,结果显示,rs9380254在千人基因组中与rs1063630等其他SNP基本无连锁,二者位于不同的block。剩余28种TagSNP中,LD=1的SNP占86.9%,无连锁TagSNP占13.1%,包括rs41554412、rs61738275、rs1063630、rs1051790、rs1051785、rs1063632、rs3819268。说明相对于中国人群,千人基因组人群MICA基因外显子2~5区各TagSNP间连锁不平衡更弱。 2.5MICA基因候选SNP在CRC组与健康对照组中的差异 通过连锁不平衡分析发现,TagSNP在中国人群的CRC组、健康对照组及千人基因组有区别,筛选7种MICA基因显著性差异的SNP位点,进一步比较发现,这些显著性差异SNP位点在104例CRC组与536例健康对照组中的差异有临界统计学意义(P<0.1)。见表1。 表1 MICA基因7种显著性差异SNP位点 CRC的一个基本特征是基因或表观遗传的不稳定性。基因或表观遗传的不稳定可以鉴别肿瘤新生物与正常的大肠上皮[14-16]。研究发现,CRC相关的基因或表观遗传学分子病理学机制主要包括:(1)染色体不稳定(CIN);(2)微卫星不稳定(MSI);(3)非MSI的超突变;(4)异常的DNA甲基化及全DNA的去甲基化[17]。深入探讨CRC分子病理学机制有利于其早期诊断及预防性治疗[18]。 健康人MICA蛋白仅微量表达于肠道上皮,当感染、应激或恶性转化时其表达可明显上调,如卵巢癌、肝癌。MICA表达量与肿瘤发展水平及肿瘤浸润程度有一定关联:癌症早期MICA表达量低,弥漫型肿瘤时表达量增高。MICA蛋白在体内有可溶性MICA(sMICA)和膜型MICA(mMICA)两种形式[19],研究发现,sMICA是肿瘤细胞死亡时由MICA分泌和金属蛋白酶水解脱落到外周血的MICA[20]。释放sMICA是肿瘤细胞削弱MICA-NKG2D系统免疫监视的重要途径[21]。mMICA和sMICA对机体免疫系统的作用方向相反[22]。有文献报道,干扰素等可上调肿瘤mMICA的表达,且MICA多态性可影响mMICA的表达及sMICA的剪切[23]。TagSNP是疾病尤其是癌症等复杂疾病关联研究的重要分析策略。因此,探索MICA多态性尤其是MICA基因TagSNP对CRC的影响十分重要。本课题组前期研究发现,104例CRC癌组织和对应癌旁组织的MICA基因外显子2~5区无体细胞突变发生[13]。基于此,本研究选择536例健康对照组的外周血标本进行MICA基因外显子2~5区的SNP进行研究。本研究发现,CRC组与健康对照组7种MICA基因TagSNP位点的差异有临界统计学意义(P<0.1)。本课题组将在以后的研究中增加CRC组及健康对照组样本量进一步研究,使数据更具有说服力。 CRC典型的恶性变过程涉及多种致病因素,如基因突变、表观遗传学改变、局部炎症改变等。早期发现这些致癌因素并做出早期诊断有利于CRC的防治。MICA基因及其受体NKG2D在肿瘤免疫监视及肿瘤细胞杀伤方面有重要作用[24-25]。MICA基因SNP多态性可影响其与NKG2D的识别与结合,进而影响其免疫监视功能。本研究中,MICA外显子2~5区7种TagSNP在CRC组及健康对照组的差异有临界统计学意义(P<0.1),且TagSNP之间强连锁。说明MICA基因TagSNP具有关联分析CRC的潜力[26-27]。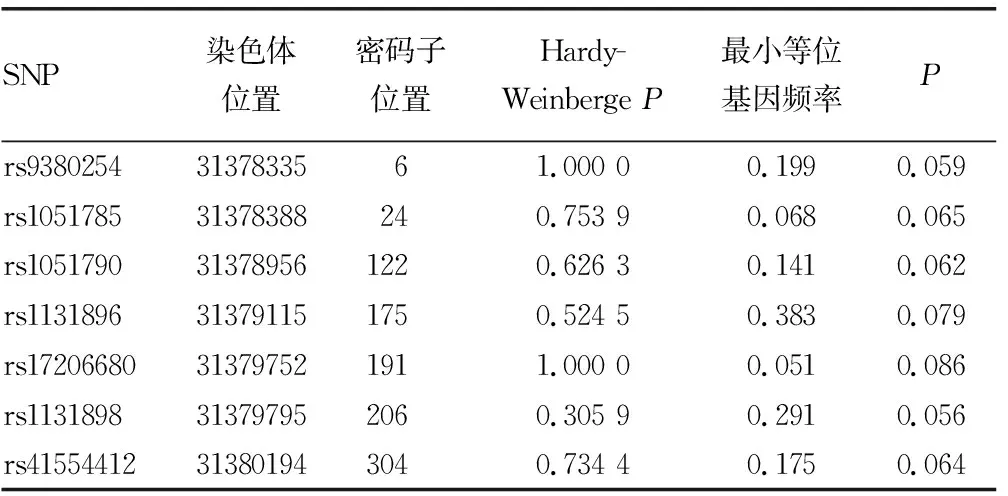
3 讨 论

