Enterobacter hormaechei Wu-15解盐促生相关基因的筛选
刘欢,徐小琳*,武占省,2,刘思琪
(1 石河子大学化学化工学院,新疆 石河子 832003;2 西安工程大学环境与化学工程学院,陕西 西安 710048)
植物根际促生菌(plant growth-promoting rhizobacteria,简称 PGPR)是指分布于植物根际周围,能够刺激宿主植物生长或抑制有害微生物的一类有益土壤细菌的统称[1]。近年来,PGPR因其良好的促生、抑菌作用成为有机农业中生物肥料和生物防治的重要组成部分[2],在现代农业生产与绿色发展中发挥着重要作用[3]。大量研究表明,植物根际促生菌不仅能通过固氮、溶磷、分泌植物生长激素等作用来促进植物生长,还可以通过诱导植物建立抵抗或忍耐机制,增强植物在盐胁迫条件下的生存能力[4]。
随着分子生物学技术的发展,对 PGPR 进行全基因组测序,为分析PGPR的生理生态功能,揭示PGPR的作用机制和代谢调控提供了有利条件。Kang等人研究表明LeifsoniaxyliSE134中trp 基因簇以及乙酰转移酶基因(ysnE)参与生长素 IAA 合成前体物色氨酸的生物合成[5]。ZipA被发现在PseudomonasaurantiacaStrainJD37基因组中,其编码的受体蛋白可能与铁载体产生有关,它可以促进植物吸收铁离子并形成特定的螯合物以预防病原体[6]。pst 基因以及 pho 基因可参与芽孢杆菌的溶磷过程,用基因工程方法使这两个基因表达量上调,将增加溶磷量,更好地促进植物生长[7]。诱导SOS1和NHX1基因编码的Na+/H+反转运蛋白的表达可以增强和适应盐分积累[8]。通过挖掘相关功能基因,解析不同菌株的主要促生特性,在分子水平上深化对各菌株生命活动的认识,能够为强化研究各菌株促生特性以及提高植物根部定殖效率、提高植物抗盐胁迫水平和菌株代谢互补等提供理论支持。
EnterobacterhormaecheiWu-15是本课题组分离自新疆盐渍化土壤的棉花根际的解盐促生菌,前期实验证明Wu-15具有良好的解盐促生能力[9]。本文以该菌株为材料,运用第二代测序技术和第三代单分子测序技术对其进行全因组测序分析,寻找菌株中与解盐促生相关的基因,以期能为进一步深入探究Wu-15解盐促生作用相关机制提供依据。
1 材料与方法
1.1 材料
EnterobacterhormaecheiWu-15 由本实验室自行筛选,16 s rDNA 鉴定为霍氏肠杆菌(Enterobacterhormaechei);曾用名为Rs-5。
1.2 基因组DNA的提取
参照 Michael 方法[10]提取Wu-15基因组DNA。
1.3 DNA质量检测
使用荧光染料(Quant-iT PicoGreen dsDNA Assay Kit)检测 DNA 总量并通过 1% 琼脂糖凝胶电泳检测 DNA 的完整性,之后将DNA 稀释的工作液保存于 4 ℃,储存液保存于-20 ℃。
1.4 全基因组测序
以EnterobacterhormaecheiWu-15 菌株为材料,基于 Illumina NovaSeq和PacBio Sequel 测序平台对Wu-15全基因组进行测序。本研究采用全基因组鸟枪法(Whole Genome Shotgun,WGS)策略[11],构建不同插入片段的文库,对其进行测序,测序工作委托给上海派森诺生物技术有限公司。
1.5 测序数据过滤及整理
二代测序采用 FastQC[11]对数据进行质量控制,SOAPec进行校正,三代下机数据用HGAP[133]和CANU[14]软件进行组装,得到contig序列。第二代数据用于校正第三代结果:将二代高质量数据用pilon[15]软件对第三代contig结果进行校正,最终得到完整的序列,相关数据已上传到NCBI,BioProject ID为PRJNA686495。
1.6 功能基因的注释
以Wu-15全基因组测序所得基因序列和通用数据库,如GO、COG和KEGG功能数据库进行BLAST比对,获得基因功能注释结果。
用BLAST2GO软件对GO功能进行了注释,并根据生物学过程、细胞组成和分子功能对Wu-15基因组中的编码基因进行了GO分类。此外,通过与COG及KEGG进行比对,对Wu-15基因组进行了全面的预测,并对其关键基因进行了详细的功能注释。
2 结果与分析
2.1 基因组DNA质量分析
2.1.1 DNA的总量
采用PROMEGA荧光定量设备进行DNA定量分析,结果如表1所示。由表可知,经测定Wu-15菌株DNA浓度为63.80 ng·μL-1,经过计算,最后得出菌株Wu-15的DNA总量为3.19 μg,样品质量合格,总量满足2次或者2次以上建库需要。

表1 Picogreen荧光检测结果
2.1.2 DNA的完整性
凝胶电泳被用来检测DNA大致的浓度,并看其是否降解、有没有污染。结果如图1所示,由图可观察到条带单一明亮,无弥散拖尾现象,说明DNA没有降解;条带明亮、不是隐隐约约的一条线,说明DNA浓度足够高;胶孔处没有出现弥散现象和条带,说明DNA没有被其它蛋白和杂质污染。
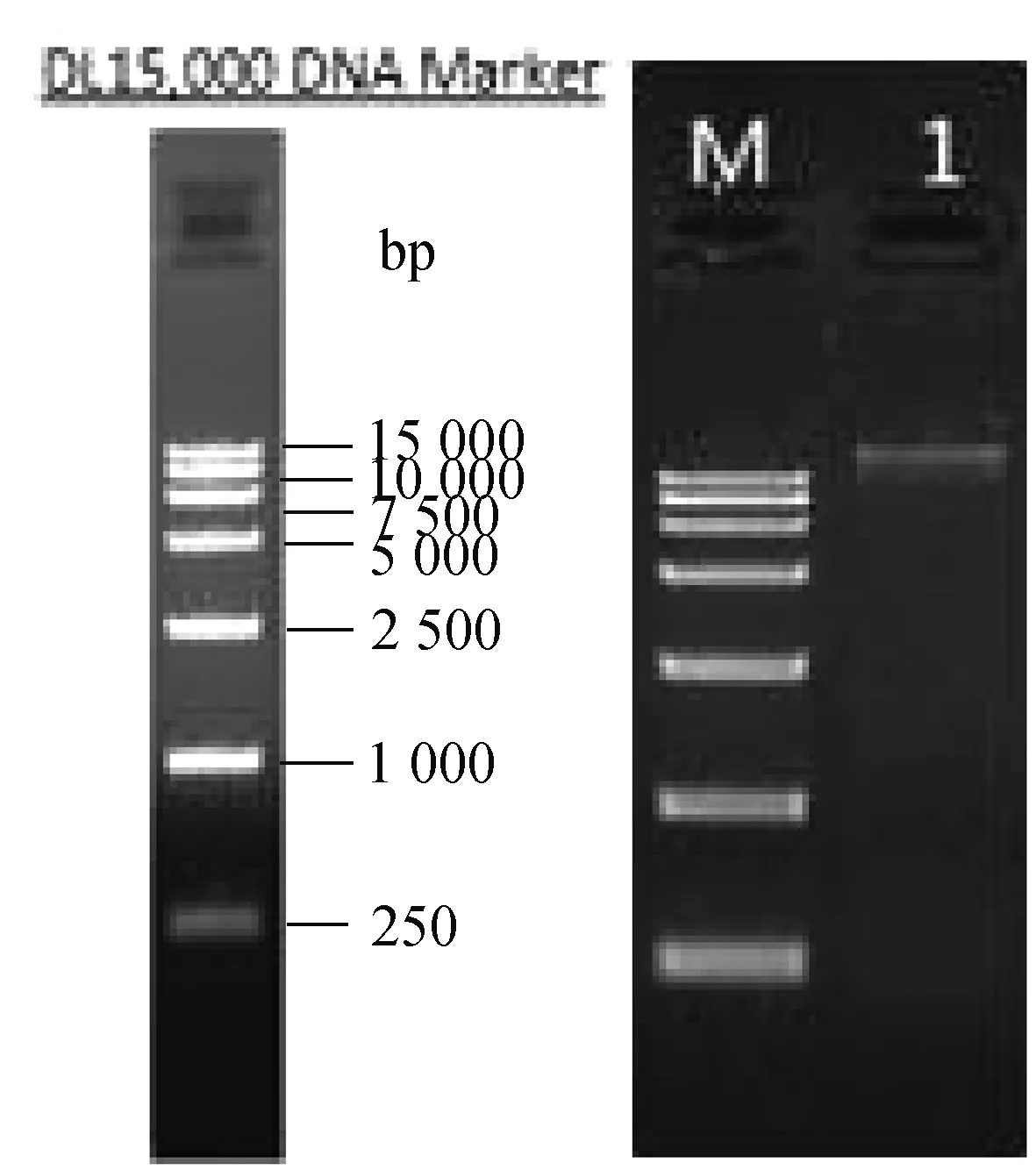
图1 Wu-15基因组DNA琼脂糖凝胶电泳图
2.2 测序数据统计分析
2.2.1 二代测序数据整理
测序数据中含有的低质量、带接头的reads,这将对后续的信息分析造成很大的干扰。为了保证后续信息分析的质量,有必要对数据进行进一步过滤。数据过滤的基本情况见表2。

表2 数据过滤统计
2.2.2 三代测序数据整理
对三代下机数据进行统计分析,统计情况见表3。

表3 三代测序数据序列长度统计
2.3 菌株Wu-15基因组的组成
2.3.1 基因组拼装结果
装配结果见表4。Wu-15菌株通过全基因组测序结果表明,Reads 总数为 13,733,028 条;碱基总数高达 4,614,006 bp;开放阅读框平均GC含量为56.96%,开放阅读框平均长度为962.03 bp,开放阅读框总长度为4 079 019 bp。有3个原噬菌体和29个基因岛。

表4 基因组拼装的数据统计
2.3.2 基因组圈图
将预测信息集成到一个标准的 GBK(GenBank)格式文件中,然后使用 cgview 绘制基因组图[16],最后使用 Photoshop CS 编辑图谱。以 chr 序列为例,最终结果如图 2所示。
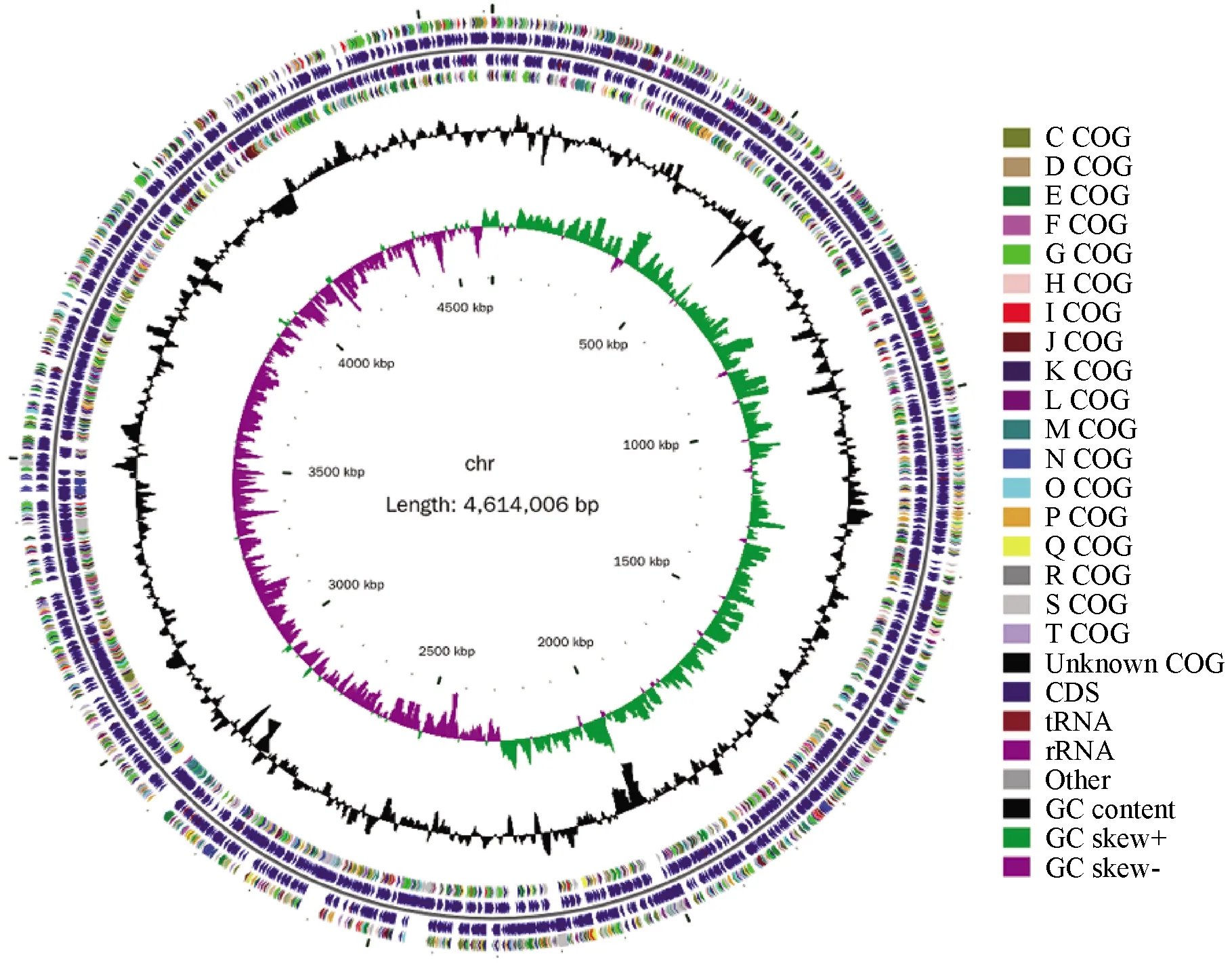
图2 Enterobacter hormaechei Wu-15基因组圈图(chr)
由内向外,第一圈是基因组大小的标识,每个刻度为100 kb;第二个圆是(G-C/G+C)值,该值为正时正链更倾向于转录编码基因,数值为负时则正好相反。第三圈代表GC含量,内部区域表明该区域的GC含量低于基因组的平均GC含量。第四圈和第七圈是基因组正负链上的基因,不同颜色代表COG的不同功能分类;第五圈和第六圈代表CDS、tRNA 和 rRNA 在基因组中的位置。
2.4 基因组注释
进行基因组功能注释主要是为了分析基因的功能,从而在分子水平上刨析该物种。GO是基因本体论的缩写,进行GO分析的主要目的是为了解决同一基因在不同数据库中的定义混乱和同一基因在不同物种中的定义混乱的问题。COG数据库全称是:直系同源基因簇数据库,把某一蛋白质与库中的蛋白进行比对,并将其归入适当的COG分类,对于预测蛋白功能很有用。KEGG全称是京都基因与基因组百科全书,它能系统性的分析基因产物在细胞中的代谢途径及该基因相关的功能。蛋白编码基因功能注释的概述性结果见表5。
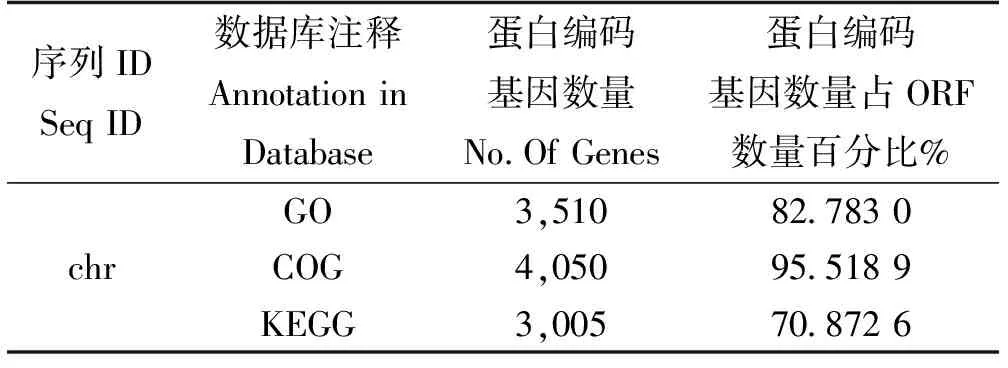
表5 蛋白编码基因功能注释概述
2.4.1 GO注释
GO注释由BLAST2GO软件完成[17]。根据GO注释方法,将其注释结果分为3大类(生物过程、分子功能、细胞组成),Wu-15基因功能集中在生物过程(图3)。在生物过程分类水平上,129个基因与信号转导有关,与代谢过程相关的基因有2 669个,与应激反应相关的基因有174个,包括磷酸盐饥饿反应(GO:0016036)、氧应激反应(GO:0006979)、活性氧物种反应(GO:0000302)等。分子功能分类中,催化活性相关基因有2 179个,表现在Wu-15的细胞内和细胞外代谢反应,129个基因与信号转导有关,22个基因与抗氧化活性有关;其中,与催化活性相关的GO注释基因数量占很大比例,说明Wu-15的分子功能主要集中催化胞内外物质生成、运输以及代谢等功能上,印证了解盐促生菌Wu-15的特性。
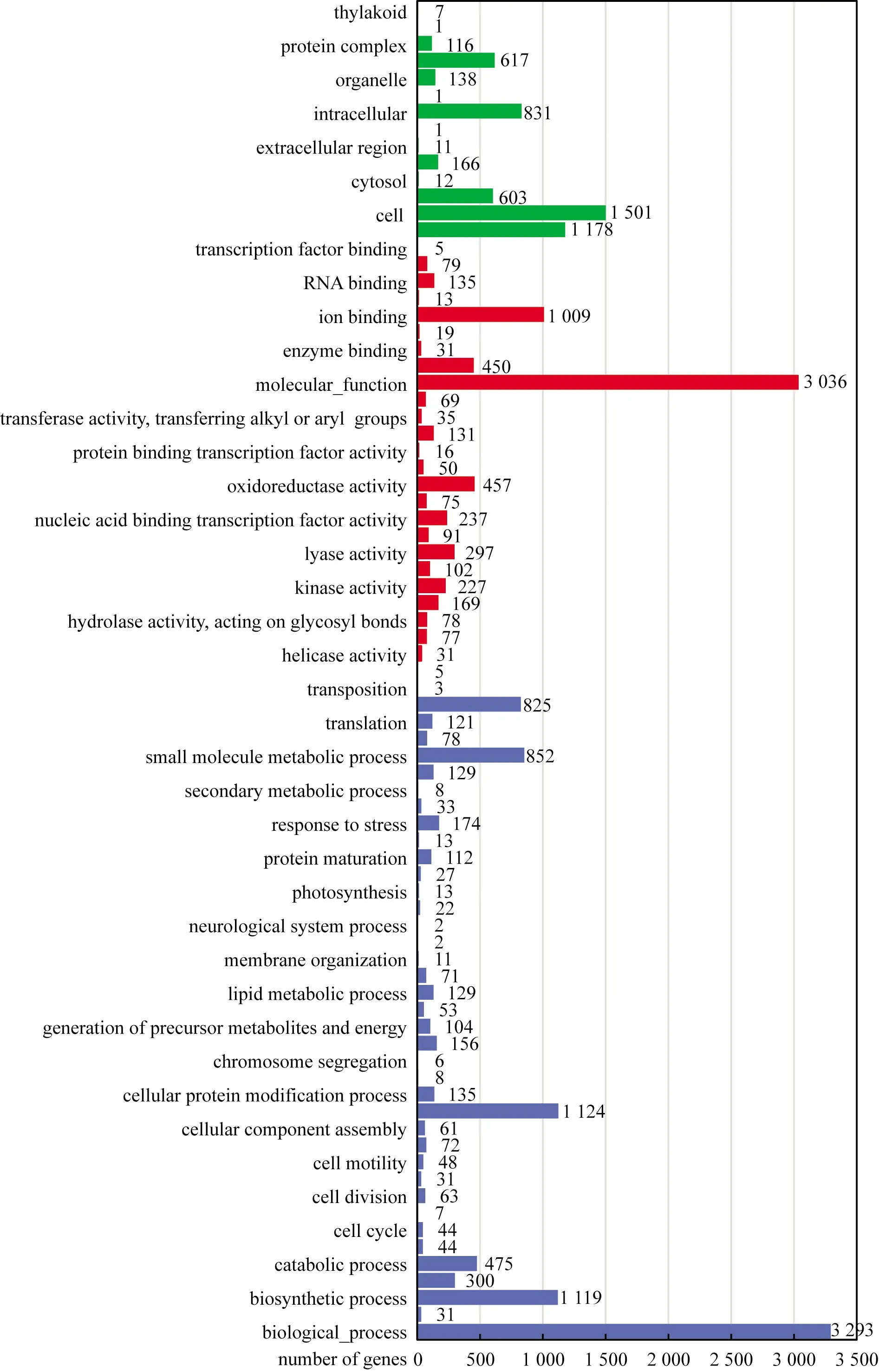
图3 Enterobacter hormaechei Wu-15的GO功能注释
2.4.2 COG注释
通过比较全基因组数据,把不同序列注释到不同的COG分类中(图4)。其中富集基因最多的S类有940个,然而这一部分基因功能未知,需进一步探索。编码蛋白质的基因共有21类,共4 050个基因,其中,有397个基因负责碳水化合物的运输和代谢,321个基因编码氨基酸运输和代谢相关蛋白,319个基因编码转录相关蛋白,说明COG注释的基因大多与细菌细胞的基本功能有关。此外,与细胞壁和膜形成有关的基因278个,与能量产生和转化有关的基因233个,脂质转运和代谢基因(1.9%),证明细菌使用了大量营养物质;与复制、重组和修复相关的基因148个,139个与辅酶转运代谢相关的基因,64个与次生代谢物的生物合成、转运和代谢有关的基因,用于合成次生代谢物,如参与不溶性磷溶解的各种有机酸,有70个与细胞运动有关的基因,说明该菌株运动性较强,有利于菌株的迁移和存活;此外,Wu-15整个基因组中没有与核质结构和动力学以及细胞骨架相关的基因。
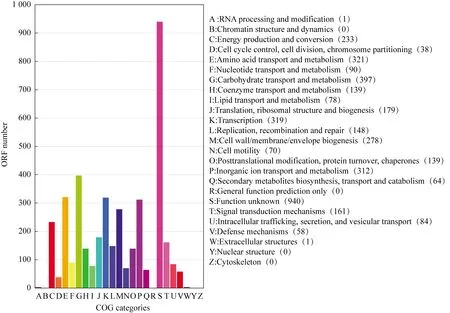
图4 Enterobacter hormaechei Wu-15的COG功能注释
COG主要确定了碳水化合物的转运与代谢、氨基酸的转运与代谢、转录与无机盐转运等功能,说明菌株可以生成多种次生代谢产物,并能调节无机盐离子的转运进而帮助植物抵抗盐胁迫。
2.4.3 KEGG注释
蛋白质编码基因的KO和pathway注释主要由KEGG的KAAS自动注释系统完成[18],以“原核生物”为基因集,进行判别。KO注释完成后,KO被映射到相应的KEGG通路上。Wu-15的KEGG注释主要体现在以下几个方面。在细胞加工过程中,与细菌运动有关的基因有118个,与原核生物细胞群落形成有关的基因201个。在代谢过程中,有515个与碳水化合物有关的基因,292个氨基酸合成基因;在环境信息处理过程中,有307个膜运输相关的基因,192个与信号传导相关的基因;在遗传信息处理过程,有54个折叠,分类和降解相关的基因,83个与翻译相关的基因,90个复制和修复相关的基因,4个与转录相关的基因(图5)。
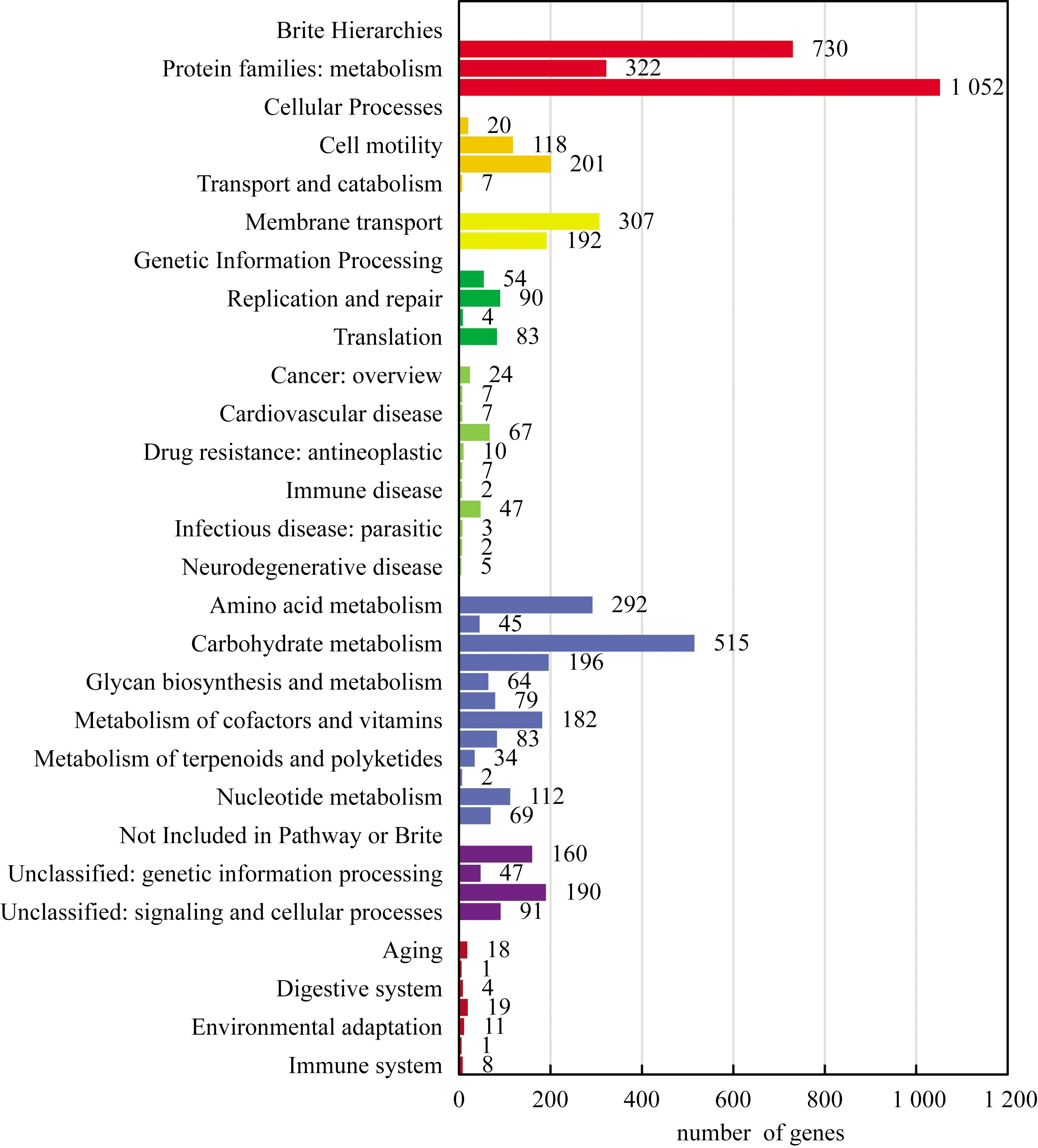
图5 Enterobacter hormaechei Wu-15的KEGG功能注释
Wu-15菌株中被注释最多的为与信号传导和细胞过程相关的基因1 052个,说明该菌株能够与外界进行跨膜运输和能量物质交换,印证了菌株调节离子浓度进而应对渗透胁迫的现象。
2.5 解盐促生功能相关基因
以Wu-15全基因组测序所得序列与不同数据库进行BLAST比对,之后对注释出的基因进行综合比对分析,得出如下结果。
2.5.1 溶磷相关的基因
溶解土壤中不溶性磷是根际促生菌促进植物生长的一种重要方式。如表6所示,本文在Wu-15菌株的基因组中鉴定了多个溶磷基因。葡萄糖酸(Gluconic acid,GA)被认为是大多数细菌的主要有机酸之一,它在无机磷酸盐的溶解中发挥了重要作用。Wu-15具有编码GDH(葡萄糖脱氢酶)活性的基因,并含有编码辅因子的pqq基因,包括pqqBCE,这些基因编码的酶可以催化合成GA;菌株中含有的磷酸酶可以水解磷酸单酯将底物分子上的磷酸基团除去,并生成磷酸根离子和羟基;三磷酸酶能将三磷酸分解成二磷酸和磷酸。由下表可知,Wu-15中与GA合成相关的基因有4个、酸性磷酸酶编码基因1个、碱性磷酸酶相关基因1个、有三磷酸酶编码基因1个。

表6 与溶磷相关基因的统计
2.5.2 渗透调节物质相关基因
渗透调节物质可以降低渗透势和水势,维持膨压,进行渗透调节。
如表7所示,在该菌株中发现了编码甜菜碱醛脱氢酶和胆碱脱氢酶的基因,这两个基因是甜菜碱合成途径中的关键基因;treY/treS和treZ是海藻糖合成的关键基因。另外,在Wu-15中发现渗透保护剂结合蛋白、脯氨酸/甜菜碱转运蛋白还有胆碱/甘氨酸/脯氨酸甜菜碱转运蛋白等在渗透胁迫中发挥作用。
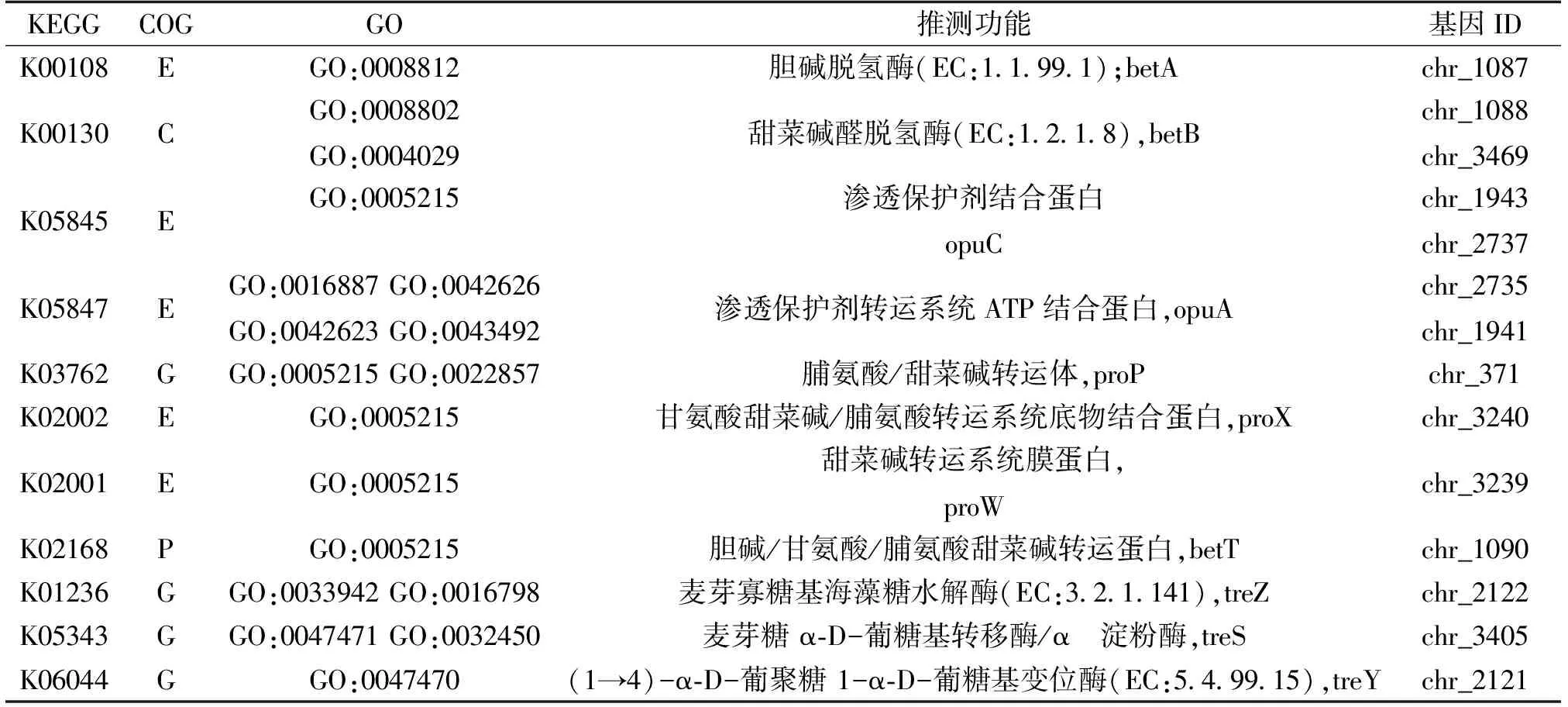
表7 Enterobacter hormaechei Wu-15耐盐渗透调节物质相关基因统计
2.5.3 逆向转运系统相关基因
Wu-15可以提高植物耐盐性并促进植物生长。Wu-15中某些基因编码组氨酸激酶,这些酶对K+摄取系统(kdp)很关键,而K+摄取系统在缓解盐胁迫中起到重要作用[19]。转运系统在抵抗渗透胁迫中起到重要作用,如K+转运系统和Na+/H+反转运系统(Nha),其用于导入H+和泵出Na+,在Wu-15基因组中也被发现(表8)。
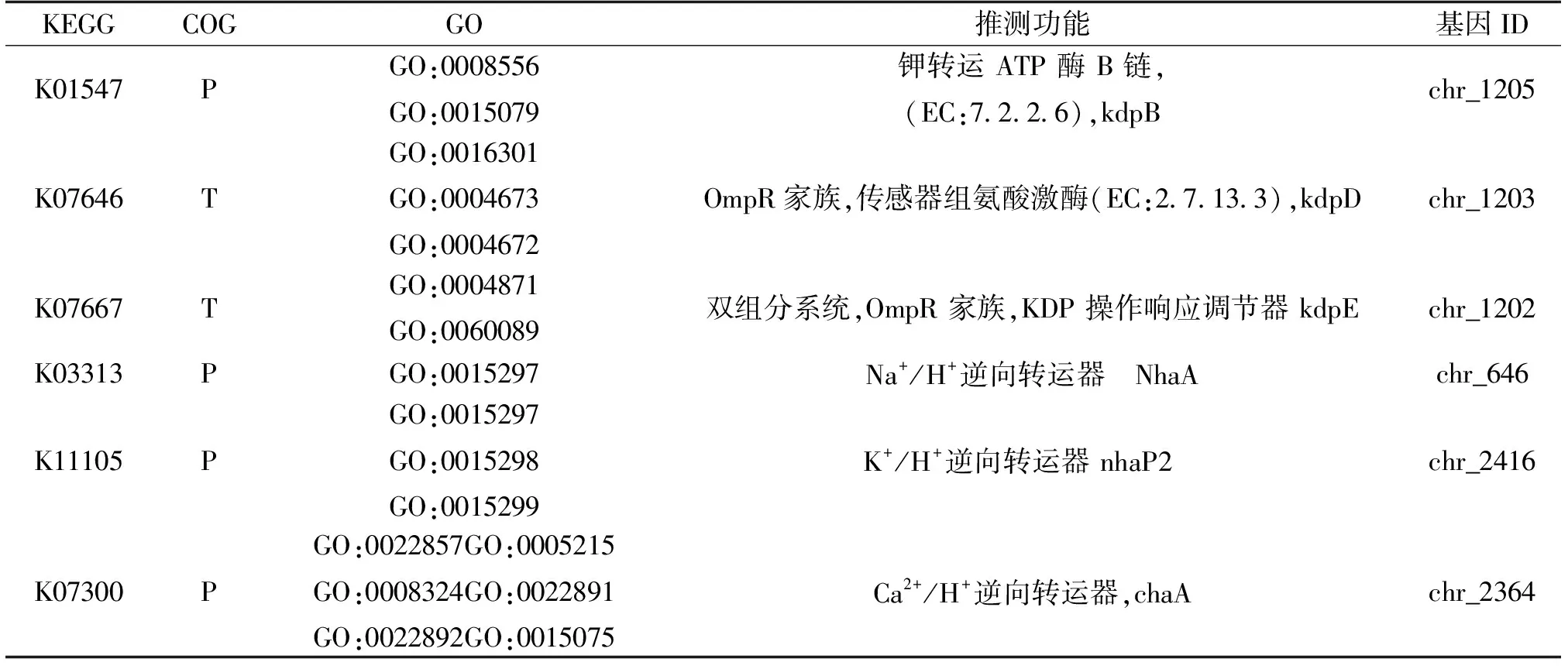
表8 与盐离子相关编码基因统计
2.5.4 IAA合成相关的基因
植物激素IAA是最丰富的天然植物激素,PGPR可以通过多种途径(吲哚乙酰胺途径、吲哚丙酮酸途径、色胺途径、吲哚乙腈途径、色氨酸侧链途径),并依赖色氨酸合成IAA。吲哚丙酮酸途径被认为是IAA生物合成的重要途径。图6是吲哚丙酮酸途径合成IAA的示例图。经过筛选,在Wu-5基因组中发现ipdC基因,该基因编码吲哚丙酮酸脱羧酶(EC:4.1.1.74),该酶能将吲哚丙酮酸脱羧生成吲哚乙醛,这一反应是吲哚丙酮酸途径的限速步骤。在Gang等人的全基因组学分析中,也发现了吲哚丙酮酸通路中的ipdC基因[20]。
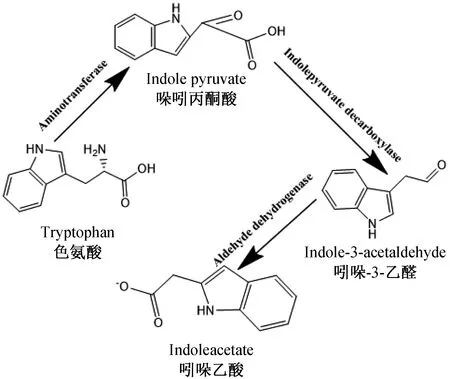
图6 IAA合成代谢途径
2.5.5 ACC脱氨酶编码基因
ACC 脱氨酶(ACCD)是一种植物生长激素,它能够减少乙烯的释放进而促进植物的生长。在Wu-15菌株的筛选过程中,没有发现编码ACC脱氨酶的基因ACDS,然而却发现了dcyD基因,该基因编码D-半胱氨酸脱巯基酶(D-Cdes)的生成,经过BLAST比对发现该酶的氨基酸序列与ACC脱氨酶序列高度一致,因此可以推测,Wu-15菌株中的dcyD并不是真正的特异性D-Cdes,它可能同时具有ACCD和D-CDes活性。
3 讨论
根际促生菌减少了合成肥料和农药的大量使用,在可持续农业发展中展现出不可或缺的作用。对根际促生菌进行全基因组测序,并对第二代和第三代测序结果进行分子水平上的解释,阐明解盐促生基因的代谢途径和功能,能为PGPR解盐促生相关机制提供有效的参考数据。
土壤中的磷以不溶性磷的形式存在,难以被植物吸收和利用。溶磷微生物是一类能够将植物难以吸收利用的磷转化为植物可吸收利用形式的微生物[21]。溶磷菌的溶磷机制因菌株不同而有所差异,目前,更多的研究集中在磷酸酶、氢质子和有机酸分泌方面[22]。本文在Wu-15菌株中鉴定了多个相关基因,如aphA,其能编码酸性磷酸酶的产生,而酸性磷酸酶可以从不同的有机磷底物上水解磷酸基团,供植物吸收利用;ygiF产生三磷酸酶;gcd编码的葡萄糖脱氢酶GDH及其辅因子可以催化葡萄糖酸合成,这些物质都在菌株的溶磷过程中发挥了重要作用。
脯氨酸、多糖、甜菜碱等渗透调节物质的积累是植物对盐碱胁迫反应的敏感指标,PGPR通过分泌这些渗透调节剂来增强植物的抗盐碱性[23]。在Wu-15菌株中筛选渗透调节物质相关基因,发现了编码甜菜碱醛脱氢酶和胆碱脱氢酶的基因,这些基因在甜菜碱的形成过程中发挥了重要作用。此外,Wu-15中发现了麦芽低聚糖海藻糖合酶和麦芽低聚糖海藻糖水解酶的编码基因,水解酶在(1→4)-α-D-葡萄糖苷与海藻糖之间的糖苷键发生作用,催化海藻糖(1→4)-α-D-葡聚糖的生成;麦芽糖α-D-葡萄糖基转移酶由treS编码,该酶能将麦芽糖转化为α-海藻糖,这些酶促反应在海藻糖合成过程中起到了关键作用。
盐胁迫扰乱了细胞内离子的稳态并减慢植物的新陈代谢,过量的Na+/K+会导致植物细胞缺水、膜功能障碍和离子毒性等[24]。因此,植物正常的细胞功能和生长发育中需维持低水平的Na+/K+浓度。从碱性土壤中分离的D5A菌株全基因组中发现了几种转运蛋白和激酶kdpD/kdpE,其能帮助植物缓解盐胁迫[25]。前期试验表明,Wu-15能显著降低根对Na+的吸收,缓解植物所受到的盐胁迫,因此探究菌株Wu-15逆向转运蛋白相关的基因,在Wu-15基因组中发现了编码K+转运系统和Na+/H+逆向转运系统等相关基因,这些基因被用来导入H+和泵出Na+,这些基因的发现从分子水平证实了Wu-15解盐促生功能(表7)。
合成IAA是许多根际促生菌的特性。以色氨酸为前体的IAA的合成可分为5类:吲哚乙腈、色胺、色氨酸侧链氧化酶、吲哚-3-丙酮酸和吲哚乙酰胺。细菌可通过一个或多个途径合成IAA。在吲哚-3-丙酮酸(IpyA)途径[26]中,色氨酸首先氧化脱氨形成吲哚丙酮酸,然后吲哚丙酮酸经过吲哚丙酮酸脱羧酶的作用形成吲哚乙醛,最后再氧化为IAA。基因ipdC(吲哚丙酮酸脱羧酶EC:4.1.1.74)被发现在Wu-15全基因组中,ipdC是吲哚丙酮酸途径合成吲哚乙酸的关键基因,说明菌株Wu-15可能通过IpyA途径合成IAA,不过还需要实验来进行进一步的验证。
ACC 脱氨酶可以降解乙烯的前体物质 ACC,生成氨和羟基丁酸,减少植物体乙烯数量,进而促进植物生长。McDonnell等人发现了编码d-半胱氨酸脱硫酶的At1g48420基因具有ACC脱氨酶活性[27]。经过筛选,在Wu-15菌株中发现了chr_2553,该基因编码d-半胱氨酸脱硫酶,然而前期的实验鉴定出该菌株有产ACC脱氨酶的能力,所以推测该基因可能也具有ACC脱氨酶活性。
4 结论与展望
本研究用BLAST比对方法从分子水平成功挖掘了EnterobacterhormaecheiWu-15与植物耐盐性相关的功能基因,其中包括溶磷相关基因7个、渗透调节物相关基因14个、反转运蛋白相关基因6个、IAA合成相关基因ipdC、ACC脱氨酶相关基因dcyD,本研究从分子生物学角度探究了根际促生菌,为菌株的实际应用提供了有价值的基因数据,也为解盐促生菌进一步的优化改良奠定了坚实的基础。

