利用CRISPR/Cas9技术定向编辑水稻OsIAA11
李兆伟,零东兰,孙聪颖,曾慧玲,刘凯基,蓝颖珊,范凯,林文雄
利用CRISPR/Cas9技术定向编辑水稻
李兆伟1,零东兰1,孙聪颖1,曾慧玲1,刘凯基1,蓝颖珊1,范凯2,林文雄1
1福建农林大学生命科学学院,福州 350002;2福建农林大学农学院,福州 350002
【】参与的生长素信号途径在水稻生长发育阶段和环境因子响应中起重要作用,并影响水稻生育后期的产量形成过程。利用CRISPR/Cas9编辑技术对粳稻中花11(ZH11)的序列进行编辑,获得突变植株,通过对突变植株的农艺性状指标开展田间调查分析,以期探索突变对水稻产量构成因子的影响。依据CRISPR/Cas9编辑原理,在第1和第2外显子区域设计2个20 bp的编辑靶点,并在水稻基因组数据库中比对分析靶点序列,排除非特异性编辑,将2个靶点核苷酸片段分别与pYLgRNA-U6a和pYLgRNA-U6b载体连接,通过2次PCR扩增,得到含特异性连接接头的U6a-IAA11-T1和U6b-IAA11-T2表达盒,再将2个表达盒连接到pYLCRISPR/Cas9-MT载体上,获得pYLCRISPR/Cas9-IAA11-T12重组表达载体,利用农杆菌介导法转化ZH11愈伤组织,再生培养得到T0代转基因幼苗,通过PCR扩增潮霉素抗性基因获得阳性株系。对T2代植株的靶点区域序列进行PCR扩增和测序分析,鉴定突变类型,并考察突变植株的田间农艺性状。pYLCRISPR/Cas9-IAA11-T12表达载体成功转化ZH11水稻愈伤组织,并获得25株转基因再生植株,经潮霉素鉴定得到20株阳性株系,从阳性植株的T2后代中鉴定出17种在2个靶点区域都发生编辑的纯合突变类型植株,除、、和在第1个靶点、以及在第2个靶点是单碱基插入突变外,其他突变植株在第1个靶点为小片段缺失突变,在第2个靶点多为单碱基缺失突变。对17种不同基因型突变体的农艺性状调查表明,与野生型水稻相比,突变体水稻的株高、有效穗数、穗长、穗粒数、结实率、千粒重、收获指数以及谷草比等性状未发生明显变化,而分蘖成穗率则显著降低,表明无效分蘖增多。采用CRISPR/Cas9技术对序列进行编辑,得到17种不同基因型的突变体水稻,其分蘖成穗率显著降低,无效分蘖变多,表明参与了生长素对分蘖芽发生的调节代谢。
水稻;基因编辑;;CRISPR/Cas9技术;突变体
0 引言
【研究意义】生长素通过调控生长素响应基因的表达而调节植物的生长发育进程,包括胚胎分化发育、侧根形成、顶端优势维持、侧枝和花的分化、向性运动感应等[1]。在生长素信号途径中,Aux/IAA基因家族成员通过与生长素响应因子(auxin response factor,ARF)互作,调控生长素信号途径下游响应基因的应激表达,在起始生长素信号途径代谢中发挥重要作用,并最终影响作物产量形成因子变化。利用CRISPR/Cas9编辑技术对Aux/IAA基因家族成员进行编辑突变,调查突变植株的农艺性状特征,对探究生长素早期响应机制,丰富和完善生长素信号途径调控水稻农艺特性及产量形成等具有重要意义。【前人研究进展】基因编辑是以核酸酶作为剪切工具,对基因组中的目标基因进行剪切修饰,实现对靶基因特定位置DNA片段的删除或插入突变,达到对目标性状的分子遗传改良[2]。较早的基因编辑手段是以锌指核酸酶(zinc finger nucleases,ZFNs)为代表的第一代编辑技术,以及类转录激活因子效应因子核酸酶(transcription activator-like effector nucleases,TALENs)为代表的第二代编辑技术,其中,ZFNs技术存在靶向载体构建复杂、难以实现多靶点编辑等局限性,TALENs技术则存在靶向串联基序较长、重复序列较多等缺陷,编辑成本较高,严重制约了其在分子设计育种与生物遗传改良中的应用[3-4]。近年来出现的成簇规律间隔短回文重复序列核酸酶(clustered regularly interspaced short palindromic repeats/CRISPR- associated nucleases,CRISPR/Cas)作为第三代基因编辑技术,具有编辑更加简单、高效、精准等优势,尤其是CRISPR/Cas9编辑技术通过导向RNA(guide RNA,gRNA)互补识别目标基因的特异靶序列,核酸酶Cas9切割与gRNA互补的基因组DNA区域,引起目标基因DNA双链断裂,生物体在修复断裂DNA的过程中引入碱基突变[5]。CRISPR/Cas9技术已成功地在拟南芥、水稻、小麦、玉米、番茄、烟草、大豆等农作物中实现了对目标基因的编辑突变[2,6-8]。Aux/IAA家族基因编码半衰期极短的蛋白,典型的Aux/IAA蛋白含有4个保守结构域:DomainⅠ、DomainⅡ、DomainⅢ和DomainⅣ,其中DomainⅠ和DomainⅡ位于氨基酸序列N端,DomainⅢ和DomainⅣ位于氨基酸序列C端,DomainⅠ能够与阻遏物结合,是转录抑制结构域;DomainⅡ区域含有5个保守氨基酸的泛素化降解靶点,该保守氨基酸的编码碱基突变会引起Aux/IAA蛋白不能被泛素化降解,增加Aux/IAA蛋白的稳定性,使ARF转录因子长时间被Aux/IAA蛋白结合阻遏,不能激活生长素下游响应基因的表达[9-10];DomainⅢ和DomainⅣ则负责与ARF蛋白C端结构域结合形成异源二聚体,抑制ARF对下游基因的调控转录,阻断生长素信号途径[11-12]。水稻基因组中共鉴定出31个Aux/IAA基因,分布在12条染色体上,在水稻生长发育不同阶段的多个组织、器官中呈现出多样化的表达模式,并受外源水杨酸、细胞分裂素、赤霉素、生长素、茉莉酸等激素影响[13-14],参与水稻多个组织器官的生长素信号途径调节的生理表型。前人研究报道,过表达转基因株系的株高变矮,植株分蘖角增大,株型变得松散,而主根变长,根系数目增加,并对外源生长素处理不敏感[15];的DomainⅡ区域点突变的过表达水稻变得对外源生长素和重力刺激不敏感,侧根和不定根变短,冠状根减少,叶片变短,维管组织发育受到影响[16];的表达则受干旱胁迫诱导,并在水稻茎基部的分裂组织中特异性表达,调节水稻对干旱的响应和分蘖芽的发生[17];参与的生长素信号途径调控水稻侧根发育起始基因表达,缺失突变的水稻植株侧根数减少,根系对重力响应缺失,侧根发育起始基因表达模式发生改变[18];则在水稻病毒感染过程中起重要调节作用,在矮缩病毒感染水稻时,矮缩病毒P2蛋白与水稻OsIAA10蛋白直接互作,抑制OsIAA10蛋白通过26S蛋白酶体介导的降解过程,使得感病水稻的OsIAA10蛋白积累显著高于健康植株,阻碍生长素信号传导,增强了矮缩病毒的复制侵染能力,而敲除突变植株则对水稻矮缩病毒的抗性显著增强[19];介导的生长素信号在水稻根尖静止中心功能维持中起重要作用,水稻突变体根尖的静止中心缺失,侧根和不定根数目减少且发育不良,植株生长迟缓,花粉育性变差,结实率降低,根对重力反应消失[20];而目前被报道参与调控水稻侧根起始,点突变严重阻碍植株侧根原基的发生,导致无侧根表型[21]。以上这些研究表明,水稻Aux/IAA基因家族介导的生长素信号途径参与了水稻侧根分化、分蘖发生、株型构建等组织生长发育过程,以及干旱抗性、病毒侵染等环境因子响应,然而,水稻仍有大多数Aux/IAA基因的生物学功能有待被进一步揭示。【本研究切入点】虽然已被报道参与水稻侧根发育起始过程,然而笔者前期通过灌浆期叶片早衰突变体水稻材料研究发现,在水稻产量形成阶段,的表达丰度显著异于野生型,早衰突变体的株高、分蘖数、结实率、千粒重、收获指数等农艺指标均显著低于野生型,丰度与产量形成因子间呈现一定的相互关系[22],而对介导的生长素信号与产量形成之间的调控机制仍不清楚。【拟解决的关键问题】本研究拟利用CRISPR/Cas9技术对粳稻中花11(ZH11)的进行编辑突变,并通过测序分析鉴定编辑植株的基因突变类型,田间试验调查突变体的农艺性状指标,以期探究突变对农艺特性和产量构成因子的影响。
1 材料与方法
1.1 试验材料
以粳稻中花11(ZH11)作为重组载体转化的受体材料,利用农杆菌介导法进行重组pYLCRISPR/ Cas9-IAA11-T12表达载体的遗传转化。转基因水稻材料种植于福建省福州市福建农林大学试验农场转基因管理区,采用适合当地温光条件的常规田间管理措施。
pYLgRNA-U6a、pYLgRNA-U6b、以及pYLCRISPR/ Cas9-MT载体由福建农林大学海峡联合研究院朱强教授惠赠,最初来源于华南农业大学刘耀光院士实验室,试验用的编辑靶点序列和引物序列(表1)、以及核酸测序服务均由生工生物工程(上海)股份有限公司实施完成,Ⅰ内切酶、T4 DNA连接酶、KOD-Plus-Neo高保真酶则分别从NEB(北京)有限公司、宝生物工程(大连)有限公司、东洋纺(上海)生物科技有限公司购买。
1.2 靶点设计和CRISPR/Cas9编辑载体构建
根据水稻(LOC_Os03g43400)序列特征,利用CCTop - CRISPR/Cas9 target online predictor在线软件分别在第1和第2外显子区域设计编辑靶点(图1-A),并将2个靶点序列在水稻基因组数据库中进行序列比对,以消除非特异性靶点干扰。在靶点1序列的2条单链5′端添加gccg和aaac酶切序列,使其与pYL-U6a-gRNA载体经Ⅰ(NBE)酶切后形成黏性末端接头,在靶点2序列的2条单链5′端添加gttg和aaac序列(表1),使其与pYL-U6b-gRNA载体经Ⅰ酶切后形成互补黏性末端。
参照MA等[23]方法构建双靶点pYLCRISPR/ Cas9-IAA11-T12表达载体,首先将含接头的2个靶点分别与pYL-U6a-gRNA和pYL-U6b-gRNA载体连接,利用引物U-F:5′-CTCCGTTTTACCTGTGGAATCG-3′和gRNA-R:5′-CGGAGGAAAATTCCATCCAC-3′经PCR扩增得到含靶点序列的U6a-IAA11-T1和U6b- IAA11-T2表达盒;通过接头引物Uctcg-B1′/gRctga-B2(上游:5′-TTCAGAGGTCTCTCTCGACTAGTGGAA TCGGCAGCAAAGG-3′,下游:5′-AGCGTGGGTCTC GTCAGGGTCCATCCACTCCAAGCTC-3′)和Uctga-B2′/ gRcggt-BL(上游:5′-TTCAGAGGTCTCTCTGACACT GGAATCGGCAGCAAAGG-3′,下游:5′-AGCGTGG GTCTCGACCGACGCGTCCATCCACTCCAAGCTC-3′)分别经PCR扩增为U6a-IAA11-T1和U6b-IAA-T2表达盒加入特异性接头,依照图1-B所示,使用Ⅰ内切酶、T4 NDA连接酶将U6a-IAA11-T1和U6b- IAA11-T2表达盒与pYLCRISPR/Cas9-MT载体连接,构建好的重组载体经热激法转化DH5α感受态细胞,经平板培养后,挑取单菌落用于测序,经测序验证正确的克隆用于转化农杆菌EHA105感受态,并侵染转化粳稻ZH11愈伤组织,获得转基因植株。通过PCR扩增潮霉素基因筛选阳性转基因株系。

表1 所用的引物序列
序列中的小写字母为Ⅰ内切酶的黏性末端接头 The lowercase letters stand for sticky end joints ofⅠ restriction endonuclease
1.3 基因编辑株系的靶点分析与鉴定
为检测的靶点突变情况,在2个靶点序列两侧分别设计引物OsIAA11-T1-F/OsIAA11-T1-R和OsIAA11-T2-F/OsIAA11-T2-R(表1),扩增产物长度为443和469 bp,通过CTAB法提取转基因植株的DNA,进行PCR扩增检测,扩增片段送交生工生物工程(上海)股份有限公司进行测序,并将测序结果与野生型DNA的扩增测序结果进行比对分析,鉴定突变植株的基因变异类型。
1.4 突变体植株的农艺性状调查
突变体水稻种子在浸种、催芽后,于2020年4月5日点播在育秧田中,待长出新叶后,对正常成苗的植株进行标记编号,每株剪取约2 cm长度叶片提取DNA,用以进行突变位点检测,鉴定出的纯合突变植株于4月30日移栽,各突变体经测序检测得到的纯合突变植株,分别于试验小区种植6行,每行栽种8株,单本插秧,株行距为25 cm×25 cm,不同突变体之间种植3行野生型ZH11作间隔,试验田边界种植3行ZH11隔离行,每个突变体材料设置2个重复试验小区,进行常规田间栽培管理和病虫草害防除。在6月上旬水稻孕穗期前2周时的分蘖高峰期,挑选长势相对一致的植株统计总分蘖数,待7月下旬水稻成熟时,考察突变体水稻的株高、有效穗、穗长、每穗粒数、种子结实率、千粒重、单株产量、生物产量等农艺性状指标,并计算成穗率、收获指数和谷草比。利用SPSS13.0软件计算农艺性状指标的平均值与标准差,使用单因素方差分析法比较数据的种间差异,以≤0.05为显著性差异。
2 结果
2.1 OsIAA11靶点设计和pYLCRISPR/Cas9-IAA11-T12载体构建
位于水稻第3染色体,DNA序列编码区全长为3 373 bp,由5个外显子和4个内含子组成,以第1和第2外显子作为突变区域设计靶点,选取第1外显子的反义链+270到+272 bp处的GGG为PAM(protospacer-adjacent motif)序列,+273到+292 bp之间的20 bp碱基作为靶点1序列,以第2外显子+1 944到+1 946 bp处的TGG碱基为PAM序列, +1 924到+1 943 bp区间的20 bp碱基作为靶点2的编辑序列(图1-A)。由图1-B的氨基酸序列结构分析表明,OsIAA11氨基酸序列具备典型的Aux/IAA蛋白结构,共包含4个结构域,其中,结构域Ι具有2个与乙烯响应因子相关的反应抑制EAR基序,以及位于2个EAR基序间的一个核定位信号(nuclear localization signal,NLS);结构域Ⅱ含有保守的TIR1/AFB识别序列“GWPPV”的降解决定子,以及一个NLS,编辑靶点1位于结构域Ⅱ核心序列“GWPPV”上游的PDKPRA氨基酸区域;结构域Ⅲ为βαα-折叠域,在Aux/IAA与ARF蛋白互作中起作用,编辑靶点2位于该结构域;结构域Ⅳ含有一个与蛋白质静电作用相关的保守“GDVP”基序,以及一个SV40型NLS片段。
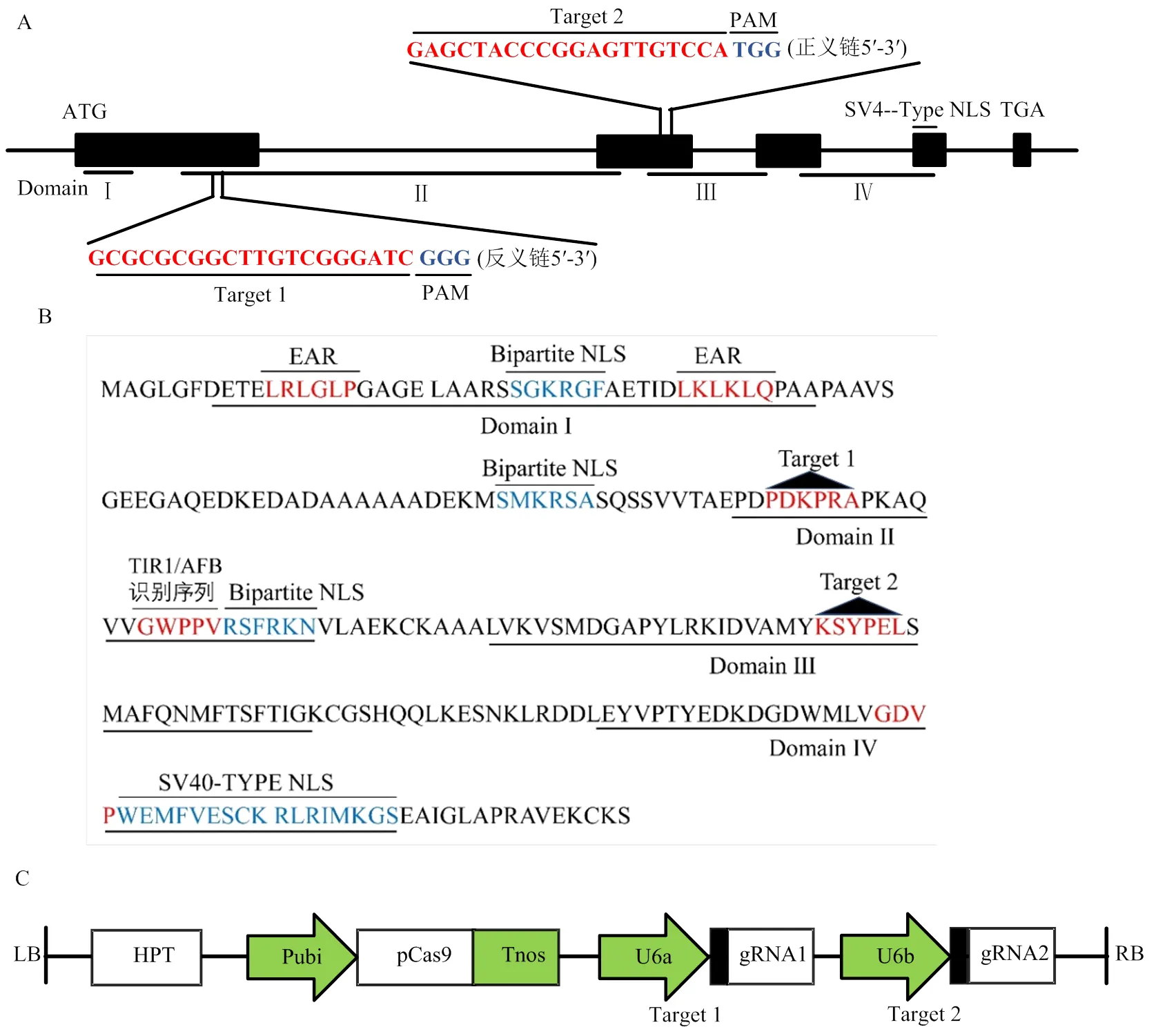
A:OsIAA11的2个gRNA靶点位置;B:OsIAA11氨基酸序列结构;C:2个靶位点gRNA表达盒与pYLCRISPR/Cas9-MT组装成的表达载体。LB:左边界;RB:右边界
为使重组CRISPR/Cas9载体中的2个靶点序列分别由OsU6a和OsU6b启动子驱动,通过酶切、连接将双链靶点序列1与pYL-U6a-gRNA载体连接,靶点序列2与pYL-U6b-gRNA载体连接,再经2次PCR扩增获得含特异性酶切位点和连接接头的启动子-gRNA1表达盒和启动子-gRNA2表达盒,依据图1-C连接方式将2个表达盒与pYLCRISPR/Cas9-MT载体连接,获得pYLCRISPR/Cas9-IAA11-T12重组表达载体,经测序分析表明,重组载体中的2个靶点序列(图2)与设计序列一致(表1),靶点准确连接到pYLCRISPR/Cas9载体上,重组载体pYLCRISPR/ Cas9-IAA11-T12适合转化水稻愈伤组织。

图2 pYLCRISPR/Cas9-IAA11-T12重组载体中2个靶点序列测序结果
2.2 获得T0代阳性植株
利用农杆菌介导法将pYLCRISPR/Cas9-IAA-T12重组表达载体转化ZH11愈伤组织,经培养再生得到25株转化株系,提取每个转基因株系的基因组DNA,采用PCR扩增检测潮霉素标记基因,由图3结果可见,共有20株转基因植株可以扩增出280 bp长度的条带,为阳性转化植株,有5株植株的DNA扩增不出潮霉素标记基因,为阴性植株,阳性率约达80%。
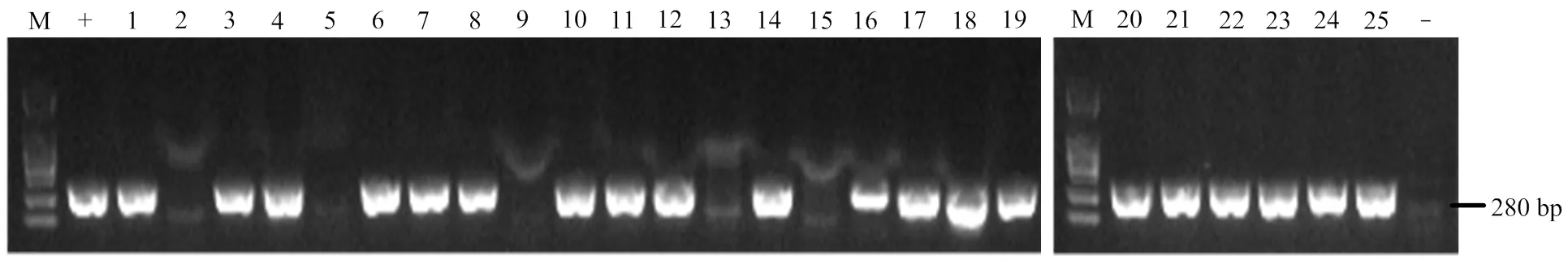
-:阴性对照;+:阳性对照;M:2000 bp的DNA marker -: negative contro; +: positive control; M: 2000 bp DNA marker
2.3 鉴定osiaa11突变体植株的基因突变类型
为了从转基因株系中筛选出纯合突变植株,对T2代植株的目标基因靶点及其上、下游序列进行PCR扩增和测序分析(图4)。以T2代突变体水稻DNA为模板的PCR反应中,2个靶点位置均能扩增出特异性条带,靶点1的PCR产物大小为443 bp,靶点2的PCR产物大小为469 bp。对PCR扩增产物进行测序分析表明,共有17种在2个靶点区域都发生突变的纯合突变类型水稻植株,除、、和在第1个靶点、以及在第2个靶点产生单碱基插入突变外,其他突变植株的靶点处均表现为碱基缺失突变。和在第1个靶点缺失24个碱基,在第2个靶点处为单碱基缺失,但2个突变体植株缺失的碱基类别有差异,易引起表达产物中氨基酸残基丢失和移码翻译;突变体则在2个靶点都缺失24个碱基,共形成外显子区域丢失48个碱基,会造成翻译产物缺失16个氨基酸;和突变体在第1靶点缺失含22个差异碱基的小片段,在第2个靶点则分别丢失单个G和T碱基;、、和突变体在第1个靶点分别缺失6、7、3和10个碱基,而在第2个靶点则缺失单个G碱基;、和突变体分别在第1个靶点丢失3、1和6个碱基,在第2个靶点则缺失2、9和2个碱基,会导致氨基酸序列移码变异。测序鉴定结果表明,采用双靶点编辑明显增加了突变类型的多样性,获得了较多碱基片段变异多样化的突变体种质材料。
为检验17种纯合突变体植株中是否含潮霉素筛选基因,利用潮霉素基因特异引物通过PCR扩增突变体基因组DNA(图5),突变体DNA能扩增出1 002 bp的潮霉素基因PCR产物,突变体DNA有较弱的潮霉素扩增产物条带,表明和突变体中有潮霉素基因残留,体内含有转基因载体DNA片段,其他突变体植株DNA均未检测出潮霉素基因残留,植株体内不含转基因载体DNA成分。
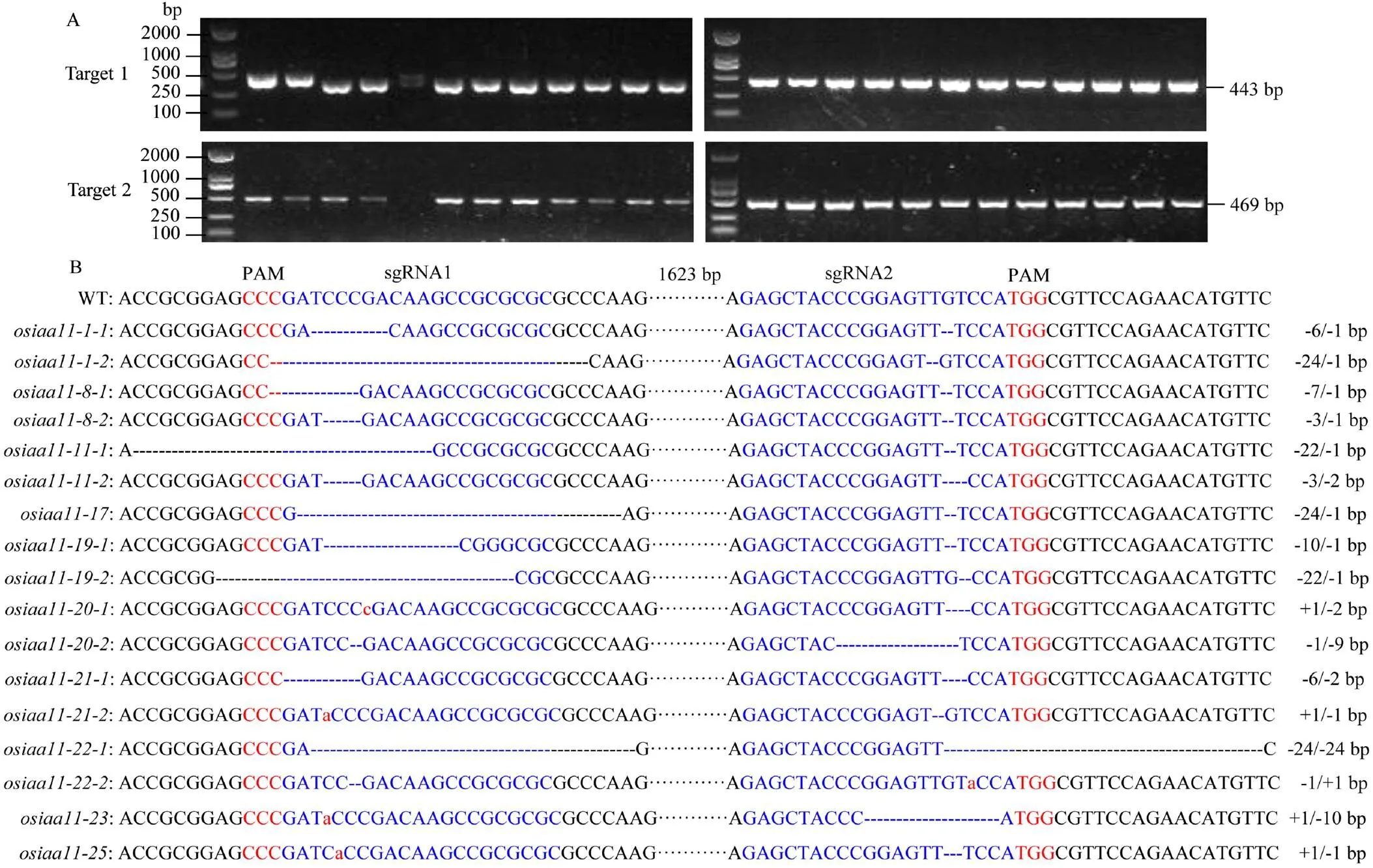
A:T2代部分转基因水稻植株OsIAA11编辑靶点附近DNA片段的PCR检测,靶点1的扩增长度为443 bp,靶点2的扩增长度为469 bp,野生型(WT)为ZH11;B:转化植株编辑靶点的PCR产物测序序列与野生型(WT)序列的比对结果,蓝色字母为靶点序列,红色大写字母表示PAM序列,删除线表示缺失碱基,红色小写字母表示插入碱基,-:缺失,+:插入,WT:野生型
2.4 osiaa11突变体农艺性状
对T2代突变体纯合植株的农艺性状进行调查,在获得的17种不同基因型突变水稻材料中,突变体植株的株高为(60.5±1.50)cm,显著低于野生型(69.6±0.8)cm,其余16种基因型突变水稻的株高与野生型无显著差异;突变体的有效穗数为(12.00±2.00)个,较野生型水稻显著增加,其他突变体的有效穗数与野生型无显著差异。野生型ZH11的分蘖成穗率为89.78%,而突变水稻植株的分蘖成穗率仅为26%—57%,显著低于野生型,表明突变植株的无效分蘖变多,降低了分蘖成穗率,而有效穗数基本维持不变,总分蘖数有所增多。此外,突变植株的穗长、穗粒数、结实率、千粒重、以及收获指数和谷草比等产量指标与野生型植株间的差异没有达到显著水平;在单株产量方面,、、和突变体植株的产量为18.3—22.45 g,比野生型提高70%以上,而其他突变植株的单株产量与野生型无显著差异。

-:阴性对照;+:阳性对照;M:2000 bp DNA marker -: negative control; +: positive control; M: 2000 bp DNA marker
3 讨论
稻米作为全球主要的粮食作物之一,养育着世界上半数以上人口,尤其是进入21世纪以来,伴随着全球人口持续增长、耕地面积和水肥资源逐步减少、极端自然灾害频发、生态环境逐步恶化,粮食短缺问题变得日益严峻[24]。解决当前粮食困境的有效措施主要有两方面:一方面是开发拓展可用耕地面积,例如当前随着中国“一带一路”和中非合作的持续开展,由中资企业提供资金和技术,在非洲地区开垦闲散农田,建立以大米种植为主、多种粮食及经济作物为辅,集农田开发、粮食生产、仓储、加工、销售为一体的综合农业产业园取得了较好的经济、生态、以及社会效益,有效缓解了地区性粮食需求和社会就业,促进区域性经济发展[25];另一方面是在现有耕地面积基础上,突破传统育种手段和栽培措施的技术瓶颈,借助当前生物科技水平,实现精准、高效的分子设计育种和精确定量化栽培,努力提高单位耕地的稻米产量[24,26]。
传统育种技术需要经历多代回交筛选,育种工作量较大,目标性状筛选繁琐,育种周期较长,效率低下,且容易受连锁基因干扰。CRISPR/Cas9编辑技术能精确、高效地编辑目标基因,快速获得目标性状改善并能稳定遗传的纯合突变植株,极大缩短优良性状的遗传改良周期。本研究利用CRISPR/Cas9技术,通过双靶点编辑方式,对进行定点突变,相对于许多研究中单靶点编辑得到较多单一碱基突变的编辑方式[27-28],极大地提高了对的编辑效率,本研究从T2代植株中筛选出了较多短片段缺失基因型突变植株(图4-B),突变种类较为丰富,极大地提高了变异的多样性。此外,鉴于CRISPR/ Cas9编辑植株的T0代转基因株系会产生纯合突变、杂合突变、双等位突变、以及未发生突变等4种基因型植株,纯合突变株系比例相对较少[29],若想获得较多的变异类型,必须对杂合突变植株、双等位突变等转基因植株的后代分离情况进行分析与鉴定,工作量相对较为繁琐,本研究直接对T0代阳性转基因株系的T2后代植株编辑靶点进行测序分析,从T2后代中筛选获得了较为丰富的纯合突变植株,如和、和、和、和、和、和、和等突变植株分别是T0代植株、、、、、、以及的T2后代分离产生的纯合植株,而、和突变植株的T0代株系为纯合突变型,在从T0代到T2代的遗传过程中没有发生基因分离,T2后代植株中未产生其他的突变类型。另外,和突变植株在靶点1区域为单碱基插入突变,在靶点2区域为单碱基缺失突变,碱基突变方式相对简单,而碱基以成片段的缺失突变则多出现在T0代中的杂合突变或双等位突变植株中,从其T2后代中能分离鉴定到纯合的片段碱基缺失突变植株。此外,对从T2代获得的17种纯合突变体植株内的潮霉素抗性基因检测发现,仅有2个突变体内有潮霉素基因残留,表明其含有转基因载体的DNA片段(图5),其余15个纯合突变体则均无载体DNA残留,可以进一步用于与其他常规品种间的杂交育种或生物功能分析。
生长素在水稻生长发育阶段和环境因子响应中发挥重要调节作用,并最终引起稻米产量形成因子改变,本研究对17个纯合突变植株的产量形成指标进行了初步调查,发现突变体植株的分蘖成穗率显著降低,有效穗数与野生型差异不显著,而穗长、穗粒数、千粒重、结实率、收获指数、谷草比等指标变化不明显(表2),表明突变引起了无效分蘖数增多,Zhu等[21]研究点突变的水稻根系时发现点突变能抑制植株侧根原基形成,引起无侧根表型,本研究表明,可能在生长素控制水稻分蘖芽形成和促进侧根发育过程中起作用,突变会导致生长素对侧芽发育的调控作用削弱,并抑制对侧根促进作用,而具体的分子生态调控机制有待进一步深入探索。在水稻栽培实践中,生育前期适当促进分蘖芽多发早发,配合相应的高产栽培措施,可使无效分蘖转化为有效分蘖,提高分蘖成穗率;在分蘖后期,合理减少水肥使用,削弱迟发分蘖的形成,能有效促使叶、茎、蘖等部位的同化物质向稻穗籽粒中转移,有利于提高稻谷的结实率和千粒重,进而达到较高的收获指数和谷草比,因此,本研究获得的突变体和观察到的突变体分蘖芽增多现象,对水稻合理株型的稻作群体构建,有效利用环境温光因子等农业生态调控具有一定的实践意义,也对探索参与的生长素调控分蘖芽发生的分子生态机理研究具有积极的理论意义。
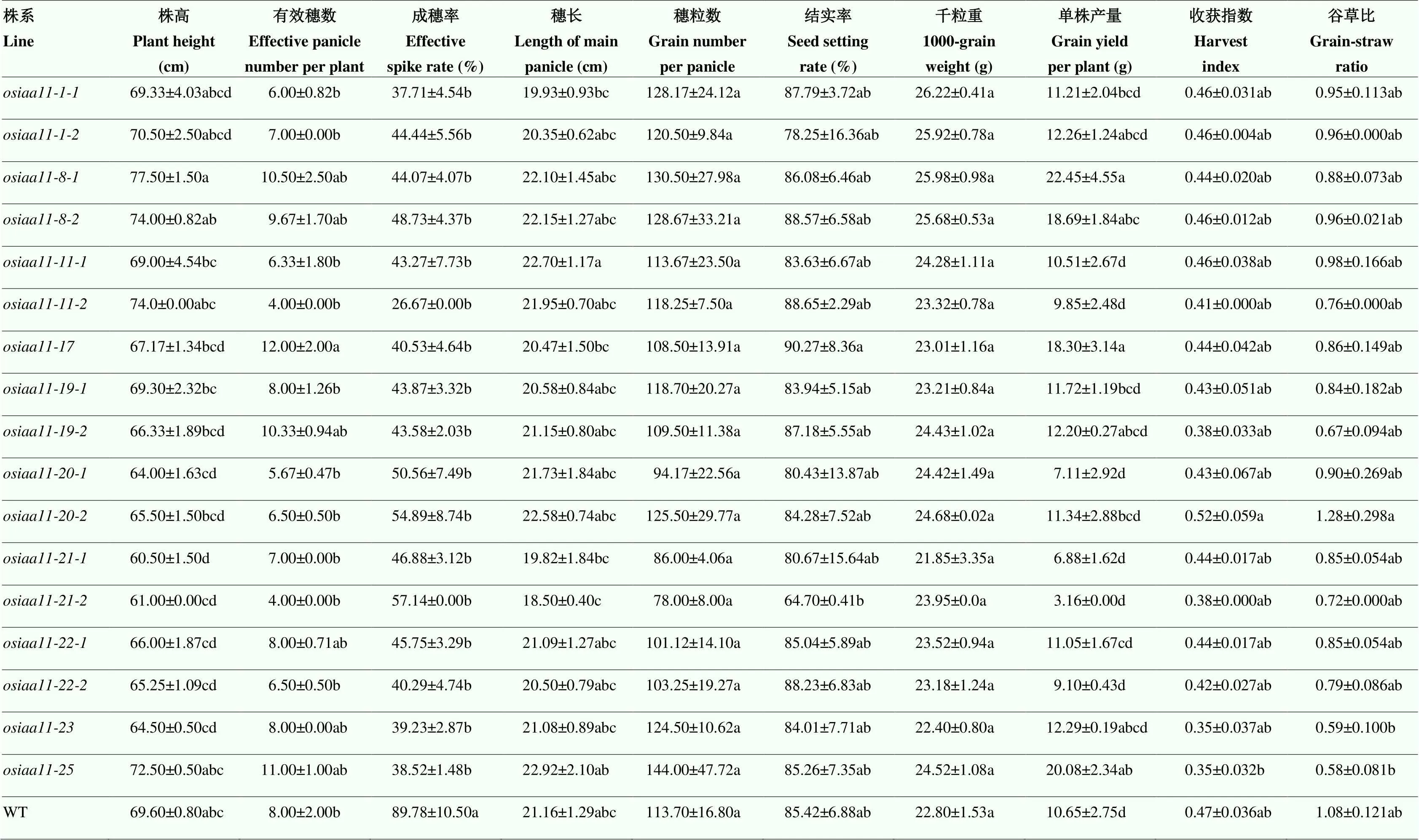
表2 不同类型osiaa11突变体植株及其野生型水稻(WT)的田间农艺性状
表中所列数据为平均值±标准误;同列不同字母表示在0.05水平上差异显著
Data listed in the table are presented as average values ± standard deviation; values followed by the different letter within the same column are significantly different at the 0.05 probability level
4 结论
利用CRISPR/Cas9编辑技术,获得17种不同基因型纯合突变植株。突变体植株的株高、有效穗、穗长、穗粒数、结实率、千粒重、收获指数、以及谷草比等农艺指标与野生型植株无显著差异,但分蘖成穗率显著降低。
[1] HAGEN G, GUILFOYLE T. Auxin-responsive gene expression: genes, promoters and regulatory factors. Plant Molecular Biology, 2002, 49: 373-385.
[2] BELHAJ K, CHAPARRO-GARCIA A, KAMOUN S, PATRON N J, NEKRASOV V. Editing plant genomes with CRISPR/Cas9. Current Opinion in Biotechnology, 2015, 32: 76-84.
[3] CARROLL D, MORTON J J, BEUMER K J, SEGAL D J. Design, construction and in vitro testing of zinc finger nucleases. Nature Protocols, 2006, 1(3): 1329-1341.
[4] LI T, LIU B, SPALDING M H, WEEKS D P, YANG B. High- efficiency TALEN-based gene editing produces disease-resistant rice. Nature Biotechnology, 2012, 30(5): 390-392.
[5] 卢俊南, 褚鑫, 潘燕平, 陈映羲, 温栾, 戴俊彪. 基因编辑技术: 进展与挑战. 中国科学院院刊, 2018, 33(11): 1184-1192.
LU J N CHU X, PAN Y P, CHEN Y X, WEN L, DAI J B. Advances and challenges in gene editing technologies. Bulletin of the Chinese Academy of Sciences, 2018, 33(11): 1184-1192. (in Chinese)
[6] WANG F J, WANG C L, LIU P Q, LEI C L, HAO W, GAO Y, LIU Y G, ZHAO K J. Enhanced rice blast resistance by CRISPR/Cas9- targeted mutagenesis of the ERF transcription factor gene. PLoS ONE, 2016, 11(4): e0154027.
[7] ZHOU H, HE M, LI J, CHEN L, HUANG Z F, ZHENG S Y, ZHU L Y, NI E, JIANG D G, ZHAO B R, ZHUANG C X. Development of commercial Thermo-sensitive genic male sterile rice accelerates hybrid rice breeding using the CRISPR/Cas9-mediatedediting system. Scientific Reports, 2016, 6: 37395-37406.
[8] ZONG Y, WANG Y P, LI C, ZHANG R, CHEN K L, RAN Y D, QIU J L, WANG D W, GAO C X. Precise base editing in rice, wheat and maize with a Cas9-cytidine deaminase fusion. Nature Biotechnology, 2017, 35(5): 438-441.
[9] ZENSER N, ELLSMORE A, LEASURE C, CALLIS J. Auxin modulates the degradation rate of Aux/IAA proteins. Proceedings of the National Academy of Sciences of the USA, 2001, 98(20): 11795-11800.
[10] KEPINSKI S, LEYSER O. Auxin-induced SCFTIR1-Aux/IAAinteraction involves stable modification of the SCFTIR1complex. Proceedings of the National Academy of Sciences of the USA, 2004, 101(33): 12381-12386.
[11] TIWARI S B, HAGEN G, GUILFOYLE T. The roles of auxin response factor domains in auxin-responsive transcription. The Plant Cell, 2003, 15(2): 533-543.
[12] OUELLET F, OVERVOORDE P J, THEOLOGIS A. IAA17/AXR3: biochemical insight into an auxin mutant phenotype. The Plant Cell, 2001, 13: 829-841.
[13] JAIN M, KAUR N, GARG R, THAKUR J K, TYAGI A K, KHURANA J P. Structure and expression analysis of early auxin- responsivegene family in rice (). Functional & Integrative Genomics, 2006, 6(1): 47-59.
[14] SONG Y, WANG L, XIONG L Z. Comprehensive expression profiling analysis ofgene family in developmental processes and in response to phytohormone and stress treatments. Planta, 2009, 229: 577-591.
[15] SONG Y, YOU J, XIONG L. Characterization ofgene, a member of rice Aux/IAA family involved in auxin and brassinosteroid hormone responses and plant morphogenesis. Plant Molecular Biology, 2009, 70(3): 297-309.
[16] NAKAMURA A, UMEMURA I, GOMI K, HASEGAWA Y, KITANO H, SAZUKA T, MATSUOKA M. Production and characterization of auxin-insensitive rice by overexpression of a mutagenized rice IAA protein. The Plant Journal, 2006, 46(2): 297-306.
[17] JUNG H, LEE D K, CHOI Y D, KIM J K., a member of the ricegene family, is involved in drought tolerance and tiller outgrowth. Plant Science, 2015, 236: 304-312.
[18] KITOMI Y, INAHASHI H, TAKEHISA H, SATO Y, INUKAI Y.-mediated auxin signaling is involved in lateral root initiation in rice. Plant Science, 2012, 190: 116-122.
[19] JIN L, QIN Q, WANG Y, PU Y, LIU L, WEN X, JI S, WU J, WEI C, DING B, LI Y. Rice dwarf virus P2 protein hijacks auxin signaling by directly targeting the rice OsIAA10 protein, enhancing viral infection and disease development.PLoS Pathogens, 2016, 12(9): e1005847.
[20] NI J, WANG G, ZHU Z, ZHANG H, WU Y, WU P. OsIAA23- mediated auxin signaling defines postembryonic maintenance of QC in rice. The Plant Journal, 2011, 68(3): 433-442.
[21] ZHU Z X, LIU Y, LIU S J, MAO C Z, WU Y R, WU P. A gain-of- function mutation inaffects lateral root development in rice. Molecular Plant, 2012, 5(1): 154-161.
[22] LI Z, PAN X, GUO X, FAN K, LIN W. Physiological and transcriptome analyses of early leaf senescence formutant rice (L.) during the grain-filling stage. International Journal of Molecular Sciences, 2019, 20: 1098.
[23] MA X, ZHANG Q, ZHU Q, LIU W, CHEN Y, QIU R, WANG B, YANG Z, LI H, LIU Y, XIE Y, SHEN R, CHEN S, WANG Z, CHEN Y, GUO J, CHEN L, ZHAO X, DONG Z, LIU Y G. A robust CRISPR/ Cas9 system for convenient, high-efficiency multiplex genome editing in monocot and dicot plants. Molecular Plant, 2015, 8(8): 1274-1284.
[24] 郭韬, 余泓, 邱杰, 李家洋, 韩斌, 林鸿宣. 中国水稻遗传学研究进展与分子设计育种. 中国科学: 生命科学, 2019, 49(10): 1185-1212.
GUO T, YU H, QIE J, LI J Y, HAN B, LIN H X. Advances in rice genetics and breeding by molecular design in China. Scientia Sinica Vitae, 2019, 49(10): 1185-1212. (in Chinese)
[25] 宫景文, 刘文超. 2030年非洲粮食问题预测及对中国的影响. 国土资源情报, 2017, 8: 31-38.
GONG J W, LIU W C. Forecast of Africa’s food problems in 2030 and its impacts on China. Land and Resources Information, 2017, 8: 31-38. (in Chinese)
[26] 凌启鸿, 张洪程, 丁艳锋, 张益彬. 水稻高产技术的新发展—精确定量栽培. 中国稻米, 2005, 1: 3-7.
LING Q H, ZHANG H C, DING Y F, ZHANG Y B. New development of rice high-yield technology-precise and quantitative cultivation. China Rice, 2005, 1: 3-7. (in Chinese)
[27] 黄忠明, 周延彪, 唐晓丹, 赵新辉, 周在为, 符星学, 王凯, 史江伟, 李艳锋, 符辰建, 杨远柱. 基于CRISPR/Cas9技术的水稻温敏不育基因突变体的构建. 作物学报, 2018, 44(6): 844-851.
HUANG Z M, ZHOU Y B, TANG X D, ZHAO X H, ZHOU Z W, FU X X, WANG K, SHI J W, LI Y F, FU C J, YANG Y Z. Construction of tms5 mutants in rice based on CRISPR/Cas9 technology. Acta Agronomica Sinica, 2018, 44(6): 844-851. (in Chinese)
[28] 季新, 李飞, 晏云, 孙红正, 张静, 李俊周, 彭廷, 杜彦修, 赵全志. 基于CRISPR/Cas9系统的水稻光敏色素互作因子基因编辑. 中国农业科学, 2017, 50(15): 2861-2871.
JI X, LI F, YAN Y, SUN H Z, ZHANG J, LI J Z, PENG T, DU Y X, ZHAO Q Z. CRISPR/Cas9 system-based editing of phytochrome- interacting factor. Scientia Agricultura Sinica, 2017, 50(15): 2861-2871. (in Chinese)
[29] 盛夏冰, 谭炎宁, 孙志忠, 余东, 汪雪峰, 袁贵龙, 袁定阳, 段美娟. 利用CRISPR/Cas9基因组编辑技术定向降低水稻落粒性. 中国农业科学, 2018, 51(14): 2631-2641.
SHENG X B, TAN Y N, SUN Z Z, YU D, WANG X F, YUAN G L, YUAN D Y, DUAN M J. Using CRISPR/Cas9-mediated targeted mutagenesis ofreduces the seed shattering in rice. Scientia Agricultura Sinica, 2018, 51(14): 2631-2641. (in Chinese)
CRISPR/Cas9 Targeted Editing of
LI ZhaoWei1, LING DongLan1, SUN CongYing1, ZENG HuiLing1, LIU KaiJi1, LAN YingShan1, FAN Kai2, LIN WenXiong1
1College of Life Sciences, Fujian Agriculture and Forestry University, Fuzhou 350002;2College of Agriculture, Fujian Agriculture and Forestry University, Fuzhou 350002
【】Auxin signaling pathway regulated byplays an important role in the plant growth and response to various environment stresses, impacting on the final grain yield during the late stage. To explore the effect ofmutation on the yield, themutant plant was obtained using CRISPR/Cas9 system, and the agronomic characters of mutant plants were investigated in the field. 【】According to the principle of CRISPR/Cas9 technology, two 20 bp targeted sequences were designed in the first exon and second exon of thegenome sequence. The gene shuffling of non-specific target sites was eliminated using the blast analysis. The oligonucleotides of two target sites were inserted into the pYLgRNA-U6a and pYLgRNA-U6b plasmid vector, and then were amplified twice by PCR technology to construct U6a-IAA11-T1 and U6b-IAA11-T2 expression-boxes. The recombinant pYLCRISPR/Cas9-IAA11-T12 vector was obtained by linking two expression-boxes to pYLCRISPR/Cas9-MT vector. The pYLCRISPR/Cas9-IAA11-T12 vector was transformed into the callus ofrice ZH11 through-mediated method. The T0generation of transgenic plants was obtained by tissue culture, and the positive transgenic plants were selected through PCR method. The target sites of each T2generation from T0positive plants were determined via PCR and sequencing method, and finally the mutated genotypes were identified. Meanwhile, agronomic traits of the T2generation were investigated in the field. 【】The pYLCRISPR/Cas9-IAA11-T12 expression vector was successfully transformed into the callus of ZH11. 25 transgenic lines were obtained, and 20 transgenic lines were identified as the positive plants. After analysis of T2transgenic plants via PCR and sequencing the target locus, 17 different homozygous mutations were identified in the two target sites of thegenomic sequence. Except for the single base insertion mutation of,,, andmutants did not present a significant difference in plant height, panicle length, grain number per panicle, seed setting rate, 1000-grain weight, harvest index, and grain-straw ratio in comparison with the wild type. However, thedisplayed a significant decrease in effective spike rate, suggesting a more ineffective tiller phenotype. 【】CRISPR/Cas9 technology successfully editedand 17 different homozygous mutations genotypes were obtained. The mutant showed the reduced effective spike rate and increased ineffective tillers, indicating thatwas related to the tiller bud appearance dominated by auxin.
rice; gene editing;; CRISPR/Cas9 technology;mutant

10.3864/j.issn.0578-1752.2021.13.001
2020-12-03;
2021-01-21
国家自然科学基金(31701329)、中国博士后科学基金(2015M580560)
李兆伟,E-mail:lizw197@163.com。通信作者林文雄,E-mail:wenxiong181@163.com
(责任编辑 李莉)

