基于支持向量机对云南常见野生食用牛肝菌中红外光谱的种类鉴别
胡翼然,李杰庆,刘鸿高,范茂攀,*,王元忠
(1.云南农业大学资源与环境学院,云南 昆明 650201;2.云南农业大学农学与生物技术学院,云南 昆明 650201;3.云南省农业科学院药用植物研究所,云南 昆明 650200)
牛肝菌(Boletaceae)是一种大型真菌,富含多糖、蛋白质、氨基酸、维生素、矿物质、膳食纤维等营养物质[1],且有抗癌、抗氧化、抗肿瘤、抗疲劳等治疗功效[2],受广大消费者的喜爱。云南省地处低纬,海拔高差大、干湿季分明、光热资源充足,为野生牛肝菌生长发育提供了适宜的生长环境[3]。云南省野生牛肝菌种类丰富,已知种类约有244 种,可食用种类占59%(144 种)[4]。在可食用野生牛肝菌中,部分野生牛肝菌具有毒性,如食用加工方法不当,易导致食物中毒[5],如华丽牛肝菌[6]、小美牛肝菌[7]、绒柄牛肝菌[6]等。新鲜野生牛肝菌外貌相似,即使是有经验的专家学者,也很难快速、准确识别其种类,同时野生食用菌不易保存,常被制成干片销售,进一步增加了辨识难度。目前,国内外野生牛肝菌混杂现象屡屡发生[8-9],严重威胁消费者的身体健康,急需一种准确、快速、廉价的常见野生食用牛肝菌种类鉴别技术。
传统鉴别方法如对形态特征法、动物检验法等,此类方法操作简单、准确性低、主观因素影响大,鉴别能力不足[10]。传统化学检测方法如紫外光谱法[11]、高效液相色谱法[12]、电感耦合等离子体-原子发射光谱法[13]、电子鼻技术[14]等,上述方法耗时、费用昂贵、操作难度大,不能实现快速、廉价的种类鉴别。傅里叶变换中红外光谱法具有操作简单、速度快捷、成本低廉的特点,近年来,化学指纹图谱结合数理统计学广泛应用于种类鉴别研究[15-17]。
虽然傅里叶变换中红外光谱表征样品的化学信息全面,但其中存在大量干扰信息和无效信息,反而会导致模型分类性能下降,故研究不同信息挖掘方法对野生牛肝菌种类鉴别具有重要意义。目前,对牛肝菌光谱信息挖掘主要集中在不同预处理方法对模型分类效果的影响[18-19]。因此,本实验采用预处理及不同特征变量提取方法(主成分、变量重要性、变量投影重要性),挖掘中红外光谱有效信息,结合支持向量机(support vector machine,SVM)建立判别模型,比较模型分类效果,探索野生牛肝菌种类鉴别方法,为野生食用菌鉴别和质量控制提供参考依据。
1 材料与方法
1.1 材料与试剂
8 种827 个云南常见野生牛肝菌样本:于2011—2014年采买于云南省,经云南农业大学刘鸿高教授鉴定为华丽牛肝菌、灰褐牛肝菌、栗色牛肝菌、美味牛肝菌、绒柄牛肝菌、双色牛肝菌、小美牛肝菌和皱盖疣柄牛肝菌(图1、表1)。
溴化钾(分析纯) 天津风船化工科技有限公司。

图1 牛肝菌样品图Fig. 1 Pictures of boletus samples
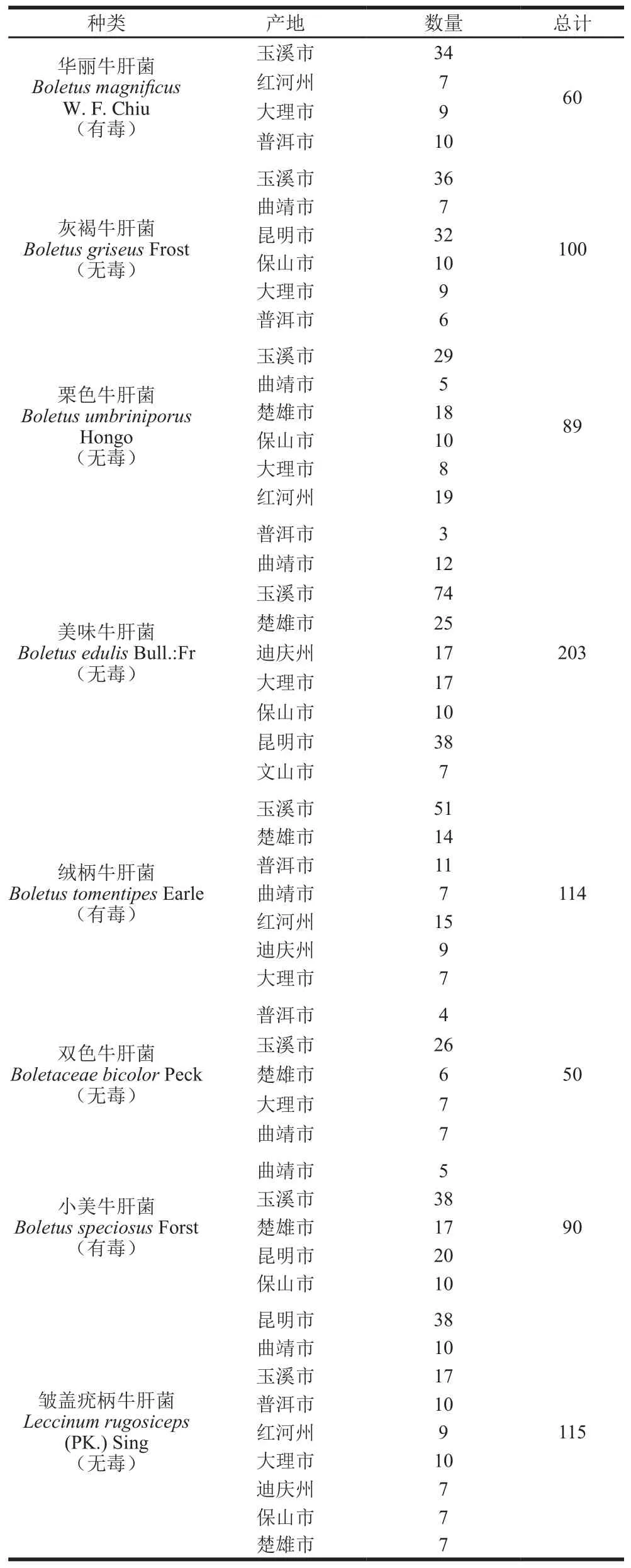
表1 牛肝菌种类信息Table 1 Information about boletus samples tested in this study
1.2 仪器与设备
Frontier傅里叶变换中红外光谱仪 美国Perkin Elmer公司;FW-100型高速粉碎机 浙江华鑫仪器厂;YP-2型压片机 上海山岳科学仪器有限公司;80 目标准筛盘 浙江绍兴市道墟五四仪器厂;AR1140型电子分析天平 上海升隆电子科技有限公司。
1.3 方法
1.3.1 样品前处理
样品前处理模拟干片的制备,样品采集后去除土样等杂质,用纯净水清洗干净,置于50 ℃烘箱烘干至质量恒定,研磨成粉过80 目标准筛盘,分别储存于自封袋中,保存于避光处。
1.3.2 傅里叶变换中红外光谱采集
取(1.5±0.2)mg野生牛肝菌样品和(150±20)mg KBr粉末在研钵中磨细混匀,再将细粉倒入磨具中压成薄片。先扫描溴化钾空白片作为基线,再扫描各样品片。扫描波数范围4 000~400 cm-1,分辨率4 cm-1,信号累计扫描次数16 次,每个样本重复扫描3 次,取平均光谱。
1.3.3 SVM算法
SVM是一种基于非线性映射的二分类监督学习方法,利用核函数将向量投影到高维特征空间,建立最优超平面,对样本分类。本研究在Matlab中使用LIBSVM包构建SVM,核函数为径向基函数,利用网格搜索算法(grid search,GS)寻找最优参数组合惩罚因子(C)和核参数(g),交叉验证次数k设置为10;惩罚参数步进距离、核参数步进距离、步进间隔距离均设置为0.5。该算法是基于一对多全分区的二叉树支持向量机,算法原理如图2所示,该算法分类速度和正确率高。
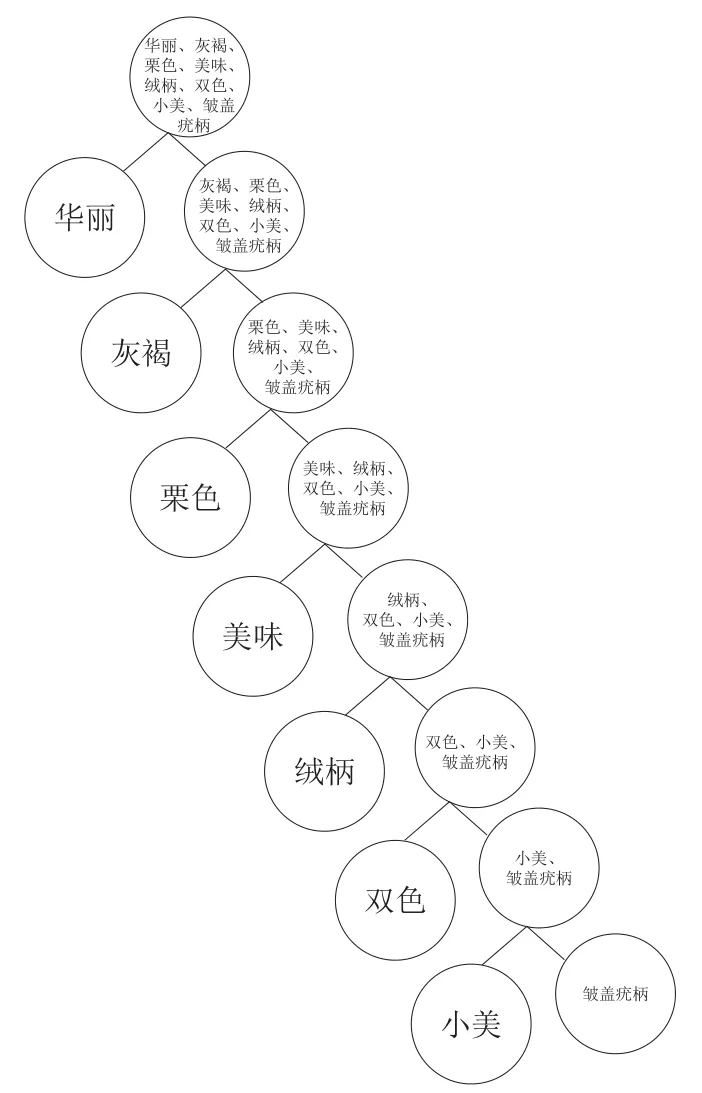
图2 基于字母顺序排列的SVM流程图Fig. 2 Flow chart of SVM classification in the alphabetical order
C是惩罚因子,代表SVM算法对异常点的重视程度,影响模型的分类精度。C越大训练集误差越小,C过大容易导致过拟合;C越小训练集误差越大,C过小容易欠拟合;C过大或过小都会导致模型泛化能力减弱。g是核函数半径,代表数据映射到高维特征空间后的分布,影响模型的分类训练速度。g越大,支持向量越少,速度越快;g越小,支持向量越多,速度越慢。利用GS寻找最优参数C和g,GS是穷举搜索,建立一个三维坐标系,将待搜索参数C和g依次排列于X轴(log2C)和Y轴(log2g),形成带网格的坐标系,坐标系上每一个点代表一种参数组合,将不同参数组合的交叉验证精确度依次排列于Z轴,最终选取交叉验证精度最高的(C、g)组合[20]。当C的范围在[2-2, 24]且g的范围在[2-4, 24]之间,模型拟合度高。
1.4 数据挖掘方法
1.4.1 预处理方法
中红外光谱数据中重叠的谱带和各种噪声信号,会降低模型的识别能力。对光谱数据进行预处理可以减少干扰信息和噪音同时提高模型的适应性,增强模型的预测能力。本实验采用多元散射校正(multiplicative scatter correction,MSC)结合多项式平滑法对光谱数据进行预处理。MSC能有效去除红外光谱中散射和粒径的乘性干扰[21]。多项式平滑法是SG平滑与导数处理的结合,可以消除光谱中的高频噪音及位移变化,通过平滑点数、导数阶数、多项式次数3 个参数调节。平滑能够衰减噪声信号,保持光谱数据特征信息。导数处理可以消除基线漂移,加强光谱特征,但也会放大噪音信号。本实验的平滑点数为15,导数阶数为1(1D),多项式次数为2。
为避免样本之间的量纲影响,利用归一化把数据映射在[-1, 1]之间,从而加快模型收敛速度,增强模型精度[22]。利用Kennard-Stone(K-S)算法将数据集(827)分为70%的训练集(549)和30%的测试集(278),K-S算法通过计算样品间欧氏距离,对样本从远到近依次排序,从而保证选取样本代表性强同时避免随机选择的不可重复性[23]。
1.4.2 提取特征变量方法
主成分(principal component,PC)通过偏最小二乘回归算法将数据降维成互不线性相关的多组PC,特征值代表PC能解释原始数据的方差大小,特征值越大能解释的方差越多。提取特征值大于1的PC为特征变量[24]。
特征重要性(feature importance,FI),通过随机森林算法得到各变量的FI,根据FI依次排列变量,用10折交叉验证对波长点进行迭代筛选,提取有效变量作为特征变量[25]。
变量投影重要性指标(variable importance in projection,VIP)值表示自变量对模型拟合程度的重要性,VIP值越高波长点对标签的解释能力越强[26],提取VIP大于1的波长点为特征变量。
2 结果与分析
2.1 中红外光谱分析
从图3a可以看出,原始光谱(raw extracted spectra,RAW)中有6 处明显的光谱峰。3 200~3 600 cm-1之间是羟基伸缩振动区,3 386 cm-1附近是O—H和N—H的伸缩振动。2 932 cm-1与N—H、CH2和CH3的伸缩振动有关。1 645 cm-1是酰胺I带和的C=O伸缩振动,可能与蛋白质有关。1 645 cm-1附近可能是亚硝基的N=O伸缩振动和酰胺II带C—N、N—H的伸缩振动,可能与蛋白质和壳聚糖有关。1 411 cm-1附近是C—H和O—H的弯曲振动有关,可能与多糖有关。在900~1 200 cm-1附近之间是多糖区域,1 080 cm-1与醚、酯等含氧化合物的C—O伸缩振动有关,可能与β-葡聚糖有关;1 022 cm-1附近与醇、酚的C—O伸缩振动有关。900 cm-1以下是指纹图谱区,可能与多糖结构有关[27-28]。图2b为8 种野生牛肝菌经MSC+SG+1D处理后的中红外平均光谱图,有18 处明显的光谱峰,集中在3 569、3 348、3 213、3 102、2 952、2 891、1 682、1 562、1 476、1 336、1 269、1 177、1 095、1 042、902、816、734、628 cm-1附近。可能原因是经MSC+SG+1D预处理后,中红外图谱的乘性干扰、噪音及无效信息被大幅去除,光谱特征被加强。
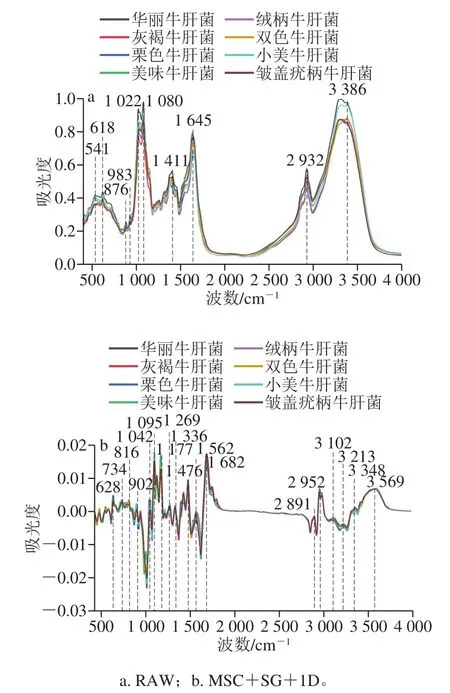
图3 8 种牛肝菌平均光谱图Fig. 3 Average mid-infrared spectra of Boletus samples from eight species
比较图3中8 种野生牛肝菌的中红外平均光谱图。8 种野生牛肝菌均有相似的峰形、峰数、峰位,表明这8 种野生牛肝菌有相似的化学成分。华丽牛肝菌和小美牛肝菌相较于其他牛肝菌吸光度更高,其余6 种野生牛肝菌的吸光度有微小差异,表明这8 种野生牛肝菌的化学成分含量不同。利用中红外光谱图对8 种野生牛肝菌进行可视化分析,可以看出不同种类野生牛肝菌存在差异,因此需进一步结合数理统计学鉴别种类。
2.2 预处理对模型分类效果的影响
RAW模型是由827 个样品×1 867 个变量组成的数据矩阵,如图4a1所示,基于GS选出原始中红外光谱最优参数组合(C=2.62×105,g=3.81×10-6),分类结果如图4a2所示,有3 个样品分类错误,其中1 个栗色牛肝菌被分类为美味牛肝菌,2 个双色牛肝菌分别被分类为栗色牛肝菌和美味牛肝菌。原始数据经MSC+SG+1D处理后,形成由827 个样品×1 839 个变量组成的数据矩阵,如图4b1所示,基于GS选出最优参数组合(C=90.5,g=3.45×10-4),分类结果如图4b2所示,有1 个分类错误,其中1 个栗色牛肝菌被分类为小美牛肝菌。研究表明,原始数据结合SVM建立判别模型,CRAW大于24,gRAW小于2-4,模型过拟合风险大,经预处理后去建立模型,CMSC+SG+1D小于CRAW,gMSC+SG+1D小于gRAW,模型拟合能力增加,但CMSC+SG+1D大于24,gMSC+SG+1D小于2-4,过拟合风险依然大。根据模型的混淆矩阵,可以计算出灵敏度、特异性参数(表2),从保护消费者身体健康的角度,不允许有毒牛肝菌错分类为无毒牛肝菌,即鉴别野生牛肝菌种类要有高灵敏度,本实验根据灵敏度判断模型分类性能,灵敏度越高模型分类性能越高。
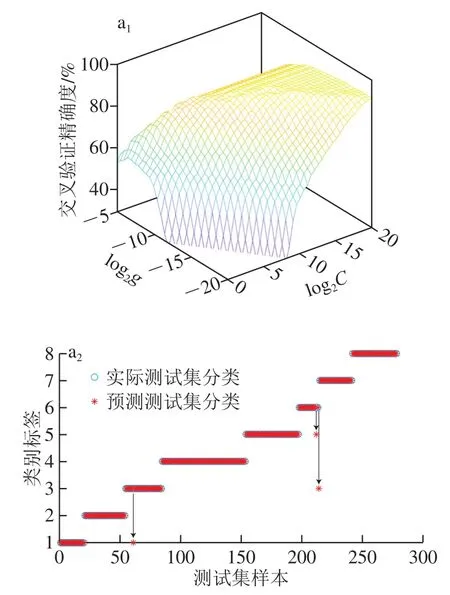
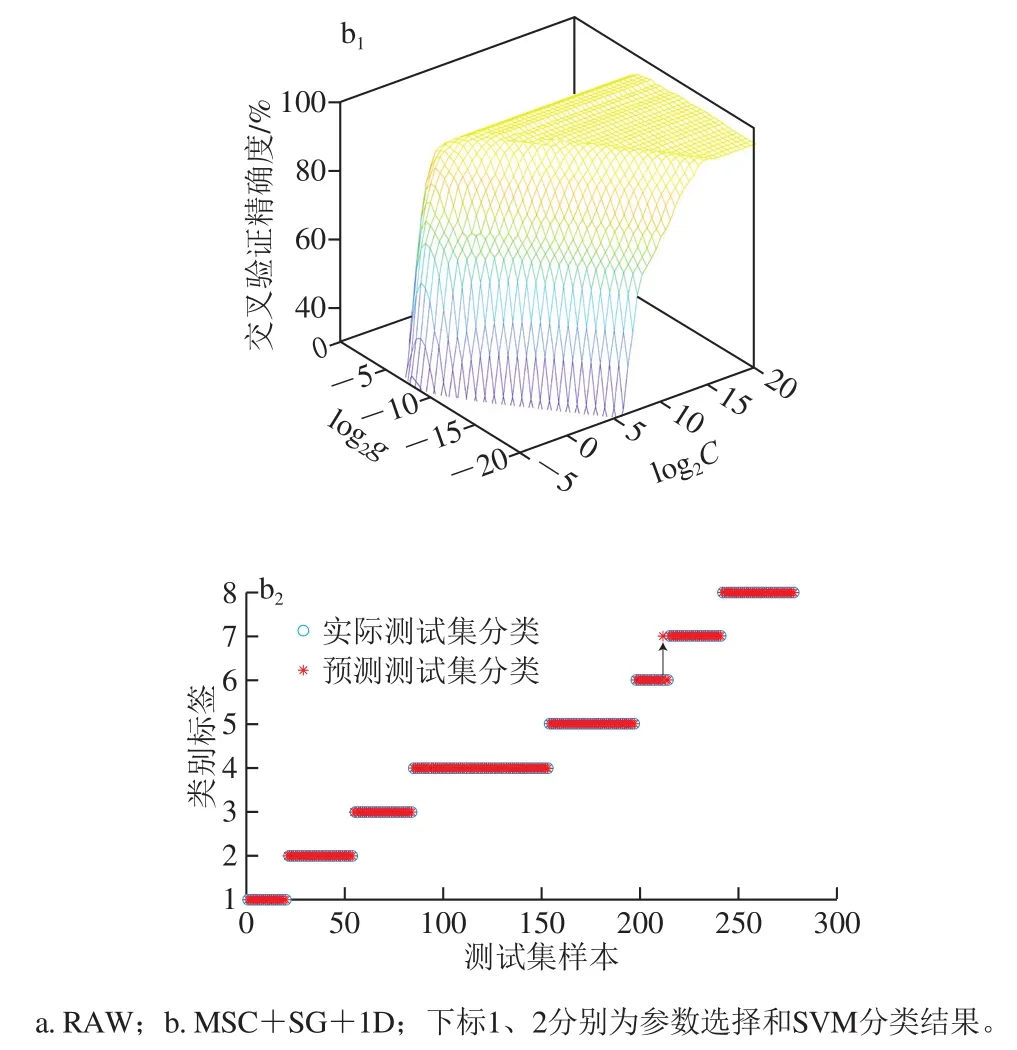
图4 参数选择图和SVM分类结果Fig. 4 Parameter selection and SVM classification results
预处理后的光谱效果较好,评价SVM模型性能的测试集分类结果如表2所示。与RAW相比,基于MSC+SG+1D预处理后光谱建立的模型相比,模型过拟合风险降低,分类精度更加准确,研究结果证实了粉末样品存在噪音和干扰因子,影响模型的分类性能。该预处理方法可去除干扰因子及增强光谱特征,但模型拟合度不理想,需进一步深入挖掘光谱信息。

表2 不同数据集SVM模型参数值Table 2 Parameters of SVM models established by different data mining methods
2.3 不同特征变量对模型分类效果的影响
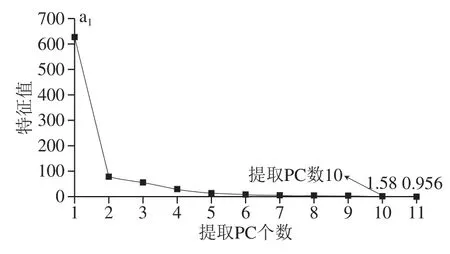
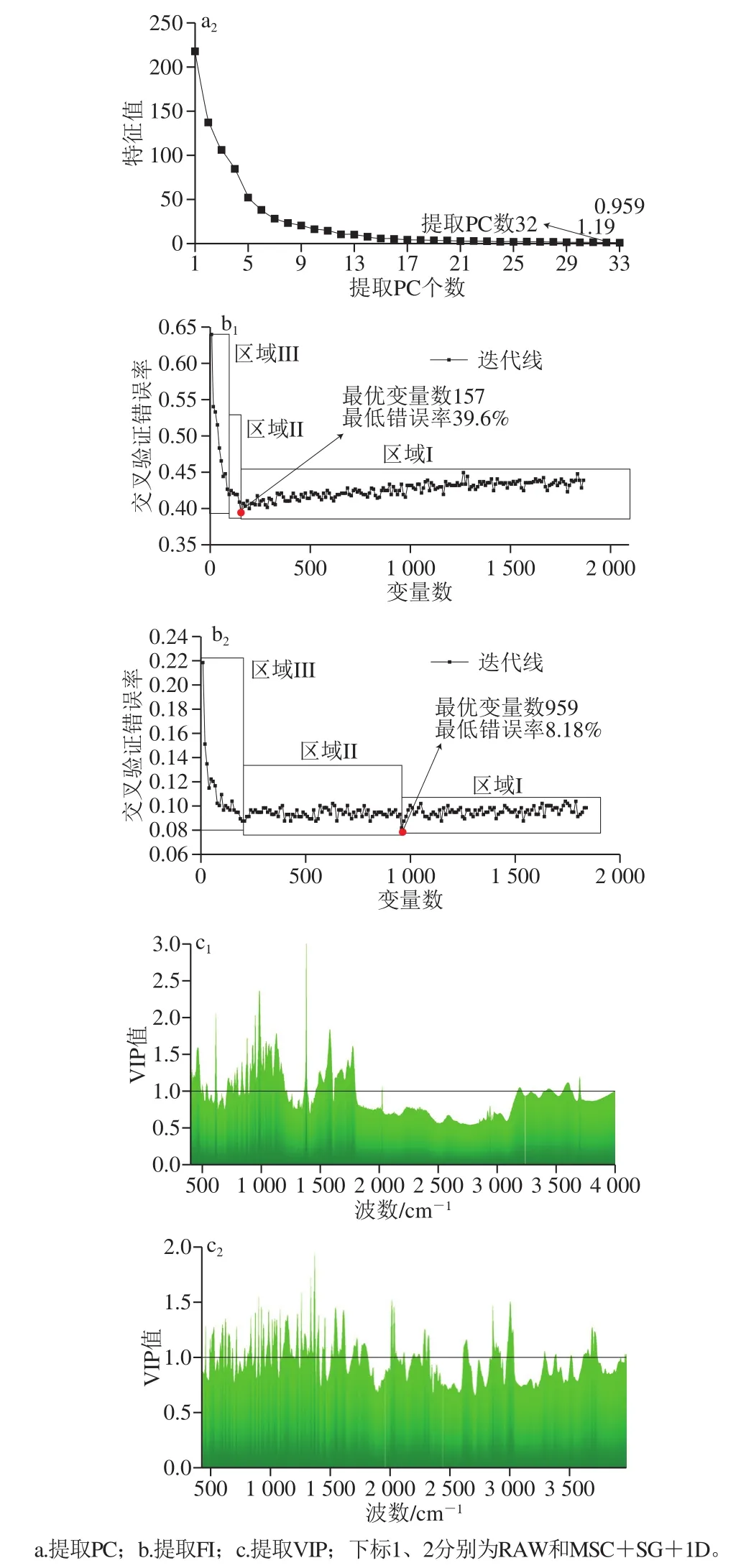
图5 特征变量提取方法Fig. 5 Comparison of feature variable extraction methods
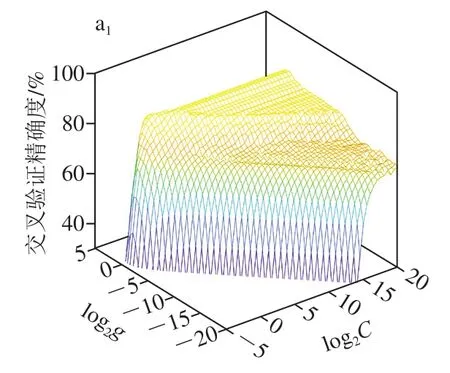

图6 参数选择图和SVM分类结果图Fig. 6 Parameter selection and SVM classification results
RAW-PC模型是由827 个样品×10 个变量形成的数据矩阵,如图5a1所示,挖掘原始数据信息,总的提取了628 个PC,前10 个PC大于1,筛选前10 个PC为特征变量,如图6a1所示,基于GS选出最优参数组合(C=2,g=0.176),图6a2有15 个样品分类错误,其中2 个华丽牛肝菌分别被错误分类到灰褐牛肝菌和小美牛肝菌,3 个灰褐牛肝菌被错误分类为绒柄牛肝菌,2 个绒柄牛肝菌被错误分类为栗色牛肝菌,3 个双色牛肝菌被错误分类为1 个栗色牛肝菌和2 个美味牛肝菌,1 个小美牛肝菌被错误分类为华丽牛肝菌,1 个皱盖疣柄牛肝菌被错误分类为美味牛肝菌。RAW-PC模型拟合程度高,分类性能弱,可用于野生牛肝菌种类鉴别。
RAW-FI模型是由827 个样品×157 个变量形成的数据矩阵,如图5b1所示,挖掘原始数据信息,区域I 1 867~157,错误率不断降低,代表负面信息被去除;区域II 157~97,错误率在同一水平上下浮动,代表无效信息被去除;区域III 157~97,错误率大幅上升,代表有效信息被去除;提取特征重要性最高的前157 个变量建立模型。如图6b1所示,基于GS选出最优参数组合(C=9.27×104,g=3.45×10-4)分类结果如图6b2所示,有7 个分类错误,其中1 个华丽牛肝菌被错误分类为灰褐牛肝菌,3 个灰褐牛肝菌被错误分类为2 个栗色牛肝菌和1 个双色牛肝菌,2 个栗色牛肝菌被错误分类为1 个灰褐牛肝菌和1 个绒柄牛肝菌,1 个双色牛肝菌被错误分类为栗色牛肝菌。RAW-FI模型的拟合程度低,过拟合风险大,不适结合SVM用于野生牛肝菌种类鉴别。
RAW-VIP模型是由827 个样品×641 个变量形成的数据矩阵,如图5c1所示,挖掘原始数据信息,按VIP排列,前641 个VIP大于1,筛选前641 个VIP为特征变量,如图6c1所示,基于GS选出最优参数组合(C=4.63×104,g=6.1×10-5),图6c2有5 个样品分类错误,其中1 个华丽牛肝菌分别被错误分类到灰褐牛肝菌,1 个栗色牛肝菌被错误分类为华丽牛肝菌,3 个双色牛肝菌被错误分类为2 个栗色牛肝菌和1 个绒柄牛肝菌。RAW-VIP模型拟合程度低,分类性能弱,不适结合SVM用于野生牛肝菌种类鉴别。
提取特征变量挖掘光谱信息后模型拟合度变好(表2)。与RAW相比,基于提取特征变量建立光谱的模型与RAW光谱建立的模型相比,模型过拟合风险均降低,分类精度均更加准确,研究结果证实了光谱信息中存在大量无效信息,混淆了算法对有效特征的识别,减弱了模型的分类性能。实验中3 种提取特征变量方法均不同程度去除了非有效信息,增加了模型拟合度,但效果不理想。模型RAW-FI、RAW-VIP过拟合风险大,原因是这两种方法基于波长点挖掘数据,可能将噪音等负面信息误判为特征变量;模型RAW-FI、RAW-VIP过拟合风险小,原因是该方法基于波长整体挖掘数据,可能减弱了负面信息和有效信息的影响,导致相较于其他模型拟合度高,分类性能弱。需进一步深入挖掘光谱信息。
2.4 预处理组合不同特征变量对模型分类效果的影响
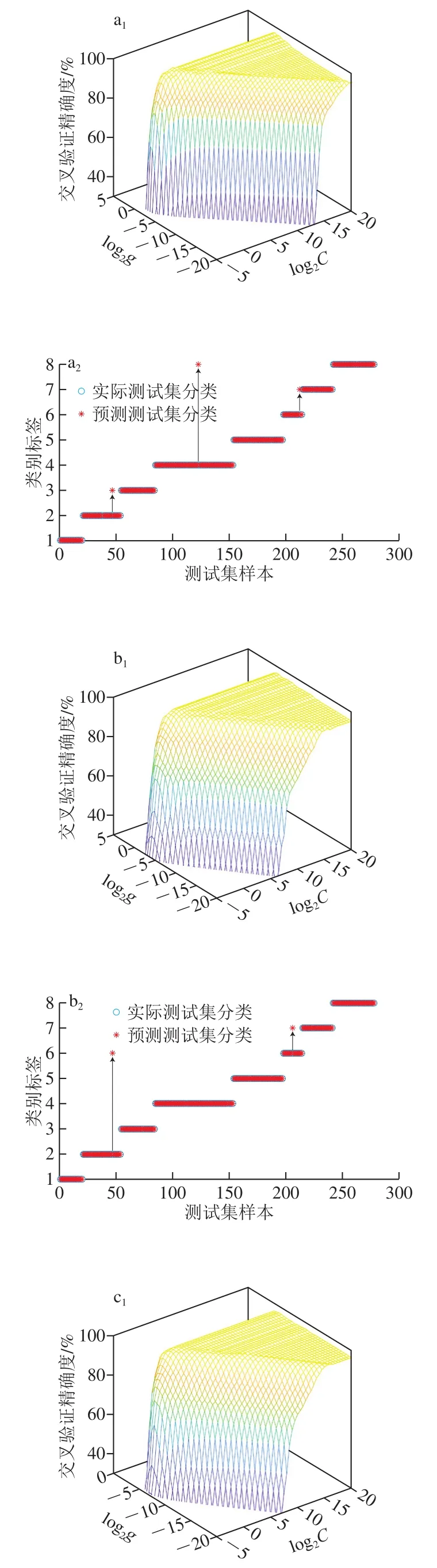
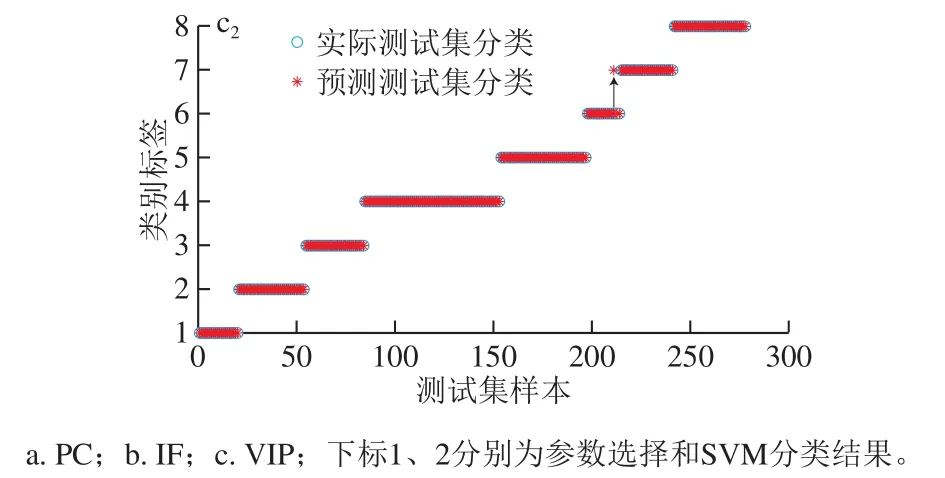
图7 参数选择图和SVM分类结果图Fig. 7 Parameter selection and SVM classification results
MSC+SG+1D-PC模型是由827 个样品×39 个变量形成的数据矩阵,如图7a1所示,基于GS选出最优参数组合(C=2.83,g=0.354)。如图5a2所示,预处理与PC组合挖掘数据信息,总的提取了137 个PC,提取前32 个PC为特征变量。分类结果如图7a2所示,有3 个分类错误,其中1 个灰褐牛肝菌被错误分类为栗色牛肝菌,1 个美味牛肝菌被错误分类为皱盖疣柄牛肝菌,1 个双色牛肝菌被错误分类为小美牛肝菌。MSC+SG+1D-PC模型的拟合程度高,分类性能强,可用于野生牛肝菌种类鉴别。
MSC+SG+1D-FI模型是由827 个样品×959 个变量形成的数据矩阵,如图7b1所示,基于GS选出最优参数组合(C=4,g=1.56×10-2),如图5b2所示,预处理与FI组合挖掘数据信息,提取FI最高的前959 个变量是最优变量。分类结果如图7b2所示,有2 个分类错误,其中1 个灰褐牛肝菌被错误分类为双色牛肝菌,1 个双色牛肝菌被错误分类为小美牛肝菌。MSC+SG+1D-FI模型的拟合程度高,分类性能强,可用于野生牛肝菌种类鉴别。
MSC+SG+1D-VIP模型是由827 个样品×780 个变量形成的数据矩阵,如图7c1所示,基于GS选出最优参数组合(C=256,g=2.44×10-4),如图5c2所示,预处理与VIP组合挖掘数据信息,提取VIP大于1的前780 个变量。分类结果如图7c2所示,有1 个分类错误,1 个双色牛肝菌被错误分类为小美牛肝菌。MSC+SG+1D-FI模型的拟合程度相较于RAW-VIP有了显著提高,但不适结合SVM用于野生牛肝菌种类鉴别。
预处理组合特征变量方法挖掘数据后的分类效果较好(表2)。与RAW、MSC+SG+1D、RAW-PC、RAWFI、RAW-VIP相比,基于预处理组合特征变量方法建立光谱的模型,模型拟合度高、分类性能好,研究结果证实了预处理与特征变量组合方法可以大幅减少非有效信息,增强有效信息,起协同作用达到准确鉴别的目的。
2.5 建模方法对比分析
为验证数据挖掘方法的可靠性和泛用性,相同条件结合偏最小二乘判别算法(partial least squaresdiscrimination algorithm,PLS-DA)建立判别模型。如表3所示,各PLS-DA模型交叉验证均方根误差(root mean square error of cross validation,RMSECV)大于预测均方根误差(root mean square error of prediction,RMSEP)且数值均较小,表明模型拟合良好[29]。特征变量FI与预处理组合的方法,模型拟合度高,分类性能最优。
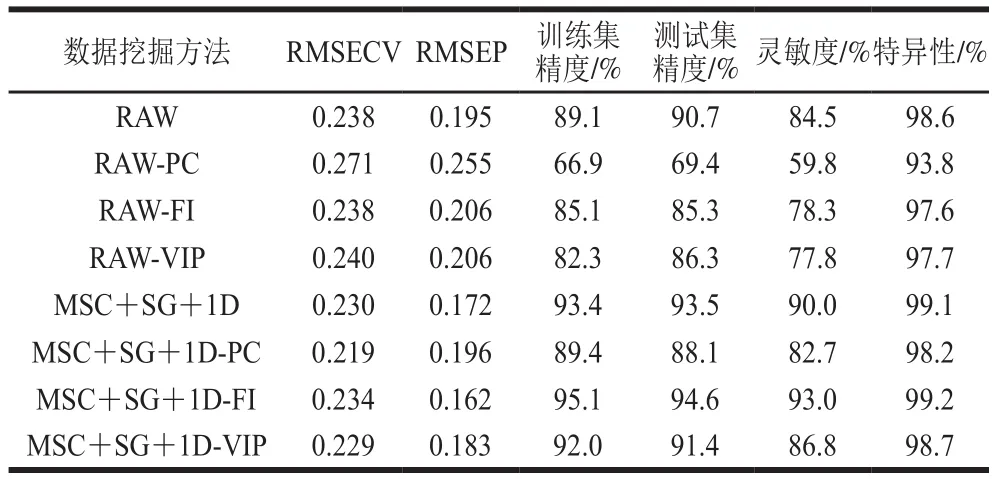
表3 不同数据集PLS-DA模型参数值Table 3 Parameters of PLS-DA models established by different data mining methods
对比表2和表3分析不同算法的模型参数。PLS-DA模型灵敏度在59.8%~93.0%之间,SVM模型灵敏度在92.2%~99.3%之间,研究表明,SVM算法的分类能力优于PLS-DA。但Wang Yuanyuan等[30]对灵芝的中红外光谱鉴别研究中有相反的结论,PLS-DA模型分类性能优于SVM模型,原因可能是其样本量少(120),本研究中样本量更大(827),研究表明,SVM模型更适用于大样本种类鉴别。预处理与特征变量组合方法挖掘光谱信息能力最强,对模型分类效果提升最大,研究表明,该方法适用范围广。
3 结 论
采用傅里叶变换中红外光谱法测定8 种827 个野生牛肝菌子实体的红外光谱,分析牛肝菌的化学信息。采用预处理、提取特征变量(PC、FI、VIP)及两者组合等方法挖掘样品光谱信息,提高模型分类效果,结果表明:预处理组合特征变量对模型信息挖掘能力最强,结合SVM建立判别模型,模型拟合好,分类精度高,实现了8 种野生牛肝菌的准确、快速鉴别,可以为野生牛肝菌种类鉴别的提供参考。

