阿尤恩地区自然发酵牛乳中乳酸菌分离鉴定及多样性研究
史迪,党娜,武岳,马腾,刘文俊,张和平
(内蒙古农业大学乳品生物技术与工程教育部重点实验室农业部奶制品加工重点实验室内蒙古乳品生物技术与工程重点实验室,呼和浩特010018)
0 引 言
阿尤恩位处非洲是一个拥有热带海洋气候的荒漠小镇,当地畜牧业和畜产品贸易兴盛,具有独特的气候特征和丰富的生物资源。这种独特的气候特征孕育着丰富的微生物资源,采集当地特色的自然发酵牛乳样品,研究其特有的乳酸菌菌群结构对于野生型乳酸菌的保护和利用具有实际意义,对于丰富不同自然发酵乳制品中乳酸菌多样性信息也有理论意义和研究价值。
自然发酵乳制品历史悠久,酸牛乳、奶酪、酸驼奶等乳制品是我国内蒙古、新疆等地重要的饮食组成部分。自然发酵乳的特点是在自然的发酵过程中营养并没有流失,同时又富含氨基酸、维生素等代谢产物,在增强口感和风味的同时,更有利于人体的吸收。目前,关于酸牛乳对乳糖不耐症的缓解、维持人体肠道平衡等益生保健的作用均有报道[1]。乳球菌属中,乳酸乳球菌经常被作为奶酪的发酵剂菌种,其次也被添加到发酵牛乳中,以促进酸牛乳浓郁的酪香味[2-4];乳杆菌属中,干酪乳杆菌1889年被分离出,是工业化生产中有应用价值的菌种之一[5];链球菌属中,检出频率较高的嗜热链球菌,被认为是在工业化应用中重要的乳品发酵剂[6-8]。
本文通过传统纯培养方法揭示了阿尤恩地区自然发酵牛乳样品中的乳酸菌组成,对于后期优良乳酸菌菌种的筛选提供了菌种资源;同时结合PacBio SMRT技术探索了阿尤恩地区自然发酵牛乳的细菌组成,对揭示该地区自然发酵牛乳样品中的细菌多样性有着重要意义。
1 材料与方法
1.1 样品来源与运输
本文的5份自然发酵牛乳样品于2018年10月采集自阿尤恩市周边牧区,如表1。采集5份样品于2 mL无菌冻存管中,用于乳酸菌的分离。再次采集5份自然发酵牛乳样品于50 mL无菌离心管中,每个样品采集20 mL,采集后加入10~15 mL的DNA TaKaRa保护液,用于宏基因组DNA提取;采集的样品临时存放4℃采样箱中,一周内送达实验室进行实验操作[9]。

表1 阿尤恩地区自然发酵牛乳样品采集信息表
1.2 试剂与仪器
DNA保护液,大连TaKaRa公司;MRS培养基、M 17培养基,英国OXOID公司;TIANamp Bacteria DNA Kit细菌基因组DNA提取试剂盒,北京天根生化科技有限公司;E.Z.N.A.®Soil DNA Kit,美国OMEGA公司;SMRTbellTMTemplate Prep Kit试剂盒,Life Technologies公司;HiFi Hot Start Ready Mix PCR Kit,美国KAPA公司。
DYY-12电泳仪,上海一恒科技有限公司;SX-700高压蒸汽灭菌器,日本KAGOSHIMA SEISAKUSYO公司;高速冷冻离心机,德国Eppendorf公司;梯度基因扩增仪,美国Life Technologies公司;UVPGDS-8000凝胶成像分析系统,美国UVP公司;ND-1000微量紫外分光光度计,美国Nanodrop公司;PacBio RS II测序仪,美国Pacific Biosciences公司。
1.3 样品乳酸菌计数
5份自然发酵牛乳样品的乳酸菌计数选取生理盐水作为稀释液(浓度为0.85%NaCl)进行10倍梯度稀释,采用倾注法对阿尤恩地区的5份自然发酵牛乳样品进行乳酸菌计数[10],5份样品均选取10-5、10-6、10-7作为乳酸菌计数梯度,吸取1 mL 5份样品的10-5、10-6、10-7梯度稀释液于灭菌的90 m L玻璃培养皿中,倾倒25~30 mL加入琼脂(1.2%Agar Powder/100 mL)的55℃MRS培养基和M 17培养基,待培养基完全凝固后倒置于37℃恒温培养箱厌氧培养48 h,活菌计数即每毫升样品的乳酸菌菌落总数。
1.4 乳酸菌的分离纯化及保存
选取与1.3中乳酸菌活菌计数相同的3个稀释梯度,每个梯度吸取0.2 mL的稀释液于与活菌计数相同成分的培养基上,使用涂布棒顺时针均匀涂布,完成操作后选择与1.3中相同的培养条件进行培养;培养结束后选择形态不同的单菌落进行划线纯化,将革兰氏染色阳性呈紫色、过氧化氢酶试验显示阴性、没有芽孢且形态唯一的纯培养物传代培养;将传至三代的分离株进行菌种保藏,吸取以脱脂乳粉与酵母粉混合的保护剂800μL(8 g脱脂乳粉、2g酵母粉/100 mL),分装在5支灭菌安瓿管和3支灭菌螺口冻存管中,(50μL/每支安瓿管、200μL/每支螺口冻存管),液氮速冻安瓿管和螺口冻存管后,将分离出的111株乳酸菌菌种于-80℃超低温冰箱保存[9]。
1.5 菌株DNA提取与扩增
传至二代的纯培养物经3 800 rpm低速离心8 min后,弃液体培养基;加入3 mL配制好的0.85%生理盐水,经3 800 rpm低速离心10 min后,弃上清液(重复此操作);完成上述操作后,再吸取1 mL 0.85%生理盐水,振荡混匀后转移至1.5 mL离心EP小管中,1 200 rpm离心2 min,吸净1.5 mL EP管中的残留液体后,使用TIANamp Bacteria DNA Kit(离心柱型)细菌基因组DNA试剂盒提取DNA,具体操作参照说明书中的11个步骤依次进行提取;提取后使用ND-1000微量紫外分光光度计和0.8%琼脂糖凝胶电泳实时检测浓度、纯度以及完整程度。
PCR扩增体系:将111株符合浓度和纯度的DNA作为扩增模板2μL;正、反引物(27F(5'-AGAGTT TGATCMTGGCTCAG-3')和1492R(5'-ACCTTGTTACGACTT-3'))各1.5μL;10×PCR Buffer(含Mg2+离子)5μL;d NTP 4μL;Taq DNA polymerase 0.5μL;dd H2O 35.5μL[1]。扩增条件为:95℃,预变性5 min;95℃,变性30 s;58℃,退火45 s;72℃,延伸1 min(进行30个循环),72℃,末端延伸10 min[10]。
1.6 自然发酵牛乳中乳酸菌菌株的鉴定
将条带清晰、明亮无拖尾的PCR产物放于装有冰袋的泡沫箱邮寄,由上海美吉生物科技有限公司接收并完成111个PCR产物的双向测序工作。在NCBI数据库中的BLAST下比较111株分离株的同源性,根据同源性结果在LPSN数据库中下载与分离株同源性>98%的模式株序列,采用Mega7.0软件数据库中的邻接法构建进化树[11]。
1.7 自然发酵牛乳样品宏基因组DNA的提取
提取样品DNA前需将5份样品充分混匀,参照说明书的42个步骤使用E.Z.N.A.®Soil DNA Kit试剂盒提取样品DNA;提取后使用ND-1000微量紫外分光光度计和0.8%琼脂糖凝胶电泳检验样品DNA的浓度、纯度及完整程度,将OD260/280为1.8~2.0之间,DNA浓度>20 ng/μL且片段化程度小的DNA置于-20℃低温冰箱保存备用[12]。
1.8 细菌16S rRNA基因片段全长扩增
DNA的扩增需要加入16个碱基长度的核苷酸序列(Barcode)用于区分5份自然发酵牛乳样品。PCR反应扩增体系为:正向、反向通用引物(27F 1492R)各1.5μL,KAPA体系25.0μL,DNA模板1.5μL,dd H2O 20.5μL。PCR反应条件为:95℃预变性3 min,98℃变性20 s,60℃退火15 s,72℃延伸30 s,(进行30次循环),72℃末端延伸2 min[13]。
1.9 PacBio SMRT三代测序及生物信息学分析
检测合格的PCR产物,使用试剂盒(Pacific Biosciences SMRT bellTMTemplate Prep Kit)构建文库,按产品说明书使用PacBio RS II仪器进行上机测序;将测序得到的原始序列依据最小循环次数为5次,预测准确度为90%,DNA插入片段为1 800 bp进行质量控制[10];在QIIME平台上对符合要求的序列进行分析;首先利用PyNAST软件对每个序列进行排齐操作,其次在相似度为97%的条件下划分OUT,再次使用RDP、Silva和Greengenes数据库对代表性序列进行比对,并确定门、属、种等水平的分类学信息,并在此基础上计算分析α多样性常用的超1指数、香农指数、发现物种数、和辛普森指数比较5份自然发酵牛乳样品中的微生物种类和相对丰度;最后采用Origin软件对摩洛哥阿尤恩地区自然发酵牛乳样品的多样性结果进行可视化处理。本研究中涉及的序列数据已提交至MG-RAST数据库,序列号为no.mgp95102。
2 结果与分析
2.1 样品乳酸菌计数结果
阿尤恩地区自然发酵中乳样品中乳酸菌计数如表2。

表2 阿尤恩地区自然发酵牛乳样品中乳酸菌计数结果
本文5份自然发酵牛乳样品的乳酸菌活菌数在5.89×105~8.33×106CFU/mL之间。呼斯楞[14]研究呼伦贝尔地区酸牛乳发现,6份酸牛乳样品活菌数都达到106CFU/mL以上,由于本文5份自然发酵牛乳样品与呼伦贝尔地区酸牛乳样品采集后的贮存方式、以及运输的时间大致相同,因此不同地区自然发酵乳的活菌数可能与样品的贮存、运输的时间有关。
2.2 分离株的表型特征
本文对阿尤恩地区5份自然发酵牛乳进行乳酸菌的分离鉴定,根据革兰氏四步染色、过氧化氢酶的试验,初步将革兰氏染色阳性呈紫色、过氧化氢酶试验显示阴性、没有芽孢且显微镜下形态唯一的111分离株暂定为乳酸菌,如图1所示,111株分离株菌落颜色呈白色或淡黄色,表面呈粗糙或光滑形态,并且在显微镜下均匀分布。
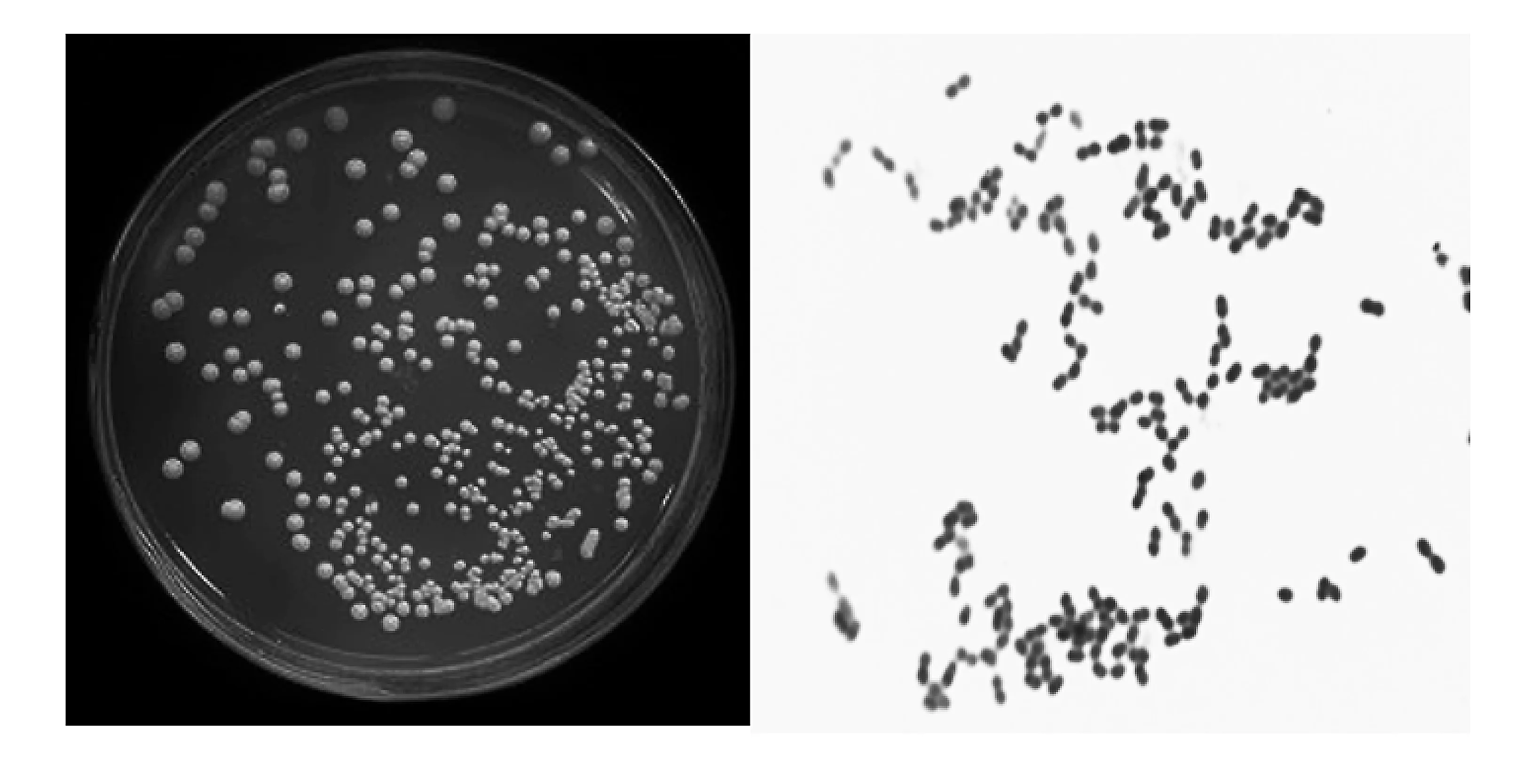
图1 分离菌株在荧光显微镜下图片
2.3 乳酸菌分离株16S rRNA基因序列扩增结果
将质量浓度<200 ng/μL,纯度值(A260/A280)均在1.8~2.0之间的111株分离株进行16S rRNA基因序列扩增。图2显示在1 500 bp处有完整性较好的明亮条带,因此PCR扩增产物满足实验要求。
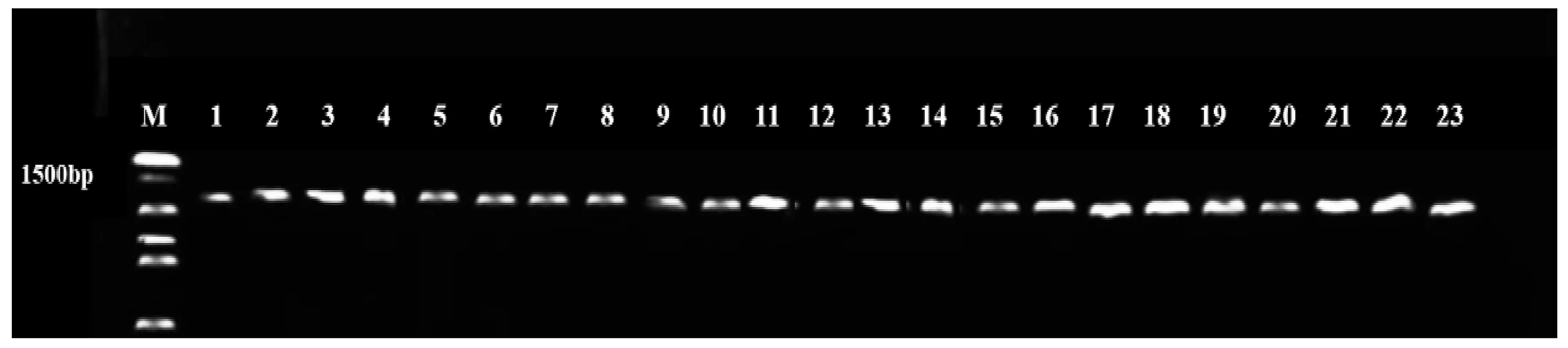
图2 部分16SrRNA基因的PCR凝胶电泳图
2.4 自然发酵牛乳中乳酸菌分离菌株鉴定结果
5份摩洛哥阿尤恩地区的自然发酵牛乳中分离出111株乳酸菌菌株,鉴定为5个属,10个种。
将每个分离株的16S rRNA序列与模式株整合后,应用MEGA7.0软件构建部分菌株与模式株的系统发育树[15]。IMAU 98277、IMAU 98255与Lactobacillus paracasei ATCC 25302T(D 79212.1)模式株聚为一类;IMAU 98296与Lactobacillus plantarum ATCC 14917T(AF 404710.1)模式株聚为一类;IMAU 98291与Lactobacillus kefiri ATCC 35411T(AJ621553.1)模式株聚为一类;IMAU 98339与Leuconostoc pseudomesenteroides ATCC 12291T(X 195979.1)模式株聚为一类;IMAU 98211与Lactobacillus helveticus ATCC 15009T(FR 683085.1)模式株聚为一类;IMAU 98275与Lactobacillus kefiranofaciens ATCC 43761T(AJ575259.1)模式株聚为一类;IMAU 98151与Enterococcusitalicus DSM 15952T(AJ582753.1)模式株聚为一类;IMAU 98249与Enterococcus faecium ATCC 19434T(AB012213.1)模式株聚为一类;IMAU 98205与Streptococcusthermophilus ATCC 19258T(X 68418.1)模式株聚为一类;IMAU 98172、IMAU 98164与Lactococcus lactis ATCC 19435T(AB100803.1)模式株聚为一类;且分离株的基因序列与模式株的同源性均大于99%。

表3 阿尤恩自然发酵牛乳中乳酸菌分离鉴定结果

图3 乳酸菌分离株16SrRNA基因序列进化树
2.5 阿尤恩地区自然发酵牛乳中的多样性分析
通过SMRT测序,5份阿尤恩地区自然发酵牛乳样品一共获得了33492条序列,平均每份自然发酵牛乳样品有6698条DNA序列。通过划分序列,一共获得2270个具有代表性的OTUs,其中样品MLG10获得的代表性OTUs最少,为179条;样品MLG7获得的代表性OTUs最多,为667条;平均每个样品获得454条代表性序列。分析阿尤恩地区自然发酵牛乳样品的稀疏曲线和香农曲线发现,MLG7样品和MLG8样品的稀疏曲线和香农曲线是基本重合的,并且测定的样品中OTUs数量随着测序深度的增加而增加,当测序量达到6 000时,一些样品中还有物种被检测到。在测序量为4 000时香农曲线已经趋于饱和,说明在此测序量下,5份样品中的多样性信息被充分的展示如图4,表4。
2.6 阿尤恩地区自然发酵牛乳的细菌群落结构
5份自然发酵牛乳样品鉴定归属于8个细菌门、66个菌属、131个菌种、细菌门水平,8个菌门分别是Firmicutes、Proteobacteria、Deinococcus-Thermus、Actinobacteria、Acidobacteria、Bacteroidetes、Fusobacteria、TM 7,其 中Firmicutes(69.63%)和Proteobacteria(29.77%)是主要菌门,其他菌门的相对含量均较低(表5)。
细菌属水平,共检测到66个细菌属,如图5(a),相对含量较高的细菌属有:Lactococcus(31.80%)、Streptococcus(18.64%)、Lactobacillus(18.35%)等。MLG10中Streptococcus含量最多,达到90.33%;MLG13中的Lactococcus的含量最高(53.26%),MLG14中的Lactobacillus含量最高(43.51%)。
细菌种水平共检测到131个细菌菌种,如图5(b),主要菌种为Lactococcus lactis(22.95%)、Streptococcus thermophilus(18.05%)、Lactobacillus helveticus(10.26%);相对含量高于1%的菌种还有Lactococcus chungangensis(7.53%)、Lactobacillus kefiranofaciens(4.32%)、Lactobacillus paracasei(1.88%)、Lactobacillus kefiri(1.42%);进一步对自然发酵牛乳样品中的主要菌种进行相对分析发现,5份样品中不同菌种的相对含量存在明显的差异,Lactococcus lactis是MLG13、MLG14样品中的优势菌种,相对含量达到50.52%;Streptococcus thermophilus是MLG10样品中的优势菌种,相对含量高达90.23%;Lactococcus chungangensis是MLG7、MLG8的优势菌种,相对含量为18.76%。
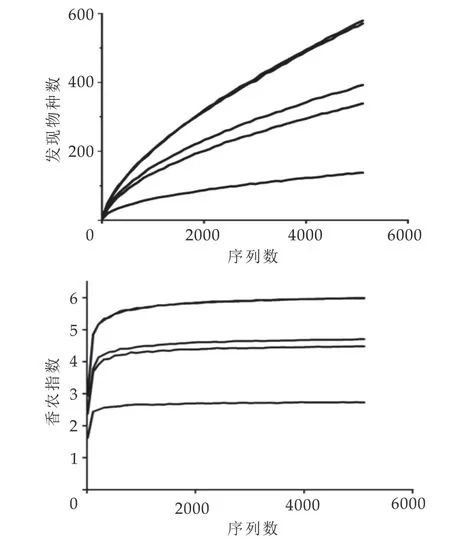
图4 阿尤恩地区自然发酵牛乳稀疏曲线及香农曲线

表4 阿尤恩地区自然发酵牛乳中序列信息和α多样性指数
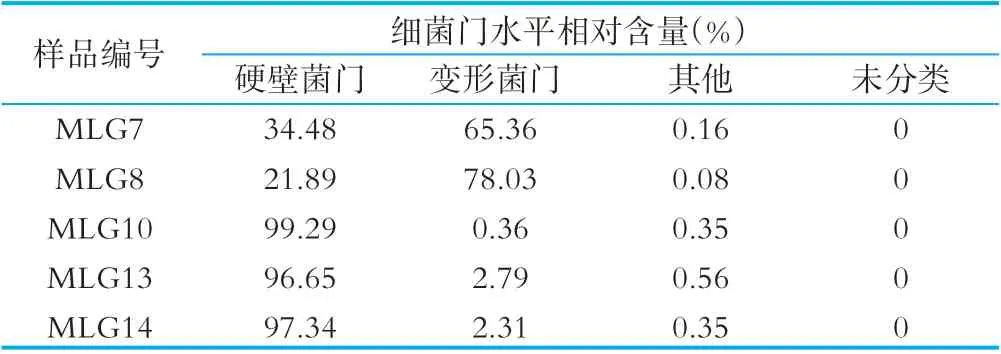
表5 阿尤恩地区自然发酵牛乳中细菌门水平的相对含量
根据表6发现,PacBio SMRT测序方法捕捉到5份自然发酵乳纯培养方法分离到的全部菌株,其中5份样品全部分离出Lactococcus lactis,共分离到51株,占总分离株的45.95%,SMRT测序方法测得Lactococcus lactis相对含量为22.95%,说明Lactococcus lactis为阿尤恩地区自然发酵牛乳中的绝对优势菌种。

图5 阿尤恩地区自然发酵牛乳中主要乳酸菌相对含量

表6 阿尤恩地区自然发酵乳中纯培养与PacBio测序方法鉴定结果
结合分离鉴定结果(表3)发现,样品MLG7、MLG8分离到的菌株类别较为单一,主要分离出Lactococcus lactis;样品MLG10主要分离出Streptococcus thermophilus;样品MLG13和MLG14分离的菌株类别比较丰富,分离出Lactococcus lactis、Lactobacillus helveticus、Lactobacillus kefiranofaciens、Lactobacillus casei等菌种。
2.7 OTU水平菌群结构分析
OTU对α多样性分析有重要的指导意义,5份自然发酵牛乳共产生1673个OTU(如图6),其中仅出现在一份样品中的OTU共有1217个,所包含的序列数为3422条,分别占OTU总数和质控合格序列数的72.74%和10.22%。
同时,5份样品中均可能存在特有的OTU,MLG14样品特有OTU最少(9个),MLG10样品特有OTU最多(54个)。MLG7样品特有乳酸菌为Lactobacillus gasseri,MLG8样品特有乳酸菌为Lactococcus taiwanensis,MLG10样品特有乳酸菌为Streptococcus salivarius,MLG13样品特有乳酸菌为Lactobacillus harbinensis、Lactobacillus zeae,MLG14特 有 乳 酸 菌 为Lactobacillus otakiensis。5份样品还存在非乳酸菌菌种,MLG7样品 特 有 菌 种 为Acinetobacter soli、Enterobacter asburiae,MLG8样品特有菌种为Acinetobacter junii,MLG13样品特有菌种为Arcobacter butzleri,MLG14样品特有菌种为Enterobacter hormaechei。
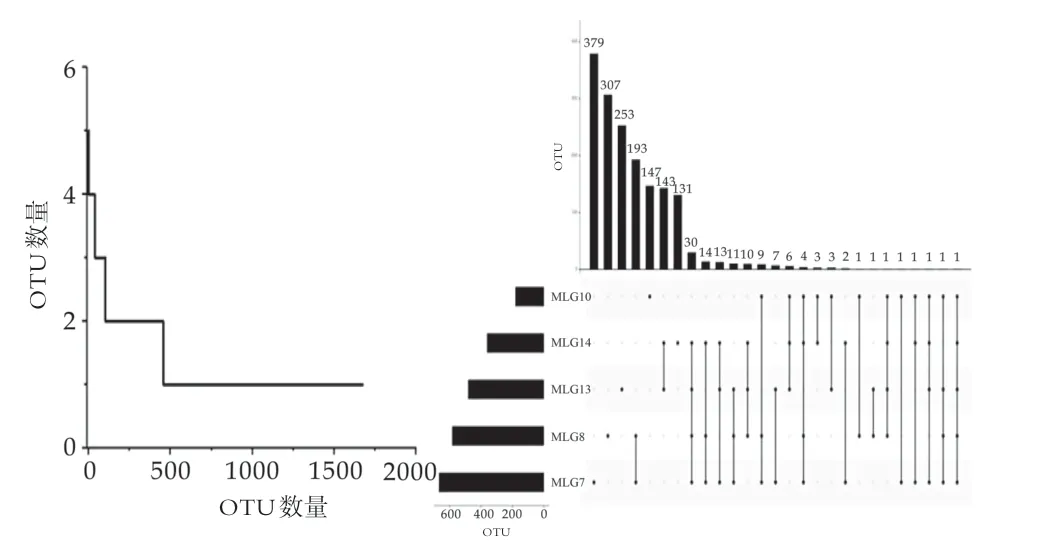
图6 阿尤恩自然发酵牛乳中OTU出现次数和各样品中特有OTU分析
2.8 自然发酵牛乳中菌群关联性分析
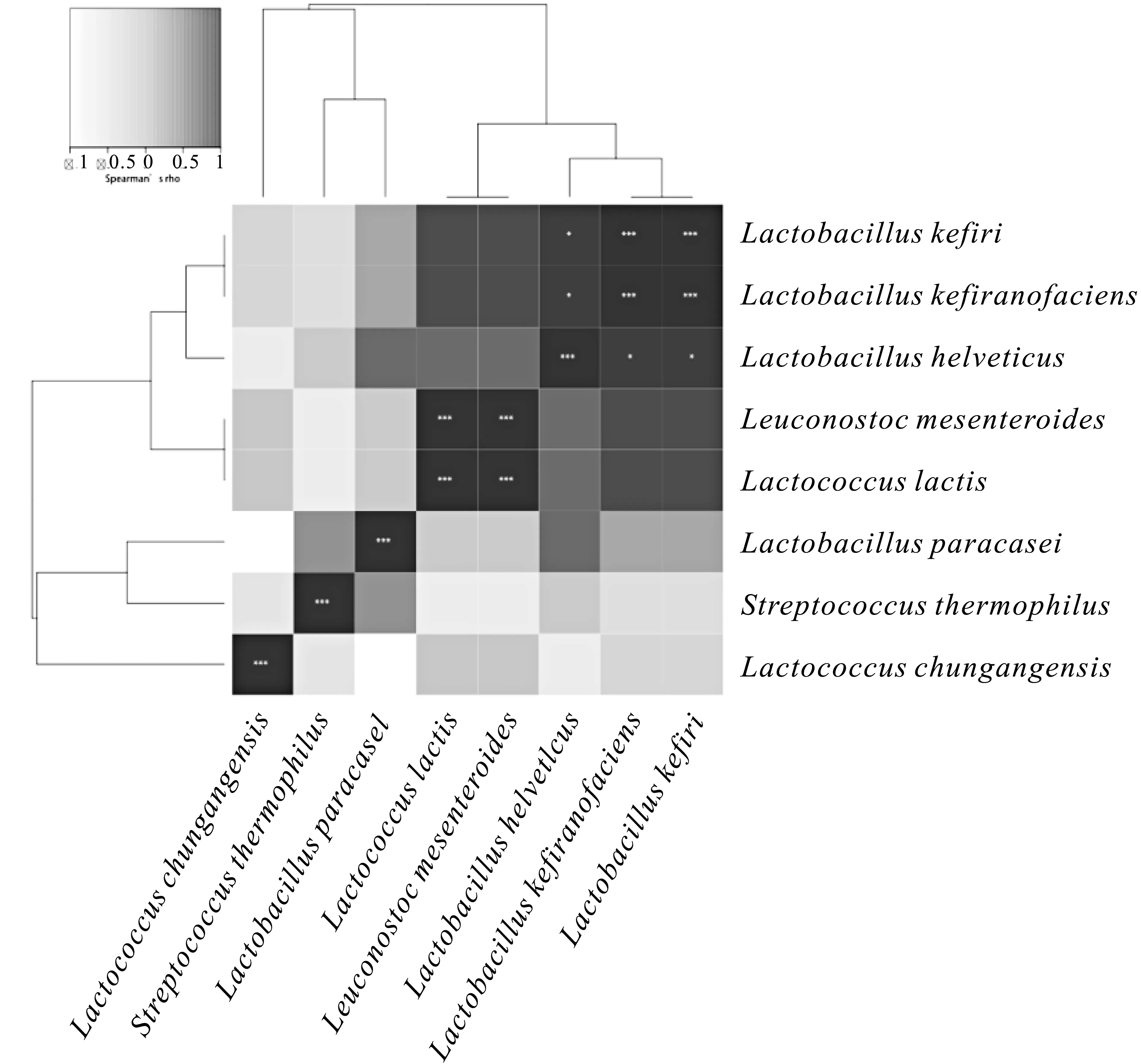
图7 阿尤恩地区自然发酵牛乳中种水平细菌之间的相关性
为揭示自然发酵牛乳细菌之间的相关关系,本研究利用Spearman相关性检验对5份样品中平均相对含量大于1%的核心乳酸菌群进行相关性分析。图7展示了优势乳酸菌种之间的相关关系,种水平结果显示:Lactobacillus kefiri与Lactobacillus kefiranofaciens呈显著正相关(R=1,P<0.001),Leuconostoc mesenteroides与Lactococcus lactis呈显著正相关(R=1,P<0.001),Lactobacillus helveticus与Lactobacillus kefiri、Lactobacillus kefiranofaciens呈显著正相关(R=0.91,R=0.91,P<0.05),Lactobacillus paracasei与Lactococcus chungangensis呈显著负相关(R=-0.97,P<0.001)。
3 讨 论
本文研究发现,Lactococcus lactis、Streptococcus thermophilus、Lactobacillus helveticus、Lactobacillus kefiranofacien被分离出的菌株数较多,其中Lactococcus lactis为阿尤恩地区自然发酵牛乳的最优势菌种。Lactococcus广泛存在于各乳制品中,例如:Quigley[16]发现,Lactococcus是爱尔兰原奶中优势菌属;V Lafarge团队[17]发现Lactococcus lactis是生牛乳中的优势菌;Liu等[18]对内蒙古自然发酵牛乳的研究发现,Lactococcus lactis subsp.Lactis、Lactobacillus casei、Lactobacillus helveticus是优势乳酸菌;Yu等[20]对蒙古国自然发酵乳的研究发现,Streptococcus thermophilus、Lactobacillus helveticus为优势菌,因此本文中最优势菌种Lactococcus lactis、Lactobacillus helveticus与上述文献的研究有相同之处。由于自然发酵乳受地理、气候、工艺等因素的影响,所以微生物组成有相同之处,同时也有差异之处[21]。
本文通过测序技术检测到的细菌种覆盖了传统纯培养方法分离到的所有菌株,进一步证实了本文通过纯培养方法得到的摩洛哥阿尤恩地区自然发酵乳的分离鉴定结论。测序技术的优点之一是在种水平可检测到样品中活的或曾经存在过的微生物,例如本研究通过测序检测到的Lactobacillus brevis、Lactobacillus gasseri等菌种,可能受菌落选择、丰度较低、菌种已死亡等因素的限制,导致在纯培养方法中未分离到。金桩等[22]认为短乳杆菌LB-02在1%接种量下,最佳培养条件是35℃24 h。因此测序结果可在一定程度上指导乳酸菌的分离鉴定。技术的局限性在一定程度上限制了传统纯培养方法的应用,但此方法的优势在于可分离得到纯培养物[23]。
因此,传统的纯培养分离方法与测序技术有效、紧密的结合,一方面可充分揭示自然发酵乳中的微生物多样性,另一方面为丰富菌种资源库奠定基础。
4 结 论
本文运用纯培养方法对摩洛哥阿尤恩地区的5份自然发酵牛乳制品进行分离鉴定,共分离出111株乳酸菌菌株,归属为5个属,10个种,Lactococcus lactis占样品总分离菌株的45.95%;应用宏基因组16S rRNA基因测序技术从5份自然发酵牛乳样品中检测出8个门,66个属,131个种的细菌,其中含量较高的乳酸菌有Lactococcus lactis、Streptococcus salivarius、Lactobacillus taiwanensis、Lactobacillus harbinensis、Lactobacillus zeae、Lactobacillus otakiensis等菌种,优势菌种为Lactococcus lactis,相对含量为22.95%。结果表明,Lactococcus lactis为阿尤恩地区5份自然发酵牛乳样品的优势菌种,并且细菌种类丰富,样品间细菌组成存在相似性和差异性。

