甲状腺乳头状癌患者BRAF基因突变及相关生物特征分析①
毛宏凯,杨 梅,吴 军
(新疆医科大学1公共卫生学院劳动卫生与环境卫生学教研室,2基础医学院生化教研室,乌鲁木齐830011)
关健词:甲状腺癌;乳头状癌;BRAF;基因突变
甲状腺癌(thyroid carcinoma,TC)是最一种常见的内分泌系统恶性肿瘤,发病率位于所有肿瘤第七位,在全球范围内增长较快[1],在我国甲状腺癌是增长速度最快的恶性肿瘤[2],2018年全球新增甲状腺癌死亡4.1万例,占所有癌症死亡的0.4%,其死亡率位居全球第6位[3],2005-2014年中国甲状腺癌死亡率及疾病负担均在增长,且以女性为主[4],对人群健康构成较大威胁。根据肿瘤起源及分化特点,TC可分为甲状腺乳头状癌(papillary thyroid carcinoma,PTC)、甲状腺滤泡癌(follicular thyroid carcinoma,FTC)、甲状腺髓样癌(medullary thyroid carcinoma,MTC)以及甲状腺未分化癌(anaplastic thyroid cancer,ATC),其中PTC最常见,占全部甲状腺癌的85%~90%,而PTC和FTC合称分化型甲状腺癌(differentiated thyroid carcinoma,DTC)[5]。BRAF基因突变是PTC最常见的遗传学改变[6],与PTC发生及更高的TNM分期呈正相关[7]。其中单因素分析提示BRAFV600E突变与甲状腺被膜外侵犯相关(χ2=5.227,P=0.022)[8]。此外,BRAF基因突变已经成为高效的风险预测分子标记物,同时也是潜在的药物治疗靶点[9],对于PTC的早期诊断、预防控制、临床治疗及预后评估具有十分重要的作用[10]。如何开发出更多的鉴别诊断的标志物可能更依赖于对BRAF基因分子机制的了解[11]。本研究在相对大PTC样本中,分析BRAF基因的突变率、突变特点,同时分析BRAF基因突变阳性患者中显著上调基因的表达特点、PPI及其所参与的生物学功能和分子过程等,分析潜在的关键基因,为阐明PTC发病机制及其预防和控制提供理论基础。
1 资料与方法
1.1 BRAF基因在PTC中的突变频率及突变位点分析 本研究通过肿瘤基因组图谱(the cancer genome atlas,TCGA)的子数据库cBioPortal中“快速搜索(quick search)”功能搜索关键词“BRAF”,选择BRAF基因在各类癌症中的变异情况,分析BRAF基因在各类癌症中的突变频率,操作方法可见使用说明[12]。然后在cBioPortal中选择“Thyroid”并勾选“Thyroid Carcinoma(TCGA,PanCancer Atlas)500samples”数据集,点击按基因查询后勾选“mRNA expression z-scores relative to diploid samples(RNA Seq V2 RSEM)”即量化基因表达指标为相对于二倍体样品的mRNA表达z得分,数据自动筛选为具有突变、拷贝数畸变(copy number alteration,CNA)和表达数据的完整样本(480例),最终获取TCGA数据库中480例完整样本的PTC患者生物学资料及基因测序结果,分析PTC中BRAF基因的突变位点。
1.2 在PTC中筛选与BRAF基因突变共发生或互斥的基因 将患者分为改变组(存在BRAF基因突变,300/480)和非改变组(无BRAF基因突变,180/480),利用“突变Mutations”功能对改变和非改变组患者进行BRAF基因所有突变位点分析,再利用“互斥Mutual exclusivity”功能对上一步找出有统计学差异的基因两两进行分析,按照有无突变构建2×2列联表,通过单侧Fisher精确检验计算显著性,Log2OR为量化所选样本中A存在或不存在与B存在或不存在关联强烈程度指标,OR=(Neither*Both)/(ANot B*BNot A)。
1.3 PTC患者中肿瘤恶性表型相关功能基因表达情况 选择“Enter Genes”列表中的不同癌相关基因集分析了细胞增殖(growth or proliferation)、侵袭和迁移(invasion and metastasis)、细胞周期(cell cycle)、生存凋亡(survival/cell death)相关基因,及在480例生物学资料完整的PTC患者中的mRNA表达情况。
1.4 改变组与非改变组患者中显著变化的基因及PPI网络构建 对两组患者组织中表达有统计学差异的基因进行筛选,共得到12 699个基因,两组中各取变化前50的基因,共计100个。随后,分别把该50个基因导入String中在线分析,将最低互作分值(minimum required interactionscore)设置成中度可信(medium confidence:0.4),同时隐藏网络中断开连接的节点(hide disconnected nodes in the network)进行分析,最终获得蛋白与蛋白之间相互作用网络(Protein-protein interaction,PPI)。
1.5 改变组中异常表达基因参与的生物学过程分析为进一步分析改变组中显著变化的前50个基因的生物学功能,在PPI分析基础上,进一步对互作基因执行了GO分析,其中包括生物学过程(biological process,BP)和分子功能(molecular function,MF)。
1.6 统计学分析 按照PTC患者数据信息的特点分别采用Kruskal-Wallis检验、χ2检验及Fisher精确检验等方法,以P<0.01或FDR(false discovery rate)<0.05为差异有统计学意义。Q值=被拒绝但却是正确的概率=假阳性/推测为阳性的个数。
2 结果
2.1 BRAF基因在PTC中的突变频率及突变位点分析 BRAF基因突变频率在PTC中最高,以点突变为主,突变率达60%左右;480例生物学资料完全的PTC癌患者BRAF基因突变情况(图1)。480例PTC者中有300例患者存在BRAF基因突变,频率为62.5%;其中错义突变285例,移码突变1例,均位于Pkinase_Tyr区域,突变位点分别为V600E(283/480)、K601E(2/480)及P490_Q494del(1/480),见图2。
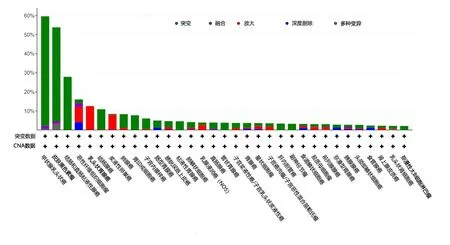
图1 BRAF基因在各类癌症中的突变频率

图2 BRAF基因在PTC中的突变位点
2.2 在PTC中筛选与BRAF基因突变共发生或互斥的基因 在非改变组,HRAS和NRAS基因表达,而改变组无HRAS和NRAS基因表达,上述HRAS和NRAS基因的表达与BRAF基因突变发生存在互斥性(P<0.001),同时HRAS和NRAS基因的表达存在共现性(P=0.568),但未发现与BRAF共发生的基因。HRAS和NRAS基因表达和HRAS、NRAS和BRAF基因的互斥性及共现性见表1~2、图3~4。
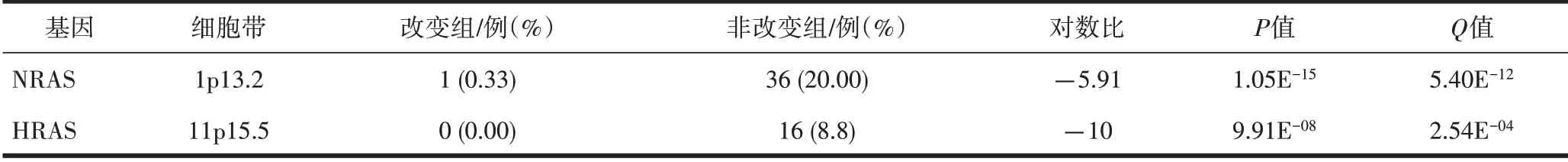
表1 HRAS和NRAS基因在改变组与非改变组中的表达情况

表2 HRAS、NRAS和BRAF基因的互斥性及共现性情况
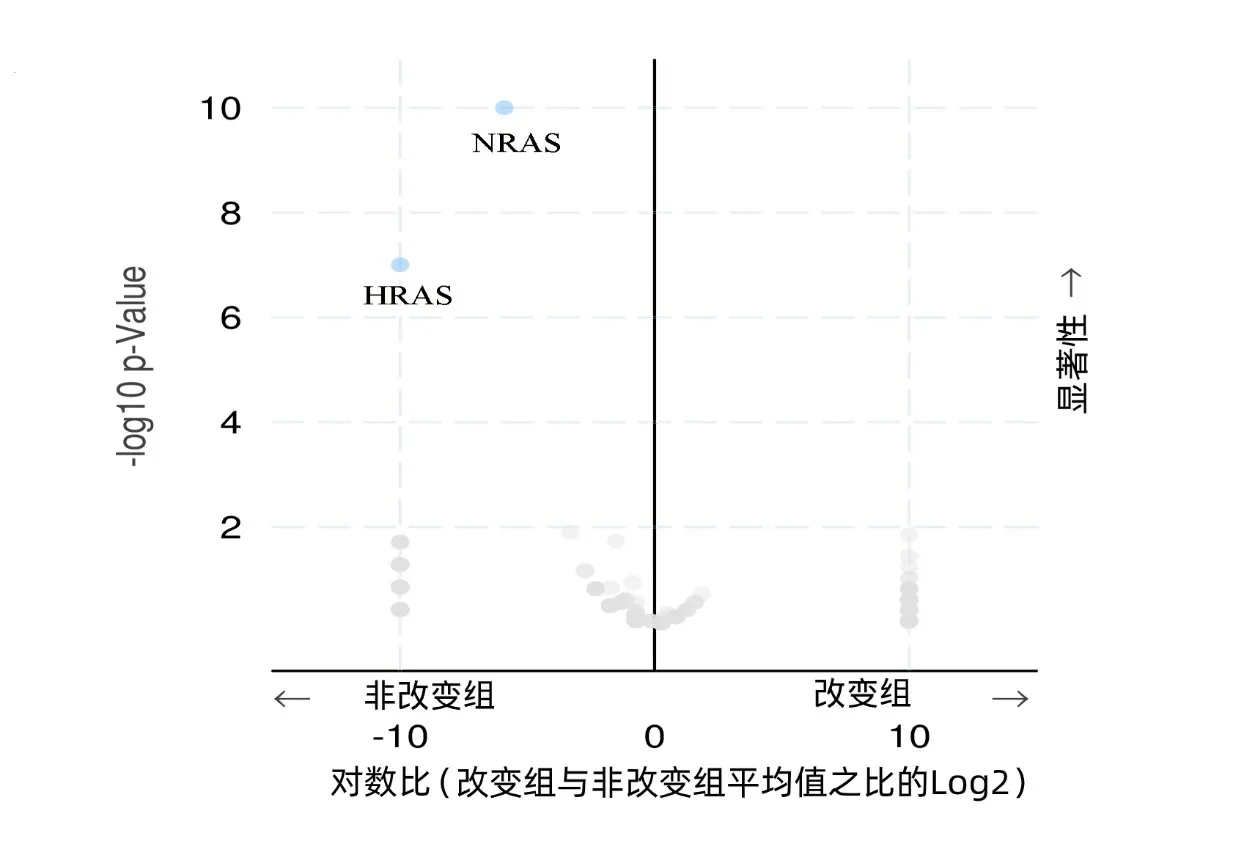
图3 PTC中改变组与非改变组中基因突变的火山图
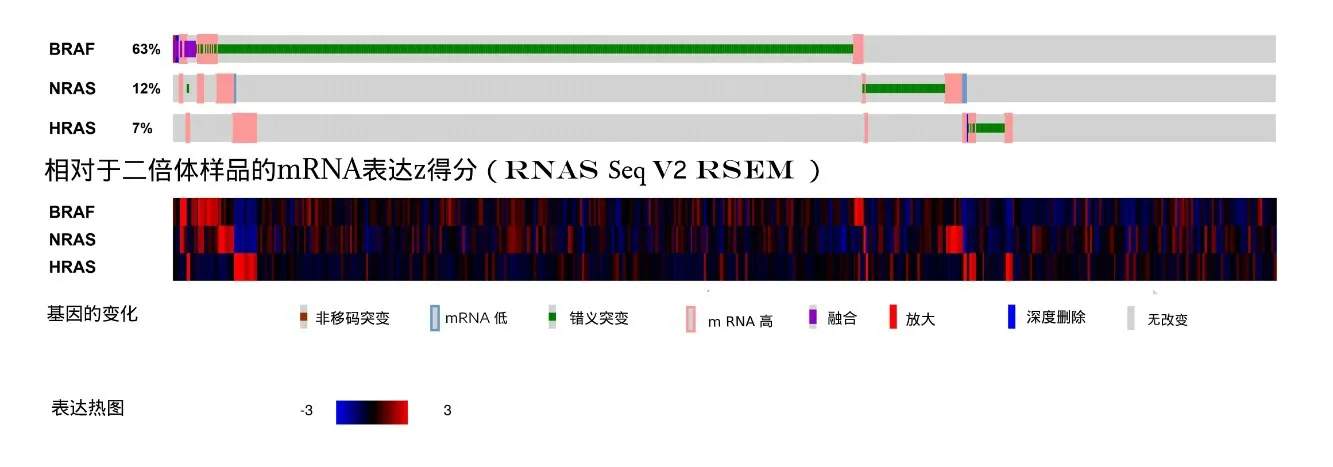
图4 BRAF与NRAS和HRAS表达情况
2.3 肿瘤恶性表型相关基因在PTC患者中的表达频率 肿瘤恶性表型相关基因中影响PTC侵袭和迁移的基因主要是MMP相关基因,影响其生存凋亡的基因主要是CASP、NFKB、TGFB等基因,影响其细胞周期的基因主要是E2F、STAT、CDK等基因。
2.4 改变组与非改变组中显著上调的基因 改变与非改变组中基因差异表达水平显著上调的前50个基因(P均<0.01),结果见图5。其中,与非改变组比较,改 变 组 中FN1、SLC34A2、ITGA3、MET、TACSTD2、PPL、ARMCX3、PROS1、RAD23B、CD55等基因表达差异有统计学意义(P均<0.01)。与改变组比较,非改变 组 中TPO、C16ORF89、MLEC、FHL1、SORBS2、MT1G、ALDH2、MT1F、FAM167A、ITPR1、NUCB2等基因表达差异有统计学意义(P均<0.01)。
2.5 改变组与非改变组中显著表达基因的PPI分析进一步分析改变组与非改变组中显著表达基因间PPI,获得关键的互作基因,分别将非改变组和改变组中表达变化的前50个基因进行了PPI分析,这些关键基因的交互作用分析结果,见图6,图中的圈代表基因,圈与圈连线代表基因控制的蛋白间存在相互作用,连线越多表示相互作用关系越密切。PPI分析结果显示,非改变组中前50个基因调控的蛋白间相互作用联系不紧密表明基因间交互作用较少,而改变组中表达上调的前50个基因中FN1、MET、TGFBR1及ERBB3等调控的蛋白间联系较为紧密,且交互作用较多。

图6 在非改变组和改变组中上调基因间的蛋白质相互作用
2.6 改变组中显著表达基因的生物学功能推测 GO分析结果表明,上述基因主要参与血管生成、阿米巴型细胞迁移、胚胎形成和发育、多细胞生物发育、信号转导调控、程序性细胞死亡、细胞迁移、细胞粘附等生物学过程;上述基因主要的分子功能与I型转化生长因子β受体活性、跨膜受体蛋白激酶活性、激活素结合有关。见表3。

表3在改变组中显著上调的互作基因可能参与的BP及MF(部分)
3 讨论
本研究中BRAF基因在PTC患者中突变频率最高,其中又主要是PTC,BRAF基因的突变位点集中在Pkinase_Tyr区域,又以V600E为主。有研究指出,BRAF基因可以激活ERK信号通路导致下游转录因子发生磷酸化,进而通过一系列调控,最终导致PTC的发生[13],而BRAF V600E突变可以引起丝氨酸/苏氨酸激酶激活,经过调控表达后促使甲状腺滤泡细胞转化成PTC细胞[14]。本研究中PTC患者中BRAF基因与HRAS、NRAS基因表达具有互斥性,且HRAS与NRAS基因表达具有共现性,且差异具有统计学意义。这方面的发现为无BRAF突变的PTC的诊断提供了新的方向,可以从另一方面提高对PTC的诊断能力。同时可以为PTCBRAF基因反馈和负反馈调控网络及基因交互作用的深入研究提供线索,如BRAF基因也可能通过驱动其他基因过表达或发生甲基化参与PTC发生与发展[15]。本研究发现肿瘤恶性表型相关基因在一定程度上会影响PTC的发生发展,如PTC组织中自噬相关基因Beclin-1的表达[16]及CD56基因表达对PTC的侵袭性和淋巴结转移有一定意义等[17]。PPI网络中改变组比非改变组中基因调控蛋白间相互作用联系更加密切,这可能与是否发生BRAF基因突变会引发不同的基因调控机制有关,改变组中FN1、MET、TGFBR1及ERBB3等为相互作用较强的基因,其中有研究提出的FN1是与散发MTC的病理生理变化有关的新型预后生物标志物[18],MET蛋白能够作为预测PTC中淋巴结转移的标记[19],抑制TGFBR1可能对治疗ATC和其他侵袭性肿瘤具有潜力[20],ERBB3可能参与了PTC的增殖、转移及复发,有望成为PTC治疗的新靶点[21]。但这些基因的相互作用关系还需进一步实验论证。基因GO分析中,这些基因可能通过上述生物学过程及分子功能参与PTC的发生与发展。本研究中PTC在BRAF基因突变阳性情况下,其PPI分析中的关键基因相互作用,以及在无BRAF基因突变的情况下,HRAS和NRAS的高表达对PTC的诊断、治疗及预后评估中具有重要的意义,为今后多个特异性分子标记物的联合应用提供了一些线索。本研究从生物信息学角度对PTC患者BRAF基因进行分析,但缺少相应的实验学验证,且该TCGA样本数据并不能很好的控制混杂因素,此为本研究的局限性。
综上所述,本研究利用生物信息学方法对PTC的BRAF基因的突变特点,影响PTC发生发展的关键基因和交互作用网络、生物学功能等生物学特征进行了探讨,为多个特异性分子标记物的联合应用提供线索,为阐明PTC分子发病机制、预防、诊断和治疗的研究提供了理论基础。

