豫南地区野生大豆种质资源的SSR遗传多样性分析
刘红云, 周 强, 李淑梅, 王付娟
(信阳农林学院农学院/河南省豫南农作物有害生物绿色防控院士工作站, 河南 信阳 464000)
野生大豆(Glycinesoja)属豆科(Leguminoase)蝶形花亚科(Papilionoideae)大豆属(Glycine) Soja 亚属[1],属于一年生草本植物,它的种子及根、茎、叶均可入药。主要分布在东亚中北地区,包括中国、朝鲜半岛、日本列岛和俄罗斯远东地区。在我国,除了青海、新疆和海南外,其余省份均发现有野生大豆的分布[2]。熊振宇等[3]用20对ISSR引物对野生大豆与栽培大豆及其10份杂种F1进行遗传多样性分析,探讨野生大豆与栽培大豆之间的遗传关系。对野生大豆的遗传多样性研究,最早的是形态学、同位酶标记,李军等[4]利用聚丙烯酰胺电泳技术检测了浙江金华南坡野生大豆种群内14个样本6种同工酶水平上的生化遗传结构及其变异。李为民等[5]为了研究陕西省不同居群野生大豆的遗传多样性,归纳了几种常见DNA分子标记技术的原理及特点,综述了不同DNA分子标记在野生大豆遗传多样性研究中的应用现状。
SSR分子标记技术重复性高、简单可靠,应用于研究各种作物的遗传结构和遗传多样性。严茂粉等[6]利用40对SSR引物对北京地区的野生大豆天然居群进行了遗传结构和遗传多样性分析。李为民等[7]利用SSR分子标记对陕西省6个野生大豆自然种群和1个栽培大豆种群的遗传多样性和遗传结构进行了分析。王果等[8]以山西省的544份野生大豆为材料,分析了质量性状、数量性状及SSR标记遗传多样性。但对河南省野生大豆遗传多样性研究比较少见,尤其是河南省豫南地区的野生大豆。因此,对河南豫南地区12份野生大豆材料进行SSR遗传多样性分析,旨在分析河南省豫南地区野生大豆资源的遗传多样性分布特点,揭示豫南地区野生大豆的亲缘关系,为河南省野生大豆种质资源的利用与开发提供参考依据。
1 材料和方法
1.1 材 料
12份野生大豆材料,于2019年10—11月分别在河南省信阳市平桥区、驻马店市正阳县、南阳市桐柏县3个地点采集,其中信阳市5份、驻马店市1份、南阳市6份。
1.2 器 材
研钵和研磨棒、1.5 mL离心管、植物组织基因组提取试剂盒、PCR仪、离心机、垂直板电泳槽、水平电泳仪、照胶仪、微波炉、水浴锅。
1.3 试 剂
液氮、200 μL RCP tube、DNase-free water、2×Taq酶Mix、20对引物浓度为10 μmol·L-1、琼脂糖、1%TBE、GelRed、EDTA。
1.4 方 法
1.4.1DNA提取
采用CTAB法,取泡水发芽的大豆组织(时间2~3周,2~3 cm)100 mg,置于液氮中在研钵里进行研磨液体状,快速盛入1.5 mL离心管中,编号,置于-20 ℃冰箱中保存,使用植物组织基因组提取试剂盒对研磨的样本进行基因组抽提;最终洗脱体积为100 μL;使用Nanodrop测定样本DNA浓度,将测定浓度后的DNA样品放置在4 ℃冰箱临时保存。
1.4.2SSR标记
PCR的反应体系为:DNA模板4.5 μL(10 μg·L-1),2×Taq酶 PCR Mix 10 μL,10 μmol·L-1正反引物各1.5 μL,加无菌水至20 μL。反应程序为95 ℃下预变性5 min,94 ℃变性30 s,55 ℃退火30 s,72 ℃循环60 s,循环35次,72 ℃延伸5 min,最后4 ℃保存。

表1 12份样本DNA浓度Table 1 DNA concentration of 12 soybean samples
1.4.3琼脂糖凝胶电泳检测
用2%琼脂糖凝胶电泳检测PCR产物,120 V电压条件下30~40 min电泳,待电泳结束后在照胶仪器上拍照保存胶图。
1.5 数据分析
对跑过的胶图条带进行统计。使用Photoshop软件对胶图进行修正处理,处理完后再用分析软件Quality one对图片进行分析,对扩增后的每个样品的片段大小进行确定,确定每个样品的位点和条带数。然后根据每个引物在各样品上显示的位点进行统计带数,有条带的标为1,无条带的标为0,将数据输入软件Excel中,创建矩阵,用Popgene 32软件进行遗传相似系数分析,然后根据遗传相似系数用Ntsys 1.0软件进行聚类分析。
2 结果与分析
2.1 SSR位点多态性分析
利用20对引物对豫南地区12份野生大豆基因组进行扩增,如表2所示,20对引物总共扩增了360条带,扩增带数最多的为42条(Satt 654),如图1所示;扩增带数最少的为8条(Satt 022)。20个位点共检测到63个等位基因变异,平均每个位点扩增3.1个等位基因。结果如图2所示,并且20对SSR引物都表现出多态性,等位变异有随着取样范围的缩小逐渐减少的趋势。
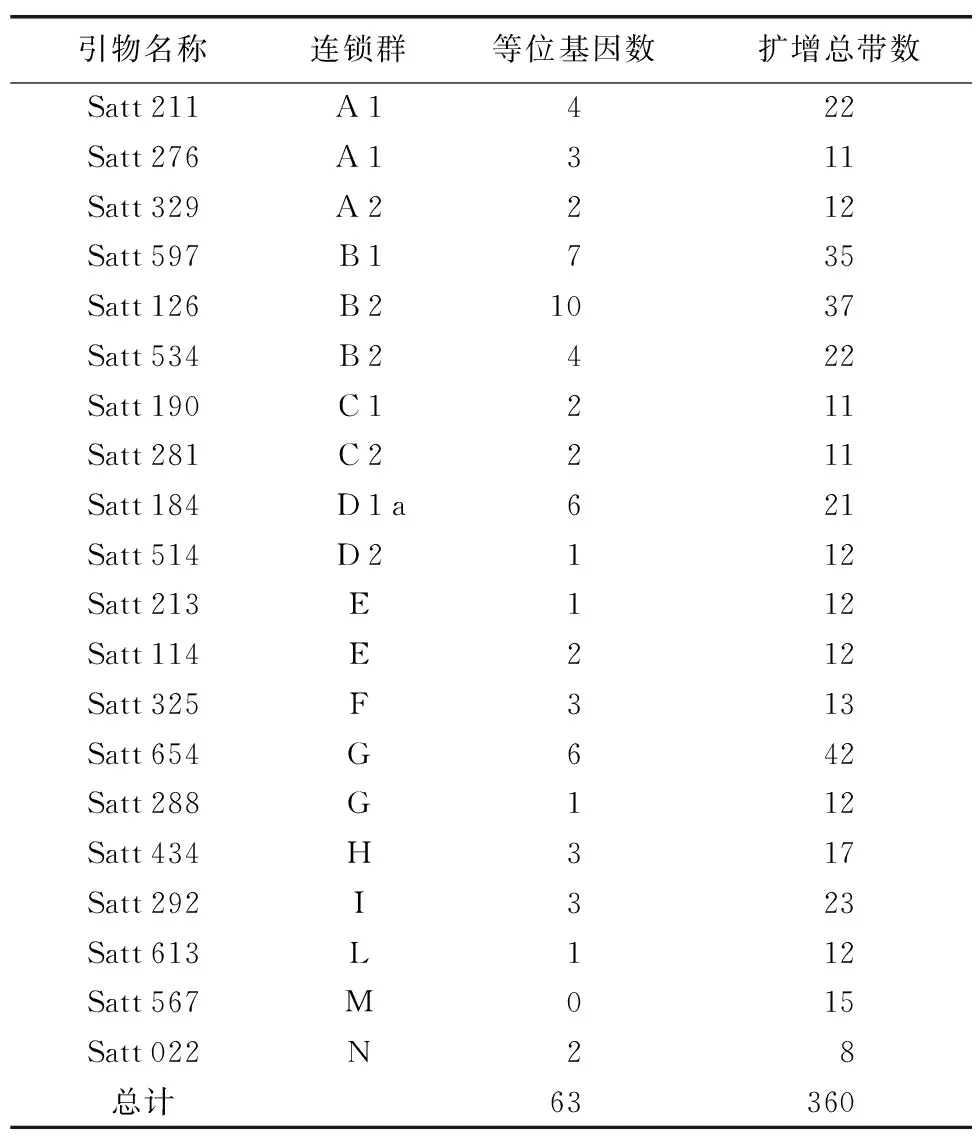
表2 20对引物扩增的结果Table 2 Amplification results of 20 pairs of primers
2.2 野生大豆的遗传相似性分析
12份野生大豆的遗传相似性如表3,每份材料的遗传相似性变化范围为0.590 9~0.954 5,平均为0.797 1,其中,XY 2与XY 4的遗传相似性最高,为0.954 5,说明XY 2与XY 4的亲缘关系较近;TB 2与TB 3的遗传相似性最低,为0.590 9,说明TB 2与TB 3亲缘关系较远,虽然地理位置相同,但亲缘关系不同,可能是种群间出现了变异。

表3 12份材料的遗传相似系数Table 3 Genetic similarity coefficient of 12 soybean materials
2.3 野生大豆的聚类分析
根据遗传相似系数建立的12份材料的树状聚类图(图3)。根据聚类图,可以将这些材料分三大类。第一大类包括10份,分别为TB 6、TB 5、TB 4、XY 5、XY 3、XY 2、XY 4、ZY、XY 1、TB 1,又可分为2个亚类;第1亚类包括2份,分别为TB 6、TB 5;第2亚类包括8份,分别是TB 4、XY 5、XY 3、XY 2、XY 4、ZY、XY 1、TB 1。第二大类1份TB 3;第三大类1份TB 2。12份材料的遗传距离都小于0.12,说明材料之间有较近的亲缘关系。从聚类结果可以看出,SSR分子标记与材料的地理来源并没有明显的相关性。
3 讨 论
李建东等[9]利用40对引物对辽宁省30份野生大豆进行多样性分析,结果表明,中部平原遗传多样性指数最高,其次为东部山地,西部丘陵最低,从聚类结果看到SSR分子标记的结果与品种的地理来源没有相关性。本研究对12份材料聚类分析,聚类结果分为三大类,三大类又分为2个亚类,从聚类结果看到,SSR分子标记与材料的地理来源并没有明显的相关性。有可能是材料的地理距离太近,环境因素相似。
利用SSR分子标记野生大豆种质资源多样性分析,得出12份野生大豆在分子水平上发生了一定的遗传变化,野生大豆亲缘关系与材料生长的地理环境、表型性状等具有一定的相关性,同一区域收集到的野生大豆也表现出遗传分化。与程春明等[10]对江西野生大豆的遗传多样性分析的结论一致。进一步了解豫南地区野生大豆种质资源的遗传规律及进化,也进一步为野生大豆杂交育种及生理栽培研究等提供指导。由此可见,在豫南地区,积极发掘野生大豆,分析和研究利用其特性,拓宽大豆育种遗传基础是十分必要的。
4 结 论
本研究利用20对引物对12份野生大豆材料进行遗传多样性研究,结果表明,20对引物对12份野生大豆共扩增63个等位基因,平均每对引物扩增3.1个等位基因。说明豫南地区具有较高的的遗传多样性,并且该地区属于亚热带季风气候,有利于喜温作物生长。基于20对SSR引物PCR结果的聚类分析能将12份材料分类出来,得知各材料之间亲缘关系较近,SSR分子标记与材料的地理来源并没有明显的相关性,进而证实SSR标记在遗传多样性分析中有很高的准确性。由于当时只是在豫南地区选择3个点进行收集野生大豆,一些野生大豆资源丰富的乡镇可能收集不够全面,只有部分野生大豆的多样性,结论仅供参考。今后将扩大收集野生大豆的地区,丰富野生大豆的资源,有针对性地进行研究,拓宽大豆育种的遗传基础。

